2. 三峡大学水利与环境学院, 宜昌 443002;
3. 中国农业大学农学与生物技术学院/生物质工程中心, 北京 100193
2. College of Hydraulic and Environmental Engineering, China Three Gorges University, Yichang 443002;
3. College of Agronomy and Biotechnology/Center of Biomass Engineering, China Agricultural University, Beijing 100193
依赖于微生物的厌氧消化作用能够将多种有机废水和固体废物转化为甲烷、氢气等清洁能源(Alvarez & Lidén,2008; Demirer & Chen,2005; Murto et al.,2004; Ueno et al.,1996),是一种环境友好型的新能源生产途径(Amon et al.,2007; Antonopoulou et al.,2008),也是目前处理工农业有机废物的一个重要手段(Grady Jr et al.,2012; Horan,1999).厌氧消化的核心是微生物群,因此,获得高活性的厌氧消化菌群是有效转化有机废弃物为能源物质的重要保障.生物反应器内的厌氧消化菌群具有复杂的组成(Hori et al.,2006; Sasaki et al.,2011; Shin et al.,2010),通常被划分为3类,即酸化菌群、产氢产乙酸菌群以及产甲烷菌群(Ahring,2003).其活性受多种因素影响,包括反应器的结构、pH值、水力停留时间等.Nadarajah等认为温度是厌氧消化菌群的重要影响因子,将影响生物反应器内的菌群结构、化学需氧量(Chemical Oxygen Demand,COD)去除率、出水悬浮固体(Effluent Suspended Solids,ESS)和污泥指数(Sludge Volume Index,SVI)等(Nadarajah et al.,2007).
通常厌氧消化作用在2个温度范围内进行,即中温厌氧消化和高温厌氧消化.良好高温厌氧消化菌群在驯化之初,菌源常取自于自然环境如土壤、河道底泥、牛羊粪便以及常温活性污泥等.在提升温度的富集培养过程中,逐渐形成稳定的厌氧消化菌群.Pettersson等的研究认为,培养温度改变会给菌群结构的调整带来新的选择压力.温度转变幅度越大,这种选择压力越强烈,并增加菌群结构调整的幅度.同时,温度可能影响菌群变化的周转速率,高的周转速率使菌群更加快速的更换掉不适应新温度的菌群(Pettersson,2004).Zheng等利用分子生物学的检测结果发现,高温的产甲烷古菌可存在于常温厌氧反应器中,反之亦然(Zheng & Raskin,2000),而其生物量取决于培养温度(van Lier et al.,1992).Ahring探索了高温厌氧消化过程温度变化对菌群生物量的影响,结果显示当将一个连续搅拌反应器的培养温度从55 ℃提高到65 ℃,细菌的生物量将有明显的下降,而古菌的生物量会有上升,当温度达到65 ℃时,氢营养型产甲烷古菌具有明显的生物活性(Ahring et al.,2001).
可见,温度的转变对厌氧消化菌群结构和功能都有强烈的影响.为了减少温度对厌氧消化菌群的冲击,在厌氧生物反应器启动时通常采用梯度升温的方法(韩育宏等,2009).本研究则利用37 ℃的中温厌氧消化菌群直接转入50 ℃培养,并通过连续进料糖蜜有机废水驯化高温厌氧消化菌群,进而考察高温驯化过程中菌群结构、多样性及功能的变化特征.研究结果对阐明厌氧发酵菌群在温度剧烈变化带来的选择压力下菌群结构与功能的变化趋势,对改进高温厌氧消化菌群富集方法具有重要意义.
2 材料与方法(Materials and methods) 2.1 厌氧反应器运行条件400 mL实验室培养的中温(37 ℃)厌氧消化秸秆、猪、牛粪便的活性污泥注入1 L自制厌氧反应器中,自来水补充至800 mL,放置于50 ℃条件下培养.高浓度糖蜜有机废水(100000 mg·L-1 COD)(李杰等,2011)作为厌氧消化原料,每2 d进料1次,每次30 mL(水力停留时间(Hydraulic Retention Time,HRT)=27 d).每次进料前从反应装置中抽出30 mL厌氧消化液,用于理化指标与微生物多样性分析,以及维持反应体系体积平衡,厌氧反应器装置见图 1.
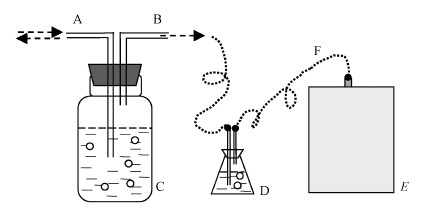 |
| 图 1 厌氧消化反应器(A:进/取料口, B:出气口, C:厌氧发酵瓶, D:缓冲装置, E:集气袋, F:连接管) Fig. 1 Anaerobic digest reactor(A: Material In/Out, B: Air out, C: Anaerobic fermentation equipment, D: Cushioning bottle, E: Gas collecting bag) |
1)pH 取待测液0.5 mL,用日本HORIBA B-212型微量pH计(Compact pH meter,model B-212,Japan)测定pH值(吕育财等,2012).
2)化学需氧量(COD)0.25 mmol·L-1 K2Cr2O7 溶液与Ag2SO4 溶液(Ag2SO4 /浓H2SO4质量体积比=1/100)按体积比1:3混合,记为溶液A.取2 mL 待测样品、3 mL 溶液A、少许HgSO4(屏蔽Cl-),加入10 mL 消解管中,在消解仪上消解(150 ℃)2 h.消解的样品冷却后利用分光光度计测定样品OD值.空白控制使用蒸馏水替代样品溶液(吕育财等,2012).
3)沼气总量及甲烷含量 每日生产沼气使用集气袋收集,利用排水集气法测定总量.沼气中甲烷含量用英国Geotech 公司的沼气成分测定仪(型号为Biogas Check)测定(袁旭峰 et al.,2011).
4)总固体(Total Solids,TS)含量和挥发性固体(Volatile Solid,VS)含量 采用重量法测定(贺延龄,1998).
5)挥发性有机酸(Volatile Fatty Acid,VFA)含量 1 mL消化液10000 r·min-1离心15 min,取0.5 mL上清,加入0.5 mL乙氰,充分混匀后,冰上保持10 min,10000 r·min-1离心15 min后过0.22 μm去除蛋白.前处理样品检测有机酸,使用LC/MS-2010A高效液相色谱(Shimadzu,Kyoto,Japan).检测条件为:10 μL样品注入仪器检测,检测柱为Aminex HPX-87H Column(Bio-Rad,CA,USA),0.05 mmol·L-1 H2SO4为流动相.洗脱程序为0.6 mL·min-1 5 min,0.4 mL·min-1 30 min,0.6 mL·min-1 5 min.柱温40 ℃,检测器为二极管阵列检测器.
2.3 微生物多样性分析1)DNA提取及PCR条件 冰冻保存样品利用淤泥基因组DNA快速提取试剂盒(BioTeke,百泰克生物技术有限公司,北京)提取总DNA,方法按说明.抽提的总DNA利用引物进行扩增,反应体系(50 μL):模板DNA 10 ng,10 ×PCR Gold Buffer 5 μL,25 mmol·L-1 MgCl2,3 μL,dNTP mix(各2 mmol·L-1)5 μL,45 μmol·L-1 引物(27f和906r)各0. 5 μL,5 Unit Ampli Taq Gold 0. 2 μL.PCR反应条件为:94 ℃预变性10 min,94 ℃变性30 s,52 ℃退火45 s,72 ℃延伸1 min 30 s,共22个循环,最后在72 ℃下延伸5 min.PCR产物用15 mg·mL-1的琼脂糖凝胶电泳检测.
2)变性梯度凝胶电泳(DGGE)提取厌氧消化菌群DNA,利用PCR仪扩增菌群16S rDNA基因片段片段(使用引物,细菌:357F-GC,517 R;古菌:A348IF,U806IR-GC).使用DcodeTM Universal Mutation Detector System(BIO-RAD Laboratories,Hercules,CA,USA)对所获DNA片段进行凝胶电泳.变性胶1 mm 厚,Polyacrylamide浓度梯度6%~12%(W/V),0.5× TAB Electrophoresis Buffer(20 mmol·L-1 Tris-HCl,pH值 8.3,10 mmol·L-1 acetic acid,0.5 mmol·L-1 EDTA),变性剂尿素梯度20%~55%(含有40%甲酰胺,7 mol·L-1尿素).电泳电压200 V,温度为61 ℃,时间5 h.电泳结束后用SYBR Green I(Molecular Probes,Eugene,OR.)染色,观察和照相使用Alpha Innotech凝胶成像仪.根据电泳条带,分析菌群结构稳定性.
3)克隆文库 将纯化后的PCR产物(使用引物,细菌:27F,907R;古菌:109F,912R)连接到pGEM-T Easy载体,4 ℃过夜,然后通过在42 ℃ 热激 45 s转化到E.coli JM109菌体内;通过X-Gal和IPTG的α-互补作用筛选出白色菌落.并将白色菌落转移到新鲜的含有抗生素的LB固体培养基上,编码建立克隆文库.利用变性梯度凝胶电泳(Denaturing Gradient Gel Eletrophoresis,DGGE,变性剂浓度范围设为20%~60%)筛选出不同种类的克隆子.对含有目标克隆子的菌体,用碱裂解法提取质粒,并测序.利用GenBank(NCBI,http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行相似菌株检索,获取近缘菌株相关序列.应用DNA序列分析软件DNA Star 7.0和MEGA 5.0软件进行序列拼接,构建系统发育树(NJ法).
4)定量PCR 古菌总量定量PCR采用Platinum ® SYBR ® Green qPCR SuperMIx-UDG试剂盒.反应体系(20μL)为:2×Platinum SYBR ® Green qPCR SuperMix-UDG,10 μL;50 mmol·L-1 MgCl2,1.2 μL;Forward and Reverse specific primers(10 pmol·μL-1),各0.4 μL;DNA,1 μL;Rox,0.4 μL;PCR grade distilled water 7.8 μL.标准曲线制作,使用含有被检测菌株目标片段克隆子的pGEM-T Easy质粒作为标准样品,用荧光定量仪精确测定标准样品浓度,并计算其拷贝数.将稀释成不同数量级拷贝数的梯度标准液,与被测样品一同使用ABI Prism ® 7500型荧光定量PCR仪(ABI,USA)进行PCR及荧光检测.扩增程序采用ABI仪器的标准程序:50 ℃持续 2 min(UDG孵育)→ 95 ℃持续2 min → 40个循环:95 ℃,15 s;60 ℃,30 s.荧光检测器在60 ℃时测定体系荧光强度.数据分析,通过Ct值确定标准曲线,评价数值的优良,最终获取待测样品拷贝数(生物量).Methanomicrobiales(MMB),Methanococcales(MCC),Methanosaetaceae(MST)分析使用TaqMan ® Universal PCR Master Mix(Applied Biosystems,USA)试剂盒分析,反应体系为PCR-grade water,5 μL;Forward and Reverse specific primers(10μmol·L-1),各1 μL;对应TaqMan探针(1 μmol·L-1)1 uL;2×TaqMan ® Universal PCR Master Mix,10 μL; DNA,1 μL.其他同Platinum ® SYBR ® Green qPCR SuperMIx-UDG试剂盒反应.其中MMB退火温度为63 ℃.(Zhang et al.,2012)
3 结果(Results) 3.1 高温驯化过程中COD、pH值和菌群产沼气量、甲烷含量变化将来自于实验室的37 ℃条件下运行的禽畜粪便厌氧消化产甲烷菌群直接转移至50 ℃条件下培养,处理高浓度有机废水.高温驯化期间,厌氧消化菌群的产气量、甲烷含量、pH值及COD等理化特征指标被检测(图 2).结果表明中温的厌氧产甲烷菌群转入高温培养的前7 d即开始产气,平均日产气量为115 mL·d-1,20 d后,产气量进入高峰期,平均日产气量超过360 mL·d-1.最高日产气量出现在培养的第31 d,达到410 mL·d-1(图 2d).培养期间总计产气量为9735 mL(图 2d),平均COD转化效率为162.5 mL·g-1.甲烷含量的变化趋势与平均日产气量相似,初期甲烷含量较低(18.9%),20 d后,甲烷含量维持在34%以上.反应器内的COD含量在产气期间稳定在6800~7700 mg·L-1(图 2a),pH值维持在7.3~8.1之间(图 2b).4个参数的检测结果表明厌氧消化菌群被转至高温条件下驯化培养,能够快速启动,并连续、稳定地处理高浓度有机废水产生沼气.
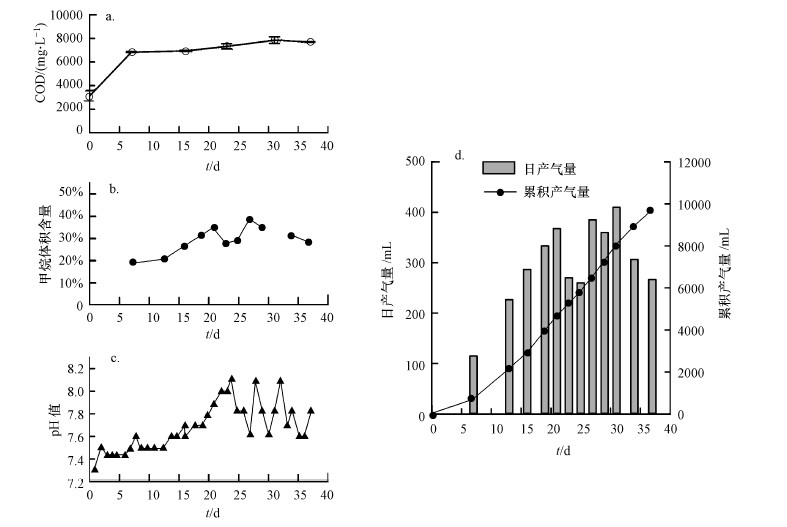 |
| 图 2 高温驯化厌氧消化菌过程中COD、pH值和菌群产沼气量、甲烷含量变化(注:本研究中0 d样品为30 ℃培养的原始厌氧消化液,其他时间为转移至50 ℃后的厌氧消化液样品,下同.) Fig. 2 COD, pH, biogas production, methane content variation during anaerobic digestion(Note: In this study, the 0 day′s samples were original anaerobic digested wastewater in 37 ℃, the other time samples were in 50 ℃, the same below.) |
丙酸和乙酸是发酵液中含量最高的两类有机酸(图 3).丙酸含量在启动前期占有明显的优势,最高值为784.7 mg·L-1(第15 d),当反应器运行状态稳定后,丙酸含量显著下降,在培养的第30 d,降低至25.3 mg·L-1.乙酸是产甲烷古菌合成甲烷的重要底物,反应器内乙酸的积累及转化速度常常反应了厌氧消化菌群生产甲烷的状态.在反应器启动前期,发酵液内出现乙酸积累现象,并随培养时间的增加而增加,在第15 d含量达到439.3 mg·L-1.进入产气高峰后,反应体系内的乙酸含量明显降低,在培养的第30 d,乙酸含量减少至145.3 mg·L-1.甲酸和乳酸在发酵液中的含量较少,其中乳酸质量浓度18~81 mg·L-1之间,甲酸仅在发酵前期被检测到,质量浓度约在16~18 mg·L-1之间.
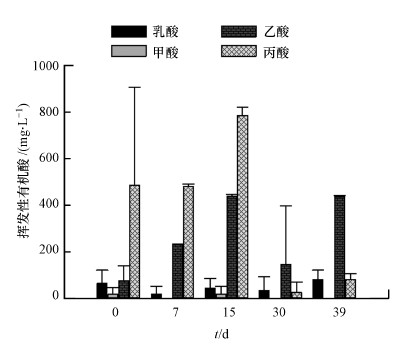 |
| 图 3 厌氧消化过程有机酸含量变化 Fig. 3 Change of Volatile Fatty Acid content during anaerobic digestion |
反应器运行期间,厌氧消化菌群中细菌和古菌的菌群结构利用DGGE技术进行分析.结果显示,在细菌菌群的DGGE图谱中,大量的高亮条带存在于每个泳道内,表明细菌菌群的多样性十分丰富(图 4a、4b),而古菌菌群的高亮条带相对于细菌菌群明显减少.在反应器运行期间,无论细菌还是古菌菌群,都存在一些条带的消失和出现的状况.在细菌菌群中约有7个主要条带出现(“△”指示部分,下同),7个主要条带消失(“
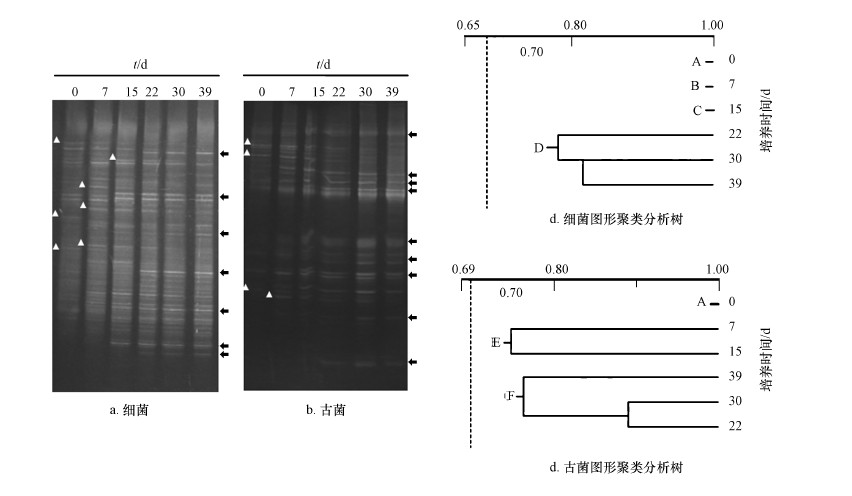 |
图 4
DGGE技术分析细菌和古菌的菌群结构(注:a. 细菌不同培养时间的DGGE图像;b. 古菌不同培养时间的DGGE图像,“△”指示新增条带,“  |
利用Quantity One软件对反应器菌群DGGE图片中的条带图形进行分析,所获数据利用UPGAMA方法进行聚类分析绘制成系统树,结果见图 4c.细菌菌群的DGGE图谱被划分入4个族,0、7、15 d图谱分别属于A、B、C 3个族,22、30、39 d图谱被划归入D族,表明在转入高温培养的前22 d,发酵体系内的菌群结构不断调整,以适应新温度带来的选择压力.22 d后,菌群结构趋于稳定.古菌菌群的DGGE图谱被划归入3个族(图 4d),0 d谱图划归为A族,7 d和15 d图谱划归入B族,22、30和39 d图谱划归入C族.表明改变温度培养后,古菌菌群结构即产生变化.菌群结构的变化程度弱于细菌,且变化主要集中在培养的前7 d.与细菌相似,古菌菌群结构自22 d后进入稳定阶段,直至培养结束,菌群结构没有出现显著的差异.通过细菌和古菌DGGE图谱变异分析结果,可发现,中温厌氧消化菌群转入高温培养后,菌群结构将会发生显著的变异,这种菌群结构的变异将会持续20 d左右,这与反应体系沼气产量相吻合(图 2d).细菌的变迁程度相对于古菌更加剧烈,导致这一现象的原因可能是由于细菌菌群的多样性较古菌菌群更为丰富所引起的.这一分析结果可由针对DGGE图谱的主成分分析更为形象的说明(图 5).
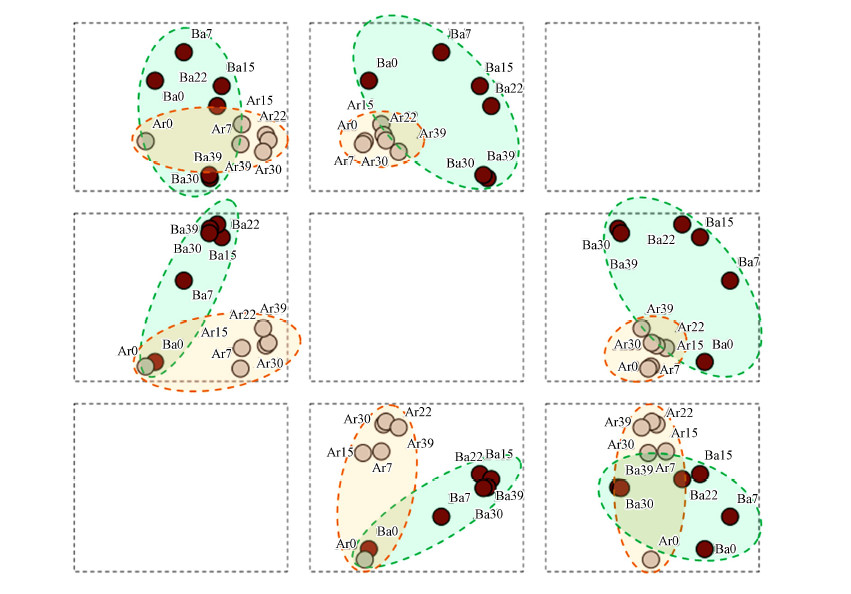 |
| 图 5 细菌和古菌DEEG条带变化的主成分分析(注:图中Ba为细菌,Ar为古菌,数值为培养时间) Fig. 5 Principal Component Analysis (PCA) of bacterial and archaeal DGGE pattern(Note: Ba, bacteria, Ar, archaea. Numbers were culture times (days)) |
主成分分析常用来分析菌群结构变化(Castro et al.,2005; Dollhopf et al.,2001; Mao et al.,2008).在本研究中,细菌和古菌菌群的分析结果被提取了3个主成分,其累积方差为73.2%(主成分1、2、3分别为32.1%、28.1%、13.0%).图 5为3个主成分的载荷图,元素类型矩阵.图中结果显示,细菌菌群和古菌群在图中能够较为明显的被区分.与古菌菌群相比,细菌菌群在转变培养温度后菌群结构方差值更为分散,表明细菌菌群的结构变异更为强烈.转入高温培养后,2组菌群的菌群结构与原始培养温度均产生了明显的变化,在图中则表现为0 d的菌菌群结构和后期培养的菌群结构均产生了较远的变异距离.
3.4 高温驯化厌氧消化菌群过程中菌群多样性分析厌氧消化菌群的16S rRNA基因片段利用克隆方法被获取,并建立克隆文库、绘制系统发育树,获取菌群内菌群多样性信息.
图 6a显示厌氧消化菌群中细菌菌群的系统发育特征.所有细菌菌群划入7个族,包括Caloramator、Thermoanaerobacter、Peptococcaceae、Thermacetogenium、Acetomicrobium、Soehngenia、Bacteroides.其中Ba4族主要为Thermacetogenium菌,在菌群中的比例最多,达到33.44%.第4族中的类似菌株Thermacetogenium phaeum strain PB.分离于55 ℃高温的厌氧反应器,能够与嗜热氢营养型产甲烷菌共生而氧化乙酸(Hattori et al.,2000).Bac5在细菌菌群中的比例约为20.99%.此族的已知代表菌株为Acetomicrobium faecal,该菌发现于下水道污泥,它能够代谢多种己糖和戊糖为乳酸、乙酸、乙醇、CO2等(Winter et al.,1987).此外,Bac3、Bac1、Bac7等族的菌株在厌氧消化菌群中占有较大比例,分别为13.5%、12.35%、12.34%.它们多数发现于高温厌氧环境.
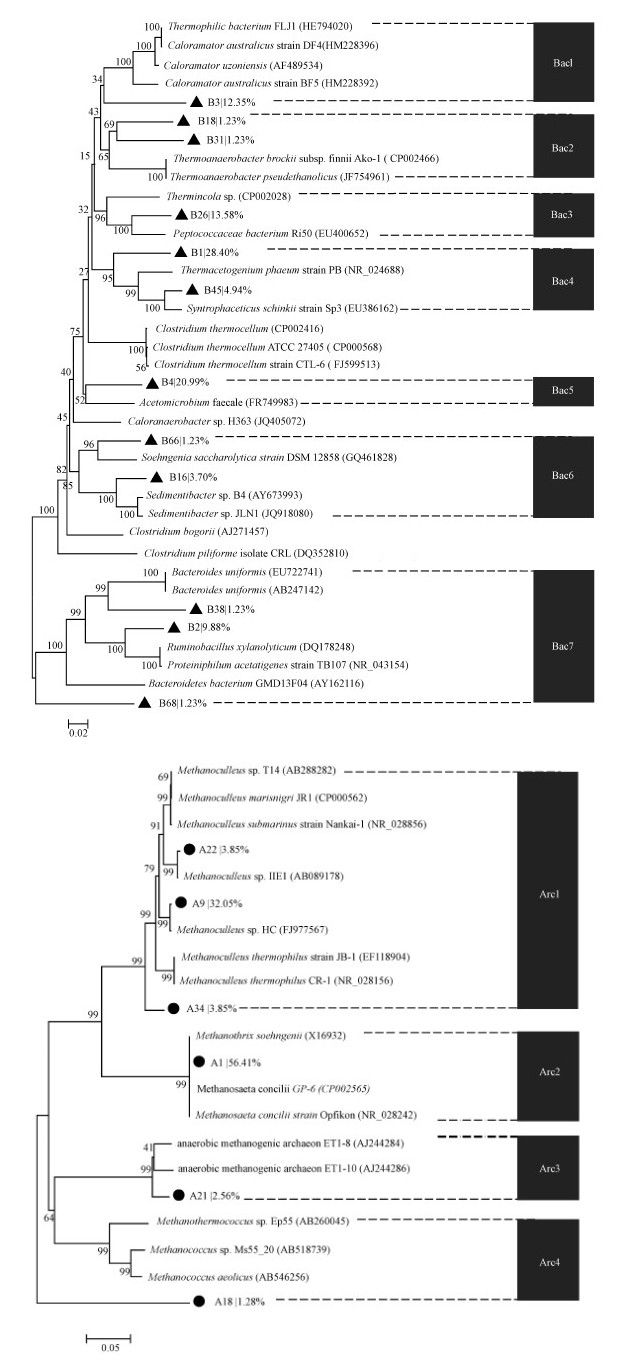 |
| 图 6 细菌、古菌菌群16S rDNA序列的系统发育树分析 Fig. 6 Phylogenetic tree analysis based on bacterial and archaeal 16S rDNA sequences |
厌氧消化菌群中的古菌菌群被划分为4个族(图 6b),其中Arc2为4族中最为丰富的古菌菌群,丰度为检测古菌总量的56.4%,代表菌株Methanosaeta concili是一类能够利用乙酸作为唯一能源底物代谢产生等量的CH4和CO2.能够利用甲酸、乙酸和CO2作为碳源而生长(PATEL & SPROTT,1990).Arc1在古菌clone文库中所占的比例为39.75%,代表菌株为Methanoculleus,该菌株能够利用H2/CO2作为底物而生长并产生甲烷(Tian et al.,2010).一些菌种还需要乙酸作为生长因子(Mikucki et al.,2003).此外,还有一些厌氧产甲烷古菌及Methanococcus(Arc3和Arc4)被检测到,它们在clone文库中的比例分别为2.56%和1.28%.Jones(Jones et al.,1983)的研究发现Methanococcus是一种产甲烷古菌,能够利用H2和CO2生成甲烷.Arc3中的近缘古菌为Chin(Chin et al.,1999)所报道,是通过克隆文库方法于水田土壤中发现的菌株.
可见,转入高温培养后,菌群中主要的构成菌株转换成为能够在高温条件下生长并与糖、多种有机酸代谢相关的细菌菌株以及产甲烷古菌菌株.正是由于这些菌株的稳定存在,才能够维持菌群在高温条件下稳定消化有机废水生成沼气.
3.5 高温驯化厌氧消化菌群过程中主要菌株的定量分析产甲烷古菌的总生物量及一些典型的产甲烷古菌的生物量可以利用荧光定量PCR方法进行定量分析.结果显示,厌氧消化菌群从37 ℃转移至50 ℃,产甲烷古菌的总生物量减少(图 7c),并在22 d后进入相对稳定状态,含量约为7.6×106 拷贝/g活性污泥,相比于中温培养减少42.2%.利用特异性荧光探针,检测了MMB,MCC,MST 3类产甲烷古菌,发现,MMB是一类在37 ℃和50 ℃均能检测到,并大量存在的产甲烷古菌类群,这与董慧峪等(2014)的研究结果相一致.MCC和MST的生物量在群体中
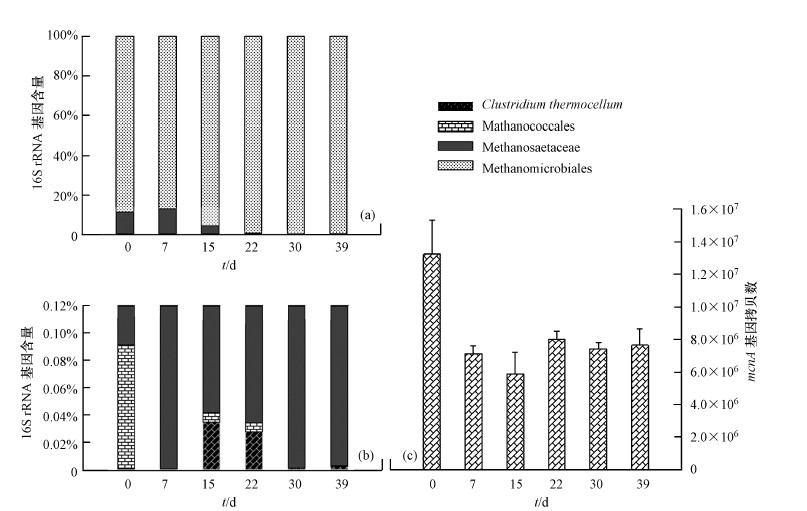 |
| 图 7 厌氧消化菌群内产甲烷古菌的生物量(注:(a).各类菌株的16S rRNA基因拷贝数占被检测总菌株拷贝数的比例;(b),(a)放大到0.12%(Y轴)尺度的图像;(c).产甲烷古菌mcrA基因的拷贝数) Fig. 7 Biomass of methanogenic archaea in anaerobic culture(Note: (a), Copy number of detectable microorganism. (b), Amplifying A to 0.12% scale. (c), Copy number of mcrA of Methanogenic archaea.) |
较少约占检测4种菌株总量的11%,并且在转入高温培养后,其生物量的急剧减少,MCC甚至在菌群稳定后消失.表明这2种产甲烷古菌并不适宜于50 ℃条件下生长.Clostridium thermocellum是一株严格厌氧的纤维素分解菌株,广泛存在于含有纤维素基质的厌氧降解环境.C. thermocellum 是一株能够在高温良好生长的菌株,在本文中,转入高温后生物量反而小于常温培养,推测为厌氧消化体系以不含纤维素糖蜜废水为主,因缺乏适宜的代谢底物,随着厌氧消化体系运行时间的延长,生物量逐渐衰退.
4 讨论(Discussion)在厌氧消化过程,温度通常是影响微生物菌群的显著因子,影响微生物的生长和生存(Levén et al.,2007).本研究将37 ℃的厌氧消化菌群直接提温至50 ℃驯化培养,在温度和底物的选择压力下,原始菌群结构经过剧烈的变化调整后逐渐适应新的温度条件,并具有产甲烷的功能.通常在厌氧消化菌群驯化中,多采用中温菌群梯度升温的方法获得高温厌氧消化菌群,如欧阳二明等(2009)等采用序梯度升温方法将中温活性污泥(35 ℃)驯化为高温处理热水解污泥的菌群.李超伟等(2014)以食堂餐厨垃圾为原料,采用中温沼液作为菌种经缓慢升温成功启动了高温处理餐厨垃圾的厌氧消化装置.在本研究中通过一步直接提温的方法也可获得高温厌氧消化高浓度有机废水菌群,这与Ahring、Tian和Raskin的观点相一致(Ahring,2003; Tian et al.,2015; Zheng & Raskin,2000).
一些中温厌氧消化菌群中常存在适宜于高温生长的功能菌株,这部分菌株并不会在中温条件下的长期培养而丢失.无论本研究中将分解禽畜粪便的中温厌氧消化菌群直接转入高温培养,获得高温厌氧消化菌群,还是利用梯度升温法获得高温厌氧消化菌群,其结果均能使存在于中温菌群中的高温功能菌株活化并富集为优势菌群而发挥功能.其区别在于菌群在温度改变过程中所面临的选择压力大小.显然,直接提升温度的方法增加了这种选择压力(Pettersson,2004).
在菌群结构变迁过程中,细菌的变化程度较古菌强烈.Gobema等研究认为,细菌具有非常高的多样性,在常温厌氧反应器内,其种类丰富程度远高于古菌(Goberna et al.,2009; Levén et al.,2007).本研究中,当培养温度提高以后,大量DGGE条带或消失、或新增,表明菌群内一些无法适应新培养温度菌株的生物量骤减,而适应高温生长细菌菌株的生物量增加并占据了活跃的主导地位.与细菌菌群相比较,古菌菌群的多样性变异较为平缓,这除了与古菌种类较少(图 3、5、6)有关,可能也和古菌生长周期长、生长缓慢有关(Ferry,1993).细菌因其生长速度快,菌群结构更新周期短,表现为细菌菌群结构变异剧烈(Pettersson,2004).
5 结论(Conclusions)1) 37 ℃培养的中温厌氧消化产甲烷菌群直接转入50 ℃高温驯化处理高浓度糖蜜有机废水,厌氧消化过程能够快速启动并生成甲烷,并在22 d后形成稳定的高温厌氧消化产甲烷菌群,平均甲烷生成效率为162.5 mL·g-1.稳定产气期间乙酸和丙酸的质量浓度分别为25.3和145.3 mg·L-1.
2) 菌群结构及多样性在高温驯化过程中发生剧烈变化,细菌强于古菌,并逐渐稳定成为以细菌(Thermacetogenium,Acetomicrobium)以及产甲烷古菌(Methanosaeta,Methanoculleus)为主要优势菌群的高温厌氧消化菌群.
3) 所获高温厌氧消化产甲烷菌群,其产甲烷古菌的总生物量明显下降,大约为7.6×106拷贝/g活性污泥.
| [1] | Ahring B. 2003. Perspectives for Anaerobic Digestion Biomethanation I, (Eds.) Ahring B, Angelidaki I, de Macario E, et al, Vol. 81, Springer Berlin/Heidelberg, 1-30 |
| [2] | Ahring B K, Ibrahim A A, Mladenovska Z. 2001.Effect of temperature increase from 55 to 65℃ on performance and microbial population dynamics of an anaerobic reactor treating cattle manure[J]. Water Research, 35 (10): 2446–2452. |
| [3] | Alvarez R, Lidén G. 2008.Semi-continuous co-digestion of solid slaughterhouse waste, manure, and fruit and vegetable waste[J]. Renewable Energy, 33 (4): 726–734. |
| [4] | Amon T, Amon B, Kryvoruchko V, et al. 2007.Methane production through anaerobic digestion of various energy crops grown in sustainable crop rotations[J]. Bioresource Technology, 98 (17): 3204–3212. |
| [5] | Antonopoulou G, Gavala H N, Skiadas I V, et al. 2008.Biofuels generation from sweet sorghum:fermentative hydrogen production and anaerobic digestion of the remaining biomass[J]. Bioresour Technol, 99 (1): 110–119. |
| [6] | Castro H, Newman S, Reddy K R, et al. 2005.Distribution and stability of sulfate-reducing prokaryotic and hydrogenotrophic methanogenic assemblages in nutrient-impacted regions of the florida everglades[J]. Applied and Environmental Microbiology, 71 (5): 2695–2704. |
| [7] | Chin K J, Lukow T, Stubner S, et al. 1999.Structure and function of the methanogenic archaeal community in stable cellulose-degrading enrichment cultures at two different temperatures (15 and 30℃)[J]. FEMS Microbiology Ecology, 30 (4): 313–326. |
| [8] | Demirer G N, Chen S. 2005.Two-phase anaerobic digestion of unscreened dairy manure[J]. Process Biochemistry, 40 (11): 3542–3549. |
| [9] | Dollhopf S L, Hashsham S A, Tiedje J M. 2001.Interpreting 16S rDNA T-RFLP Data:Application of Self-Organizing Maps and Principal Component Analysis to Describe Community Dynamics and Convergence[J]. Microbial Ecology, 42 (4): 495–505. |
| [10] | Ferry J G. 1993. Methanogenesis:ecology, physiology, biochemistry & genetics[M]. New York: Springer US . |
| [11] | Goberna M, Insam H, Franke-Whittle I H. 2009.Effect of Biowaste Sludge Maturation on the Diversity of Thermophilic Bacteria and Archaea in an Anaerobic Reactor[J]. Applied and Environmental Microbiology, 75 (8): 2566–2572. |
| [12] | Grady Jr C L, Daigger G T, Love N G, et al. 2012. Biological wastewater treatment (Third Edition)[M]. New York: CRC Press . |
| [13] | Hattori S, Kamagata Y, Hanada S, et al. 2000.Thermacetogenium phaeum gen[J]. nov, sp. nov, a strictly anaerobic, thermophilic, syntrophic acetate-oxidizing bacterium[J]. International Journal of Systematic and Evolutionary Microbiology, 50 (4): 1601–1609. |
| [14] | 贺延龄. 1998. 废水厌氧生物处理[M]. 北京: 中国轻工业出版社 . |
| [15] | Horan N J. 1999. Biological wastewater treatment systems:theory and operation (First Edition)[M]. New York: John Wiley & Sons . |
| [16] | Hori T, Haruta S, Ueno Y, et al. 2006.Dynamic transition of a methanogenic population in response to the concentration of volatile fatty acids in a thermophilic anaerobic digester[J]. Appl Environ Microbiol, 72 (2): 1623–1630. |
| [17] | Jones W J, Leigh J A, Mayer F, et al. 1983.Methanococcus jannaschii sp[J]. nov, an extremely thermophilic methanogen from a submarine hydrothermal vent[J]. Archives of Microbiology, 136 (4): 254–261. |
| [18] | Levén L, Eriksson A R B, Schnürer A. 2007.Effect of process temperature on bacterial and archaeal communities in two methanogenic bioreactors treating organic household waste[J]. FEMS Microbiology Ecology, 59 (3): 683–693. |
| [19] | 李超伟, 康勇, 高磊, 等.2014.高温厌氧消化处理餐厨垃圾试验研究[J].中国沼气, (03):18–21+7. |
| [20] | 李杰, 张冬冬, 袁旭峰, 等.2011.固定载体卧式厌氧反应器处理糖蜜废水的快速启动[J].微生物学通报, 38 (4):474–480. |
| [21] | 吕育财, 崔宗均, 王小芬, 等.2012.三组微生物菌群分解油菜秸秆的消化液性质比较[J].农业工程学报, 28 (3):210–214. |
| [22] | Mao Y, Zhang X, Yan X, et al. 2008.Development of group-specific PCR-DGGE fingerprinting for monitoring structural changes of Thauera sppin an industrial wastewater treatment plant responding to operational perturbations[J]. J Microbiol Methods, 75 (2): 231–236. |
| [23] | Mikucki J A, Liu Y, Delwiche M, et al. 2003.Isolation of a methanogen from deep marine sediments that contain methane hydrates, and description of methanoculleus submarinus spnov[J]. Applied and Environmental Microbiology, 69 (6): 3311–3316. |
| [24] | Murto M, Bj rnsson L, Mattiasson B. 2004.Impact of food industrial waste on anaerobic co-digestion of sewage sludge and pig manure[J]. Journal of Environmental Management, 70 (2): 101–107. |
| [25] | Nadarajah N, Allen D G, Fulthorpe R R. 2007.Effects of transient temperature conditions on the divergence of activated sludge bacterial community structure and function[J]. Water Res, 41 (12): 2563–2571. |
| [26] | 欧阳二明, 王伟, 等.2009.高温ASBR处理热水解污泥的梯度升温法启动试验研究[J].环境科学, 30 (4):1105–1109. |
| [27] | Patel G B, Sprott G D. 1990.Methanosaeta concilii gen,nov, sp. nov. ("Methanothrix concilii") and Methanosaeta thermoacetophila nom. rev, comb. nov[J]. International Journal of Systematic Bacteriology, 40 (1): 79–82. |
| [28] | Pettersson M. 2004. Factors affecting rates of change in soil bacterial communities[D]. Lund:Department of Biology, Lund University |
| [29] | Sasaki D, Hori T, Haruta S, et al. 2011.Methanogenic pathway and community structure in a thermophilic anaerobic digestion process of organic solid waste[J]. J Biosci Bioeng, 111 (1): 41–46. |
| [30] | Shin S G, Lee S, Lee C, et al. 2010.Qualitative and quantitative assessment of microbial community in batch anaerobic digestion of secondary sludge[J]. Bioresour Technol, 101 (24): 9461–9470. |
| [31] | Tian J, Wang Y, Dong X. 2010.Methanoculleus hydrogenitrophicus sp. nov, a methanogenic archaeon isolated from wetland soil[J]. International Journal of Systematic and Evolutionary Microbiology, 60 (9): 2165–2169. |
| [32] | Tian Z, Zhang Y, Li Y, et al. 2015.Rapid establishment of thermophilic anaerobic microbial community during the one-step startup of thermophilic anaerobic digestion from a mesophilic digester[J]. Water Research, 69 : 9–19. |
| [33] | Ueno Y, Otsuka S, Morimoto M. 1996.Hydrogen Production from Industrial Wastewater by Anaerobic Microflora in Chemostat Culture[J]. Journal of Fermentation and Bioengineering, 82 (2): 194–197. |
| [34] | van Lier J, Grolle K F, Stams A M, et al. 1992.Start-up of a thermophilic upflow anaerobic sludge bed (UASB) reactor with mesophilic granular sludge[J]. Applied Microbiology and Biotechnology, 37 (1): 130–135. |
| [35] | Winter J, Braun E, Zabel H P. 1987.Acetomicrobium faecalis spec, nov, a strictly anaerobic bacterium from sewage sludge, producing ethanol from pentoses[J]. Systematic and Applied Microbiology, 9 (1/2): 71–76. |
| [36] | 袁旭峰, 高瑞芳, 李培培, 等.2011.复合菌系MC1预处理对玉米秸秆厌氧发酵产甲烷效率的提高[J].农业工程学报, 27 (9):266–270. |
| [37] | Zhang D, Zhu W, Tang C, et al. 2012.Bioreactor performance and methanogenic population dynamics in a low-temperature (5~18℃) anaerobic fixed-bed reactor[J]. Bioresource Technology, 104 : 136–143. |
| [38] | Zheng D, Raskin L. 2000.Quantification of methanosaeta species in anaerobic bioreactors using genus-and species-specific hybridization probes[J]. Microbial Ecology, 39 (3): 246–262. |
 2016, Vol. 36
2016, Vol. 36


