2. 中国环境科学研究院 环境基准与风险评估国家重点实验室, 北京 100012;
3. 中国环境科学研究院 城市水环境科技创新基地, 北京 100012
2. State Key Laboratory of Environmental Criteria and Risk Assessment, Chinese Research Academy of Environmental Sciences, Beijing 100012;
3. Department of Urban Water Environmental Research, Chinese Research Academy of Environmental Sciences, Beijing 100012
Corresponding author, E-mail: pjf1995@163.com
温室气体在大气中浓度的增加越来越受到人们的重视.在所有温室气体中,甲烷(CH4)对温室气体效应的功效仅次于CO2,但是其增温潜势却为CO2的23倍(IPCC,2001).近200年来CH4在大气中的含量平均以每年1%的速度增加(Cicerone et al., 1998),主要的原因是CH4排放源的增加和CH4汇的减少(Blake et al., 1998),所以对于甲烷来源的研究迫在眉睫,而甲烷的来源主要是天然湿地、稻田、河流等.微生物产甲烷是一种普遍现象,而产甲烷菌又是介导天然湿地土壤和河流底泥中甲烷循环重要的功能菌群之一(佘晨兴等,2012),深入探索天然湿地和河流产甲烷菌的多样性有助于了解自然环境中甲烷的代谢循环.河流在地球上的分布并不少于天然湿地,有着不同于天然湿地的特殊环境,研究其产甲烷菌的多样性是有必要的.而且目前对于产甲烷的研究主要集中在稻田(陈中云等,2001;王殳屹等,2006),河口沼泽地(佘晨兴等,2013;曾志华等,2014),泥炭地(Narihiro et al., 2011;Kotsyurbenko et al., 2004;Cadillo-Quiroz et al., 2008;Yavitt et al., 2012)等,对于河流底泥的研究鲜有报道.
产甲烷菌是一类能够将无机或有机化合物厌氧消化转化成甲烷和二氧化碳的古细菌,它是专性厌氧菌,属于广古生菌门(Euryarchaeota).从Wolfe(1993)等第一个得到了产甲烷菌纯培养物并对产甲烷菌的产甲烷代谢过程进行研究开始,人们对于产甲烷菌的研究进入了一个全新的时代(李煜珊等,2014).之后随着厌氧培养技术的进步,许多学者在厌氧生物降解的环境分离了大量的产甲烷菌(Simankova et al., 2003;Bruer et al., 2006;Jones et al., 1983;Miller et al., 1982).而近年来由于分子生物学技术的提高,PCR-RFLP分子生物学检测技术广泛应用于包括河口、沼泽、湖泊在内的各种天然湿地土壤和河流底泥产甲烷古菌多样性的检测.浑河流经辽宁中部城市群,重工业相对发达、人口密集,是沿岸城市废水和废弃物排放的主要渠道(王博涵等,2015).大伙房水库位于浑河上游,目前是抚顺和沈阳市区供水的重要来源(张鸿龄等,2011),其水质情况直接影响着周边居民的生命安全.
因此,本文利用PCR-RFLP技术及测序分析技术对浑河底泥产甲烷的多样性进行研究,扩大产甲烷菌的研究领域,使人们加深对产甲烷菌在自然界广泛分布现象的认识,有助于了解河流底泥中产甲烷菌的群落结构特点,为人类最终有效调控甲烷代谢,减少甲烷排放量提供科学基础.
2 材料与方法(Materials and methods) 2.1 研究区域概况与样品采集浑河地处辽宁省中部(122°20′E~125°20′E,40°00′N~42°20′N),南邻太子河,北邻辽河,东邻浑江.浑河发源于抚顺市清原县湾甸子乡的长白山支脉滚马岭,河流自东北向西南,流经抚顺、沈阳、辽阳、鞍山4个市,全长415.4 km,在海城古城子附近三岔河与太子河相汇进入大辽河,向南流至营口市附近入辽东湾(张静等,2011).根据浑河的地理位置和自然条件,本研究共取6个采样点,具体分布见图 1.
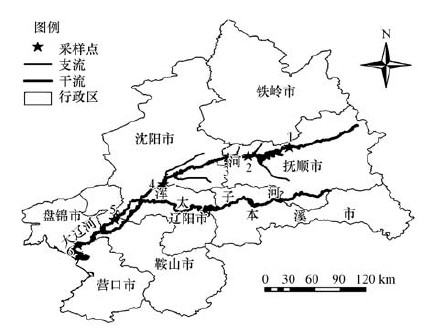 |
| 图 1 采样地点示意图(1.南杂木,2.章党大桥,3.葛布大桥,4.黄腊坨,5.对坨,6.田庄台) Fig. 1 Descriptions of sampling sites |
样品于2014年9月12日至16日采集水样和底泥样品.水样的采集: 用不锈钢采水器采集水样,采集深度约在底泥上方2 cm处,水样保存在500 mL聚乙烯采样瓶中,放入带有冰块的保温箱,运回实验室储存于4 ℃冰箱内;底泥的采集:用灭菌的柱状采泥器采集表层底泥(0~10 cm),样品采集后置于无菌自封袋中,置于干冰上运回实验室,于-80 ℃冰箱中保存.
2.2 理化性质的测定使用便携式水质测定仪(Thermo Orion Star,赛默飞世尔科技有限公司)现场测定水温、pH和溶解氧(DO).于实验室内,按照文献(国家环境保护总局,2002)测定以下主要水质污染指标:五日生化需氧量(BOD5)、总磷(TP)、氨氮(NH4+-N)、硝氮(NO3--N)和亚硝氮(NO2--N),底泥检测的指标为溶解性有机碳(DOC).
2.3 土壤总DNA 的提取及mcrA 基因的扩增使用 Power Soil DNA Kit(Mo Bio Laboratories,Carlsbad,CA)对浑河底泥样品进行总DNA的提取,具体步骤按其说明书进行.DNA提取结果经1%的琼脂糖凝胶电泳检测.
mcrA基因的扩增是采用产甲烷古菌的特异性引物对ME1(5′-GCMATGCARATHGGWATGTC-3′)和ME2(5′-TCATKGCRTAGTTDGGRTAGT-3′)(Hales et al., 1996),以提取的土壤总DNA作为模板进行扩增.2×PCR GoTaq® Green Master Mix(Promega,USA)12.5 μL,上下引物各1 μL(10 μmol·L-1),DNA模版2 μL(1~10 ng),最后用ddH2O补足至25 μL.PCR反应条件:94 ℃预变性3 min;94 ℃变性40 s,50 ℃退火1 min 30 s,72 ℃延伸2 min,30个循环;最后72 ℃延伸8 min.接着以PCR扩增产物作为DNA模板进行第二次扩增,程序和引物同上.PCR扩增产物用2%琼脂糖凝胶电泳进行检测.
2.4 mcrA 基因克隆文库的构建及RFLP分析将经2%的琼脂糖电泳检验确认正确后的产甲烷菌的mcrA功能基因的二轮PCR产物连接到pGM-T(Promega,USA)载体上,具体操作步骤按其说明书进行.然后转化到感受态细胞E.coli DH5α(Promega,USA)中进行涂板,筛选白斑.每个样品随机挑选100个克隆子,送到上海生工生物工程股份有限公司(sangon,上海)进行测序.
在所构建的每个克隆文库中随机挑取100个白色克隆子进行菌落PCR验证,以检查整个克隆文库的有效性.利用限制性内切酶PST I对阳性克隆的PCR产物进行酶切.酶切体系10 μL:PCR产物5 μL,10×T Buffer 1 μL,限制性内切酶PST I 0.5 μL,灭菌的ddH2O补充至10 μL;37 ℃酶切2 h.酶切消化产物用2%的琼脂糖凝胶分离酶切片段,凝胶成像仪采集照片.利用Quantity One(Bio-Rad,USA)软件根据酶切图谱进行初步分型.
2.5 测序及系统发育分析将经上海生工测序返回所得的序列利用BLAST在GenBank数据库中进行对比,确认为目标序列后进行多重序列对齐.利用DOTUR软件对产甲烷菌的mcrA功能基因进行OTU分类(Schloss et al., 2005)最后利用MEGA 4.0软件以Neighbor-Joining(Bootstrap=1000)的方法分别构建产甲烷菌的mcrA功能基因的系统发育树.
2.6 多样性分析指数根据文库酶切及测序分析结果计算克隆文库的库容C,并进行产甲烷古菌的多样性分析,多样性指数包括OTUs 类型数、克隆数、Shannon多样性指数(H′)和Simpson多样性指教(D).库容C计算公式如下:
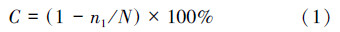
式中,N代表mcrA克隆文库总克隆数,n1代表在文库仅出现1次的OTU数量.Shannon多样性指数(H′)和Simpson多样性指数(D)利用Bio~Dap软件进行计算分析.
3 结果(Results) 3.1 水质理化特征浑河各采样点水质的理化指标见表 1.可知,浑河水质总体呈弱碱性至碱性.BOD5的值基本位于5~14 mg·L-1之间,依据地表水环境质量标准(GB3838-2002),浑河受有机污染程度较为严重(IV类至劣V类).氮磷浓度都比较低,且最大值基本出现在位于城市段之后河流中游的采样点黄腊坨,中下游明显高于上游.BOD5浓度均从上游到下游呈现出先上升后下降的趋势.底泥的溶解性有机碳含量(DOC)各采样点较为接近.河流中游的黄腊坨采样点,位于城市下游,受城市影响BOD5和氮磷含量要高于其他采样点,随着河流的流动,污染物得到稀释,有机污染程度得到稍微改善.
| 表 1 水质和底泥理化指标 Table 1 Physicochemical properties of water and sediments at sampling points |
以ME1/ME2为引物,6个底泥DNA样品经PCR扩增均可得到单一清晰的mcrA基因片段,大小约为760 bp,与所设计的目标片段(760 bp)结果相符.构建的6个mcrA基因克隆文库经菌落PCR验证共检测到592个阳性克隆,阳性率为98.67%.利用限制性内切酶PST I对阳性克隆PCR产物进行初步分型(图 2).对于每一独特的酶切类型选取一单克隆进行测序,共得到30种不同的操作分类单元(OTUs)(编号: OTU-1~OTU-30).6个克隆文库的多样性分析结果见表 2.结果表明,浑河底泥的OTUs类型数呈现“少-多-中”的规律.此外,从图 3可以看出,OTU-3和OTU-4是克隆文库M1的主要OTUs类型;OTU-3和OTU-24是克隆文库M2的主要OTUs类型;OTU-3和OTU-5是克隆文库M3、M6的主要OTUs类型;OTU-1、OTU-24和OTU-30是克隆文库M4的主要OTUs类型;OTU-15和OTU-26是克隆文库M5的主要OTUs类型;OTU-3是河流上游和下游的主要OTUs类型,而与河流中游的OTUs类型存在较大差异.
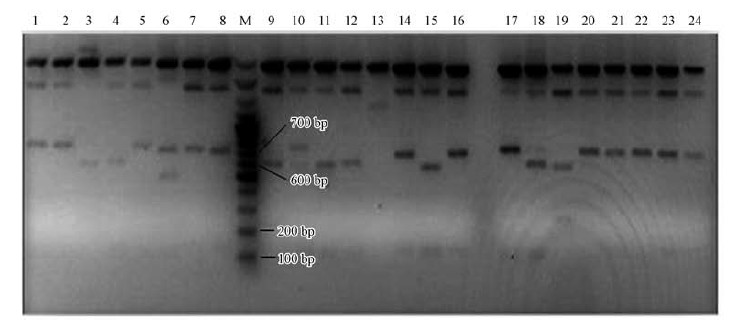 |
| 图 2 部分阳性克隆子的PST Ⅰ酶切图谱(部分) Fig. 2 mcrA genes of positive clones digested by PSTⅠ(partial) |
| 表 2 浑河底泥6个克隆文库的多样性分析 Table 2 Diversity analysis of 6 clone libraries from the sediments in the Hun River |
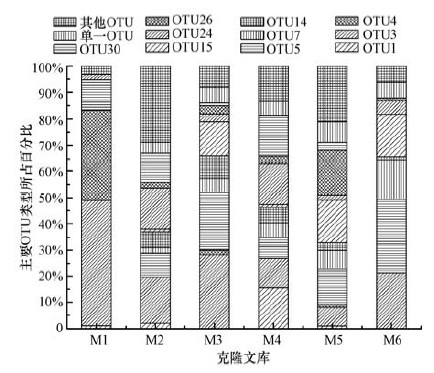 |
| 图 3 6个克隆文库主要OTUs类型的百分比 Fig. 3 Percentage of main OTUs in 6 clone libraries |
克隆文库的库容理论上代表样品中微生物种类的多样性.理论上,当库容C为100%时可以代表样品中的所有微生物的种类,库容含量越高说明库的覆盖率越高.本研究所构建的6个克隆文库的库容值均在91%以上(表 2),说明所构建的文库的覆盖程度已经较高,能够比较真实地代表该底泥中产甲烷古菌群落结构的多样性.Shannon多样性指数(H′)和Simpson多样性指数(D)计算结果(表 2)表明,河流上游(H′=1.56,D=0.33)和中游(H′=1.79,D=0.26)产甲烷古菌的多样性与下游游(H′=1.65,D=0.32)均较为接近,这与底泥中溶解性有机碳含量(DOC)的差异性结果相符.但特别指出,位于中游的M3文库Shannon指数(H′=1.38)要明显低于同样位于中游的M4文库(H′=2.21),而Simpson多样性指数(D=0.38)却高于M4文库(D=0.13),这可能与河流中游流经城市段有关,城市大部分污水都排放于河流,造成了河流水质的复杂性,污染物不断沉积到底泥中,进而改变了河流底泥中产甲烷古菌的生存环境,可能导致了产甲烷古菌群落组成发生变化.
利用Canoco for windows 4.5软件对多样性指数和环境因子进行DCA分析,得到样品矩阵第一轴的最大梯度长度小于3,因此选用冗余梯度分析方法(RDA)来分析环境因子与底泥古菌群落组成变化的相关性.分析结果显示,第一轴分别与DOC、BOD5和NO2--N的相关性好,相关系数分别为0.4603、-0.6155和-0.7341;第二轴与DO的相关性较好,相关系数为-0.5740.
3.3 mcrA基因序列的系统发育分析选取每一OTUs类型相对应的mcrA基因序列(编号:OTU1~OTU30)与GenBank数据库中已知序列进行BLAST比较,鉴定与克隆序列亲缘关系最近的种属.根据测序结果和聚类同源性分析(图 4)比较发现,浑河底泥中产甲烷古菌可以聚成4大类群:甲烷杆菌目(Methanobacteriales)、甲烷微菌目(Methanomirobiales)、甲烷胞菌目(Methanocellales)和甲烷八叠球菌目(Methanosarcinales).检测到的菌属包括甲烷食甲基菌属(Methanomethylovorans)、甲烷杆菌属(Methanobacterium)、甲烷绳菌属(Methanolinea)、甲烷胞菌属(Methanocella)、甲烷螺菌属(Methanospirillum)、甲烷囊菌属(Methanoculleus)、甲烷细菌属(Methanosphaerula)和甲烷八叠球菌属(Methanosarcina)等8个属.与互花米草沼泽湿地和闽江河口芦苇沼泽湿地的检测结果类似(佘晨兴等,2013;佘晨兴等,2012).
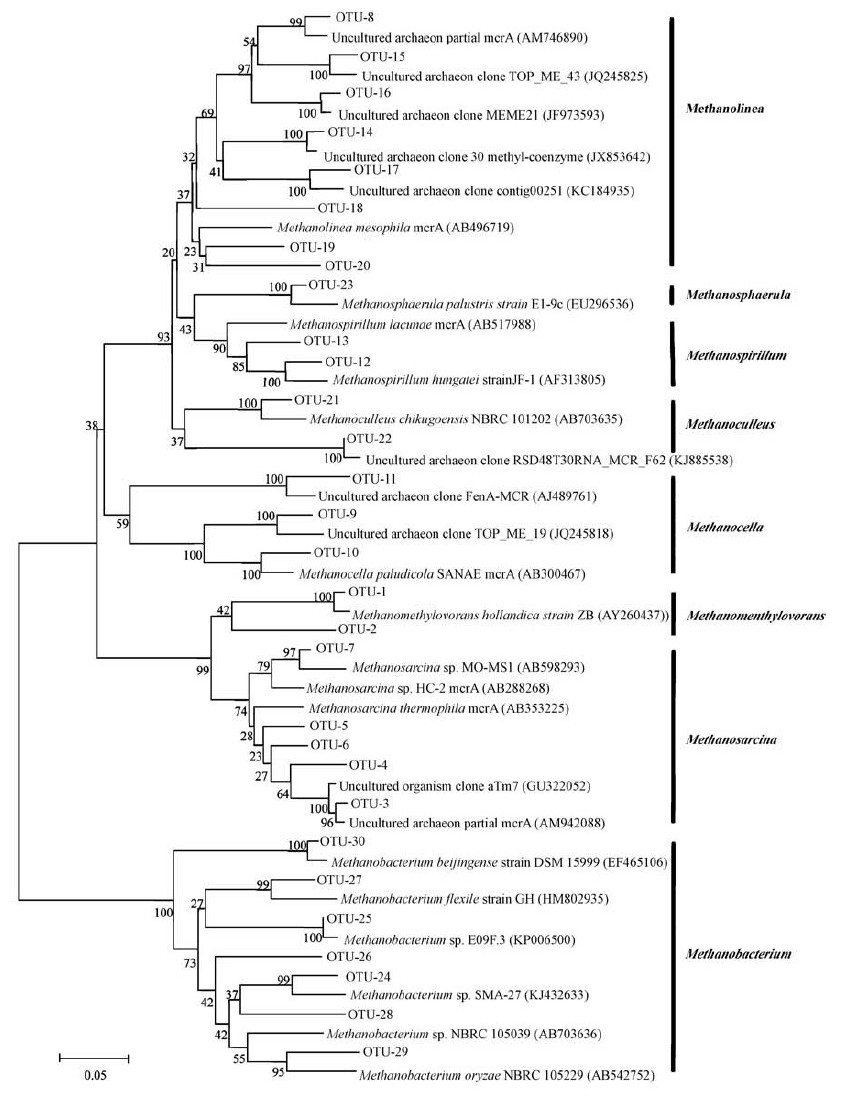 |
| 图 4 基于mcrA序列构建的浑河底泥产甲烷古菌系统发育树 Fig. 4 Phylogenetic tree of methanogens in the sediments of the Hun River based on mcrA sequence |
从浑河上游到下游,产甲烷古菌的群落结构呈现出空间变异的特征.河流上游占优势的产甲烷古菌菌属为Methanosarcina和Methanospirillum,分别约占46.2%和20.8%;中游主要菌属为Methanosarcina、Methanobacterium和Methanomethylovorans,分别约占40.9%、20.7%和15.2%;下游主要菌属为Methanosarcina和Methanobacterium,分别约占46.7%和22.1%.浑河底泥中Methanosarcina为优势菌属.这可能与浑河的水质环境有关.甲烷八叠球菌通常不要求有机生长因子,能量代谢表现为甲基胺或甲醇歧化为CH4、CO2和NH3(当利用甲基胺时).其他的代谢底物还有H2和CO2或乙酸,但从不利用甲酸.大多数可在缺氧的海水沉积物,湖水沉积物或厌氧消化器中分离到.本次在河流底泥中却能检测到甲烷八叠球菌,说明河流底泥也是其适宜的生存环境.
曾志华等(2014)通过对闽江河口区淡水和半咸水潮汐沼泽湿地土壤产甲烷菌多样性的研究发现,闽江河口区短叶茳芏潮汐淡水沼泽湿地土壤主要的优势产甲烷菌有Methanoregula、Methanosarcina和Methanobacterium,而短叶茳芏潮汐半咸水沼泽湿地土壤主要的优势产甲烷菌则转化为仅以Methanoregula为主.而本研究结果为浑河底泥中主要的优势产甲烷菌为Methanosarcina,通过对比了解到,不同的自然生态环境导致产甲烷优势菌种发生变化.
4 讨论(Results)本研究在浑河底泥中产甲烷古菌检测到4大类群:甲烷杆菌目(Methanobacteriales)、甲烷微菌目(Methanomirobiales)、甲烷胞菌目(Methanocellales)和甲烷八叠球菌目(Methanosarcinales).这与先前在其它天然湿地检出的产甲烷古菌类群基本一致(Freitag et al., 2010;Zhang et al., 2008;David et al., 2012;Galand et al., 2002;Parkes et al., 2012;Liu et al., 2012).
不同天然湿地土壤产甲烷古菌群落的垂向分布特征已有相关报道(Galand et al., 2002;Parkes et al., 2012).而对于河流产甲烷古菌群落的分布研究却很少,河流底泥中含有各种微生物和其他物质,就很有可能会存在产甲烷菌.河流周边的污染物也会通过地表径流等方式进入河流,经过长时间的积累沉淀到底泥中.河流是定向流动的,不同于湖泊等其他天然湿地,而浑河底泥中优势菌属只有一种,且产甲烷菌的群落结构会呈现空间变异的特征,说明河流的定向流动并不是驱动产甲烷菌的群落结构发生变化的主导因素,而是受到周边环境的影响.浑河上游环境主要是森林,占优势的菌种为Methanosarcina和Methanospirillum,而中游途经城市,受生活和生产的影响,主要菌属变为Methanosarcina、Methanobacterium和Methanomethylovorans,下游主要以农田和村庄为主,主要菌属又变为Methanosarcina和Methanobacterium,浑河底泥中产甲烷菌的群落结构的差异性主要是由于周边环境的影响.而为了进一步更全面准确地揭示浑河底泥产甲烷古菌的多样性及其影响因子,我们今后将结合使用其它分子生物学技术及分离、培养等常规方法对浑河底泥产甲烷古菌群落结构及其影响因子进行近一步研究.
5 结论(Conclusions)1)浑河底泥产甲烷古菌的多样性划分为4大类群: 甲烷杆菌目(Methanobacteriales)、甲烷微菌目(Methanomirobiales)、甲烷胞菌目(Methanocellales)和甲烷八叠球菌目(Methanosarcinales).
2)从河流上游到下游,产甲烷古菌的群落结构呈现出空间变异的特征.河流上游占优势的产甲烷古菌菌属为Methanosarcina和Methanospirillum,分别约占46.2%和20.8%;中游主要菌属为Methanosarcina、Methanobacterium和Methanomethylovorans,分别约占40.9%、20.7%和15.2%;下游主要菌属为Methanosarcina和Methanobacterium,分别约占46.7%和22.1%.
3)浑河底泥中Methanosarcina为优势菌属.
| [1] | Blake D R, Rowland R S. 1998.Continuing world wild increase in tropospheric methane, 1978-1987[J]. Science, 239:1129-1131 |
| [2] | Bruer S L, Cadillo-Quiroz H, Yashiro E, et al. 2006.Isolation of a novel acidophilic methanogen from an acidic peat bog[J].Nature, 442(7099):192-194 |
| [3] | Cadillo-Quiroz H, Yashiro E, Yavitt J B, et al. 2008.Characterization of the archaeal community in a minerotrophicfen and T-RFLP-directed isolation of a novel hydrogenotrophic methanogen[J].Applied and Environmental Microbiology, 74(7):2059-2068 |
| [4] | Cicerone R J, Oremland R S. 1998.Biogeochemical aspects of atmospheric methane[J]. Global Biogeochem Cycles, 2:299-327 |
| [5] | 陈中云, 闵航, 陈美慈, 等.2001.不同水稻土甲烷氧化菌和产甲烷菌数量与甲烷排放量之间相关性的研究[J].生态学报, 21(9):1493-1505 |
| [6] | David E, Wendy B, Jason K K, et al. 2012.Anaerobic metabolism in tidal fresh water wetlands:Ⅱ.Effects of plant removal on archaeal microbial communities[J].Estuaries and Coasts, doi:10.1007/s12237-012-9496-9 |
| [7] | Freitag T E, Toet S, Ineson P, et al.2010.Links between methane flux and transcriptional activities of methanogens and methane oxidizers in a blanket peat bog[J].FEMS Microbiology Ecology, 73(1):157-165 |
| [8] | Galand P E, Saarnio S, Fritze H, et al. 2002.Depth related diversity of methanogen Archaea in Finnish oligotrophic fen[J].FEMS Microbiology Ecology, 42(3):441-449 |
| [9] | 国家环境保护总局.水和废水监测分析方法编委会. 2002.水和废水监测分析方法(第4版)[M].北京:中国环境科学出版社, 227-671 |
| [10] | Hales B A, Edwards C, Ritchie D A, et al. 1996.Isolation and identification of methanogen-specific DNA from blanket bog peat by PCR amplification and sequence analysis[J].Applied and Environmental Microbiology, 62(2):668-675 |
| [11] | Intergovernmental Panel on Climate Change (IPCC).2001. Climate Change 2001:The Scientific Basis[R]. Chapter 4:Atmosphere Chemistry and Greenhouse Gases. Cambridge:Cambridge University Press |
| [12] | Jones W J, Paynter M J B, Gupta R. 1983.Characterization of Methanococcus maripaludis sp.nov., a new methanogen isolated from salt marsh sediment[J].Archives of Microbiology, 135(2):91-97 |
| [13] | Kotsyurbenko O R, Chin K J, Glagolev M V, et al. 2004.Acetoclastic and hydrogenotrophic methane production and methanogenic populations in an acidic West-Siberian peat bog[J].Environmental Microbiology, 6(11):1159-1173 |
| [14] | Liu D Y, Ding W X, Jia Z J, et al. 2012.The impact of dissolved organic carbon on the spatial variability of methanogenic archaea communities in natural wetland ecosystems across China[J].Applied Microbiology and Biotechnology, 96(1):253-256 |
| [15] | 李煜珊, 李耀明, 欧阳志云.2014.产甲烷微生物研究概况[J].环境科学, 35(5):2025-2030 |
| [16] | Miller T L, Wolin M J, De Macario E C, et al. 1982.Isolation of Methanobrevibacter smith ii from human feces[J].Applied and Environmental Microbiology, 43(1):227-232 |
| [17] | Narihiro T, Hori T, Nagata O, et al. 2011. The impact of aridification and vegetation type on changes in the community structure of methane cycling microorganisms in Japanese wetland soils[J].Bioscience, Biotechnology and Biochemistry, 75(9):1727-1734 |
| [18] | Parkes R J, Brock F, Banning N, et al. 2012.Changes in methanogenic substrate utilization and communities with depth in a salt-marsh, creek sediment in southern England[J].Estuarine, Coastal and Shelf Science, 96(1):170-178 |
| [19] | Schloss PD, Handelsman J. 2005.Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness[J]. Applied and environmental microbiology, 71(3):1501-1506 |
| [20] | Simankova M V, Kotsyurbenko O R, Lueders T, et al. 2003.Isolation and characterization of new strains of methanogens from cold terrestrial habitats[J].Systematic and Applied Microbiology, 26(2):312-318 |
| [21] | 佘晨兴, 仝川, 王维奇. 2013.互花米草沼泽湿地产甲烷古菌的多样性及垂向分布[J].环境科学学报, 34(1):186-193 |
| [22] | 佘晨兴, 仝川.2012.闽江口芦苇沼泽湿地土壤产甲烷菌群落结构的垂直分布[J].生态学报, 32(17):5299-5308 |
| [23] | Wolfe R S.An historical overview of methanogenesis[A]. 1993.In:Methanogenesis:Ecology, Physiology, Biochemistry & Genetics[M].US:Springer, 1-32 |
| [24] | 王博涵, 朱美桦, 项珍龙, 等.2015.浑河流域硅藻群落多样性的研究[J].河北渔业, 2015(1):13-16 |
| [25] | 王殳屹, 韩琳, 史弈, 等.2006.FACE对水稻土产甲烷菌和甲烷氧化菌种群及其活性的影响[J].土壤, 38 (6):768-773 |
| [26] | Yavitt J B, Yashiro E, Cadillo-Quiroz H, et al. 2012.Methanogen diversity and community composition in peatlands of the central to northern Appalachian Mountain region, North America[J].Biogeochemistry, 109(1/3):117-131 |
| [27] | Zhang G S, Tian J Q, Jiang N, et al. 2008.Methanogen community in Zoige wetland of Tibetan plateau and phenotypic characterization of a dominant uncultured methanogen cluster ZC-I[J].Environmental Microbiology, 10(7):1850-1860 |
| [28] | 张鸿龄, 孙丽娜, 罗庆. 2011. 浑河流域水体污染的季节性变化及来源[J]. 生态学杂志, 30(1):119-125 |
| [29] | 张静, 何俊仕, 周飞, 等.2011.浑河流域非点源污染负荷估算与分析[J].南水北调与水利科技, 9(6):69-73 |
| [30] | 曾志华, 杨民和, 佘晨兴, 等.2014.闽江河口区淡水和半咸水潮汐沼泽湿地土壤产甲烷菌多样性[J].生态学报, 34(10):2674-2681 |
 2016, Vol. 36
2016, Vol. 36


