2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085;
3. 中国科学院生态环境研究中心环境水质学国家重点实验室, 北京 100085
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085;
3. State Key Laboratory of Environmental Aquatic Chemistry, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085
蛋白质是生命活动的主要执行者,许多蛋白质的活性是通过翻译后的修饰作用进行调节的,其中磷酸化修饰是蛋白质最主要的翻译后修饰形式之一,哺乳动物细胞内有1/3 以上的蛋白质可以被磷酸化,在脊椎动物基因组中,有5%的基因编码的蛋白质是参与磷酸化和去磷酸化过程的蛋白激酶和磷酸(酯)酶(Alonso et al., 2004; Cohen,2002; 姜铮等,2009; 隋少卉等,2007).蛋白质磷酸化可以通过改变蛋白质的活性、 细胞定位以及与其他蛋白的相互作用来调节蛋白质的功能,在信号传导、基因表达、细胞分裂等许多生物学过程的调控中起着重要作用.例如,胞外刺激信号可以通过细胞膜上受体酪氨酸激酶和配体介导的蛋白质的磷酸化和去磷酸化,引起下游蛋白的响应,从而形成一系列信号通路,将信号由细胞膜传导至细胞核,从而调控细胞的生物学过程(Seo et al., 2004).因此,研究蛋白质的磷酸化修饰对于更好地理解机体内一些受磷酸化影响的信号通路机制是十分必要的(Nilsson,2011).
由于磷酸化蛋白质的总量低,磷酰键容易断裂,且磷酸化和去磷酸化处在动态平衡之中,因此分析蛋白质的磷酸化要比普通的蛋白质的鉴定困难得多,需要高灵敏度的方法以及大量的生物样品,而传统的分析方法如放射性自显影、免疫印迹等不仅工作量大,而且一次能够分析的磷酸化蛋白质数量有限,限制了蛋白质磷酸化的分析(Seo et al., 2004).另外,大多数磷酸化蛋白质含有一个以上的磷酸化位点,并且以不同磷酸化形式的混合物存在,因此蛋白质磷酸化的定量研究和磷酸化位点的鉴定在进一步探讨磷酸化蛋白质的功能时尤为重要(Nilsson,2011; 姜铮等,2009; Seo et al., 2004).用蛋白质组学的理念和分析方法研究蛋白质磷酸化修饰,可以高通量地识别和鉴定生物体内磷酸化蛋白质的表达及其变化,从整体上观察细胞或组织中磷酸化修饰的状态及其变化,为探讨药物刺激和环境应激下生物体受损的生物学过程提供新的视角.近年来磷酸化蛋白质组学逐渐被应用于环境毒理学的研究中,本文就磷酸化蛋白质组学技术的发展以及其在环境毒理研究中的应用展开综述.
2 磷酸化蛋白质组学的研究内容和分析策略 (Research contents and analytical strategy of phosphoproteomics)目前系统的磷酸化蛋白质组学研究包括对磷酸化蛋白的种类、磷酸化位点的鉴定以及不同条件下各种磷酸化蛋白的定量分析.一般来说,传统的蛋白质组学分析方法可以用来分析磷酸化修饰,但是由于磷酸化蛋白特性的限制,磷酸化蛋白质组分析又具有其自身的特点.首先磷酸化蛋白质表达丰度低,磷酸化的肽段很容易淹没在大量非磷酸化的肽段中,因此磷酸化蛋白质组的分析常常需要先进行磷酸化蛋白或肽段的富集以及染色或标记;其次,磷酸化肽段本身所具有的负电性使其在正离子模式的质谱分析中信号受到抑制,在MALDI(matrix assisted laser desorption ionization,基质辅助激光解析电离)源的质谱仪中磷酸化肽段的信号丰度会比其相应非磷酸化肽段的信号强度要低很多,因此,如果用这项技术分析蛋白质的磷酸化必须进行蛋白质或肽段的预分离,如一维或二维凝胶电泳分离或高效液相色谱(high performance liquid chromatography,HPLC)分离;第三,磷酰键较肽键容易断裂,使磷酸化蛋白比非磷酸化蛋白鉴定困难得多.使用常规质谱仪所配置的碰撞诱导解离(collision induced dissociation,CID)碎裂时易发生磷酸基团的中性丢失,导致碎裂不完全,造成二级谱图质量变差,需要采取其他碎裂方式,如电子捕获解离(electron capturedissociation,ECD)、电子转运解离(electrontransfer dissociation,ETD)等,以使磷酸化肽段中的磷脂键在碎裂过程中保持较好的完整性,提高二级谱图质量,增加磷酸化肽段位点鉴定的可靠性(Leitner et al., 2011; 隋少卉等,2007; 衣泰龙等,2014).下面具体介绍磷酸化蛋白质组学研究中的分析策略(图 1):
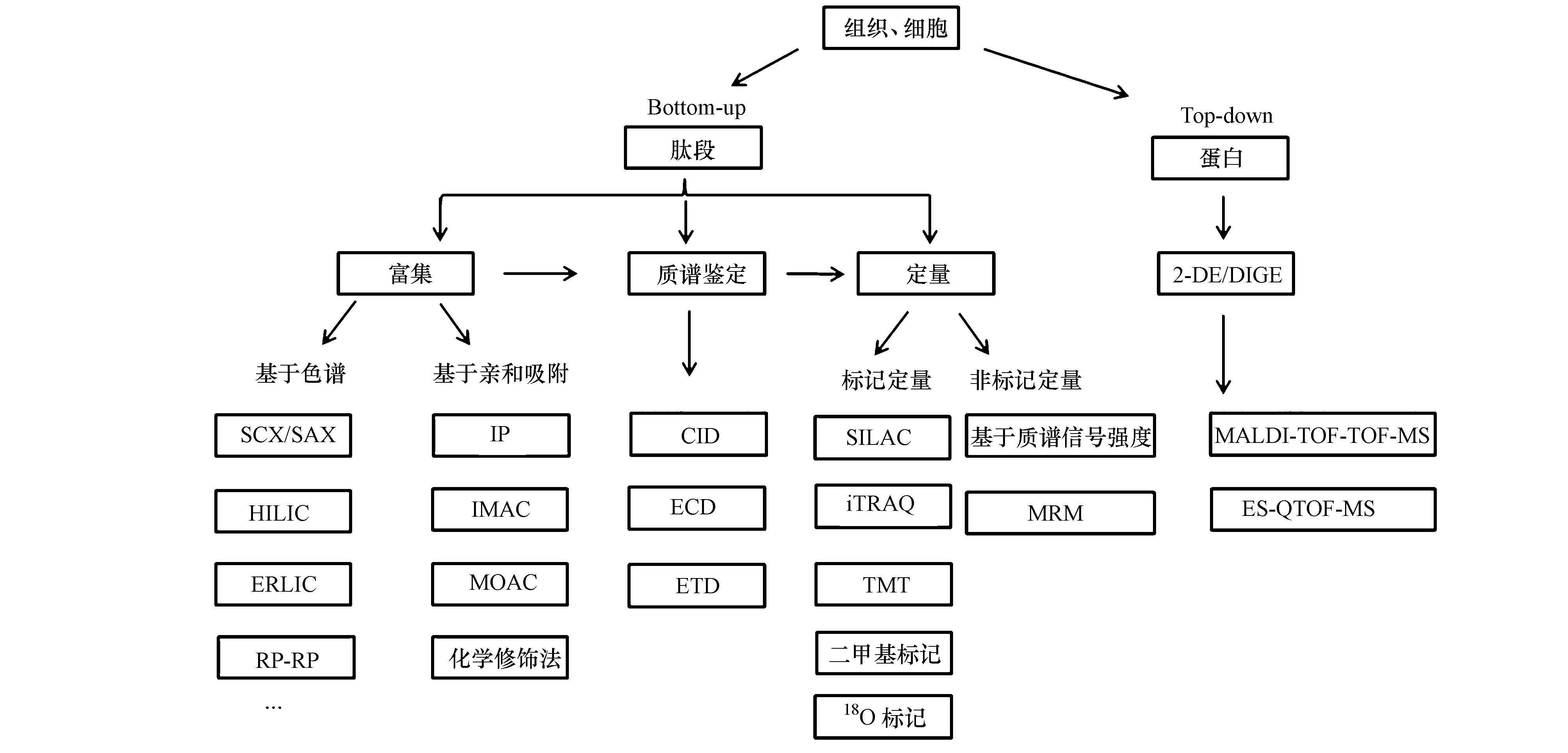 |
| 图1 磷酸化蛋白质组富集、质谱鉴定及定量策略示意图 Fig.1 trategies for phospho-specific enrichment,MS scanning and quantification |
目前常用的磷酸化肽段富集手段主要包括抗体富集法、固相金属离子亲和色谱(Immobilized metal ion affinity chromatography,IMAC)、金属氧化物亲和色谱(Metal oxide affinity chromatography,MOAC)、化学修饰法以及基于色谱技术的分离富集.
2.1.1 抗体富集法检测酪氨酸磷酸化蛋白质的单克隆抗体是已知的较好的检测磷酸化蛋白质的抗体,它具有较强的亲和力,可以有效地免疫沉淀酪氨酸磷酸化的蛋白质,可用于酪氨酸磷酸化蛋白质组学研究(Pandey et al., 2000).而针对丝氨酸、苏氨酸磷酸化修饰肽段的抗体特异性较差,但这种低亲和力的抗体用于免疫印迹(Western blot)分析中,也可取得很好的效果(Grønborg et al., 2002).然而用这一方法检测磷酸化蛋白质的局限性在于同一蛋白质可能会因为其不同的磷酸化状态而分布于二维电泳图的不同位置,从而降低了蛋白质的浓度; 同时,用非序列特异性的抗体检测丝氨酸磷酸化和苏氨酸磷酸化的蛋白质可能会产生假阳性结果(姜颖等,2003).用免疫沉淀(immunoprecipitation,IP)对磷酸化的蛋白质先进行富集,可大大降低非磷酸化蛋白质的干扰,提高检测的正确性,但如果要达到可鉴定水平,它需要大量的抗体和样品,而且鉴定出的蛋白质多为高丰度蛋白质(Collins et al., 2007).
2.1.2 IMAC富集法IMAC是利用磷酸基团与固相化的Fe3+、Ga3+、Cu2+等金属离子的高亲和力来富集磷酸肽(Posewitz et al., 1999).该方法的优点在于对每个可溶磷酸肽,不管其长度如何,都有富集作用,而且IMAC 柱洗脱下的样品可直接用于反相高效液相色谱(reversed phase high performance liquid chromatography,RP-HPLC)分析,但这种方法的局限性在于一些与IMAC柱结合能力较弱的磷酸肽或某些因有多个磷酸化位点而难以洗脱的磷酸肽有可能丢失;某些酸性基团(如天冬氨酸和谷氨酸)、电子供体(如组氨酸)也会结合到柱上,造成非特异性吸附(Posewitz et al., 1999).Ficarro等(2002)在亲和提取前对肽混合物进行甲酯化反应,使肽链中的羧基都转化为羧甲酯,不仅抑制了非特异性吸附,还减少了游离羧基对亲和位点的竞争,使磷酸肽的提取更完全.Zou研究小组(Feng et al., 2007; Zhou et al., 2008)发明了一种新的IMAC树脂,该材料是利用Zr4+或Ti4+螯合到磷酸酯聚合物上所构成,将这种材料应用于小鼠肝脏磷酸化蛋白富集,有效性及选择性均明显优于Fe3+-IMAC.
2.1.3 MOAC富集法90年代初期,以TiO2和ZrO2为代表的金属氧化物在磷酸化肽段富集的研究中逐渐兴起(Leitner et al., 2011).这种MOAC方法是利用金属氧化物与磷酸肽上的磷酸基团的亲和能力实现对磷酸肽的相对富集,该技术在对磷酸化肽段富集时的选择性和灵敏度方面都优于IMAC技术,而且其化学稳定性也比IMAC技术高,对缓冲液添加剂如盐、表面活性剂、变性组分等的耐受力更强(Leitner et al., 2011; Pinkse et al., 2004).将金属氧化物与磁性材料相结合组成的核-壳复合微球,既便于分离又节省时间,近年来得到了广泛的应用.Li等(2008)发展了一种Fe3O4@TiO2复合微球并用于小鼠肝脏磷酸化蛋白的富集,共鉴定56个磷酸化肽(65个磷酸化位点),显示了该雌性复合材料的高特异性和高灵敏度.另外,纳米材料因为具有较大的比表面积,也被应用到了IMAC和MOAC中,进一步增强了该技术对磷酸化肽段富集的巨大潜力.例如,Nelson等(2010)使用ZrO2和HfO2纳米材料对复杂样品进行磷酸化肽富集,特异性大于99%,优于TiO2纳米材料,而且这些材料经过简单再生就可以被重新利用,在磷酸化蛋白质组学领域具有良好的应用前景.此外,研究人员也发展了一些新的金属氧化物材料用于磷酸化肽的富集,取得了很好的效果,如Nb2O5(Ficarro et al., 2008),SiO2-La2O3(Jabeen et al., 2012)等.
2.1.4 化学修饰法化学修饰法是一种将磷酸肽上的磷酸基团用另一种亲和配基取代,再用亲和提取的方法从混合物中分离富集磷酸肽的方法.主要有两种取代方式:一种是使磷酸基团在碱性条件下发生β-消除反应生成双键,再用巯基乙醇通过加成反应取代磷酸基团的位置,最后通过交联剂在巯基上连接生物素,并用亲和素色谱柱分离,这种方法只适合于磷酸化丝氨酸和苏氨酸的取代(McLachlin et al., 2003);另一种是通过化学反应将胱氨加到磷酸基团上,再通过巯基乙胺与碘乙酰树脂柱的共价结合使磷酸化肽得以纯化,这种方法同时适合于磷酸化丝氨酸肽段、苏氨酸肽段和酪氨酸肽段的取代,但是为了避免副反应的影响,需要对肽段中的一些活性基团进行保护,整个反应过程比较复杂,容易造成大量的样品损失,同时该方法只能用于磷酸肽的富集,而不适用于磷酸化蛋白(Zhou et al., 2001).
2.1.5 基于色谱技术的分离富集由于磷酸集团的强负电性,磷酸化肽在离子交换色谱中的保留特性与其他未修饰的肽段形成明显对比,因此尽管离子色谱的峰容量较其他色谱低,但仍得到了广泛的应用(Nilsson,2011).在强阳离子交换色谱(strong cation-exchange chromatography,SCX)中,肽段在pH 2.7的条件下进行酸化,再利用盐度和pH的增加进行洗脱.在pH 2.7时,磷酸化肽由于本身所带的负电荷,经胰蛋白酶水解后,比其他肽段少一个正电荷,净电荷为+1,因此是SCX柱子上最早洗脱下来的成分(Hennrich et al., 2011; Hennrich et al., 2012).与SCX相反,在强阴离子交换色谱(strong anion-exchange chromatography,SAX)中,磷酸化肽保留时间较其他肽段长,同时SAX不仅可以富集磷酸化肽,而且可以分离具有不同数量的磷酸基团的磷酸化肽(Wang et al., 2010).但是SCX和SAX除了富集磷酸化肽也可以富集其他酸性肽段(如乙酰化肽),造成非特异性吸附.
亲水交互作用色谱(hydrophilic interaction chromatography,HILIC)根据蛋白/肽段的极性对样品进行分离,与反相色谱相反,它由极性固定相和亲水性流动相组成(Jandera, 2011).磷酸化肽由于极性高可以较强地结合在HILIC的固定相而与其他肽段分离(Singer et al., 2010).将HILIC和IMAC结合可以极大地提高对磷酸化肽的选择性,对海拉细胞磷酸化肽的分离中,HILIC-IMAC对磷酸化肽的选择性几乎达到100%,共鉴定914个磷酸化肽(1000个磷酸化位点),显示了极强的富集能力(McNulty et al., 2008).
静电排斥-亲水交互作用液相色谱(electrostatic repulsion-hydrophilic interaction liquid chromatography,ERLIC)与HILIC类似,但它在原有固定相加入了静电排斥特性来优化色谱的选择性.这种色谱性质的叠加通过改变有机相或溶剂的pH以及使用盐度梯度就可以实现对磷酸化肽选择性的调整(Alpert,2007).Gan等(2008)比较了ERLIC、SCX、SCX-IMAC对磷酸化肽的富集效果,在对人上皮癌细胞A431中磷酸化蛋白的分离鉴定中,共鉴定2058个磷酸化肽,其中ERLIC、SCX、SCX-IMAC鉴定得到的磷酸化肽分别占38%、5%、57%. ERLIC和SCX -IMAC同时鉴定到的磷酸化肽占二者鉴定总数的12%.由此,不同富集方法的联用有利于提高磷酸化肽的鉴定率.
2.2 磷酸化蛋白的鉴定和磷酸化位点的预测样品制备完成后,需要利用质谱技术对磷酸化肽进行检测,并进一步确定蛋白质的磷酸化位点.在真核生物中,磷酸化主要发生于丝氨酸、苏氨酸和酪氨酸等残基;而在细菌中,蛋白质主要通过天冬氨酸、谷氨酸和组氨酸等残基被磷酸化(Leitner et al., 2011; 姜颖等,2003).在磷酸化蛋白质组学研究中,磷酸肽会产生特异的离子片段,如H2PO4-,PO3-,PO2-(相对分子质量分别为97、79和63),质谱中这些离子的出现表明样品中含有磷酸肽.此外,多肽骨架片段化技术可使肽链序列重构,如果序列中包含磷酸化残基,则可以对其进行明确定位(Boersema et al., 2009; Seo et al., 2004).目前主要利用串联质谱(MS/ MS)对磷酸化位点进行确认,而在MS/ MS鉴定过程中由于低的电离效率以及中性丢失导致磷酸化肽序列的鉴定仍面临困难.MS/ MS的前体离子扫描、中性丢失扫描等分析方式可以通过分析磷酸肽产生的特征离子直接从肽混合物中找到磷酸化肽段(Boersema et al., 2009; 隋少卉等,2007).
2.2.1 撞诱导解离(collisioninduced dissociation,CID)CID碎裂方式下,可通过产生的b、y特征离子来解析肽序列(图 2).磷酸化丝氨酸和磷酸化苏氨酸残基上的磷酸基团通常不稳定,会丢失一个中性的磷酸(H3PO4),产生98 Da 质量数的变化.但是,CID模式下大部分能量用于此项碎裂,导致肽骨架的碎裂程度不够,限制了肽序列信息的获取,从而也不利于磷酸化位点的鉴定(Boersema et al., 2009).不过,中性丢失后产生的离子可以在三级质谱中进一步碎裂,这样可以获得更详细的磷酸化肽序列和磷酸化位点的信息,但是这种方法对离子的丰度有要求而且也相应地降低了碎裂肽段的总数(Engholm-Keller et al., 2013; Leitner et al., 2011).相对于丝、苏氨酸,磷酸化酪氨酸的磷酸基团比较稳定,通常产生80 Da质量数的变化(丢失HPO3).碎片离子谱上质荷比(m/z)为216的亚胺离子可以作为磷酸化酪氨酸的特征离子(Engholm-Keller et al., 2013).
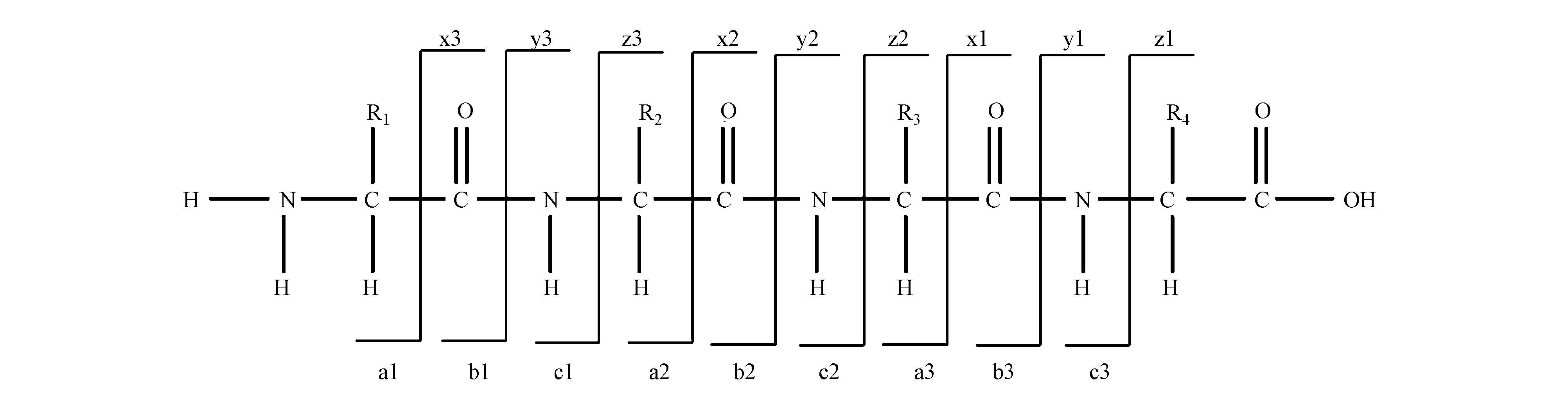 |
| 图2 肽链碎裂点示意图 Fig.2 Cleavage sites of the peptide bonds |
capturedissociation,ECD)ECD是通过低能量的自由电子与质子化的多电荷蛋白质或肽离子,在相互作用的过程中由于放热而瞬间产生碎裂,主要产生由主链上的N—C(a)键断裂而形成的c、z离子(图 2)(贾伟等,2007).ECD优先断裂二硫键,但对蛋白质和多肽的主链断裂无偏好性,主链断裂覆盖度高,保留翻译后修饰基团,基本无中性丢失.这些优点使ECD在多磷酸修饰的肽段分析中成为极具有效性的碎裂手段,可应用于大片段肽段、整体蛋白的鉴定(Top-down技术).但是,ECD只能在傅立叶变换离子回旋共振质谱(Fourier transform-ion cyclotron resonance mass spectrometry,FTICR)中应用,费用昂贵(Boersema et al., 2009; Eyrich et al., 2011; 贾伟等,2007).
2.2.3 电子转运解离(electrontransferdissociation,ETD)ETD通过将一个电子从活泼的阴离子基团上转移到带正电荷的肽段上,诱发肽段主链发生类似于ECD中的N—C(a)键的断裂,产生互补性的c和z离子(图 2)(刘科辉等,2008).ETD与ECD的碎裂机理是类似的,二者都是电子与带多电荷的肽段或蛋白质阳离子进行相互反应的过程中而产生碎裂的,电子在碎裂过程中起到了至关重要的核心作用,它们之间的主要差别是ECD的电子是自由电子,而ETD中的电子是通过小分子的阴离子提供的.另外,ETD使用四极杆离子阱而不是FTICR作为离子捕集和检测设备,离子阱质谱相对于FTICR是一种廉价、易于维护并广泛应用的质谱仪,因此基于ETD的质谱分析可以更为广泛的应用于蛋白质组学领域中(Boersema et al., 2009; 孙瑞祥等,2010).
2.3 定量磷酸化蛋白质组学研究对于各种环境条件或药物处理等外界刺激下磷酸化蛋白量的变化,目前的蛋白质组学定量技术包括基于凝胶的定量技术和基于质谱的定量技术.基于凝胶的定量方法主要以荧光染料染色Pro-Q Diamond结合双向电泳(two dimensional electrophoresis,2-DE)和差异凝胶电泳(differential in gel electrophoresis,DIGE)技术为基础.基于质谱的定量方法主要包括:代谢标记技术(metabolic labeling),化学标记技术(chemical modification with isobaric tags),无标记定量技术(label-free)等,近年来基于质谱的定量技术逐渐成为主流.
2.3.1 基于凝胶的定量技术该方法利用荧光染料Pro-Q Diamond直接对聚丙烯酰胺凝胶中的磷酸化蛋白质进行选择性染色,无需同位素或特异性抗体,通过荧光扫描仪检测可以直接显示出一维或二维凝胶电泳胶上分离的磷酸化蛋白质.Pro-Q Diamond对非磷酸化蛋白质的反应性很低,荧光强度会随着蛋白质磷酸化程度的不同而呈现出一定的量的变化.这种染料与质谱兼容,胶上的蛋白质点可以通过胶上酶切进行质谱鉴定.快速简单,易观测,成本较低,但是灵敏度低,难以检测疏水性膜蛋白和低丰度蛋白(隋少卉等,2007).
2.3.2 基于质谱的定量技术(1)代谢标记技术
细胞培养同位素标记法(stable isotope labeling with amino acids in cell culture,SILAC)是一种典型的代谢标记技术,采用含有轻、重同位素型必需氨基酸的培养基进行细胞培养,细胞传代若干代后,细胞内蛋白被同位素稳定标记,将蛋白质等量混合后进行分离和质谱鉴定,根据一级质谱图中两个同位素型肽段的面积比较进行相对定量(隋少卉等,2007).SILAC方法最主要的优点是可以实现不同的样品在细胞水平上进行混合,因而在后续步骤中减少了样品处理带来的误差.但是,并不是所有的细胞系都适合在SILAC的特殊培养基中培养;而且为了使蛋白稳定标记,SILAC技术对细胞的传代数有一定限制,母细胞系或在体外不能分裂的细胞不能进行SILAC标记(Eyrich et al., 2011; Nilsson,2011).
(2)化学标记技术
同位素标记相对和绝对定量(isobaric tags for relative and absolute quantitation,iTRAQ)和串联质谱标记(t and em mass tags,TMT)是分别由美国应用生物系统公司(Applied biosystems Inc,ABI)和Thermo公司发展的用于体外标记蛋白质或多肽的试剂.每个同重标签包括肽反应基团、平衡基团和报告基团(4重iTRAQ 试剂分子量:114-117 Da;8重iTRAQ 试剂分子量:113-119 Da;6重TMT试剂分子量:126-131 Da),在二级质谱中碎裂释放出报告离子进行蛋白质的定量(Nilsson,2011).iTRAQ技术可以对肽段进行体外标记,因此对样品来源没有限制.缺点是蛋白经过酶解后再进行标记,这个过程使得从蛋白质提取到消化为肽段的样品处理过程无法监控,可能引入误差,影响实验精度(Eyrich et al., 2011; Nilsson,2011; Wang et al., 2011).
二甲基标记(dimethyl labeling)利用甲醛与多肽N-端氨基或赖氨酸(Lys)e-氨基发生还原胺化反应,生成的席夫碱再被硼氢化钠还原,实现氨基的二甲基化,使肽段质量数相差4Da(Hsu et al., 2003).与酰基化标记的iTRAQ方法不同,二甲基标记提高了氨基的碱性,在一定程度上避免了对某些酶解肽段的离子化的抑制.二甲基标记技术快速、高效且价格低廉,可用于标记细胞或组织,目前该方法有三重同位素标记可同时分析3个样品(Nilsson,2011).二甲基标记已经广泛应用在全蛋白质组定量、蛋白翻译后修饰和蛋白相互作用的研究中(Pan et al., 2013; Wu et al., 2011).
18O标记技术(oxygen-18 labeling)是在蛋白质酶切时或酶切后加入H218O,在蛋白酶催化作用下将羧基上的2个16O替换成18O,使肽段质量数相差4 Da(隋少卉等,2007).18O标记技术不产生副产物,并能与多种磷酸化肽段富集方法连用,适于低丰度磷酸化肽段的定量标记,但是用18O进行同位素标记经常产生标记一个氧原子和两个氧原子不等的现象,即产生分子质量+2和+4两种混合的产物,一般选用全扫描的离子色谱面积(EIC)或峰强度定量,EICs在分辨率上有一定的局限性而且经常会与其他共洗脱的峰重叠.此外,18O与16O交换速率也依肽段的大小,氨基酸的类型,酶的类型以及肽段序列而有所变化(Eyrich et al., 2011; Nilsson,2011; Wang et al., 2011).
(3)无标记定量技术
基于标记的定量方法受制于样品数量的限制和复杂的标记步骤,而无标记定量技术基于质谱数据即可进行定量,其依据是蛋白质丰度与质谱数据一级谱图肽段峰强度或二级谱图数目呈现一定相关性.无标记定量技术对质谱稳定性有严格要求,稳定性差的质谱平台难以得到有统计学意义的定量信息(Soderblom et al., 2011).
质谱多反应监测(multiple reaction monitoring,MRM)技术是利用三重四级杆对特定肽段或蛋白进行定量的一种无标记定量技术(Unwin et al., 2005).MRM的原理是在第一级四极杆(Q1)中选择性检测特定母离子,母离子在第二级四极杆(Q2)中碰撞解离,在第三级四极杆(Q3)中选择性检测特定子离子,这样,只有符合设定值的母离子-子离子信号被特异性地检测到,因此MRM可以用来特异性地检测磷酸化蛋白(Eyrich et al., 2011; Sherrod et al., 2012).MRM有效地去除了大量干扰离子,具有灵敏度高、重现性好、准确度高等优点(Unwin et al., 2005).
3 磷酸化蛋白质组学在环境毒理研究中的应用 (Applications of phosphoproteomic technologies in environmental toxicology)磷酸化蛋白质组学技术的发展为研究有毒物质对生物体的毒性作用提供了有力的工具,为阐明环境污染物的毒性作用通路和作用机制提供了新的视角.目前,磷酸化蛋白质组学不仅在离体(in vitro)实验中得到开展(表 1),而且已经应用在活体(in vivo)实验中(表 2).研究的毒性作用对象主要是免疫系统、神经系统和肝脏.研究内容主要包括差异磷酸化蛋白及磷酸化位点的鉴定、生物标志物的筛选,以及有毒物质对生物体信号通路的影响和作用机制的解析.
| 表1 磷酸化蛋白质组学在离体毒理学研究中的应用 Table 1 Application of phospherproteomic technologies in in vitro toxicological studies |
| 表2 磷酸化蛋白质组学在活体毒理学研究中的应用 Table 2 Application of phospherproteomic technologies in in vivo toxicological studies |
离体实验的实验对象主要来自哺乳动物细胞,包括小鼠、大鼠和人的免疫细胞、神经细胞和癌细胞.采用的磷酸化蛋白质组学技术主要包括2-DE/DIGE结合MALDI-TOF/TOF-MS,IMAC或TiO2富集技术结合SILAC等定量技术或无标记定量技术(表 1).通过高通量的磷酸化蛋白鉴定和分析,探讨有毒物质对生物体信号通路的影响和作用机制,发展新的生物标志物.Caruso等(2014)在研究Hg2+对小鼠B淋巴细胞系WEHI-231的毒性作用时,首先利用TiO2富集结合LC-MS/MS的方法,鉴定得到样品中的磷酸化蛋白并通过生物信息学分析得到B细胞受体信号通路是Hg2+的主要作用通路且Lyn蛋白(Src家族一种蛋白磷酸激酶,参与B细胞受体信号通路)是蛋白相互作用网络中的关键蛋白,同时鉴定了7个Lyn磷酸化位点;之后利用MRM对磷酸化的Lyn蛋白进行了定量,结果表明低剂量的Hg2+可以通过对Lyn蛋白的磷酸化来刺激B细胞受体信号通路,而高剂量的Hg2+导致Lyn蛋白193、194、501、508位点的酪氨酸残基磷酸化增加,同时指出磷酸化Lyn蛋白可作为Hg2+暴露的潜在生物标志物.
SILAC是离体测试中较为常用的标记定量技术,在环境毒理学研究中也有应用.Schulz等(2013)用IMAC-HILIC-TiO2富集技术结合SILAC定量技术对1 nmol · L-1四氯二苯并-p-二 英(2,3,7,8-tetrachlorodibenzo-p-dioxin,TCDD)暴露的大鼠肝癌细胞系5L进行磷酸化蛋白质组研究,共得到5648个磷酸化肽段,2156个磷酸化蛋白;在暴露阶段(0~2 h)共有30个磷酸化蛋白发生显著变化,这些蛋白主要与基因转录调节和小GTP酶相关通路相关,其中芳香烃受体核转位蛋白(aryl hydrocarbon receptor(AhR)nuclear translocator,ARNT)在Ser77位点的磷酸化水平的增加表明TCDD对基因表达的影响存在一种新的毒性机制.
此外,其他定量技术如二甲基标记(Wu et al., 2011)、18O标记技术(Ding et al., 2008)等也有应用,可见磷酸化蛋白质组学在毒理学离体应用中的发展较为充分,并且正在不断完善.
3.2 活体(in vivo)实验活体实验的受试生物主要包括一些水生生物和哺乳动物(小鼠、大鼠).对水生生物的磷酸化蛋白质组研究采用的主要是以2-DE或DIGE技术为基础的蛋白分离手段,而对小鼠和大鼠的研究多采用基于质谱的磷酸化蛋白质组学技术(表 2).微囊藻毒素microcystin-LR(MC-LR)是水华发生过程中释放的主要次级代谢物,对蛋白磷酸酶PP1和PP2A具有潜在的抑制作用,通过经口喂食或水中投药的方式对青鳉鱼(medaka fish,Oryzias latipes)进行暴露,证明了MC-LR主要积累在生物肝脏,对青鳉鱼的肝脏、肝脏细胞质、细胞器和细胞膜组分的蛋白质组及磷酸化蛋白质组进行研究后发现若干与氧化应激、细胞骨架、信号转导以及代谢等相关的蛋白发生显著变化(上调或下调),为进一步研究MC-LR的肝脏毒性并发展生物标志物提供了重要的组学依据(Malécot et al., 2009; Mezhoud et al., 2008a; 2008b).Dai的研究小组(Shi et al., 2013; Zhang et al., 2013)用TiO2富集结合LC-MS-MS分离鉴定技术对全氟十二酸(perfluorododecanoic acid,PFDoA)暴露下的大鼠肝脏和睾丸磷酸化蛋白质组进行了分析,分别鉴定了769和1777个磷酸化蛋白,其中多数蛋白与肝脏损伤和肝脏疾病,精子发生和精子功能相关;在PFDoA肝脏毒性的研究中,糖原合成酶激酶3(glycogen synthase kinase-3,GSK3)可能是PFDoA的一个重要靶点,GSK3的抑制可能引起血脂水平的增加,同时长期暴露可能造成胰岛素信号通路的抑制;在PFDoA睾丸毒性的研究中,通路分析表明丝裂原激活蛋白激酶信号通路和细胞分裂周期蛋白2对精子活性和睾丸功能有重要的影响.
相对于离体实验,磷酸化蛋白质组学技术在毒理学活体应用中的发展还不充分,尤其在水生生物的毒理学研究中,目前的研究主要采用基于凝胶的定量技术,且一般不进行磷酸化蛋白的富集,获得的磷酸化蛋白数量和位点信息十分有限,同时不利于获得低丰度蛋白,限制了对化合物毒性机制的进一步探讨.
3.3 磷酸化蛋白质组学技术在毒性作用通路分析中的应用细胞内的大部分信号转导途径都依赖蛋白的磷酸化使得信号通过级联放大或调控效应蛋白(如激酶、转录因子、泛素连接酶等)来传递并最终激活通路.细胞的增殖、分化、免疫、传染以及对外界环境的适应都依靠蛋白的磷酸化来调节.从整体上研究信号作用网络对于揭示动态且复杂的信号转导机制是磷酸化蛋白质组学技术发展的迫切要求和必然方向(Rigbolt et al., 2012).Gruhler(2005)等利用定量磷酸蛋白质组技术IMAC-SILAC-MS3研究酵母对交配信息素的响应,发现促分裂原活化蛋白激酶信号通路及其下游的转录调节、细胞极性生长的建立以及细胞周期调节等信号通路是信息素主要的作用通路.除离体研究外,磷酸蛋白质组技术也被用于活体.在研究斑马鱼胚胎发育过程中酪氨酸磷酸化信号通路的变化时,研究人员发现黏着斑、细胞粘附、细胞分选(轴突导向和组织边界的形成)等是其中最主要的信号通路.在毒理学研究中,利用磷酸化蛋白质组技术可以快速找到化学品的靶通路,筛选出关键的作用蛋白,有助于阐明毒性作用机制(Lemeer et al., 2007).为了研究炭疽热对宿主信号响应的影响,Manes(2011)等用非标签定量技术对炭疽杆菌孢子感染的小鼠脾脏进行了磷酸化蛋白的分析,在感染后48~72 h,小鼠脾脏的磷酸化蛋白质组发生了全面的变化,感染后24 h内,有26个磷酸化肽段发生显著变化,其中8个磷酸化蛋白在宿主免疫应答通路中的作用已有明确注释,这些蛋白可能成为炭疽热病发的早期预警生物标志物.但是在这26个显著响应的磷酸化肽段有16个肽段(61.5%)的功能不明确,一定程度上限制了炭疽杆菌毒性作用通路的进一步分析,因此发展磷酸化蛋白质组的功能特征化分析方法和生物信息学工具对磷酸化蛋白质组学研究具有重要的意义.
4 展望(Future prospects)1)目前磷酸化蛋白质组学在环境毒理学研究中的研究方法上多采用基于凝胶的定量技术,且使用该技术分离鉴定磷酸化蛋白前一般不进行磷酸化蛋白的富集,获得的磷酸化蛋白数量和位点信息有限,今后可以在已有研究的基础上发展基于质谱的定量技术,以获得更加充分的信息.
2)现有的磷酸化修饰研究技术只能发现或分析那些结构性磷酸化位点,而蛋白质功能的变化往往要借助于瞬时的磷酸化变化,因此蛋白质磷酸化的动态变化研究将是在成熟的磷酸化蛋白质组技术基础上的必然选择.
3)对于多肽水平(bottom-up)的磷酸化蛋白质组学策略,很难对复杂的磷酸化情况给予准确的定量信息,如一个蛋白的磷酸化是否受到另一位点的磷酸化或其他修饰作用调控,同一蛋白的两个位点是否互斥等.通过使用不同的蛋白水解酶有助于克服这个缺陷,然而样品消耗和分析时间会大量增加.在深入研究化合物的毒性作用通路时,有必要针对目标蛋白进行磷酸化的进一步验证.
4)目前磷酸化蛋白质组学在磷酸化位点的研究上仍存在一定的难度,这不仅源于磷酸化位点鉴定的难度,而且源于解析磷酸化位点功能的难度,在研究化合物的毒性作用时,常有相当一部分蛋白的磷酸化位点功能未知而影响对其毒性机制的进一步探讨,因此进一步发展更完善的功能特征化分析方法和生物信息学工具是未来磷酸化蛋白质组学研究的一个重要方向.
5)随着翻译后修饰蛋白质组学研究方法的不断成熟,不同的翻译后修饰的相互作用(如磷酸化与泛素化、糖基化、乙酰化等的相互作用)研究正在成为研究热点,因此多种修饰化的作用网络研究对于探讨和揭示污染物对复杂生物体功能影响及其作用机制的又一方向.
术语表(Abbreviations)CID,collision induced dissociation; 碰撞诱导解离
2-DE,two dimensional electrophoresis; 双向电泳
DIGE,differential in gel electrophoresis; 差异凝胶电泳
ECD,electron capturedissociation; 电子捕获解离
ERLIC,electrostatic repulsion-hydrophilic interaction liquid chromatography; 静电排斥-亲水交互作用液相色谱
ESI,electrospray ionization; 电喷雾电离
ETD,electrontransfer dissociation; 电子转运解离
FTICR,Fourier transform-ion cyclotron resonance mass spectrometry; 傅里叶变换-离子回旋共振质谱
HILIC,hydrophilic interaction chromatography; 亲水交互作用色谱
IMAC,Immobilized metal ion affinity chromatography; 固相金属离子亲和色谱
IP,immunoprecipitation; 免疫沉淀
iTRAQ,isotope tags for relative and absolute quantitation; 同位素标签相对绝对定量
LC,liquid chromatography; 液相色谱
LC-MS/MS,liquid chromatography coupled to t and em mass spectrometry; 液相质谱-串联质谱联用
MALDI,matrix assisted laser desorption ionization; 基质辅助激光解析电离
MOAC,Metal oxide affinity chromatography; 金属氧化物亲和色谱
MRM,multiple reaction monitoring; 质谱多反应监测
MS,mass spectrometry; 质谱
RP,reverse phase chromatography; 反相色谱
SAX,strong anion-exchange chromatography; 强阴离子交换色谱
SCX,strong cation-exchange chromatography; 强阳离子交换色谱
SILAC,stable isotope labeling with amino acids in cell culture; 细胞培养同位素标记
TMT,t and em mass tags; 串联质谱标记
TOF,time-of-flight; 飞行时间
| [1] | Alonso A, Sasin J, Bottini N, et al. 2004. Protein tyrosine phosphatases in the human genome[J]. Cell, 117 (6):699-711 |
| [2] | Alpert A J. 2007. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides[J]. Analytical Chemistry, 80 (1):62-76 |
| [3] | Boersema P J, Mohammed S, Heck A J R. 2009. Phosphopeptide fragmentation and analysis by mass spectrometry[J]. Journal of Mass Spectrometry, 44 (6):861-878 |
| [4] | Caruso J A, Stemmer P M, Dombkowski A, et al. 2014. A systems toxicology approach identifies Lyn as a key signaling phosphoprotein modulated by mercury in a B lymphocyte cell model[J]. Toxicology and Applied Pharmacology, 276 (1):47-54 |
| [5] | Chitta K R, Landero Figueroa J A, Caruso J A, et al. 2013. Selenium mediated arsenic toxicity modifies cytotoxicity, reactive oxygen species and phosphorylated proteins[J]. Metallomics, 5 (6):673-685 |
| [6] | Cohen P. 2002. The origins of protein phosphorylation[J]. Nature Cell Biology, 4 (5):E127-130 |
| [7] | Collins M O, Yu L, Choudhary J S. 2007. Analysis of protein phosphorylation on a proteome-scale[J]. Proteomics, 7 (16):2751-2768 |
| [8] | D'Andrea G, Venditti S, Di Francesco L, et al. 2012. Differential phosphoproteome analysis of K562 cells exposed to 3'-azido-3'-deoxythymidine (AZT)[J]. Current Proteomics, 9 (1):40-54 |
| [9] | Dash S, Chandramouli K H, Zhang Y, et al. 2012. Effects of poly-ether B on proteome and phosphoproteome expression in biofouling Balanus amphitrite cyprids[J]. Biofouling, 28 (4):405-415 |
| [10] | Ding S J, Wang Y, Jacobs J M, et al. 2008. Quantitative phosphoproteome analysis of lysophosphatidic acid induced chemotaxis applying dual-step 18O labeling coupled with immobilized metal-ion affinity chromatography[J]. Journal of Proteome Research, 7 (10):4215-4224 |
| [11] | Engholm-Keller K, Larsen M R. 2013. Technologies and challenges in large-scale phosphoproteomics[J]. Proteomics, 13 (6):910-931 |
| [12] | Eyrich B, Sickmann A, Zahedi R P. 2011. Catch me if you can:mass spectrometry-based phosphoproteomics and quantification strategies[J]. Proteomics, 11 (4):554-570 |
| [13] | Feng S, Ye M, Zhou H, et al. 2007. Immobilized zirconium ion affinity chromatography for specific enrichment of phosphopeptides in phosphoproteome analysis[J]. Molecular & Cellular Proteomics, 6 (9):1656-1665 |
| [14] | Ficarro S B, McCleland M L, Stukenberg P T, et al. 2002. Phosphoproteome analysis by mass spectrometry and its application to Saccharomyces cerevisiae[J]. Nature Biotechnology, 20 (3):301-305 |
| [15] | Ficarro S B, Parikh J R, Blank N C, et al. 2008. Niobium(V) oxide (Nb2O5):application to phosphoproteomics[J]. Analytical Chemistry, 80 (12):4606-4613 |
| [16] | Gan C S, Guo T, Zhang H, et al. 2008. A comparative study of electrostatic repulsion-hydrophilic interaction chromatography (ERLIC) versus SCX-IMAC-based methods for phosphopeptide isolation/enrichment[J]. Journal of Proteome Research, 7 (11):4869-4877 |
| [17] | Grønborg M, Kristiansen T Z, Stensballe A, et al. 2002. A mass spectrometry-based proteomic approach for identification of serine/threonine-phosphorylated proteins by enrichment with phospho-specific antibodies:identification of a novel protein, Frigg, as a protein kinase A substrate[J]. Molecular & Cellular Proteomics, 1 (7):517-527 |
| [18] | Gramage E, Herradón G, Martín Y B, et al. 2013. Differential phosphoproteome of the striatum from pleiotrophin knockout and midkine knockout mice treated with amphetamine:correlations with amphetamine-induced neurotoxicity[J]. Toxicology, 306:147-156 |
| [19] | Gratia S, Kay L, Michelland S, et al. 2012. Cardiac phosphoproteome reveals cell signaling events involved in doxorubicin cardiotoxicity[J]. Journal of Proteomics, 75 (15):4705-4716 |
| [20] | Gruhler A, Olsen J V, Mohammed S, et al. 2005. Quantitative phosphoproteomics applied to the yeast pheromone signaling pathway[J]. Molecular & Cellular Proteomics, 4 (3):310-327 |
| [21] | Harder L M, Bunkenborg J, Andersen J S. 2013. Inducing autophagy[J]. Autophagy, 10 (2):339-355 |
| [22] | Hennrich M L, Groenewold V, Kops G J P L, et al. 2011. Improving depth in phosphoproteomics by using a strong cation exchange-weak anion exchange-reversed phase multidimensional separation approach[J]. Analytical Chemistry, 83 (18):7137-7143 |
| [23] | Hennrich M L, van den Toorn H W P, Groenewold V, et al. 2012. Ultra acidic strong cation exchange enabling the efficient enrichment of basic phosphopeptides[J]. Analytical Chemistry, 84 (4):1804-1808 |
| [24] | Hsu J L, Huang S Y, Chow N H, et al. 2003. Stable-isotope dimethyl labeling for quantitative proteomics[J]. Analytical Chemistry, 75 (24):6843-6852 |
| [25] | Huang Z, Ichihara S, Oikawa S, et al. 2015. Hippocampal phosphoproteomics of F344 rats exposed to 1-bromopropane[J]. Toxicology and Applied Pharmacology, 282 (2):151-160 |
| [26] | Imanishi S, Harada K. 2004. Proteomics approach on microcystin binding proteins in mouse liver for investigation of microcystin toxicity[J]. Toxicon, 43 (6):651-659 |
| [27] | Jabeen F, Hussain D, Fatima B, et al. 2012. Silica-lanthanum oxide:pioneer composite of rare-earth metal oxide in selective phosphopeptides enrichment[J]. Analytical Chemistry, 84 (23):10180-10185 |
| [28] | Jandera P. 2011. Stationary and mobile phases in hydrophilic interaction chromatography:a review[J]. Analytica Chimica Acta, 692 (1/2):1-25 |
| [29] | 贾伟, 应万涛, 钱小红. 2007. 另辟蹊径——串联质谱肽段断裂的新军"电子捕获解离"[J]. 质谱学报, 28 (1):55-64 |
| [30] | 姜颖, 徐朗莱, 贺福初. 2003. 质谱技术解析磷酸化蛋白质组[J]. 生物化学与生物物理进展, 30 (3):350-356 |
| [31] | 姜铮, 王芳, 何湘, 等. 2009. 蛋白质磷酸化修饰的研究进展[J]. 生物技术通讯, 20 (2):233-237 |
| [32] | Kang T H, Bae K H, Yu M, et al. 2007. Phosphoproteomic analysis of neuronal cell death by glutamate-induced oxidative stress[J]. Proteomics, 7 (15):2624-2635 |
| [33] | Leitner A, Sturm M, Lindner W. 2011. Tools for analyzing the phosphoproteome and other phosphorylated biomolecules:a review[J]. Analytica Chimica Acta, 703 (1):19-30 |
| [34] | Lemeer S, Ruijtenbeek R, Pinkse M W H, et al. 2007. Endogenous phosphotyrosine signaling in zebrafish embryos[J]. Molecular & Cellular Proteomics, 6 (12):2088-2099 |
| [35] | Li Y, Xu X, Qi D, et al. 2008. Novel Fe3O4@TiO2 core-shell microspheres for selective enrichment of phosphopeptides in phosphoproteome analysis[J]. Journal of Proteome Research, 7 (6):2526-2538 |
| [36] | 刘科辉, 钱小红. 2008. 串联质谱肽段断裂新技术-电子转移解离及其在蛋白质组学中的应用[J]. 质谱学报, 29 (2):115-119 |
| [37] | Malécot M, Mezhoud K, Marie A, et al. 2009. Proteomic study of the effects of microcystin-LR on organelle and membrane proteins in medaka fish liver[J]. Aquatic Toxicology, 94 (2):153-161 |
| [38] | Manes N P, Dong L, Zhou W, et al. 2011. Discovery of mouse spleen signaling responses to anthrax using label-free quantitative phosphoproteomics via mass spectrometry[J]. Molecular & Cellular Proteomics, 10 (3):M110.000927 |
| [39] | McLachlin D T, Chait B T. 2003. Improved β-elimination-based affinity purification strategy for enrichment of phosphopeptides[J]. Analytical Chemistry, 75 (24):6826-6836 |
| [40] | McNulty D E, Annan R S. 2008. Hydrophilic interaction chromatography reduces the complexity of the phosphoproteome and improves global phosphopeptide isolation and detection[J]. Molecular & Cellular Proteomics, 7 (5):971-980 |
| [41] | Mezhoud K, Bauchet A L, Château-Joubert S, et al. 2008a. Proteomic and phosphoproteomic analysis of cellular responses in medaka fish (Oryzias latipes) following oral gavage with microcystin-LR[J]. Toxicon, 51 (8):1431-1439 |
| [42] | Mezhoud K, Praseuth D, Puiseux-Dao S, et al. 2008b. Global quantitative analysis of protein expression and phosphorylation status in the liver of the medaka fish (Oryzias latipes) exposed to microcystin-LR:I. Balneation study[J]. Aquatic Toxicology, 86 (2):166-175 |
| [43] | Nelson C A, Szczech J R, Dooley C J, et al. 2010. Effective enrichment and mass spectrometry analysis of phosphopeptides using mesoporous metal oxide nanomaterials[J]. Analytical Chemistry, 82 (17):7193-7201 |
| [44] | Nilsson C L. 2011. Advances in quantitative phosphoproteomics[J]. Analytical Chemistry, 84 (2):735-746 |
| [45] | Nirujogi R S, D Wright J, Manda S S, et al. 2014. Phosphoproteomic analysis reveals compensatory effects in the piriform cortex of VX nerve agent exposed rats[J]. Proteomics, 15 (2/3):487-499 |
| [46] | Nogueira da Costa A, Keen J N, Wild C P, et al. 2011. An analysis of the phosphoproteome of immune cell lines exposed to the immunomodulatory mycotoxin deoxynivalenol[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 1814 (7):850-857 |
| [47] | Osman A M, van Loveren H. 2012. Phosphoproteomic analysis of mouse thymoma cells treated with tributyltin oxide:TBTO affects proliferation and energy sensing pathways[J]. Toxicological Sciences, 126 (1):84-100 |
| [48] | Pan X, Whitten D A, Wu M, et al. 2013. Early phosphoproteomic changes in the mouse spleen during deoxynivalenol-induced ribotoxic stress[J]. Toxicological Sciences, 135 (1):129-143 |
| [49] | Pandey A, Podtelejnikov A V, Blagoev B, et al. 2000. Analysis of receptor signaling pathways by mass spectrometry:identification of Vav-2 as a substrate of the epidermal and platelet-derived growth factor receptors[J]. Proceedings of the National Academy of Sciences, 97 (1):179-184 |
| [50] | Pinkse M W H, Uitto P M, Hilhorst M J, et al. 2004. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-nanoLC-ESI-MS/MS and titanium oxide precolumns[J]. Analytical Chemistry, 76 (14):3935-3943 |
| [51] | Pollio G, Hoozemans J J M, Andersen C A, et al. 2008. Increased expression of the oligopeptidase THOP1 is a neuroprotective response to Aβ toxicity[J]. Neurobiology of Disease, 31 (1):145-158 |
| [52] | Posewitz M C, Tempst P. 1999. Immobilized gallium(III) affinity chromatography of phosphopeptides[J]. Analytical Chemistry, 71 (14):2883-2892 |
| [53] | Qian P Y, Wong Y H, Zhang Y. 2010. Changes in the proteome and phosphoproteome expression in the bryozoan Bugula neritina larvae in response to the antifouling agent butenolide[J]. Proteomics, 10 (19):3435-3446 |
| [54] | Rigbolt K T G, Blagoev B. 2012. Quantitative phosphoproteomics to characterize signaling networks[J]. Seminars in Cell & Developmental Biology, 23 (8):863-871 |
| [55] | Schulz M, Brandner S, Eberhagen C, et al. 2013. Quantitative phosphoproteomic analysis of early alterations in protein phosphorylation by 2,3,7,8-tetrachlorodibenzo-p-dioxin[J]. Journal of Proteome Research, 12 (2):866-882 |
| [56] | Seo J, Lee K J. 2004. Post-translational modifications and their biological functions:proteomic analysis and systematic approaches[J]. Journal of Biochemistry and Molecular Biology, 37 (1):35-44 |
| [57] | Sherrod S D, Myers M V, Li M, et al. 2012. Label-free quantitation of protein modifications by pseudo selected reaction monitoring with internal reference peptides[J]. Journal of Proteome Research, 11 (6):3467-3479 |
| [58] | Shi Z, Hou J, Guo X, et al. 2013. Testicular phosphoproteome in perfluorododecanoic acid-exposed rats[J]. Toxicology Letters, 221 (2):91-101 |
| [59] | Singer D, Kuhlmann J, Muschket M, et al. 2010. Separation of multiphosphorylated peptide isomers by hydrophilic interaction chromatography on an aminopropyl phase[J]. Analytical Chemistry, 82 (15):6409-6414 |
| [60] | Soderblom E J, Philipp M, Thompson J W, et al. 2011. Quantitative label-free phosphoproteomics strategy for multifaceted experimental designs[J]. Analytical Chemistry, 83 (10):3758-3764 |
| [61] | 隋少卉, 王京兰, 蔡耘, 等. 2007. 磷酸化蛋白质组学分析和定量技术的研究进展[J]. 生物化学与生物物理进展, 34 (3):240-245 |
| [62] | 孙瑞祥, 董梦秋, 迟浩, 等. 2010. 基于电子捕获裂解/电子转运裂解串联质谱技术的蛋白质组学研究[J]. 生物化学与生物物理进展, 37 (1):94-102 |
| [63] | Unwin R D, Griffiths J R, Leverentz M K, et al. 2005. Multiple reaction monitoring to identify sites of protein phosphorylation with high sensitivity[J]. Molecular & Cellular Proteomics, 4 (8):1134-1144 |
| [64] | Vicente-Rodríguez M, Gramage E, Herradón G, et al. 2013. Phosphoproteomic analysis of the striatum from pleiotrophin knockout and midkine knockout mice treated with cocaine reveals regulation of oxidative stress-related proteins potentially underlying cocaine-induced neurotoxicity and neurodegeneration[J]. Toxicology, 314 (1):166-173 |
| [65] | Wang F, Han G, Yu Z, et al. 2010. Fractionation of phosphopeptides on strong anion-exchange capillary trap column for large-scale phosphoproteome analysis of microgram samples[J]. Journal of Separation Science, 33 (13):1879-1887 |
| [66] | Wang F, Song C, Cheng K, et al. 2011. Perspectives of comprehensive phosphoproteome analysis using shotgun strategy[J]. Analytical Chemistry, 83 (21):8078-8085 |
| [67] | Wen G, Hong M, Calaf G M, et al. 2010. Phosphoproteomic profiling of arsenite-treated human small airway epithelial cells.[J]. Oncology Reports, 23 (2):405-412 |
| [68] | Wu C J, Chen Y W, Tai J H, et al. 2011. Quantitative phosphoproteomics studies using stable isotope dimethyl labeling coupled with IMAC-HILIC-nanoLC-MS/MS for estrogen-induced transcriptional regulation[J]. Journal of Proteome Research, 10 (3):1088-1097 |
| [69] | 衣泰龙, 田苗苗, 杨晓明, 等. 2014. 磷酸化蛋白质组学的新进展及其在肝脏生理和病理机制中的应用[J]. 生物工程学报, 30 (7):1004-1017 |
| [70] | Zhang H, Hou J, Cui R, et al. 2013. Phosphoproteome analysis reveals an important role for glycogen synthase kinase-3 in perfluorododecanoic acid-induced rat liver toxicity[J]. Toxicology Letters, 218 (1):61-69 |
| [71] | Zhou H, Watts J D, Aebersold R. 2001. A systematic approach to the analysis of protein phosphorylation[J]. Nature Biotechnology, 19 (4):375-378 |
| [72] | Zhou H, Ye M, Dong J, et al. 2008. Specific phosphopeptide enrichment with immobilized titanium ion affinity chromatography adsorbent for phosphoproteome analysis[J]. Journal of Proteome Research, 7 (9):3957-3967 |
 2016, Vol. 36
2016, Vol. 36


