多氯联苯(Polychlorinated Biphenyls,PCBs)作为首批被列入《斯德哥尔摩公约》的持久性有机污染物,具有环境持久性、生物蓄积性、半挥发性和“三致”毒性等特点,严重威胁着人类健康和生态环境安全.在各类环境介质中,土壤被认为是PCBs最大的库和汇(Dalla et al., 2005),对PCBs污染土壤的修复已成为国内外研究者共同关注的问题.
植物-微生物联合修复因费用低、环境友好而被认为是最有前景的修复手段.PCBs的好氧降解主要以共代谢的方式进行,该过程涉及的关键酶为联苯双加氧酶(bphA),联苯可作为共代谢底物诱导bphA基因的表达(Brunner et al., 1985),但本身的毒性限制了其在PCBs原位修复中的应用.植物次生代谢产物(Secondary Plant Metabolites,SPMEs)是一类与外源有机污染物具有相似分子结构的天然物质,可由植物根系分泌,可能作为共代谢底物促进PCBs等持久性有机污染物的降解(Singer et al., 2003).研究表明,多种SPMEs,如酚类、萜烯类和黄酮类均能促进 PCBs的降解(Dudášová et al., 2012;Pham et al., 2012;Toussaint et al., 2012).
水杨酸作为一种植物次生代谢产物,能够促进萘、菲及除草剂等外源有机物的降解(Powell et al., 2008;Gottfried et al., 2010;Lu et al., 2015),但对PCBs的作用却存在争议.Singer等(2000)发现水杨酸能够促进Ralstonia eutrophus H850对PCBs的降解,但又有研究表明,水杨酸对PCBs的降解没有促进作用(Luo et al., 2008; Slater et al., 2011),以上3个研究都基于实验室的微宇宙培养.而最近Hu等(2014)对Ralstonia eutrophus H850和Pseudomonas fluorescence P2W两株PCB降解菌的纯培养研究发现,水杨酸能促进该两株菌对低氯代PCBs的降解.
为更接近污染土壤植物修复的实际情况,本文以浙江台州电子垃圾拆解地附近PCBs污染土壤为对象,在温室中模拟盆栽实验.鉴于污染土壤的植物修复研究中,通常会施肥促进植物和微生物生长,本文设置了施肥处理,考察在施肥基础上添加不同剂量水杨酸对土中PCBs的去除、脱氢酶活、微生物群落结构及bphA基因相对丰度的影响.以期为今后进一步揭示水杨酸影响PCBs降解的分子机制,以及实现植物-微生物原位修复PCBs污染土壤奠定基础.
2 材料与方法(Materials and methods) 2.1 供试材料供试土壤采自浙江台州电子垃圾拆解地附近农田.土壤的基本理化性质如下:pH为5.96,有机质含量为2.1%,阳离子交换量为7.67 cmol · kg-1,PCBs总含量为 2602.25 μg · kg-1.
实验在浙江大学华家池校区温室中进行,设置5个处理:A0(空白对照)、A1(施肥)、A2(施肥+5 mmol · kg-1水杨酸)、A3(施肥+10 mmol · kg-1水杨酸)、A4(施肥+20 mmol · kg-1水杨酸).施肥方式为:NH4NO3 285 mg · kg-1,KH2PO4 350 mg · kg-1.每个处理设置3个平行,采用称重法补给水分,30 d后采0~10 cm表层土为研究土壤.
PCBs混标(包括PCB 18、16/32、31、28/33、22、45、52、47/48/49、41/64、37/42/44、40、74、66/70、56/60、101、99、83、97、87、85/136、110、82、108/118/149、146/153、141、137、129/138、178、175、159/187、128、185、177、180和170)和回收率指示物PCB 209购自美国Accust and ard公司,丙酮、正己烷(色谱纯)购自美国TEDIA公司,其他试剂为国产分析纯.
2.2 PCB分析PCBs的分析方法参考文献(Li et al., 2013).准确称取过筛后样品5 g,加入回收率指示物PCB209,用200 mL正己烷和丙酮的混合溶液(V/V=1 ∶ 1)恒温水浴24 h.提取液在旋转蒸发仪上浓缩后经弗罗里土/无水硫酸钠层析柱净化,将洗脱液浓缩后氮吹定容至1 mL,待分析.
色谱条件:气相色谱GC-μECD(Agilent 7890N),HP-5石英毛细管柱(30 m×0.25 mm×0.25 μm),检测器放射源为63Ni,进样口温度和检测器温度为300 ℃,载气为高纯氮气(恒流1.0 mL · min-1),不分流进样1 μL.升温程序:初温90 ℃,以15 ℃ · min-1升至180 ℃,之后以1 ℃ · min-1升至220 ℃并保持5 min,以7 ℃ · min-1升至290 ℃并保持2 min(总时间63 min).
2.3 土壤脱氢酶活性的测定土壤脱氢酶活测定参考《土壤酶及其研究法》(关荫松,1986),脱氢酶活性以每克土壤中TPF质量(μg)表示.
2.4 PCR-DGGE 2.4.1 PCR扩增土壤DNA的提取采用美国MO-BIO试剂盒法,于-20 ℃冰箱保存.所用正向引物为F357:5′-CCTACGGGAGGCAGCAG-3′,反向引物R518:5′-ATTACCGCGGCTGCTGG-3′,40个GC 夹(5′-CGCCCGCCGCGCCCCGCGC CCGGCCCGCCGCC CCCGCCCC-3′)加到F357前.PCR反应体系如下:1 μL浓度为10 ng · μL-1的DNA模板,1 μL正、反两种引物(各10 μmol),4 μL的dNTPs(每种2.5 mmol),5 μL的10×PCR Buffer(含MgCl2),5 U的TaqDNA聚合酶和适量的双蒸水补足到50 μL.PCR扩增条件:94 ℃预变性5 min;94 ℃变性50 s,58 ℃退火50 s,72 ℃延伸40 s,30个循环;最后72 ℃延伸10 min.PCR反应产物用1.0%琼脂糖凝胶电泳检测.
2.4.2 变性梯度凝胶电泳利用DGGE分离PCR产物,变性梯度为30%~60%,缓冲液为1×TAE,上样量为40~45 μL的PCR产物加入20 μL 6×溴酚蓝二甲苯氰溶液,电压160 V,温度60 ℃,时间300~350 min.电泳后凝胶采用SYBR-Green1Ⅰ进行染色.
2.4.3 DNA片段回收、克隆与测序将DGGE 图谱上的主要条带切胶回收,纯化提取其中DNA模板.使用不含GC 夹子的相同引物进行PCR 扩增.纯化后的扩增片段连接至pMD19-T vector 载体,转至大肠杆菌感受态细胞并进行蓝白斑筛选.经鉴定分子克隆成功的样品进行测序(上海英捷公司),序列结果通过BLAST与GenBank中核酸数据进行比对分析.
2.5 荧光定量PCR以V3区域总细菌通用引物作绝对定量,引物参照文献(Li et al., 2013).20 μL反应体系为:10 μL的SYBR-GreenⅠ、0.4 μL的10 μmol · L-1双向引物、2 μL的DNA、7.2 μL的H2O.反应程序为:95 ℃预变性1 min;95 ℃预变性5 s,55.6 ℃退火15 s,72 ℃延伸15 s,36个循环;溶解曲线为72 ℃逐渐升温至95 ℃.扩增效率(E)和R2分别为101.2%与0.991.
2.6 数据分析方法本文用MS Excel分析实验数据,SPSS 16.0单因素方差分析(ANOVA)差异显著性.
Shannon指数:使用Quantity One软件(Bio-Rad)分析DGGE 指纹图谱中微生物群落结构,Shannon 指数(H)的计算公式为:
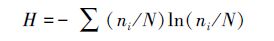
主成分分析:根据分析软件得到DGGE的条带配对图,在条带出现的地方定义1,未出现的地方记为0,得到DGGE条带配对的二次矩阵后,用SPSS 16.0进行主成分分析.
3 结果与讨论(Results and discussion) 3.1 土壤中PCBs的去除温室处理30 d后PCBs总去除率见表 1,不同氯代PCBs去除率见图 1.与对照相比,施肥和施肥基础上添加水杨酸显著提高了PCBs的去除率(p<0.05),且对三氯联苯的去除效果最好(28.98%~34.53%),而对七氯联苯的去除效果则相对较低(21.33%~26.01%).随着水杨酸添加量的增加,PCBs的去除率呈先增加后减小的趋势.
| 表1 土壤PCBs总去除率、脱氢酶活性及Shannon指数 Table 1 Total PCB removal,soil dehydrogenase activity and Shannon index in soils |
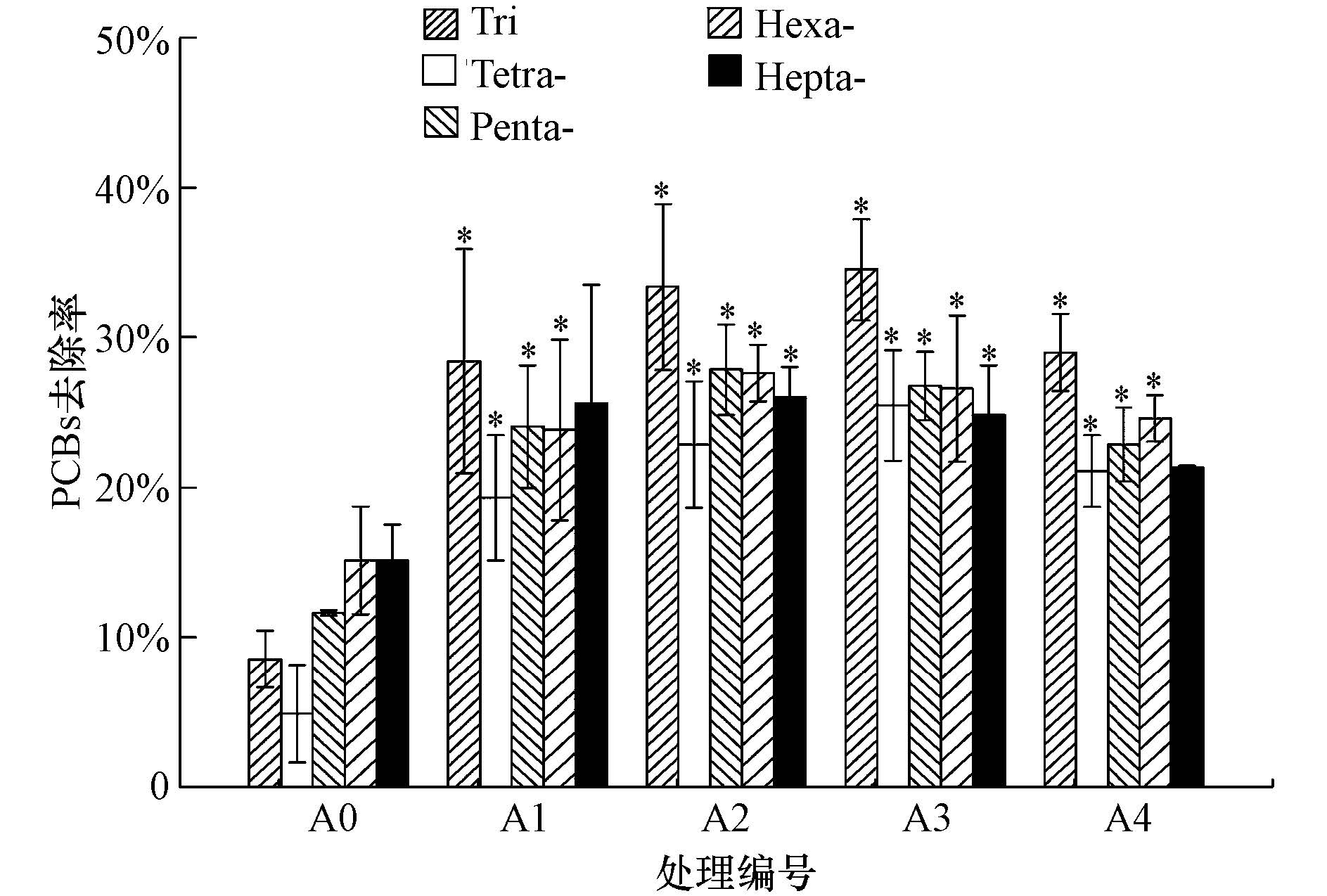 |
| 图1 不同氯代PCBs去除率(*p<0.05,下同) Fig.1 Removal of tri-,tetra-,penta-,hexa-, and hepta-CBs in different soil treatments |
本研究中水杨酸对PCBs的去除有一定促进作用,且水杨酸浓度是影响PCBs去除效果的因素之一.有关水杨酸对PCBs降解的促进(Singer et al., 2000)和抑制作用(Slater et al., 2011)前人均有报道.本研究发现,水杨酸对低氯代PCBs的去除效果更好,这与Hu等(2014)的研究结果一致.这一方面是由于好氧条件下低氯代PCBs比高氯代PCBs更易降解(Field et al., 2008),另一方面可能是由于水杨酸能够选择性地促进某些低氯代PCBs降解菌的代谢.
3.2 土壤脱氢酶活性分析土壤脱氢酶是土壤中重要的氧化还原酶,在外源有机化合物的分解转化中起重要作用(Margesin et al., 2000).与对照相比,施肥显著提高了土壤脱氢酶的活性(p<0.05),施肥基础上添加水杨酸则极显著提高了土壤脱氢酶活(p<0.001),是单施肥的2.34~2.89倍(表 1).土壤脱氢酶活性与 PCBs去除率具有相同趋势,均随水杨酸添加量的升高先增大后减小,当水杨酸添加量为10 mmol · kg-1时最高.以往的研究发现,土壤脱氢酶活与PCBs去除率存在一定的线性相关性(Chekol et al., 2004).本实验室用高羊茅和紫花苜蓿对长期受PCBs污染土壤的植物修复研究发现,植物根际土壤的脱氢酶活性显著大于施肥和空白对照,PCBs去除率和土壤脱氢酶活有同样的变化趋势,但相关性分析并没有达到显著水平(Li et al., 2013).
3.3 土壤中微生物群落结构多样性分析 3.3.1 总细菌DGGE图谱由PCR-DGGE可知(图 2),空白对照组中检测到条带3、5、6、7、8,当水杨酸添加量为5 mmol · kg-1时,条带5、6明显变亮;水杨酸添加量为20 mmol · kg-1时,条带3变亮,条带8消失.单施肥的处理组新增条带1、2,条带2在添加水杨酸后亮度减弱.施肥基础上添加水杨酸后新增条带4、9、10、11、12、13、14,其中,条带4、9、10、11在3种水杨酸添加量处理下均被检测到,条带12的亮度随水杨酸添加量的增加而减弱,条带13则呈相反趋势,条带14仅在10 mmol · kg-1的水杨酸处理中出现.土壤细菌多样性分析结果显示,当水杨酸添加量为10 mmol · kg-1时,Shannon指数最高(表 1).以上结果表明,水杨酸及其剂量水平对PCBs污染土壤微生物群落结构存在明显影响.
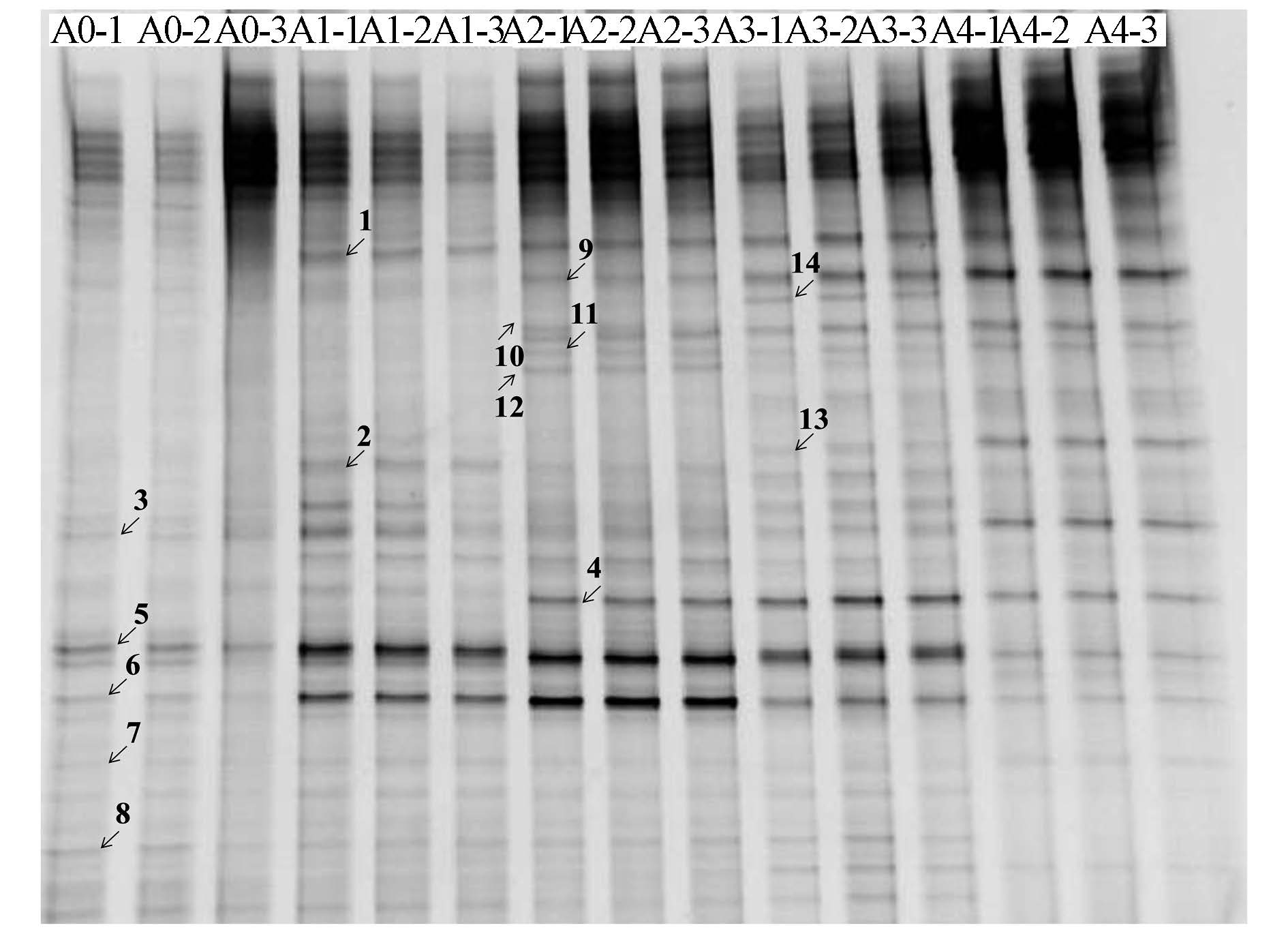 |
| 图2 不同处理土壤总细菌DGGE图 Fig.2 DGGE profiles of soil bacteria community in different treatments |
对土壤总细菌的DGGE图谱进行PCA分析,结果如图 3所示.主成份1和主成份2的贡献率分别为45.76%和30.72%,可以较充分地解释土壤微生物群落结构的变化.在主成份1的方向上,添加水杨酸的处理正向远离对照和单施肥的处理,表明水杨酸可能是影响土壤微生物结构多样性的首要因素.在主成份2的方向上,水杨酸添加量为5 mmol · kg-1和10 mmol · kg-1的处理正向远离水杨酸添加量为20 mmol · kg-1的处理,可见水杨酸剂量也是影响土壤微生物群落结构多样性的重要因素.
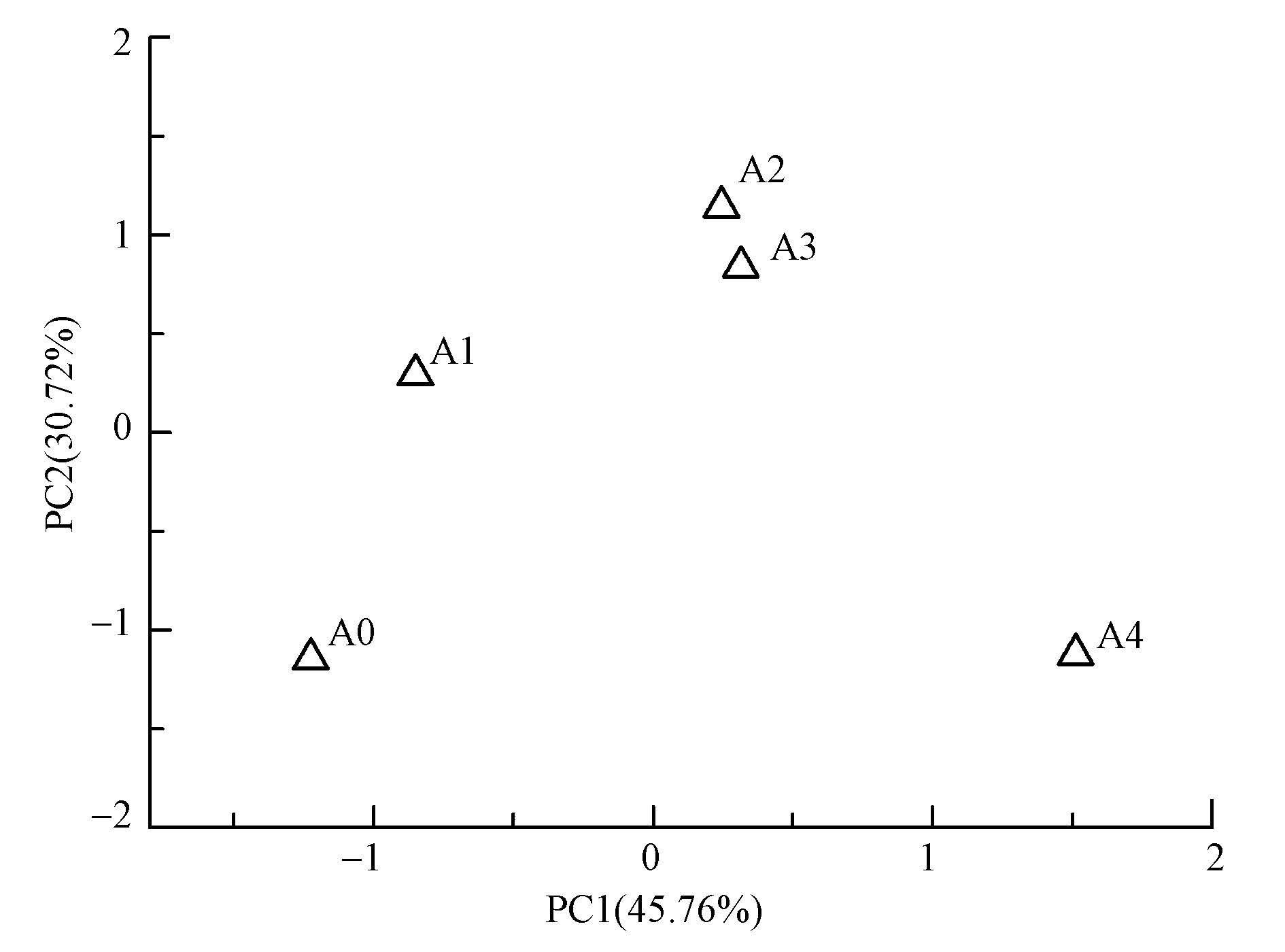 |
| 图3 不同处理条件下DGGE图谱主成分分析 Fig.3 PCA analysis based on DGGE profiles in different treatments |
根据各样品的PCR-DGGE指纹图谱,对优势条带进行切胶回收,所得序列与BLAST比对鉴定,寻找与序列相似性最高的菌株,结果见表 2.全部条带中将近65%的条带最相近的菌株属于变形菌门(Proteobacteria),其次为拟杆菌门(Bacteroidetes),只有条带8的相近菌株属于酸杆菌门(Acidobacteria).
| 表2 与污染物降解相关物种信息 Table 2 Species related with degradation of organic pollutants |
空白对照组中存在与PCBs降解相关的菌属.条带3代表的菌群在高剂量(20 mmol · kg-1)水杨酸的处理下成为优势种群,与产黄杆菌属(Rhodanobacter)相似度高达99%,Uhlik等(2012)在长期受到PCBs污染的土壤中发现产黄杆菌属可利用联苯和苯甲酸进行生长代谢.条带5与6的丰度均随水杨酸添加量的增加而降低,与条带5最接近的菌株属于鞘脂杆菌目(Sphingobacteriales),研究表明,鞘脂杆菌目具有较强的环境适应能力,能够利用一些特殊物质如除草剂、抗菌物质及溴系阻燃剂作为生长基质(Drury et al., 2013; Gaza et al., 2015);条带6与鞘氨醇单胞菌属(Sphingomonas)相似性为98%,这类菌株的一个显著特征是能降解多环或单环芳香族化合物如苯甲酸、水杨酸等(Basta et al., 2005),并具有降解PCBs的能力(Pieper et al., 2005).与条带7最接近的菌株为黄单胞菌属(Xanthomonadaceae),Klankeo等(2009)的研究结果表明,黄单胞菌属能够利用芘作为唯一的碳源.条带8指示的菌群在高剂量水杨酸的处理下消失,与条带8最接近的菌株为酸杆菌属(Acidobacteriaceae),多位研究者在PCBs和PAHs污染的土壤中检测到酸杆菌属的存在(Bertin et al., 2011; Pérez-Leblic et al., 2012).
单施肥后新增的条带1和2分别与鞘脂杆菌目(Sphingobacteriales)和热单胞菌属(Thermomonas)最为相似.其中,条带2的亮度在水杨酸的处理下减弱,表明该条带指示的菌群丰度下降.Koubek等(2013)运用稳定性同位素探针检测到土壤中一种热单胞菌能够利用4-氯联苯作为生长基质.
添加水杨酸后新增条带4、9、10、11、12、13、14,其中,所有剂量水杨酸处理下均出现条带4、9、10、11,4个条带的相似菌株分别为伯克氏菌属(Burkholderia)、潘多拉菌属(P and oraea)、噬油脂极小单胞菌属(Pusillimonas)与鞘脂菌属(Sphingobium).据报道,伯克氏菌属、潘多拉菌属及鞘脂菌属中均存在能够降解PCBs的典型菌株(Bedard et al., 1986;Pham et al., 2012;Hu et al., 2015),噬油脂极小单胞菌属则被发现能够存在于PAHs污染土壤中(Liang et al., 2010).水杨酸添加量为10 mmol · kg-1和20 mmol · kg-1的处理下出现特异性条带13,与甲基杆菌属(Methylobacterium)最为相似,其丰度随水杨酸添加量的增加而增加.水杨酸添加量为5 mmol · kg-1和10 mmol · kg-1的处理下出现特异性条带12,与Ralstonia eutropha JMP134相似度达到96%,其丰度随水杨酸添加量的增加而降低.条带14仅在水杨酸添加量为10 mmol · kg-1时出现,与Burkholderia xenovorans LB400相似度为96%.
水杨酸能够显著影响PCBs污染土壤的微生物群落结构,这与Luo等(2008)的结果类似.本研究中高剂量的水杨酸能够促进土壤中原有菌属产黄杆菌的生长,然而Uhlik等(2013)向PCBs污染土壤中添加柚皮苷、咖啡酸和柠檬烯后发现,土壤中原有的产黄杆菌消失.此外,3种剂量的水杨酸对土壤中黄单胞菌属的生长没有明显的促进作用,而Leigh等(2010)发现柳树根系分泌物能够增强黄单胞菌属降解PCBs的能力.值得注意的是,菌株Ralstonia eutropha与Burkholderia xenovorans仅在水杨酸的处理下出现,Luo等(2008)在添加水杨酸处理的PCBs污染土壤中同样发现了Ralstonia eutropha 与Burkholderia cepacia的存在.Ralstonia eutropha H850与Burkholderia xenovorans LB400作为两种高效广谱的PCBs降解菌从20世纪80年代以来一直被广泛关注.
水杨酸对PCBs降解的相关菌属可能具有特异性作用,即只对某些菌属起促进作用.不同剂量的水杨酸对PCBs降解菌的诱导也存在差异.类似的实验结果也出现在Pham等(2012)的研究中,他们比较了黄酮对两种PCBs降解菌的诱导能力,发现黄酮能显著提高P and oraea pnomenusa B356的降解能力,而对Burkholderia xenovorans LB400的作用不明显.Toussaint等(2012)比较了3个浓度水平的黄酮与联苯对PCBs降解菌Rhodococcus erythropolis U23A的诱导能力,结果显示,随着浓度的升高,黄酮对比联苯的优势更为明显.
3.4 土壤中bphA基因多样性分析联苯双加氧酶的大亚基(bphA1)主要负责底物的识别和选择.根据大亚基的序列相似性,来自革兰氏阴性菌和阳性菌的联苯双加氧酶分别划分为D.1.B、D.1.C(Sylvestre et al. 1996)和D.2.A、D.2.B(Baldwin et al. 2003).本研究运用荧光定量PCR测定了bphA、bphD.1.B、bphD.1.C、bphD.2.A和bphD.2.A/B基因相对于16S rRNA 基因的丰度,结果显示(图 4),不同处理间bphA基因相对丰度没有显著差异.单施肥及在施肥基础上添加10、20 mmol · kg-1水杨酸的土壤中,bphD.1.B、bphD.1.C、bphD.2.A和bphD.2.A/B基因相对丰度显著高于对照(p<0.05).施肥基础上添加水杨酸后,土壤中bph基因相对丰度与单施肥相比没有显著变化或反而有所下降.这与Hu等(2014)的研究结果类似:水杨酸处理后PCBs去除率增加,但bphA基因丰度却降低.Tiedje实验室对长期受PCBs污染的松树根际土壤中的bphA1基因扩增后,进行高通量测序,发现绝大多数为新的序列,并且还发现萘和苯甲酸双加氧酶基因(Iwai et al., 2010).该结果表明,目前已知的bphA1基因的信息远远不能代表环境中的真实情况,其它有机碳降解基因可能也存在相同问题,今后需要用新的手段来评估环境中的基因谱系(Iwai et al., 2011).
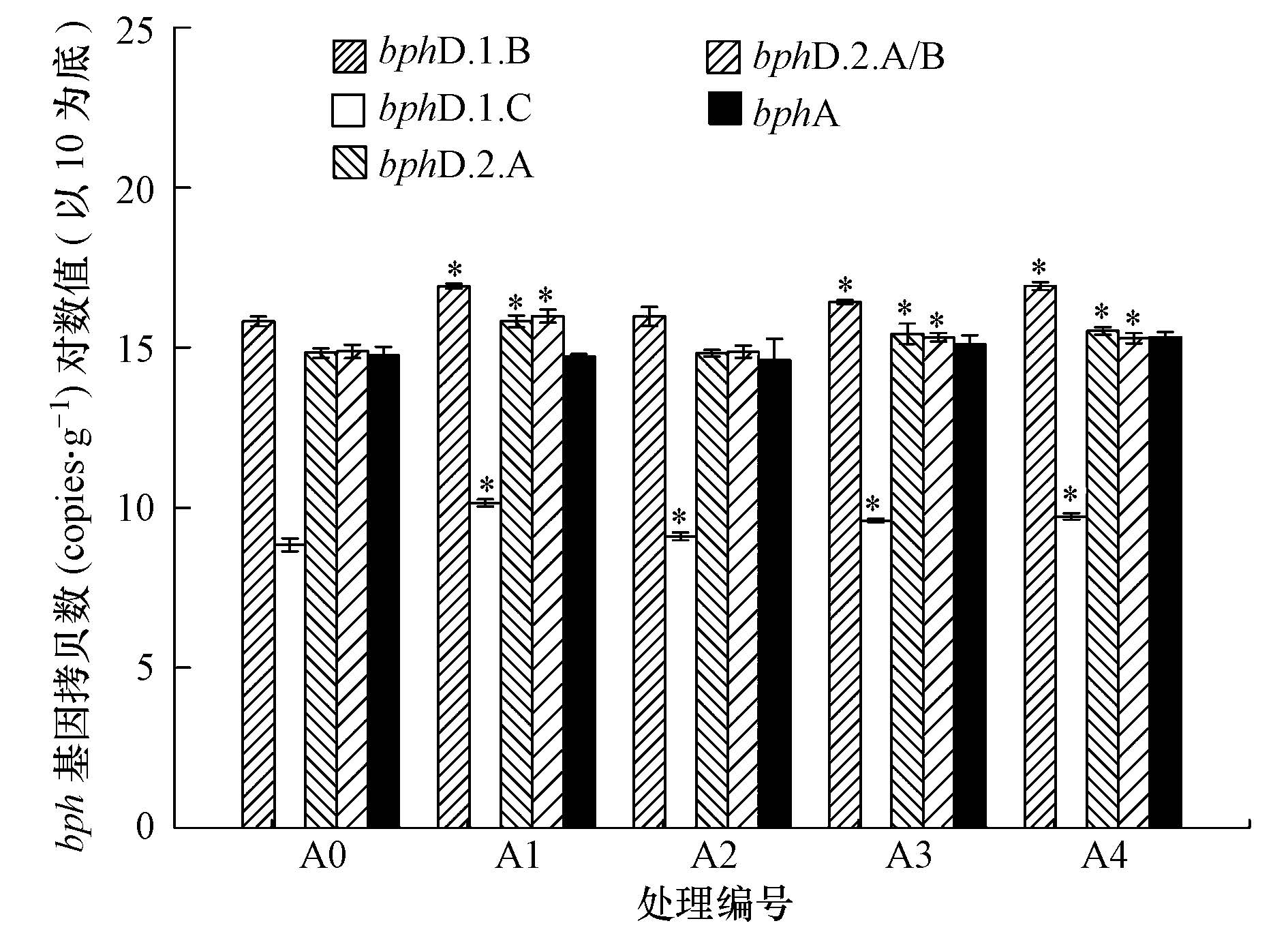 |
| 图4 bph基因的相对丰度 Fig.4 Relative abundance of the bph genes encoding biphenyl dioxygenases |
1)施肥和在施肥基础上添加水杨酸均能显著提高PCBs去除率,其中,三氯联苯的去除率最高.水杨酸添加量为10 mmol · kg-1时,PCBs去除率、土壤脱氢酶活和微生物群落多样性均最大.
2)水杨酸能显著影响PCBs污染土壤的微生物群落结构,对PCBs相关降解菌属的诱导存在特异性.水杨酸处理后新增Burkholderia sp.、P and oraea sp.、Pusillimonas sp.、Sphingobium sp.、Methylobacterium sp.和Ralstonia sp..
3)不同处理间bphA基因丰度没有显著差异,但施肥和施肥基础上添加10、20 mmol · kg-1水杨酸的处理都显著提高了bphD.1.B、bphD.1.C、bphD.2.A和bphD.2.A/B的相对丰度.
| [1] | Baldwin B R.2003.Detection and enumeration of aromatic oxygenase genes by multiplex and real-time PCR[J]. Applied and Environmental Microbiology, 69(6):3350-3358 |
| [2] | Basta T, Buerger S, Stolz A.2005.Structural and replicative diversity of large plasmids from Sphingomonads that degrade polycyclic aromatic compounds and xenobiotics[J].Microbiology,151(6):2025-2037 |
| [3] | Bedard D L,Unterman R,Bopp L H,et al.1986.Rapid assay for screening and characterizing microorganisms for the ability to degrade polychlorinated biphenyls[J].Applied and Environmental Microbiology,51(4):761-768 |
| [4] | Bertin L,Capodicasa S,Fedi S,et al.2011.Biotransformation of a highly chlorinated PCB mixture in an activated sludge collected from a membrane biological reactor (MBR) subjected to anaerobic digestion[J].Journal of Hazardous Materials,186(2):2060-2067 |
| [5] | Brunner W,Sutherland F H,Focht D D.1985.Enhanced biodegradation of polychlorinated biphenyls in soil by analog enrichment and bacterial inoculation[J].Journal of Environmental Quality,14(3):324-328 |
| [6] | Chekol T,Vough L R,Chaney R L.2004.Phytoremediation of polychlorinated biphenyl-contaminated soils:the rhizosphere effect[J].Environment International,30(6):799-804 |
| [7] | Dalla Valle M,Jurado E,Dachs J,et al.2005.The maximum reservoir capacity of soils for persistent organic pollutants:implications for global cycling[J].Environmental Pollution,134(1):153-164 |
| [8] | Drury B,Rosi-Marshall E,Kelly J J.2013.Wastewater treatment effluent reduces the abundance and diversity of benthic bacterial communities in urban and suburban rivers[J].Applied and Environmental Microbiology,79(6):1897-1905 |
| [9] | Dudášová H,Lukáčová L,Murínová S,et al.2012.Effects of plant terpenes on biodegradation of polychlorinated biphenyls (PCBs)[J].International Biodeterioration and Biodegradation,69:23-27 |
| [10] | Field J A,Sierra-Alvarez R.2008.Microbial transformation and degradation of polychlorinated biphenyls[J].Environmental Pollution,155(1):1-12 |
| [11] | Gaza S,Felgner A,Otto J,et al.2015.Biodegradation of chloro-and bromobenzoic acids:Effect of milieu conditions and microbial community analysis[J].Journal of Hazardous Materials,287(28):24-31 |
| [12] | Gottfried A,Singhal N,Elliot R,et al.2010.The role of salicylate and biosurfactant in inducing phenanthrene degradation in batch soil slurries[J].Applied Microbiology and Biotechnology,86(5):1563-1571 |
| [13] | 关荫松.1986.土壤酶及其研究方法[M].北京:中国农业出版社 |
| [14] | Hu C,Zhang Y,Tang X,et al.2014.PCB biodegradation and bphA1 gene expression induced by salicylic acid and biphenyl with Pseudomonas fluorescence P2W and Ralstoniaeutropha H850[J].Polish Journal of Environmental Studies,23(5):1591-1598 |
| [15] | Hu J,Qian M,Zhang Q,et al.2015.Sphingobium fuliginis HC3:A novel and robust isolated biphenyl-and polychlorinated biphenyls-degrading bacterium without dead-end intermediates accumulation[J].PloS One,10(4):e0122740 |
| [16] | Iwai S.2010.Gene-targeted-metagenomics reveals extensive diversity of aromatic dioxygenase genes in the environment[J].ISME Journal,4(2):279-285 |
| [17] | Iwai S,Johnson T A,Chai B,et al.2011.Comparison of the specificities and efficacies of primers for aromatic dioxygenase gene analysis of environmental samples[J].Applied and Environmental Microbiology,77(11):3551-3557 |
| [18] | Klankeo P,Nopcharoenkul W,Pinyakong O.2009.Two novel pyrene-degrading Diaphorobacter sp.and Pseudoxanthomonas sp.isolated from soil[J].Journal of Bioscience and Bioengineering,108(6):488-495 |
| [19] | Koubek J,Mackova M,Macek T,et al.2013.Diversity of chlorobiphenyl-metabolizing bacteria and their biphenyl dioxygenases in contaminated sediment[J].Chemosphere,93(8):1548-1555 |
| [20] | Li Y,Liang F,Zhu Y,et al.2013.Phytoremediation of a PCB-contaminated soil by alfalfa and tall fescue single and mixed plants cultivation[J].Journal of Soils and Sediments,13(5):925-931 |
| [21] | Liang Q,Lloyd-Jones G.2010.Sphingobium scionense sp.nov.,an aromatic hydrocarbon-degrading bacterium isolated from contaminated sawmill soil[J].International Journal of Systematic and Evolutionary Microbiology,60:413-416 |
| [22] | Leigh M B,Prouzová P,Macková M,et al.2010.Polychlorinated biphenyl (PCB)-degrading bacteria associated with trees in a PCB-contaminated site[J].Applied and Environmental Microbiology,72(4):2331-2342 |
| [23] | Luo W,D 'Angelo E M,Coyne M S.2008.Organic carbon effects on aerobic polychlorinated biphenyl removal and bacterial community composition in soils and sediments[J].Chemosphere,70(3):364-373 |
| [24] | Luo W,Hu C.2008.Interaction of plant secondary metabolites and organic carbon substrates affected on biodegradation of polychlorinated biphenyl[J].Journal of Environmental Biology,34:337-343 |
| [25] | Lu Y C,Zhang S,Miao S S,et al.2015.Enhanced degradation of herbicide isoproturon in wheat rhizosphere by salicylic acid[J].Journal of Agricultural and Food Chemistry,63:92-103 |
| [26] | Margesin R.2000.Monitoring of bioremediation by soil biological activities[J].Chemosphere,40(4):339-346 |
| [27] | Pérez-Leblic M I,Turmero A,Hernández M,et al.2012.Influence of xenobiotic contaminants on landfill soil microbial activity and diversity[J].Journal of Environmental Management,95:S285-S290 |
| [28] | Pham T T M.2012.Remarkable ability of Pandoraeapnomenusa B356 biphenyl dioxygenase to metabolize simple flavonoids[J].Applied and Environmental Microbiology,78(10):3560-3570 |
| [29] | Pieper D H.2005.Aerobic degradation of polychlorinated biphenyls[J].Applied Microbiology and Biotechnology,67(6):170-191 |
| [30] | Powell S N,Singleton D R,Aitken M D.2008.Effects of enrichment with salicylate on bacterial selection and PAH mineralization in a microbial community from a bioreactor treating contaminated soil[J].Environmental Science and Technology,42(11):4099-4105 |
| [31] | Singer A C,Gilbert E S,Luepromchai E,et al.2000.Bioremediation of polychlorinated biphenyl-contaminated soil using carvone and surfactant-grown bacteria[J].Applied Microbiology and Biotechnology,54(6):838-843 |
| [32] | Singer A C,Crowley D E,Thompson I P.2003.Secondary plant metabolites in phytoremediation and biotransformation[J].Trends in Biotechnology,21(3):123-130 |
| [33] | Slater H,Gouin T,Leigh M B.2011.Assessing the potential for rhizoremediation of PCB contaminated soils in northern regions using native tree species[J].Chemosphere,84(2):199-206 |
| [34] | Sylvestre M,Sirois M,Hurtubise Y,et al.1996.Sequencing of Comamonas testosteroni strain B-356-biphenyl/chlorobiphenyl dioxygenase genes:evolutionary relationships among Gram-negative bacterial biphenyl dioxygenases[J].Gene,174(2):195-202 |
| [35] | Toussaint J P,Pham T T M,Barriault D,et al.2012.Plant exudates promote PCB degradation by a Rhodococcal rhizobacteria[J].Applied Microbiology and Biotechnology,95(6):1589-1603 |
| [36] | Uhlik O,Wald J,Strejcek M,et al.2012.Identification of bacteria utilizing biphenyl,benzoate,and naphthalene in long-term contaminated soil[J].PloS One,7(7):e40653 |
| [37] | Uhlik O,Musilova L,Ridl J,et al.2013.Plant secondary metabolite-induced shifts in bacterial community structure and degradative ability in contaminated soil[J].Applied Microbiology and Biotechnology,97(20):9245-9256 |
 2016, Vol. 36
2016, Vol. 36


