2. 华南理工大学广东省大气环境与污染控制重点实验室, 广州 510006;
3. 工业聚集区污染控制与生态修复教育部重点实验室, 广州 510006
2. Guangdong Provincial Key Laboratory of Atmospheric Environment and Pollution Control, South China University of Technology, Guangzhou 510006;
3. Key Laboratory of Pollution Control and Ecosystem Restoration in Industry Clusters, Ministry of Education, Guangdong, Guangzhou 510006
近年来,随着抗生素的大量使用,其在环境介质中的残留已引起人们广泛关注(王冰等,2007;Gbel et al., 2004;Onesios et al., 2009).虽然所检出的抗生素残留浓度水平未达到致死浓度,不足以抑制或者杀死微生物(Davies et al., 2006;Yim et al., 2006),但由于这些物质大多数仍具有较强的生物反应活性和富集放大效应(Fent et al., 2006;Daughton et al., 1997),当达到最低抑菌浓度水平时,可能会促进耐药菌的发展和传播(Kümmerer,2003;Fan et al., 2011),已有研究表明,长期施用含有四环素的猪粪是环境中具有抗药性致病菌的重要来源,同时,也是促进其生成的主要推力(张俊等,2014).更为重要的是,耐药菌所携带的耐药基因可以长期存在于土壤和水生细菌,甚至可能会传递至致病性更强的细菌(肖斌等,2012),对人类健康造成极大威胁,因此,环境中抗生素的环境后效应以及生态效应成为近年环境领域的研究热点之一.红霉素(Erythromycin,ERY)是一类大环内酯类广谱抗生素,具有强大的微生物抑制作用.人们已在污水、水源水和饮用水中检测到ng · L-1~μg · L-1浓度的红霉素污染(毛菲菲等,2013).同时,残留在自然水体中的抗生素有75%沉积在水底沉积物中,可形成水体底泥的抗生素蓄积性污染,进而对底泥微生态造成影响(Samuelsen et al., 1992;Hansen et al., 1992),甚至诱导生物群体对红霉素产生耐药性(MariyaMorar et al., 2012).耐药菌的存在不仅可加剧残留抗生素的环境影响(Fard et al., 2011),更为重要的是能大大降低抗生素药物生物抑制的有效性.目前,针对红霉素耐药性方面的研究大多侧重于研究某单一菌种的耐药行为以及红霉素对土著微生物群落结构的影响,鲜有关于耐红霉素菌群多样性的研究,而关于耐药菌群的研究也大多集中在磺胺类(高盼盼等,2011)和四环素类(刘冲等,2012)等抗生素.作为一种环境残留率较高且广泛使用的抗生素,研究环境中红霉素耐受菌群的结构特点,不仅有助于人们正确评价红霉素的生态风险,而且可为红霉素药物污染的防治提供理论依据.
2 材料与方法(Materials and methods) 2.1 主要试剂及仪器红霉素购自广州华奇盛生物科技公司.配置100 μg · mL-1脱水红霉素母液(用3 mol · L-1 H2SO4调节红霉素溶液的pH值到3.0,在室温下搅拌4 h),4 ℃避光保存.
营养肉汤培养基:10 g蛋白胨,5 g牛肉膏,5 g NaCl,1 L蒸馏水,调pH为7.0,固体培养基加20%的琼脂粉.
其他生化试剂包括:细菌基因组DNA提取试剂盒(TIANamp Bacteria DNA Kit),购自天根生化科技(北京)有限公司;Taq DNA聚合酶连接酶(BioReadyTaq Mix),购自杭州博日科技有限公司;UNIQ-10柱式胶回收试剂盒,T-载体PCR产物克隆试剂盒,均购自生工生物工程(上海)股份有限公司.相关引物合成,由生工生物工程(上海)股份有限公司完成.
主要仪器:紫外分光光度计(岛津UV-2550),PCR仪(Bio-Rad S 1000TM Thermal Cycler);电泳仪(Bio-Rad PowerPac Basic);小型台式离心机(LabogeneScanSpeed mini).
2.2 样品采集底泥样品采自长期受低浓度红霉素污染的模拟水生生态系统,该系统的构建参考美国环保署的标准方法(Acute Dermal Toxicity,1996).向室内静水养殖箱(80 cm×60 cm×40 cm)内加入约20 kg的沉积物和60 L水,植入水蕴草和斑马鱼.实验期间室内温度控制在(30±2)℃,pH值控制在7.0±0.2、溶氧> 4 mg · L-1.系统稳定后,每天向系统内加入2.5 μg · L-1脱水红霉素溶液,持续运行1年时间,采集底泥样品进行分析.
2.3 实验方法 2.3.1 可培养耐红霉素细菌的培养和计数分别取3份1.5 g新鲜底泥分别加入含有150 mL无菌水的三角瓶中,摇床振荡3 h,制备成菌悬液,然后分别吸取适量,进行混合.采用平板菌落计数法,取适量混合均匀的菌悬液进行梯度稀释后,分别涂布在制备好的红霉素终浓度为16 μg · mL-1的营养肉汤固体培养基上,每一浓度均设置3个重复,30 ℃培养48 h,进行抗性细菌计数.
2.3.2 可培养耐红霉素菌的分离与鉴定选取2.3.1节中稀释度适当的平板挑取单菌落,分别在30 ℃,150 r · min-1进行液体培养24 h,再稀释至适当浓度进行涂布,重复3~4次,直至平板上只生长单一菌落.
提取纯菌的细菌基因组DNA,利用细菌通用引物27F和1492R进行PCR扩增,PCR产物的纯化及测序均由生工生物工程(上海)股份有限公司完成.
2.3.3 样品总DNA的提取将2.3.1节中的菌悬液取适量加入到已灭菌的含有16 μg · mL-1红霉素的营养肉汤培养基中,150 r · min-1,30 ℃过夜培养后提取基因组DNA,得到的DNA样品为底泥中耐红霉素微生物的总DNA.
2.3.4 PCR扩增PCR扩增引物:27F(5′-AGAGTTTGATCCTGGCTCAG-3′)及1492R(5′-GGTTACCTTGTTACGACTT-3′)(Karnachuk et al., 2009).
PCR反应体系(25 μL):DNA模板1 μL,ddH2O 10.5 μL,引物27F和1492R各0.5 μL,Taq酶12.5 μL.
PCR程序:95 ℃预变性3 min,95 ℃变性30 s,50 ℃退火1 min,72 ℃延伸1 min,30个循环,最后72 ℃延伸10 min,4 ℃保存.
将PCR产物用琼脂糖凝胶电泳检验后进行切胶回收,用UNIQ-10柱式DNA胶回收试剂盒纯化进行纯化.
2.3.5 克隆文库的构建利用细菌通用引物27F及1492R对样品DNA进行PCR扩增.将纯化后的PCR产物用pUCm-T载体进行目的基因连接,然后将其转入大肠杆菌DH5α感受态中,37 ℃振荡培养1 h;完后将其涂在含有IPTG和X-gal的氨苄青霉素培养基平板上,37 ℃恒温培养过夜后挑取白色的阳性克隆子,再用相同的方法进行培养;利用PstI 单酶切进行阳性克隆验证.验证后的阳性克隆子测序由上海生工生物工程技术服务有限公司完成.测序结果通过CD-hit软件基于序列相似度进行聚类分析,划分操作分类单元(Operational Taxonomic Unit,OTU);使用Estimate软件绘制微生物稀有性曲线.利用稀有性曲线和文库覆盖率C评价文库,文库覆盖率C计算公式为

所有测序结果在GenBank数据库进行Blast比对后下载同源性序列,系统发育树的绘制借助MEGA5.0软件完成.
3 结果与讨论(Results and discussion) 3.1 可培养耐红霉素细菌的计数与分离鉴定根据抗菌药物敏感性实验执行标准(NCCLS,2009),红霉素的最低抑菌浓度(MIC)为8 μg · mL-1,本研究选择略高于其MIC值的抗生素浓度,即红霉素的终浓度为16 μg · mL-1.利用平板菌落计数法对抗性细菌的计数结果为9.86×108 CFU · g-1.将平板上形态各异的耐药菌株进行反复纯化分离后,得到了3株独立的菌株,其各自的形态如表 1中所示.
| 表1 可培养耐红霉素菌菌落形态 Table 1 morphology of Culturable erythromycin resistant bacterial colonies |
分别将3株菌株的16S rDNA序列在NCBI核酸数据库中进行BLAST同源性比对,确定与其最接近的种系群体.结合各菌株形态学特征及16S rDNA 序列分析,初步鉴定为赖氨酸芽孢杆菌(Lysinibacillus sp.)、土壤芽孢杆菌(Solibacillus silvestris)以及蜡样芽孢杆菌(Bacillus cereus),各自在GenBank中的登录号分为KP694229、KP694230、KP694231,且上述3种可培养菌均属于芽孢杆菌纲.需要注意的是,蜡样芽孢杆菌在自然界分布广泛,是一种食源性致病菌,容易引起食物中毒,对红霉素极度敏感(李浇等,2012),对青霉素耐药.然而,本文的耐药实验发现,从底泥中分离出的蜡样芽孢杆菌GD2C对红霉素已表现出一定的耐药性,这可能是菌株分离自长期受低浓度红霉素污染的环境,这意味着低浓度红霉素的诱导作用可使蜡样芽孢杆菌具备一定的耐药性.
3.2 16S rDNA克隆文库的分析随机挑取83个阳性克隆子进行测序(序列长均为1500 bp左右),将测序结果在NCBI的核酸数据库中进行Blast比对,将序列比对结果相似度大于等于97%的定义为一个操作单元(Operational Taxonomic Unit,OTU),共得到6个OTU,其中有2个OTU类型代表单克隆,最多的OTU类型包括36个克隆子.随后以文库覆盖率C及稀有性曲线来评价所构建的文库,发现该文库覆盖率为97.6%,且稀有性曲线趋于平缓,这意味着所构建的克隆文库库容较大,所挑取的克隆子数量能够较为完整的反映样品中微生物的多样性(王新新等,2011).上述两项分析表明该克隆文库所含83个克隆子能够较全面地反映底泥中耐红霉素细菌种群结构的多样性.
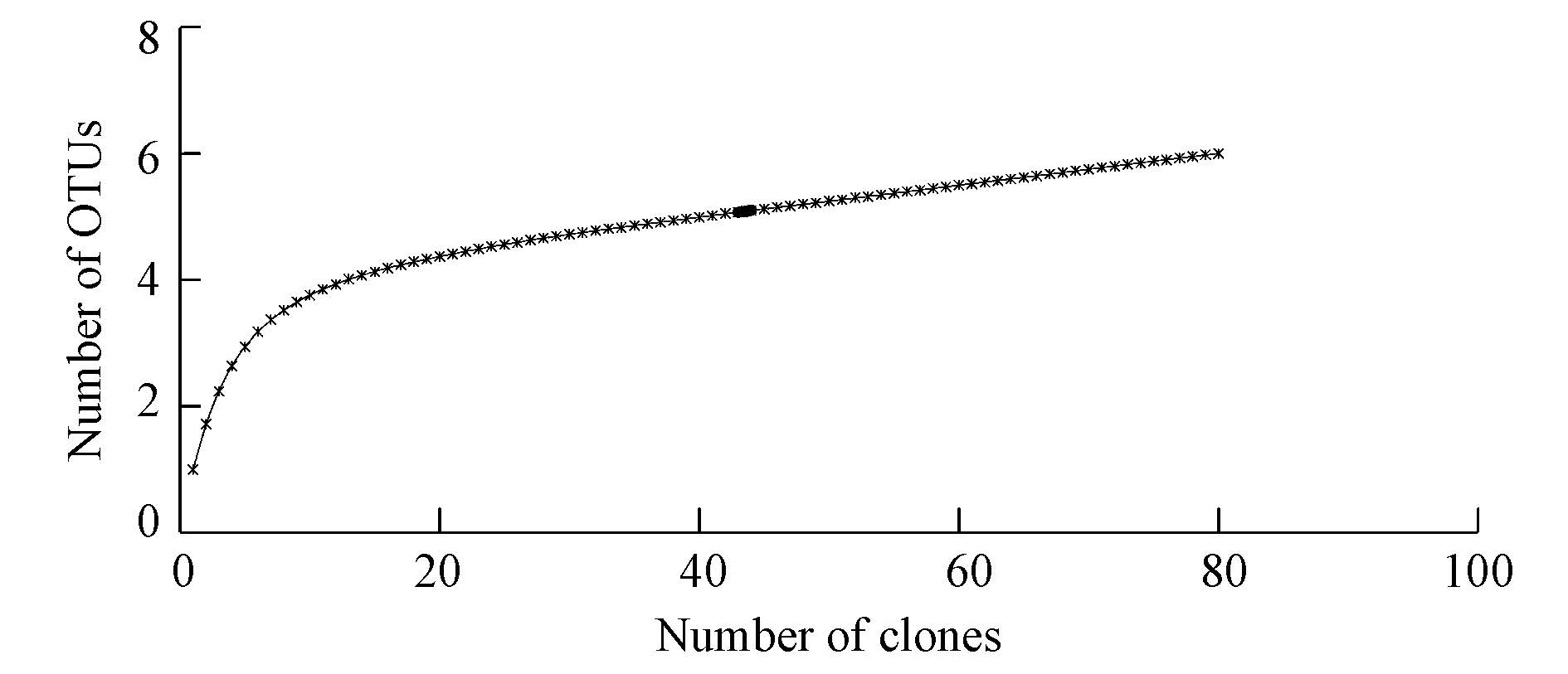 |
| 图1 底泥中耐红霉素细菌16S rDNA克隆文库的稀缺性曲线 Fig.1 Rarefaction curves generated for 16S rDNA clone library of the sediment. Clones were grouped into phylotypes based on sequence similarity≥97% |
样品中16S rDNA克隆文库结果如表 2所示,其中4个OTU的克隆子序列与NCBI核酸数据库中已知细菌的16S rDNA序列相似度在97%以上,2个OTU的相似度低于97%.由比对结果可知,长期受低浓度红霉素污染的底泥中,耐红霉素的细菌类群可分为三大类群,即:未获培养细菌(Uncultured bacterium)、芽孢杆菌纲细菌(Bacilli)和梭菌纲细菌(Clostridia),各涉及到2个OTU(54个克隆子)、3个OTU(28个克隆子)和1个OTU(1个克隆子).底泥样品中耐红霉素微生物中的优势菌群为某种未获培养的细菌,在文库中占43.37%,其次是苏云金芽孢杆菌,占文库的24.10%.
| 表2 底泥中耐红霉素细菌16S rDNA克隆文库结果 Table 2 Cloning of Erythromycin resistant bacterial 16S rDNA in the sediment |
苏云金芽孢杆菌是一种有益杆菌,多用做杀虫剂,目前对于其耐药性的报道较少,但从文库结果来看,苏云金芽孢杆菌已对红霉素产生了一定的耐药性;另外,红霉素原本对梭状芽孢杆菌有较强的抑制作用,在本研究中也表现出了一定的耐药性,这可能也是因为环境中残留的低浓度红霉素对其诱导,使其对红霉素产生了一定的抗性,这也进一步说明了环境中残留抗生素存在潜在影响.由克隆文库结果可以看出,在已知菌属中,芽孢杆菌属所占比例最大,这与上述所筛选分离出的可培养的耐药菌均为芽孢杆菌属细菌的结果是互相吻合的.
另外,构建的16S rDNA文库中有25.3%(21个克隆子)的细菌与GenBank中已知细菌的16S rDNA序列同源性低于97%,而在序列同源性研究中,一般认为细菌16S rDNA序列同源性低于97%即属于不同的种(王海燕等,2006),这说明底泥中蕴藏着许多未知的耐药细菌,有待于进一步深入研究.
3.3 底泥样品16S rDNA系统发育分析分子系统发育分析,特别是对细菌16S rDNA基因的同源性进行分析有助于了解微生物群落结构的多样性(田春杰等,2003).将表 1和表 2中的3株单菌和6个OTU的碱基序列及其相应的同源性序列进行聚类分析后,以嗜热链球菌Streptococcus thermophilus(NR042778)为外类群,绘制出底泥中耐红霉素细菌系统发育树,见图 2.
由图 2可见,土壤芽孢杆菌GD2B的碱基序列与OTU4的碱基序列之间相似度较高,且与OTU1存在较好的亲缘关系.因而将OTU1和OTU4的序列进一步在RDP数据库中进行比对,发现其最大相似菌株Uncultured bacterium clone(DQ337010)和Uncultured bacterium clone(AB506332)可以进一步划分到某未分类的动性球菌属(Unclassified Planococcaceae),而土壤芽孢杆菌和动性球菌属均属于动球菌科(Planococcaceae),所以OTU1、GD2B以及OTU4在系统发育树中聚集到一起.蜡样芽孢杆菌GD2C与OTU5的最大相似菌株苏云金芽孢杆菌存在较好的亲缘关系,这是由于苏云金芽孢杆菌和蜡样芽孢杆菌均属于蜡样杆菌属,两者之间具有高度的同源性(Qi et al., 2001).
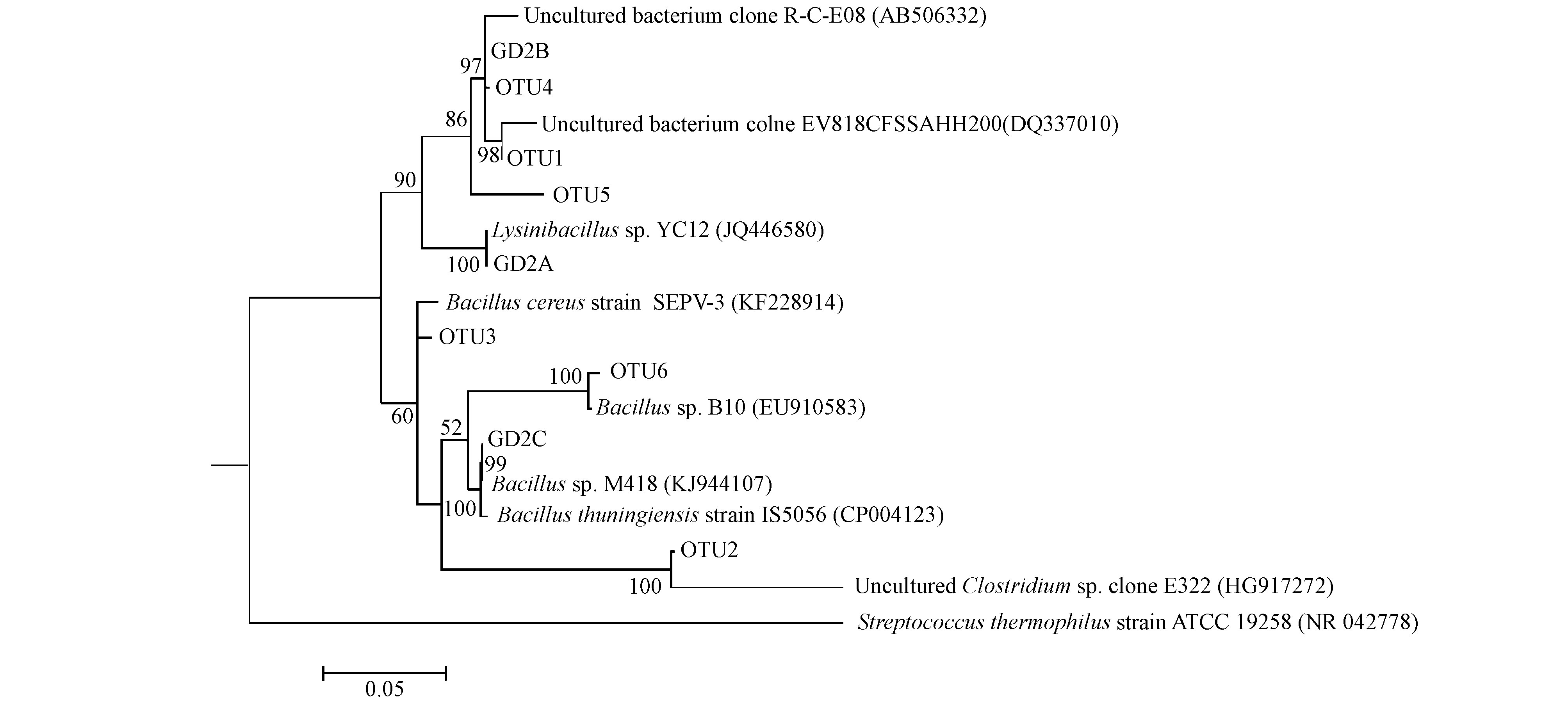 |
| 图2 底泥中耐红霉素细菌系统发育树 Fig.2 The phylogenetic tree of the erythromycin resistant bacteria in the sediment |
红霉素作为第一代大环内酯类抗生素,自1952年应用于临床以来,一直被人们长时间大剂量使用,使得多种原本敏感的微生物对其产生了抗性(李喆宇等,2013),比如肺炎链球菌、金黄色葡萄球菌、粪肠球菌等.而本研究发现的芽孢杆菌,相关报道尚不多见.于雅琼等(2007)发现,分离自土壤、饲料和肥料的芽孢杆菌对大环内酯类抗生素表现出一定的敏感性.本研究显示,经过为期1年的低浓度红霉素胁迫,芽孢杆菌对红霉素已表现出一定的耐药性,3株可培养的红霉素耐药菌及红霉素耐药菌克隆文库中有33.73%均属于芽孢杆菌.这可能是由于:①不同剂量使用大环内酯类药物,在机体内存在诱导出耐药株的可能性(刘禧杰,2008);②微生物细胞可以通过基因的水平转移或者可移动遗传因子获得抵抗力(李晶,2013),通过抗性基因的水平转移获得耐受红霉素基因,从而获得对红霉素的抵抗力.当细菌具有抗生素抗性后,可能会对细菌代谢产生特定的改变(如抗重金属污染等特性),甚至可使细菌在某些环境中更具有生存优势;而存在于基因转移单位的抗性基因,则可以自我复制,从而一直存在于微生物群落里(杨颖,2010),这必将引发巨大的潜在危害,并威胁到环境及人类健康.
4 结论(Conclusions)1)用传统方法从底泥中筛选出的3株芽孢杆菌纲耐红霉素菌株,结合各菌株形态学特征及16S rDNA序列分析,初步鉴定为赖氨酸芽孢杆菌(Lysinibacillus sp.)、土壤芽孢杆菌(Solibacillus silvestris)以及蜡样芽孢杆菌(Bacillus cereus).其中原本对红霉素极度敏感的蜡样芽孢杆菌,由于低浓度红霉素的驯化,对红霉素产生了一定的耐药性.
2)长期受低浓度红霉素污染的底泥中,耐红霉素的细菌类群分三大类群,即:未获培养的细菌、芽孢杆菌纲和梭状芽孢杆菌纲.其中未获培养的细菌在整个文库中比例最大,可培养的耐药菌的优势类群为芽孢杆菌纲.结合系统发育树的分析,发现所占比例最大的某未获培养的细菌为某种未分类的动性球菌(43.37%),可初步划分到芽孢杆菌纲(Bacilli),尚需更多地分类鉴定.在16S rDNA克隆文库中,有25.3%的细菌与GenBank中已知细菌的序列同源性低于97%,这说明底泥中蕴藏着许多未知的耐药细菌,有待于进一步深入研究.
3)通过对16S rDNA文库研究发现,对红霉素具有一定耐药性的细菌既有致病菌(例如蜡样芽孢杆菌),也有有益菌(例如苏云金杆菌).随着各种抗生素使用量的增加,一方面,使得原本对抗生素有一定敏感度的致病菌表现出了一定的耐药性,对疾病的防治加大了难度;另一方面,也使得环境中某些有益菌获得了一定的耐药性,使其可以更好的生存.在抗生素污染问题越来越严重的今天,有益的芽孢杆菌的应用研究,可能是解决抗生素问题的一个有效方案.
| [1] | Acute Dermal Toxicity.1996. EPA 712-C-96-134. Ecological Effects Test Guidelines-OPPTS 850.1900 Generic Freshwater Microcosm Test,Laboratory[S].United States:Environmental Protection Agency |
| [2] | Davies J,Spiegelman G B,Yim G. 2006. The world of subinhibitory antibiotic concentrations[J]. Curr Opin Microbiol,9(5):445-453 |
| [3] | Daughton C G,Ternes T A. 1999. Pharmaceuticals and personal care products in the environment:agents of subtle change[J]. Environmental Health Perspectives,107:907-938 |
| [4] | Fan C,He J. 2011. Proliferation of antibiotic resistance genes in microbial consortia of sequencing batch reactors(SBRs)upon exposure to trace erythromycin or erythromycin-H2O[J]. Water Res,45:3098-3106 |
| [5] | Fent K,Weston A,Caminada D. 2006. Ecotoxicology of human pharmaceuticals[J]. Aquatic Toxicology,76:122-129 |
| [6] | 高盼盼,罗义,毛大庆. 2011.天津水产业磺胺类耐药细菌及其分布[J].生态毒理学报,6(1):74-79 |
| [7] | Gbel A,McArdell C S,Suter M J F, et al. 2004. Determination of macrolide and sulfonamide antimicrobials,a human sulfonamide metabolite,and trimethoprim in wastewater using liquid chromatography coupled to electrospray tandem mass spectrometry[J]. Analytical Chemistry,76:4756-4764 |
| [8] | Granizo J J,Aguilar L,Casal J,et al. 2000. Streptococcus pneumonia resistance to erythromycin and penicillin in relation to macrolide and beta-lactam consumption in Spain(1979-1997)[J]. Antimicrob Chemother,46:767-773 |
| [9] | Hansen P K,Lunestad B T,Samuelsen O B. 1992. Ecological effects of antibiotics and chemotherapeutants from fish farming[A].Michel,C. and Alderman D J,Chemotherapy in Aquaculture Today:From Theory to Reality.Office International Des Epizooties[C].Paris.174-178 |
| [10] | Karnachuk O V,Gerasimchuk A L,Banks D,et al. 2009. Bacteria of the sulfur cycle in the sediments of gold mine tailings,Kuznetsk Basin,Russia[J]. Microbiology,78:483-491 |
| [11] | Kümmerer K. 2003. Significance of antibiotics in the environment[J]. J Antimicrob Chemother,52:5-7 |
| [12] | 李浇,郑之琬,何娟,等. 2012.重庆荣昌某奶牛场子宫内膜炎蜡样芽孢杆菌的分离鉴定及药敏试验[J].中国兽医杂志,48(6):52-54 |
| [13] | 李晶,刘玉荣,贺纪正,等. 2013.土壤微生物对环境胁迫的响应机制[J].环境科学学报,33(4):959-967 |
| [14] | 李喆宇,崔玉彬,张静霞,等. 2013.大环内酯类抗生素的研究新进展[J].国外医药抗生素分册,34(1):6-15 |
| [15] | 刘冲,黄满红,肖兵. 2012.城市污水处理厂污水处理系统中四环素类药物耐药菌的分布特性研究[J].环境污染与防治,12:5-8 |
| [16] | 刘禧杰,辛德莉,李靖,等. 2008.肺炎支原体对大环内酯类抗生素的耐药分子机制[J].实用儿科临床杂志,23(22):1730-1733 |
| [17] | 毛菲菲,刘畅,何梦琦,等. 2013.红霉素降解菌的筛分及其降解特性的研究[J].环境科学与技术, 36(7):9-12 |
| [18] | Morar M,Pengelly K, Koteva K, et al. 2012. Mechanism and Diversity of the Erythromycin Esterase Family of Enzymes[J]. Biochemistry,51(8):1740-1751 |
| [19] | NCCLS.2009. M100-S19. Performance Standards for Antimicrobial Susceptibility Testing,Nineteenth Informational Supplement[S]. United States:Clinical and Laboratory Standards Institute |
| [20] | Onesios K M,Yu J T,Bouwer E J.2009. Biodegradation and removal of pharmaceuticals and personal care products in treatment systems:a review[J]. Biodegradation,20:441-466 |
| [21] | Qi Y,Putra G,Liang X,et al. 2001. Utilization of the rpoB gene as a specific chromosomal marker for real-time PCR detection of Bacillus anthracis[J]. Apply Environ Microbio,67:3720-3727 |
| [22] | Fard R M,Heuzenroeder M W,M D. 2011. Barton Antimicrobial and heavy metal resistance in commensal enterococci isolated from pigs[J]. Vet Microbiol,148:276-282 |
| [23] | Samuelsen O B,Torsvik V,Ervik A. 1992. Long-range changes in oxytetracycline concentration and bacterial resistance towards oxytetracycline in a fish farm sediment after medication[J]. Science of the Total Environment,114:25-36 |
| [24] | 田春杰,陈家宽,钟扬. 2003.微生物系统发育多样性及其保护生物学意义[J].应用生态学报,14(4):609-612 |
| [25] | Wagner M,Loy A. 2002. Bacterial community composition and function in sewage treatment systems[J]. Curr Opin Biotechnol,13:218-227 |
| [26] | 王冰,孙成,胡冠九. 2007.环境中抗生素残留潜在风险及其研究进展[J].环境科学与技术,30(3):108-111 |
| [27] | 王海燕,周岳溪,戴欣,等. 2006. 16S rDNA克隆文库方法分析MDAT-IAT同步脱氮除磷系统细菌多样性研究[J].环境科学学报,26(6):903-911 |
| [28] | 王新新,韩祯,白志辉,等. 2011.含油污泥的堆肥处理对微生物群落结构的分析[J].农业环境科学学报,30(7):1413-1421 |
| [29] | 肖斌,刘冲. 2012.环境中耐药菌的分布现状及其耐药机制的研究[J].广州化工,40(13):5-7 |
| [30] | Yim G,Wang H H M,Davies J. 2006. The truth about antibiotics[J]. Int J Med Microbiol,296:163-170 |
| [31] | 于雅琼,陈红艳,李平兰,等. 2007.不同生境中芽孢杆菌的分离鉴定及药敏性检测[J].食品科学,28(7):324-330 |
| [32] | 杨颖. 2010.北江水环境中抗生素抗性基因污染分析[D].广州:中山大学. 6-7 |
| [33] | 张俊,杨晓洪,葛峰,等. 2014.长期施用四环素残留猪粪对土壤中耐药菌及抗性基因形成的影响[J].环境科学,35(6):2374-2380 |
 2016, Vol. 36
2016, Vol. 36


