植物纤维原料是地球上最丰富的可再生资源(宋颖琦等,2002).我国有十分丰富的农林废弃物资源,每年仅农业生产中形成的农作物残渣(稻草、秸秆等)就约有7亿t(刘睿倩等,2000),此外还有大量的木材采伐后丢弃的树枝以及工业生产中产生的纤维素废弃物,其中绝大部分都被焚烧掉.因此,如果不合理有效地利用这一丰富的资源,不仅会造成巨大的资源浪费,还会污染环境.在自然界中纤维质的分解是在多种微生物的协同作用下完成的(Haq et al., 2005;陈耀宁,2007),据报道复合菌系混合培养相对于单菌的纯培养来说具有多酶的协同互补作用(Yang et al., 2003)、能够避免单菌培养过程中的底物抑制等问题(蒲一涛等,2000).此外,复合菌系中微生物间还可形成互利共生关系(涂漩等,2004),因此根据自然中微生物转化纤维质的模式来构建纤维素降解复合菌系是实现纤维质的高效利用的有效方法.
在前期的研究中,本课题组构建了一组嗜热木薯渣纤维质高效降解复合菌系,该菌系拥有较强的纤维素降解能力,可在培养40 h后使滤纸完全降解,且经60多次传代后仍具有高效的降解性能(Zhang et al., 2010).通过PCR-DGGE(变性梯度凝胶电泳)分析,发现该复合菌系主要由20多种微生物组成,包括纤维素降解菌和非纤维素降解菌,其中许多微生物属于梭菌属的厌氧菌和少量的好氧菌,进一步研究表明该复合菌系实现纤维素高效降解的关键功能菌为Paenibacillus sp和C. clariflavum (Zhang et al., 2013).此外,用该复合菌系处理木薯渣后再进行厌氧发酵,沼气的产量和发酵效率明显提高(Zhang et al., 2011).但是,该复合菌系高效的纤维素降解机制仍不清楚,为了更好地阐明该复合菌系的高效降解机制,研究其产生的纤维素结合蛋白就显得尤为重要.由于天然纤维素的结构和组成的复杂性,仅靠单一的酶将难以实现纤维素的高效降解,微生物之所以能够高效降解天然纤维素,主要是由于能够产生内切葡聚糖酶、外切葡聚糖酶以及β-葡萄糖苷酶等多种纤维素降解酶类以及半纤维素酶等,并且组成一个复杂的酶系来实现纤维质的降解.自然界中微生物所分泌的纤维素酶由于来源微生物种类的不同,其组分往往也有所差异,进而形成了各具特色的纤维素降解酶系.据文献报道,真菌是通过分泌游离的纤维素酶系来实现纤维素的协同降解(Medve et al., 1998);厌氧的纤维素降解菌则分泌纤维素降解复合酶系(即纤维小体)并通过其中的纤维结合蛋白结合于不溶的纤维基质上,最终实现纤维素的降解(Waeonukul et al., 2009).为此,要阐明纤维素降解复合菌群的高效降解机制则必须弄清复合菌群分泌的复合酶系(即纤维小体)的组成情况,因此必须要对复合菌群所产生的纤维小体中的酶蛋白进行分离纯化与鉴定.
传统的蛋白质分离纯化方法有等电点法、盐析法、溶剂法、双水相萃取法、层析法等多种方法(吴少辉和刘光明,2012),其中这些分离方法普遍存在成本高,操作繁琐、目标蛋白的回收率低等问题(Hong et al., 2008),难以实现如纤维小体等复合酶系的分离纯化,且可能导致复合酶系中组分的缺失.近年来一种新颖的分离纯化方法——亲和消化法已成功地应用于复合菌系中纤维结合蛋白的分离纯化(Wongwilaiwalin et al., 2010;Morag et al., 1992),该方法利用酶与底物的亲和性及底物的不溶性,经低温专一结合后,通过简单离心把吸附纤维小体的底物与不需要的部分直接分离开来,然后酶会将底物消化,释放出被亲和吸附上的酶,具有操作简单,回收率高等优点,可有效地避免功能蛋白的缺失,受到了广泛的关注.
在本研究中,我们采用亲和消化法分离纯化发酵液中的纤维素结合蛋白,经SDS-PAGE分离及酶谱分析后,再利用MALDI TOF/TOF串联质谱对分离出的条带进行了鉴定,并对纤维素结合蛋白的结构域进行了预测.该研究为更好地阐明该复合菌系的高效降解机制奠定了基础,也为破解特殊功能蛋白的“第二遗传密码”提供了更进一步的信息.
2 材料与方法(Materials and methods) 2.1 实验材料 2.1.1 菌种与培养基复合菌系是从稻田淤泥、高温堆肥、牛粪堆、腐木等富含腐败的木质纤维素物质的土壤中进行取样,经筛选、定向富集培养并驯化所得(Zhang et al., 2010).
纤维素降解培养基(g · L-1):胰蛋白胨 5.0、酵母粉 1.0、CaCO3 2.0、NaCl 5.0、木薯渣20.0,滤纸 4.0,初始pH 8.0.
2.1.2 主要仪器恒温培养箱(上海光都仪器设备有限公司);超净工作台(苏净集团安泰公司);立式压力蒸汽灭菌器(上海化线医用核子仪器有限公司);AB204-N型电子天平(上海第二天平仪器厂);pH电极(瑞士梅特勒公司);蛋白电泳仪(北京荣阳经典科技有限公司);4800 Plus MALDI TOF/TOF TM Analyzer(Applied Biosystems,USA).
2.2 试验方法 2.2.1 复合菌系中粗酶液的制备将复合菌系接种于150 mL纤维素降解培养基中(接种量为10%,体积分数),于55 ℃恒温培养箱中静置培养至滤纸完全降解,随后用8层纱布过滤,滤液于10000 r · min-1离心15 min.将上清液经0.45 μm的微孔膜过滤后,再用10 kDa的超滤管浓缩(4000 r · min-1,20 min),即得粗酶液.
2.2.2 无定型纤维素的制备将10 g微晶纤维素加入至500 mL 85%的浓磷酸中混匀后于25 ℃恒温水浴锅中溶解2 h,后加入5倍体积的蒸馏水搅拌均匀,再经5000 r · min-1离心15 min后,弃去上清液,再加入蒸馏水重新悬浮并再次离心,如此重复洗涤至pH接近中性,即得无定型纤维素.
2.2.3 亲和消化法提取纯化复合菌系中的纤维结合蛋白将上述无定型纤维素悬浮于粗酶液中,并于4 ℃,150 r · min-1振荡24 h,随后离心(6000 r · min-1,15 min)收集沉淀,并用缓冲溶液(CaCl2 12 mmol · L-1,EDTA 2 mmol · L-1,sodium phosphate 50 mmol · L-1,pH 6.0)重悬,然后转入透析袋(12000~14000 Da)并置于上述缓冲溶液中于50 ℃条件下透析24 h; 透析完后于10000 r · min-1离心10 min,收集上清液,并用10 kDa的超滤管浓缩(4000 r · min-1,20 min),即得纯化后的纤维结合蛋白浓缩液.
2.2.4 SDS-PAGE及酶谱分析SDS-PAGE电泳条件为:分离胶浓度10%,浓缩胶5%,电压120 V,电流30 mA;酶谱分析是在上述SDS-PAGE的分离胶中分别加入0.1%的木聚糖或CMC进行电泳,电泳后用加有25%异丙醇的缓冲溶液(sodium phosphate 100 mmol · L-1,pH=6.0)于室温条件下浸泡30 min,2次;随后再用不含有异丙醇的磷酸缓冲溶液清洗2次,每次30 min,使蛋白复性.然后将复性后的胶浸在pH=6.0的磷酸缓冲溶液中在50 ℃保温60 min,反应后将胶置于0.1%的刚果红中染色30 min,再浸在NaCL(1 mol · L-1)溶液中脱色60 min,最后将其置于0.5%的醋酸溶液中至显现透明的条带.
2.2.5 纤维结合蛋白的质谱鉴定将SDS-PAGE胶条上的蛋白条带进行切割回收,随后用胰蛋白酶进行胶内酶解.酶解液经脱盐、冻干处理后,再用5 μL 20%乙腈溶液复溶,取1 μL复溶样品与1 μL α-氰基-4-羟基肉桂酸基质进行均匀混合后,点靶,放在质谱仪上进行一级和二级质谱分析.将获得的二级质谱结果利用MASCOT软件搜索NCBI-nr数据库,寻求匹配的相关蛋白质.
2.2.6 纤维结合蛋白结构域分析采用pfam软件分析纤维结合蛋白的结构域:http://pfam.sanger.ac.uk/.
3 结果与讨论(Results and discussion) 3.1 SDS-PAGE及酶谱分析纤维素结合蛋白的SDS-PAGE及酶谱分析如图 1所示,得到的13条蛋白条带的分子量在50~200 kDa之间,且多数具有木聚糖酶(CBP1~4,CBP6和CBP8)或CMC酶(CBP1~9,CBP12)活性,有些条带(CBP1~4,CBP6和CBP8)兼具木聚糖和CMC酶活性,分子量大于66 kDa的蛋白条带的木聚糖酶及CMC酶活性较强,其中纤维结合蛋白CBP6的CMC酶和木聚糖酶活性最强,而分子量较小的条带主要表现CMC酶活性,其木聚糖酶活性很弱.此外,通过比较酶谱图可以看出,发酵液中的活性蛋白经浓缩、提取纯化后,酶活性明显增强,该结果表明采用亲和消化法提取发酵液中的纤维结合蛋白可防止其组分的缺失,并有效地实现了蛋白的回收.
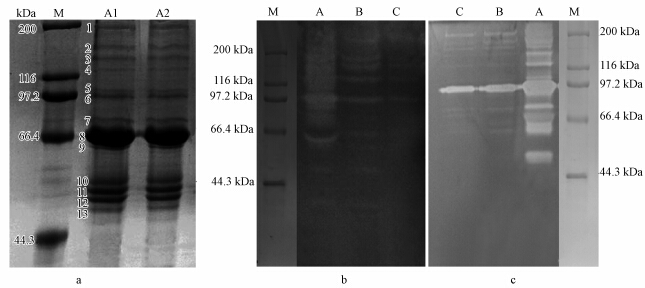 |
| 图 1 复合菌系中蛋白液的SDS-PAGE及酶谱分析(M: marker;A1、A2、A:蛋白纯化液;B:未纯化浓缩液;C:发酵原液;a. SDS-PAGE电泳;b. 木聚糖酶谱; c. CMC酶谱) Fig. 1 SDS-PAGE and spectrum analysis of protein solution of the microbial consortium |
将分离所得的条带切胶回收后进行质谱鉴定,结果如表 1所示.经鉴定这些电泳条带由8种蛋白所组成,分别来源于复合菌系中的Paenibacillus sp.和C. clariflavum DSM 19732,该结果与复合菌系中微生物组成鉴定结论一致(Zhang et al., 2010).其中,除条带4是由菌株Paenibacillus sp.产生的木聚糖酶5蛋白的外,其余均来源于梭菌C. clariflavum DSM 19732,其中条带1、2、3对应的是β-1,4-木聚糖酶,条带5和6对应的是碳水化合物结合蛋白,而条带7、8、9、11和12对应的是对接蛋白,条带10、13对应的则是未知功能的蛋白.各条带与相应蛋白的匹配分数均高于53,说明其匹配是显著的,理论等电点在4.46~5.60之间,理论分子量在50654至221017Da之间,但部分蛋白分子量的理论值与实际值存在差异,甚至分子量不同的蛋白被鉴定为同种蛋白(如CBP5和6、CBP7~9和11~12),这可能是由于蛋白在翻译后加工修饰有所不同而使其等电点或分子量发生改变,此外,同源性极高的相似性蛋白也可能会导致上述现象的发生(邱雁临,2001).
| 表 1 纤维素结合蛋白的质谱鉴定结果 Table 1 Mass spectrum identification of cellulose binding proteins |
由图 2可见,纤维结合蛋白CBP1~4均拥有第10家族糖苷水解酶的结构域,结合图 1还可发现这些蛋白均具有较强的木聚糖酶活性.此外,蛋白CBP1、CBP2及CBP4还拥有典型的碳水化合物结合域CBM,该结构域可加强酶与底物的接触,进而促进底物的水解.研究表明碳水化合域结合模块CBM可以大幅度提高结晶纤维素的降解率,除去CBM后的酶分子仍能降解可溶或无定型底物,但降解结晶纤维素的能力会下降甚至丧失(吕睿瑞,2010; Tomme et al., 1988),将催化结构域与CBM连接可显著增强其对结晶纤维素的降解活性(Gilkes et al., 1988);此外,S层同源结构域也被发现存在于纤维结合蛋白CBP1、CBP2及CBP4中,该结构域能与次生壁上的N-乙酰葡萄糖胺、N-乙酰甘露糖胺和丙酮酸相结合,增强蛋白质对细胞表面的附着(Maglione et al., 1992; Lemaire et al., 1998; Lemaire et al., 1995).而在蛋白CBP3中还发现含拥有细菌免疫球蛋白类结构域,该结构域通常被发现存在于细菌蛋白中(如intimin),通过嵌合在膜里对蛋白起到锚定的作用;纤维结合蛋白CBP5~6均拥有第9家族糖苷水解酶的结构域,能够分泌内切葡聚糖苷酶及纤维二糖酶,从而促进纤维素的降解;蛋白CBP7~9和CBP11~12均拥有第48家族糖苷水解酶的结构域,能够分泌内切-β-1,4-葡聚糖苷酶及纤维二糖酶;研究表明,第48家族的糖苷水解酶通常被发现存在于细菌纤维素酶系统中且被公认为是细菌分解纤维素基质的关键组分(Mesnage et al., 1999),且第9和48家族糖苷水解酶同时存在可促进纤维素的降解(Irwin et al.,2000; Berger et al., 2007);纤维结合蛋白CBP10还拥有第8家族糖苷水解酶的结构域,具有较强分泌纤维素酶及内切-β-1,4-木聚糖酶的能力.此外,锚定蛋白的存在(CBP5~12)进一步证实了该复合菌系是通过分泌纤维小体的方式来实现纤维素降解的.结合图 1c的酶谱图可见,纤维结合蛋白CPB6拥有较强的CMC酶活性,该蛋白具有GH9的催化结构域,据文献报道,GH9家族的酶是梭菌纤维素小体中活性最高的内切纤维素酶(Izquierdo et al., 2010),这与我们的研究结果是一致的.
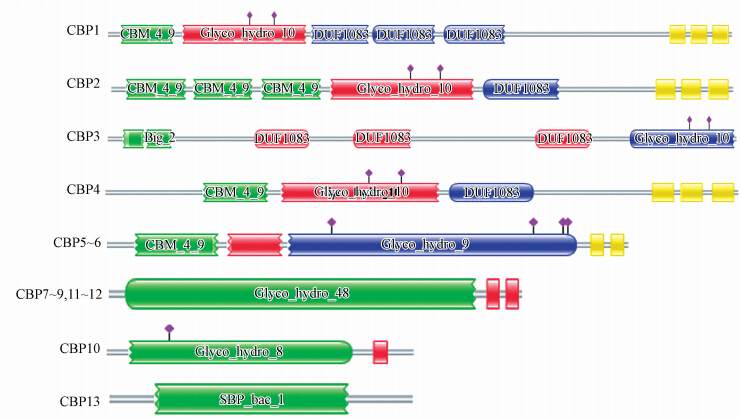 |
图 2
纤维结合蛋白的结构域预测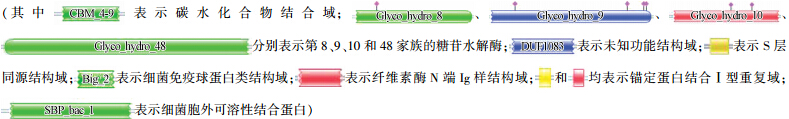 Fig. 2
Structure domain prediction of cellulose binding proteins
Fig. 2
Structure domain prediction of cellulose binding proteins
|
1)采用亲和消化法能有效地实现纤维结合蛋白的分离纯化并防止其功能组分的缺失,酶谱分析表明复合菌系中高分子量(66~200 kDa)的纤维结合蛋白具有木聚糖酶和CMC酶活性,其中蛋白CBP6表现出的木聚糖和CMC活性最强,而低分子量的蛋白主要表现CMC酶活性.
2)纤维结合蛋白经鉴定由8种蛋白所组成,其中蛋白CBP4为Paenibacillus sp.分泌的外,其余均是由C. clariflavum DSM 19732所产生的内切与外切纤维素酶及木聚糖酶所组成.
3)通过结构域预测,发现除纤维结合蛋白CBP13外所有的蛋白均具有一种催化结构域,分别隶属于第8、9、10及48家族的糖苷水解酶,而部分蛋白还具有碳水化合物结合域(CBP1,2,4~6)以及锚定蛋白结合域(CBP5~12).上述结果表明,纤维素降解复合菌系是通过形成纤维小体的模式来实现纤维素的高效降解.
| [1] | Berger E, Zhang D, Zverlov V V, et al. 2007. Two noncellulosomal cellulases of Clostridium thermocellum, Cel9I and Cel48Y, hydrolyse crystalline cellulose synergistically [J]. FEMS Microbiology Letters, 268(2): 194-201 |
| [2] | 陈耀宁. 2007. 堆肥化中协同降解木质纤维素的混合菌筛选及其培养. 长沙: 湖南大学. 1-166 |
| [3] | García-Campayo V, Béguin P. 1997. Synergism between the cellulosome-integrating protein CipA and endoglucanase CelD of Clostridium thermocellum [J]. Journal of Biotechnology, 57(1/3): 39-47 |
| [4] | Gilkes N R, Warren R A, Miller R C Jr, et al. 1988. Precise excision of the cellulose binding domains from two cellulomonas fimi cellulases by a homologous protease and the effect on catalysis [J]. Journal of Biological Chemistry, 263: 10401-10407 |
| [5] | Haq I U, Javed M M, Khan T S, et al. 2005. Cotton saccharifying activity of cellulases produced by co-culture of Aspergillus niger and Trichoderma viride [J]. Research Journal of Agriculture and Biological Sciences, 1(3): 241-245 |
| [6] | Hong J, Ye X H, Wang Y R, et al. 2008. Bioseparation of recombinant cellulose-binding module-proteins by affinity adsorption on an ultra-high-capacity cellulosic adsorbent [J]. Analytica Chimica Acta, 621(2): 193-199 |
| [7] | Irwin D C, Zhang S, Wilson D B. 2000. Cloning, expression and characterization of a family 48 exocellulase, Cel48A, from Thermobifida fusca [J]. European Journal of Biochemistry, 267(16): 4988-4997 |
| [8] | Izquierdo J A, Sizova M V, Lynd L R. 2010. Diversity of bacteria and glycosyl hydrolase family 48 Genes in cellulolytic consortia enriched from thermophilic biocompost [J]. Applied and Environmental Microbiology, 76(11): 3545-3553 |
| [9] | Lemaire M, Ohayon H, Gounon P, et al. 1995. OlpB, a new outer layer protein of Clostridium thermocellum, and binding of its S-layer-like domains to components of the cell envelope [J]. Journal of Bacteriology, 177(9): 2451-2459 |
| [10] | Lemaire M, Miras I, Gounon P, et al. 1998. Identification of a region responsible for binding to the cell wall within the S-layer protein of Clostridium thermocellum [J]. Microbiology, 144(1): 211-217 |
| [11] | 刘睿倩, 宋金柱, 宋颖琦, 等. 2000. 影响玉米秸秆微生物降解制肥的因素浅析[J]. 环境卫生工程, 10(3): 103-105 |
| [12] | 吕睿瑞. 2010. 天然木质纤维素降解相关酶的宏蛋白质组学分析与鉴定. 福州: 福建师范大学. 1-147 |
| [13] | Maglione G, Matsushita O, Russel J B, et al. 1992. Properties of a genetically reconstructed Prevotella ruminicola endoglucanase [J]. Applied and Environmental Microbiology, 58(11): 3593-3597 |
| [14] | Medve J, Karlsson J, Lee D, et al. 1998. Hydrolysis of microcrystalline cellulose by cellobiohydrolase I and endoglucanase II from Trichoderma reesei: adsorption, sugar production pattern, and synergism of the enzymes [J]. Biotechnology and Bioengineering, 59(5): 621-634 |
| [15] | Mesnage S, Tosi-Couture E, Fouet A. 1999. Production and cell surface anchoring of functional fusions between the SLH motifs of the Bacillus anthracis S-layer proteins and the Bacillus subtilis levansucrase [J]. Molecular Microbiology, 31(3): 927-936 |
| [16] | Morag E, Bayer E A, Lamed R. 1992. Affinity digestion for the near-total recovery of purified cellulosome from Clostridium thermocellum [J]. Enzyme and Microbial Technology, 14(4): 289-292 |
| [17] | 蒲一涛, 钟毅沪, 郑宗坤. 2000. 混合培养对固氮菌和纤维素分解菌生长及固氮的影响[J]. 氨基酸和生物资源, 22(1): 1-4 |
| [18] | 邱雁临. 2001. 纤维素酶的研究和应用前景[J]. 粮食与饲料工业, (8): 30-31 |
| [19] | 宋颖琦, 刘睿倩, 杨谦, 等. 2002. 纤维素降解菌的筛选及其降解特征的研究[J]. 哈尔滨工业大学学报, 34(2): 197-200 |
| [20] | Tomme P, Van Tilbeurgh H, Pettersson G, et al. 1988. Studies of the cellulolytic system of Trichoderma reesei QM 9414 [J]. European Journal of Biochemistry, 170(3): 575-581 |
| [21] | 涂漩, 薛泉宏, 司美茹, 等. 2004. 多元混菌发酵对纤维素酶活性的影响[J]. 工业微生物, 34(1): 30-34 |
| [22] | Waeonukul R, Kyu K L, Sakka K, et al. 2009. Isolation and characterization of a multienzyme complex (cellulosome) of the Paenibacillus curdlanolyticus B-6 grown on Avicel under aerobic conditions [J]. Journal of Bioscience and Bioengineering, 107(6): 610-614 |
| [23] | Wongwilaiwalin S, Rattanachomsri U, Laothanachareon T, et al. 2010. Analysis of a thermophilic lignocellulose degrading microbial consortium and multi-species lignocellulolytic enzyme system [J]. Enzyme and Microbial Technology, 47(6): 283-290 |
| [24] | 吴少辉, 刘光明. 2012. 蛋白质分离纯化方法研究进展[J]. 中国药业, 21(1): 1-3 |
| [25] | Yang Y H, Wang B C, Xiang L J, et al. 2003. Construction of a microbial consortium used for solid-state fermentation on rice chaff [J]. Colloids and Surfaces B: Biointerfaces, 32(1): 51-56 |
| [26] | Zhang Q H, Lu X, Tang L, et al. 2010. A novel full recycling process through two-stage anaerobic treatment of distillery wastewater for bioethanol production from cassava [J]. Journal of Hazardous Materials, 179(1/3): 635-641 |
| [27] | Zhang Q H, He J, Tian M, et al. 2011. Enhancement of methane production from cassava residues by biological pretreatment using a constructed microbial consortium [J]. Bioresource Technology, 102(19): 8899-8906 |
| [28] | Zhang Q H, Tian M, Tang L, et al. 2013. Exploration of the key microbes involved in the cellulolytic activity of a microbial consortium by serial dilution [J]. Bioresource Technology, 132: 395-400 |
 2015, Vol. 35
2015, Vol. 35


