2. 中国矿业大学(北京)化学与环境工程学院, 北京 100083
2. School of Chemical & Environmental Engineering, China University of Mining & Technology, Beijing 100083
甲烷(CH4)是一种重要的温室气体,其潜在增温效应是CO2的25倍(Cicerone and Oreml and ,1988).很多年来,人们认为CH4主要是在好氧条件下被氧化的,在厌氧环境中CH4的氧化仅限于同硫酸盐还原的协作反应中(Barnes and Goldberg, 1976).2006年,荷兰学者Raghoebarsing等首次在反应器富集培养物中证实了n-damo反应的存在(Raghoebarsing et al., 2006),改变了对甲烷循环的认识.n-damo反应是由反硝化厌氧甲烷氧化菌(nitrite-dependent anaerobic methane-oxidizing bacteria,n-damo菌)主导发生的,即甲烷在厌氧条件下以亚硝酸盐为电子受体发生氧化反应.2010年,n-damo菌的全基因序列第一次被获取,并被命名为“C and idatus Methylomirabilis oxyfera”,为隶属于NC10门的细菌(Ettwig et al., 2010),具有特殊的内部好氧机制,其生理特性和分子生物学特性已有相关报道(袁梦冬等,2014).通过对“Candidatus Methylomirabilis oxyfera”菌全基因序列的组装和分析,发现该菌在厌氧环境中所特有的代谢方式是由其所含有的pmoCBA基因编码的甲烷单加氧酶催化完成的.近年来,随着针对pmoA基因的特异性引物的开发,M.oxyfera-like菌被发现广泛存在于各种自然生态环境中,如德国的Constance湖(Deutzmann and Schink, 2011),日本的Biwa湖(Kojima et al., 2012),荷兰的Brunssummerheide泥炭(Zhu et al., 2012),中国的嘉兴高污染稻田(Wang et al., 2012)和钱塘江沉积物(Shen et al., 2013)等.
水稻田是甲烷形成和释放的重要生态环境之一(Bouwman,1989),全球每年由水稻田排放到大气的CH4约为甲烷总排放量的4%~19%(Le Mer and Roger, 2001).水稻田中的n-damo反应可以在一定程度上减少CH4释放,而有关水稻田中M.oxyfera-like菌分布的报道还较少.本研究以夏季和冬季江阴稻田土壤柱状样品为研究对象,旨在通过分子生物学技术,分析M.oxyfera-like菌在稻田土壤中的时空分布和群落结构特征,为水稻田甲烷的减排提供一定的理论依.
2 材料与方法(Materials and methods) 2.1 样品的采集与保存江苏省无锡市江阴县地处长江流域,属北亚热带季风性落叶阔叶、常绿阔叶林气候,夏季和冬季温差较大,平均温度分别为0 ℃和25 ℃,土壤类型为黄棕壤、黄褐土带.本研究选择了一处距离长江河岸500 m的典型稻田(120°3′31″ E,31°56′40″ N)作为研究对象,分别进行了夏季和冬季的土壤柱状样品的采集(2012年9月,2013年2月).夏季和冬季稻田土壤采样深度分别为140 cm和200 cm(均为每20 cm 1个样品),每个季节的样品经3点采集后均匀混合贮存于无菌自封袋中密封,4 ℃保存至实验室,立即进行理化性质测定,其余样品冻干后存放于-80 ℃超低温冰箱中,用于DNA提取和后续分子生物学实.
2.2 理化指标的测定土壤相关指标依照《土壤农化分析》中方法测定(鲍士旦,2000).其中NH+4、NO-3、NO-2含量是将土壤样品经浓度为2 mol · L-1的KCl溶液浸提1 h并抽滤后,用连续流动分析仪测定(SAN plus,Skalar Analytical B.V.,Breda,the Netherl and s).测定LOI550(550 ℃的烧失量)以代表土壤总有机质(张文河和穆桂金,2007).所有样品均进行3次平行测定以控制数据质量.
2.3 DNA的提取和聚合酶链式反应(PCR)称取约0.33 g经冷冻干燥后的土壤样品,利用FastDNA Spin kit for soil提取试剂盒(MP Biomedicals,USA)提取样品中的总DNA,得到的75 μL DNA溶液于-20 ℃保存待用.
M. oxyfera-like菌的16S rRNA基因通过巢式PCR进行扩增,第一步使用引物202F(Ettwig et al., 2009)和630R(Juretschko et al., 1998),第二步使用引物p1F/ p2R(Ettwig et al., 2009).利用针对M. oxyfera-like菌的pmoA功能基因的特异性引物对其进行巢式PCR扩增,第一步使用引物A189F/cmo682(Luesken et al., 2011),第二步使用引物cmo182/cmo568(Luesken et al., 2011),所用引物序列和PCR扩增程序见表 1.
| 表1 本论文中用到的引物及相应PCR程序 Table 1 Primers used in this study and correspondent thermal profiles |
PCR扩增产物用Promega Agarose Gel DNA(Promega,Madison,WI)纯化试剂盒进行切胶纯化,将回收的PCR产物连接到pGEM-Teasy载体(Promega,Madison,WI),然后转入JM109感受态细胞(TAKARA,大连),最后进行蓝白斑筛选.从每个克隆文库中随机挑选96个白色克隆,采用PCR扩增的方法鉴定阳性克隆,所用引物为载体通用引物T7、SP6.之后使用限制性内切酶Hha I对阳性克隆PCR产物进行酶切分型,每个酶切类型挑选代表菌株进行测序(Tsingke生物技术有限公司,北京),将得到的序列在Genbank中进行BLAST比对并选取参比序列,运用Mega 4.1软件以邻接法(Neighbor-Joining)构建系统发育树.独立操作单元(operational taxonomic unit,OTU)、Chao1丰富度指数(chao1 richness estimate)和香农多样性指数(shannon diversity index)通过DOTUR软件在3%(16S rRNA基因序列)或2%(pmoA基因序列)的差异性水平下进行计算得出(Schloss and Handelsman, 2005).
2.5 M.oxyfera-like菌和全细菌丰度测定采用实时荧光定量PCR方法分别对M.oxyfera-like菌和全细菌进行定量分析,定量PCR均采用SYBR Green法.由于没有针对M.oxyfera-like菌功能基因的定量引物,本研究中通过对M.oxyfera-like菌的16S rRNA基因进行定量来测定其丰度.使用两对不同的引物(p1F/R,p2F/R)对各个样品进行定量PCR以提高丰度测定的准确性,这两对引物是根据从富集培养物中获取的16S rRNA序列设计的(Ettwig et al., 2009).对于全细菌,使用引物341F/534R对其16S rRNA基因进行定量PCR(Linares and Sundbäck,2006),引物序列和扩增程序均列于表 1中.将引物与SYBR Green ExTaq 酶试剂盒(TAKARA,大连)按比例配制成20 μL反应体系,使用ABI 7300 Real-Time PCR System扩增仪(Applied Biosystems,CA,USA)进行定量,并将拷贝数已知的质粒DNA按10倍梯度稀释,得到6个标准样品同时进行定量扩增,得到标准曲线.定量PCR结果用7300 SDS System 配套软件进行分析运算,得出M.oxyfera-like菌16S rRNA基因的丰度值.每个样品做3次平行,要求结果扩增效率和可决系数R2分别大于95%和0.98,溶解曲线为单一峰.
2.6 数据分析利用PASW Statistics 18.0软件(Predictive Analytics Software Statistics)进行t检验;利用Origin 8.0软件作图.
3 结果(Results) 3.1 稻田土壤柱状样品的理化性质江阴稻田土壤柱状样品的主要理化性质如图 1所示.夏季和冬季土壤柱状样品中NOx-的含量分别为0.27~3.10 mg · kg-1和0.31~1.11 mg · kg-1,最高值分别出现在20~40 cm和100~120 cm.夏季和冬季样品中氨氮的含量分别为0.85~1.75 mg · kg-1和0.63~7.24 mg · kg-1,且在0~160 cm土壤中含量很低.夏季和冬季土壤pH值分别为7.2~7.4和7.0~7.5,呈弱碱性.TC、TN、LOI550、TP、TS和含水率的最大值均出现在表层土壤中,其中冬季的TS含量明显高于夏季.
 |
| 图 1 江阴稻田土壤柱状样品理化性质 Fig. 1 Chemical characteristics of paddy soil cores in Jiangyin |
采用实时荧光定量PCR技术,同时利用两对引物p1F/R和p2F/R(Ettwig et al., 2009)对M. oxyfera-like菌的16S rRNA基因进行定量,由图 2可看出两对引物的定量结果相近,因而取其平均值进行分析.夏季稻田土壤柱状样品中 M. oxyfera-like菌的丰度值为5.97×105~1.01×108 copies · g-1(以干土计,下同),冬季稻田土壤柱状样品中M. oxyfera-like菌的丰度值为2.61×105~8.48×107 copies · g-1.由图 2可以看出,在冬季和夏季,稻田土壤柱状样中M. oxyfera-like菌的丰度值均随着土壤深度的增加而呈增加趋势,且最大值均出现在100~120 cm处,最小值均出现在表层土壤中.此外,M. oxyfera-like菌的丰度没有明显的季节性变化(t-test,sig>0.05).
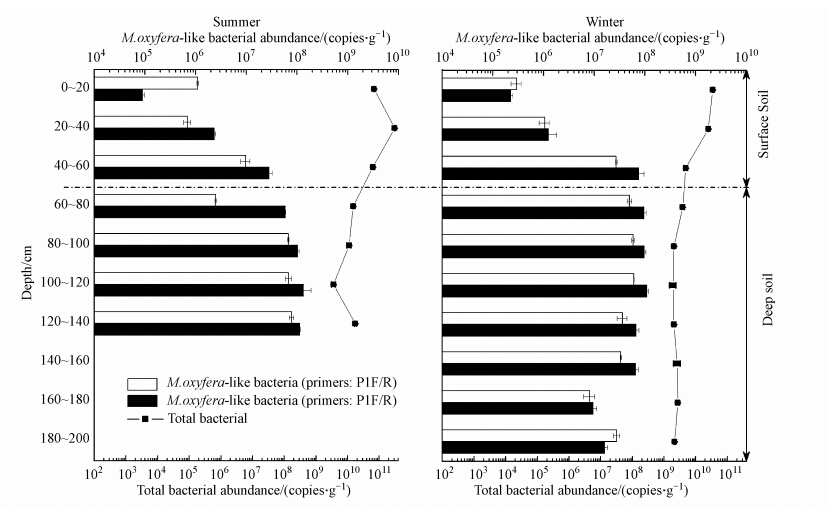 |
| 图 2 江阴稻田土壤柱状样品M.oxyfera-like菌和全细菌的丰度 Fig. 2 Abundance of M.oxyfera-like bacteria and total bacteria in paddy soil cores |
为了考察稻田土壤中M.oxyfera-like菌在全细菌中所占的比例,同时对稻田土壤样品中全细菌的16S rRNA基因进行定量.由图 2可知,全细菌的丰度随土壤深度的增加逐渐降低,夏季和冬季稻田土壤样品M.oxyfera-like菌的丰度在全细菌中所占的比例的最大值分别为2.80%和4.41%,均出现在100~120 cm,最低值均出现在表层土壤中.
鉴于用来定量分析的pF1/R和pF2/R两对引物是根据富集样品的克隆序列设计得出,有关其在环境样品中的应用的报道较少,因此对其所针对的16S rRNA基因进行定性克隆测序和系统发育分析来进一步考察M.oxyfera-like菌定量结果的准确性.构建克隆文库后得到41个夏季序列和42个冬季序列,利用DOTUR软件在3%的差异性水平下将此83个序列划分为17个OTU,将每个OTU的代表序列与从Genbank中挑选的参比序列一同构建系统发育树,如图 3Ⅰ所示.
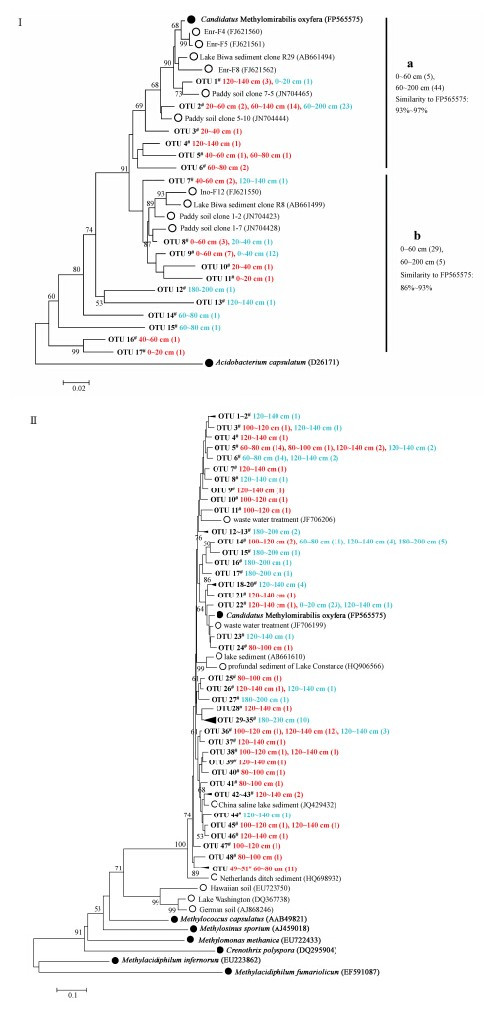 |
| 图 3 稻田土壤中M.oxyfera-like菌的16S rRNA基因(Ⅰ)和pmoA基因(Ⅱ)的系统发育树(每个OTU之后列出属于该OTU的序列所来自的土壤深度及序列数,红色和蓝色分别代表来自夏季和冬季的序列) Fig. 3 Phylogenetic tree of M.oxyfera-like bacterial 16S rRNA gene sequences(Ⅰ) and pmoA gene sequences(Ⅱ)from paddy soils. The depth of soils where one OTU came from were listed behind each OTU,with sequence numbers behind the depth. Red color and blue color represent sequences from summer and winter respectively |
通过系统发育分析可得,属于OTU 1#~OTU 6#的16S rRNA序列与NC10门的a组类(Ettwig et al., 2009)序列聚为一个分支,且与已知的M. oxyfera菌(FP565575)具有较高的亲缘性(93%~97%).OTU 1#~OTU 6#包含的49个序列中有90%来自于60~200 cm深度的土壤,仅有5个序列来自于0~60 cm深度的土壤.然而,OTU 7#~OTU 17#的34个16S rRNA序列则与NC10门的b组类(Ettwig et al., 2009)序列聚为一个分支,且与已知的M. oxyfera菌(FP565575)的亲缘关系较远(86%~93%),其中来自0~60 cm深度的土壤样品的序列占85%,来自60~200 cm深度土壤的序列仅为5个.
这一结果说明了由于目前针对M. oxyfera菌16S rRNA基因的引物特异性不足,之前的依据16S rRNA的定量结果有可能对其丰度有过高的估计,特别是对于表层土壤.因此,综合qPCR分析与系统发育分析的结果,说明60~200 cm深度的深层土壤可能是M. oxyfera-like菌的高丰度区域.
3.3 M.oxyfera-like菌pmoA序列的群落结构分析为了研究稻田柱状样品中M.oxyfera-like菌的群落结构特征,对夏季和冬季土壤柱状样品的M.oxyfera-like菌的pmoA基因通过巢式PCR进行扩增.夏季0~60 cm和冬季20~40 cm深度土壤样品的DNA的扩增在第二步PCR中未获得阳性结果.
选取夏季60~80 cm、80~100 cm、100~120 cm、120~140 cm 4个深度和冬季0~20 cm、60~80 cm、120~140 cm、180~200 cm 4个深度的土壤样品的pmoA基因PCR阳性产物构建克隆文库.共得到来自夏季土壤样品的68个pmoA序列和来自冬季土壤样品的92个pmoA序列.利用DOTUR软件将这160个pmoA序列在2%的差异性水平上划分为51个OTU,将每个OTU的代表序列与从Genbank中挑选的参比序列一同构建系统发育树,如图 3(Ⅱ)所示.可以看出,得到的pmoA序列与好氧甲烷菌的亲缘关系较远,而与M.oxyfera-like菌的亲缘关系较近;来自不同季节或不同深度的土壤样品的pmoA序列较少出现在同一个OTU中,说明M.oxyfera-like菌在稻田土壤中的群落结构具有明显的时空异质性.将每个OTU的代表序列与已知的M.oxyfera菌(FP565575)进行BlAST比对,得到的同源性为86%~97%.其中夏季样品的pmoA序列与M.oxyfera(FP565575)的同源性为89%~95%,冬季样品的pmoA序列与M.oxyfera (FP565575)的同源性为86%~97%.
利用DOTUR软件,用混乱度估计值的方法对克隆文库的物种丰富度进行估算,得到Chao1丰富度指数为125,通过计算香农多样性指数来分析该菌的多样性,得到指数为3.04.为研究不同季节和深度土壤中M.oxyfera-like菌的多样性,分别对来自夏季和冬季不同深度土壤样品的M.oxyfera-like菌pmoA序列在2%的差异性水平下进行多样性分析,得到的生物多样性指数如图 4所示.
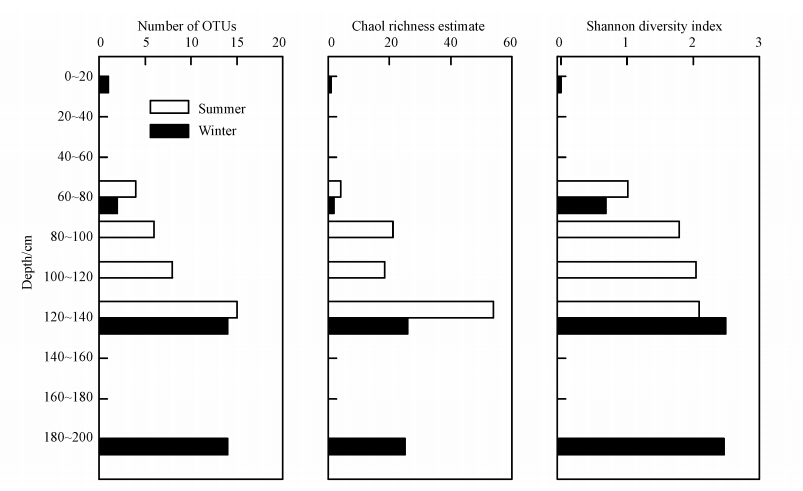 |
| 图 4 不同季节和深度土壤样品中M.oxyfera-like菌pmoA序列的生物多样性指数 Fig. 4 Biodiversity indexes of M.oxyfera-like bacterial pmoA gene sequences from soil samples of different seasons and depths |
可以看出OTU、Chao1丰富度指数和香农多样性指数均随着土壤深度的增加呈递增的趋势.来自夏季和冬季土壤样品的pmoA基因的OTU数分别为4~15和1~14,夏季和冬季最大值分别出现在120~140 cm和120~140 cm、180~200 cm深度处;夏季和冬季Chao1丰富度指数分别为4~54和1~26,两季最大值均出现在120~140 cm;夏季和冬季香农多样性指数分别为1.02~2.09和0~2.49,两季最大值也均出现在120~140 cm.
4 讨论(Discussion)本文通过定量PCR技术考察了M.oxyfera-like菌在江阴稻田土壤中的时空分布,得到夏季和冬季M.oxyfera-like菌的丰度值分别为5.97×105~1.01×108 copies · g-1和2.61×105~8.48×107 copies · g-1,明显高于之前报道的其他自然环境,如日本的Biwa湖泊(2.0×104~1.0×106 copies · mL-1(Sediment)),荷兰Brunssumerheide泥炭(1.3×107~3.2×107 copies · g-1(wet soil))、嘉兴高污染稻田(<1.0×105 copies · g-1)、钱塘江沉积物(1.32×106~1.03×107 copies · g-1)等(Kojima et al., 2012; Zhu et al., 2012; Wang et al., 2012; Shen et al., 2013),是目前自然环境中所报道的最高值.
M. oxyfera-like菌的丰度没有明显的季节性变化(t-test,sig>0.05),这说明在一定范围内,温度可能不是影响自然环境中M. oxyfera-like菌丰度的关键环境因子.此外,随着土壤深度的增加,M.oxyfera-like菌的丰度呈增加趋势,结合M. oxyfera-like菌的16S rRNA基因的系统发育分析,在60~200 cm深度的土壤中得到了M.oxyfera-like菌的高丰度区域.深层土壤中M.oxyfera-like菌高丰度区域(60~200 cm)的产生可能归因于深层土壤严格的厌氧环境和充足的反应底物CH4和NO-2.首先,深层土壤较表层土壤来说,不受翻耕活动的影响,且与大气中的氧气接触较少,具有更为严格的厌氧环境,有利于M.oxyfera-like菌这一厌氧微生物的生长.而表层土壤中存在的微量氧气对于M.oxyfera-like菌的生长有抑制作用(Luesken et al., 2012),这同时也解释了为何在夏季0~60 cm和冬季20~40 cm的稻田土壤样品中未获得M.oxyfera-like菌pmoA基因的阳性PCR产物.其次,稻田土壤中富含有机质,包括水稻根系分泌物、植物凋落物等,这些有机物在厌氧条件下由产甲烷古菌作用生成CH4(张广斌,2011),为M.oxyfera-like菌的生长提供了充足的反应底物.此外,氮肥的使用向稻田土壤中输入了大量无机氮,包括NH+4和NO-3.在氨氧化细菌(AOB)、氨氧化古菌(AOA)和硝酸盐还原菌的作用下,NH+4的氧化和NO-3的还原均可为M.oxyfera-like菌提供电子受体NO-2(Wang et al., 2011; Kojima et al., 2012; Wang et al., 2013; 2014).
本文还通过对M.oxyfera-like菌pmoA序列的系统发育分析和生物多样性分析,考察了稻田土壤中M.oxyfera-like菌的群落结构.pmoA基因系统发育分析显示在稻田土壤中M.oxyfera-like菌的群落结构具有明显的时空异质性,反应了pmoA功能基因的相对保守性;生物多样性分析显示江阴稻田土壤中的M.oxyfera-like菌具有很高的生物多样性,OTU、Chao1丰富度指数和香农多样性指数分别为51、125和3.04,明显高于其他已报道的自然环境,如德国Constance湖泊(15个pmoA序列在2%的差异性水平下划分为1个OTU,Chao1丰富度指数为1,香农多样性指数为0),日本Biwa湖泊(21个pmoA序列在1%的差异性水平下划分为2个OTU,Chao1丰富度指数为2,香农多样性指数为0.49)等(Deutzmann and Schink, 2011; Kojima et al., 2012).此外,随着土壤深度的增加,OTU、Chao1丰富度指数和香农多样性指数均增加,说明M.oxyfera-like菌的多样性可能随着深度的增加而增加,进一步说明了深层土壤是适宜M.oxyfera-like菌生存的生态环境.
5 结论(Conclusions)1)江阴稻田中60~200 cm深度的土壤是M.oxyfera-like菌的高丰度区域,得到了至今自然环境中已报道的M.oxyfera-like菌丰度的最高值.
2)江阴稻田土壤中M.oxyfera-like菌的群落结构具有一定的时空异质性,且生物多样性明显高于其他已报道的自然环境.
3)M.oxyfera-like菌的丰度和生物多样性均随着土壤深度的增加而增加,说明稻田深层土壤(60~80 cm)是适宜M.oxyfera-like菌生存的生境.
| [1] | 鲍士旦. 2000. 土壤农化分析[M]. 北京: 中国农业出版社 |
| [2] | Barnes R O, Goldberg E D. 1976. Methane production and consumption in anoxic marine sediments [J]. Geology, 4(5): 297-300 |
| [3] | Bouwman A F, 1989. The role of soils and land use in the greenhouse effect [J]. Netherlands Journal of Agricultural Science, 37(1989): 13-19 |
| [4] | Cicerone R J, Oremland R S. 1988. Biogeochemical aspects of atmospheric methane [J]. Global Biogeochemical Cycles, 2(4): 299-327 |
| [5] | Deutzmann J S, Schink B. 2011. Anaerobic oxidation of methane in sediments of Lake Constance, an oligotrophic Freshwater Lake [J]. Applied and Environmental Microbiology, 77(13): 4429-4436 |
| [6] | Ettwig K F, van Alen T, van de Pas-Schoonen K T, et al. 2009. Enrichment and molecular detection of denitrifying methanotrophic bacteria of the NC10 phylum [J]. Applied and Environmental Microbiology, 75(11): 3656-3662 |
| [7] | Ettwig K F, Butler M K, Le Paslier D, et al. 2010. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria [J]. Nature, 464(7288): 543-548 |
| [8] | Juretschko S, Timmermann G, Schmid M, et al. 1998. Combined molecular and conventional analyses of nitrifying bacterium diversity in activated sludge: Nitrosococcus mobilis and Nitrospira-like bacteria as dominant populations [J]. Applied and Environmental Microbiology, 64(8): 3042-3051 |
| [9] | Kojima H, Tsutsumi M, Ishikawa K, et al. 2012. Distribution of putative denitrifying methane oxidizing bacteria in sediment of a freshwater lake, Lake Biwa [J]. Systematic and Applied Microbiology, 35(4): 233-238 |
| [10] | Le Mer J, Roger P. 2001. Production, oxidation, emission and consumption of methane by soils: A review [J]. European Journal of Soil Biology, 37(1): 25-50 |
| [11] | Linares F, Sundbäck K. 2006. Uptake of dissolved free amino acids (DFAA) by microphytobenthic communities [J]. Aquatic Microbial Ecology, 42(2): 175-186 |
| [12] | Luesken F A, Zhu B L, van Alen T A, et al. 2011. pmoA primers for detection of anaerobic methanotrophs [J]. Applied and Environmental Microbiology, 77(11): 3877-3880 |
| [13] | Luesken F A, Wu M L, Op den Camp H J M, et al. 2012. Effect of oxygen on the anaerobic methanotroph 'Candidatus Methylomirabilis oxyfera': kinetic and transcriptional analysis[J]. Environmental Microbiology, 14(4): 1024-1034 |
| [14] | Raghoebarsing A A, Pol A, van de Pas-Schoonen K T, et al. 2006. A microbial consortium couples anaerobic methane oxidation to denitrification [J]. Nature, 440(7086): 918-921 |
| [15] | Schloss P D, Handelsman J. 2005. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness [J]. Applied and Environmental Microbiology, 71(3): 1501-1506 |
| [16] | Shen L D, Liu S, Zhu Q, et al. 2013. Distribution and diversity of nitrite-dependent anaerobic methane-oxidising bacteria in the sediments of the Qiantang River [J]. Microbial Ecology, 67(2): 341-349 |
| [17] | Wang S Y, Wang Y, Feng X J, et al. 2011. Quantitative analyses of ammonia-oxidizing Archaea and bacteria in the sediments of four nitrogen-rich wetlands in China [J]. Applied Microbiology and Biotechnology, 90(2): 779-787 |
| [18] | Wang Y, Zhu G B, Harhangi H R, et al. 2012. Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil [J]. FEMS Microbiology Letters, 336(2): 79-88 |
| [19] | Wang C X, Zhu G B, Wang W D, et al. 2013. Preliminary study on the distribution of ammonia oxidizers and their contribution to potential ammonia oxidation in the plant-bed/ditch system of a constructed wetland [J]. Journal of Soils and Sediments, 13(9): 1626-1635 |
| [20] | Wang Y, Zhu G B, Song L Y, et al. 2014. Manure fertilization alters the population of ammonia-oxidizing bacteria rather than ammonia-oxidizing archaea in a paddy soil [J]. Journal of Basic Microbiology, 54(3): 190-197 |
| [21] | 袁梦冬, 朱静, 吴伟祥. 2014. 微生物厌氧甲烷氧化反硝化研究进展[J]. 微生物学报, 54(2): 129-138 |
| [22] | 张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理, 30(3): 455-459 |
| [23] | 张广斌, 马静, 徐华, 等. 2011. 稻田甲烷产生途径研究进展[J]. 土壤, 43(1): 6-11 |
| [24] | Zhu B L, van Dijk G, Fritz C, et al. 2012. Anaerobic oxidization of methane in a minerotrophic peatland: enrichment of nitrite-dependent methane-oxidizing bacteria [J]. Applied and Environmental Microbiology, 78(24): 8657-8665 |
 2015, Vol. 35
2015, Vol. 35


 , 周磊榴1, 邹雨璇1, 王春荣2
, 周磊榴1, 邹雨璇1, 王春荣2