植物正常有氧代谢会产生活性氧(Reactive Oxygen Species,ROS),包括超氧阴离子、羟自由基、H2O2和单线态氧等,其中,H2O2在胞内的寿命较长.逆境胁迫时,植物体内ROS水平升高,会引发生物大分子如核酸、蛋白、脂类等氧化损伤,影响细胞结构和功能,因此,ROS长期以来被认为是一类具有损伤效应的毒性物质.近期研究发现,ROS可作为信号分子参与对多种细胞过程的信号转导,从而对一些重要的细胞活动和植物生理过程起调控作用(程艳丽等,2005).胁迫诱发的胞内H2O2增高可能参与调节相关基因的转录表达,继而影响植物生理代谢过程,提高植株对环境的适应性.
DNA甲基化是基因组最重要的表观遗传修饰之一,在调控重要功能基因表达、基因组防御及细胞发育分化等方面具有重要作用(Jaenisch et al., 2003; Lukens et al., 2007; Chinnusamy et al., 2009).盐、干旱和重金属等能诱发植物DNA甲基化特征改变(Wang et al., 2011;杨金兰等,2007;彭海等,2011),胁迫诱发的DNA甲基化变异在植物适应环境的过程中发挥了重要作用,某些甲基化变异的继代遗传可能是产生物种多样性的一个原因(Mirouze et al., 2011).由于逆境胁迫时植物细胞内ROS增高与甲基化变异同时发生,推测ROS可能对DNA甲基化修饰产生了某种影响,但具体机制还不清楚.
植物生长素参与植物体内多种生理过程,腈水解酶2(Nitrilase2,NIT2)是生长素合成途径中的一个关键酶,对植物生长素的合成具有重要作用(Park et al., 2003; Vorwerk et al., 2001).一些生物和非生物胁迫可导致NIT2基因的差异表达,通过调节生长素的生物合成来调控植物对逆境胁迫的应答反应(Grsic-Rausch et al., 2000).研究发现,拟南芥NIT2基因在甲基转移酶突变体中表达增强(Zhang et al., 2006),暗示胁迫条件下基因表达水平的变化可能与DNA甲基化状态有关.我们近期的研究发现,SO2胁迫后NIT2基因表达上调,基因启动子区甲基化水平降低,证明了胞嘧啶甲基化修饰对该基因转录的影响(李利红等,2012).但胁迫生理中NIT2基因甲基化特征及转录水平改变是否与胞内ROS升高有关,有待进一步研究.为此,本文采用外源H2O2对拟南芥植株进行胁迫处理,研究ROS升高对拟南芥NIT2基因DNA甲基化特征和转录水平的影响,以期为植物逆境适应中表观遗传学机制的研究提供依据.
2 材料与方法(Materials and methods) 2.1 植株培养拟南芥(Arabidopsis thaliana L.)Columbia生态型(Col-0),4 ℃春化2 d后播种于泥炭藓、蛭石、珍珠岩按 1 ∶ 1 ∶ 1混合的营养土中;然后放置于培养室培养,光照度≥ 3000 lx,光/暗周期为16 h/8 h,培养温度22 ℃,相对湿度约70%.
2.2 H2O2处理取4周龄拟南芥植株,用1、10和100 µmol · L-1的H2O2进行预实验,从顶部喷淋植株至全株湿透,置于培养室.每隔1 h喷1次,连续喷施3次,设置喷施等量蒸馏水的拟南芥植株为对照.第3次喷施后1 h取材,检测拟南芥叶组织内ROS水平,比较H2O2处理组与对照组的差异,选用可使叶组织ROS明显升高的100 µmol · L-1 H2O2处理组拟南芥用于下述研究.通过3次相同条件独立实验提供后期所需实验材料.
2.3 DNA和RNA提取取对照和100 µmol · L-1H2O2处理组拟南芥植株地上组织,CTAB法提取基因组DNA,TaKaRa RNAiso Reagent提取总RNA.
2.4 亚硫酸氢盐修饰后测序参照Henderson等(2010)的方法,取2 μg DNA,超声波处理后经亚硫酸氢钠转化(55 ℃,16 h)、过柱纯化(Promega Wizard DNA Clean-up System,A7280)和乙醇沉淀后作为后续扩增模板.NIT2基因的引物序列为:5′-ATTTGAATTATATATTTTTTAAATTAAAGTATTATTAAAATAAA AATAATAAAAAT-3′和5′-TTAAT ATACAACTCTTATATTTATATATTTAATATACAATTACTATTTTAAT A-3′.扩增条件:94 ℃预变性3 min;94 ℃变性10s,60 ℃退火90 s,60 ℃延伸3 min,2个循环;94 ℃变性10 s,59 ℃退火90 s,60 ℃ 延伸3 min,2个循环;94 ℃变性10 s,58 ℃退火90 s,60 ℃延伸3 min,2个循环;94 ℃变性10 s,57 ℃ 退火90 s,60 ℃延伸3 min,32个循环;最后60 ℃延伸5 min.以无甲基化的基因组区域为亚硫酸氢钠处理完全的对照(Hetzl et al., 2007).将PCR产物克隆到pGEM-T Easy载体(Promega)上,随机挑取20个阳性克隆,提取质粒进行酶切和PCR 鉴定后,送北京华大基因公司进行序列测定,测序结果通过网站(http://www.gmi.oeaw.ac.at/CyMATE/)进行甲基化分析.
2.5 RT-PCR分析以总RNA为模板,使用M-MLV反转录酶(TaKaRa)合成cDNA.以Actin2基因作内参,引物序列为:5′-TTCCTCATGCCATCCTCCGTCTT-3′和5′-CAGCGATACCTGAGAACAT AGTGG-3′;扩增程序: 94 ℃预变性 3 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min.NIT2基因引物序列为:5′-CAACGATACTCCCGCCACT-3′和5′-CGAGGA TAACCACCGATAAA-3′;扩增条件:94℃预变性3 min;94 ℃变性30 s,52 ℃退火30 s,72 ℃延伸1 min,25个循环;72 ℃延伸10 min.1%琼脂糖电泳检测PCR产物,每份样品都经两次生物学重复验证实验结果.
3 结果(Results) 3.1 拟南芥NIT2基因启动子区甲基化特征用亚硫酸氢盐修饰后测序法定量分析拟南芥植株地上组织细胞中NIT2基因启动子区的胞嘧啶甲基化特征.研究区域为15993439~15993888(450 bp),共有31个胞嘧啶,包括9个CG、2个CHG和20个CHH位点.以亚硫酸氢盐修饰后的DNA为模板,经PCR扩增得到了与目标序列大小一致的片段;对PCR产物克隆后测序,将测序结果与NCBI数据库中NIT2基因启动子区序列比对,目标序列与数据库中的DNA序列完全一致.依据亚硫酸氢盐修饰过程中未甲基化胞嘧啶转化为尿嘧啶,经PCR扩增转变为胸腺嘧啶的原则,比对亚硫酸氢盐修饰组与未修饰对照组的序列特征,发现该启动子区段有9个CG、2个CHG和17个CHH位点存在甲基化,还有3个CHH位点未发生甲基化(图 1).
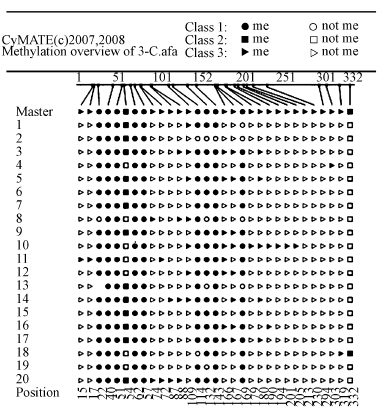 |
| 图 1 拟南芥地上组织NIT2(At3g44300)基因启动子区序列甲基化特征(Class 1: CG site; Class 2: CHG site; Class 3: CHH site) Fig.1 Methylation pattern in the promoter region of NIT2(At3g44300)gene in Arabidopsis shoots |
用亚硫酸氢盐修饰后测序法分析发现,H2O2胁迫可导致拟南芥植株NIT2基因启动子区胞嘧啶甲基化修饰特征改变,表现为不同胞嘧啶位点甲基化水平的升降或去甲基化.在对照组中,CG、CHG和CHH位点甲基化比率分别为93.3%、35.0%和12.0%,胞嘧啶总的甲基化比率为37.0%.在H2O2处理组中,胞嘧啶总的甲基化比率为37.8%,与对照组接近;CG位点的甲基化比率为96.9%,增加了3.6%,9个CG位点中有3个位点胞嘧啶甲基化水平与对照相同,5个位点甲基化水平升高,1个位点甲基化水平降低;CHG位点胞嘧啶甲基化比率为50.0%,上升15.0%,2个CHG位点甲基化水平均升高;CHH位点甲基化比率为10.6%,20个CHH位点中有6个位点甲基化水平与对照相近,6个位点甲基化水平提高,4个位点甲基化水平降低,4个位点发生去甲基化(图 2).
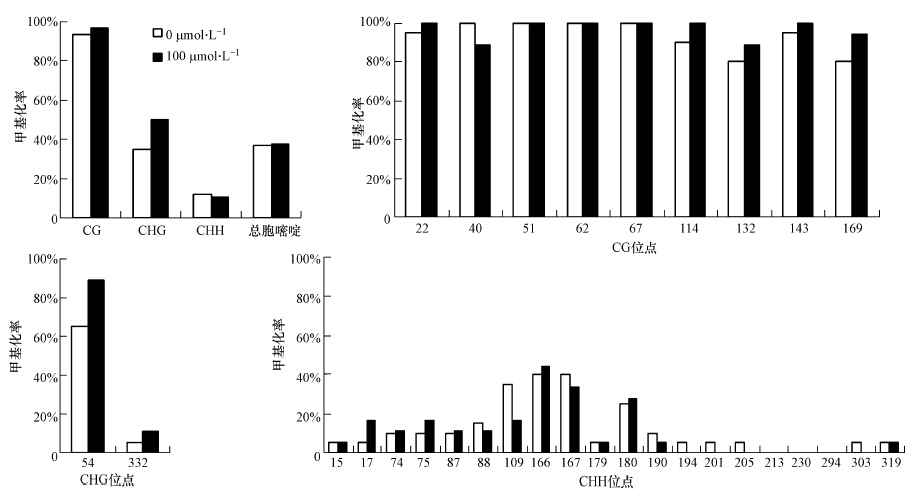 |
| 图 2 H2O2处理对拟南芥NIT2基因启动子区甲基化特征的影响 Fig.2 DNA methylation alteration in NIT2 promoter region in H2O2-treated Arabidopsis plants |
RT-PCR检测发现,用100 µmol · L-1的H2O2处理拟南芥植株3 h后,地上组织细胞中NIT2基因的表达量明显增加,表明H2O2胁迫能诱导NIT2基因转录水平提高(图 3).
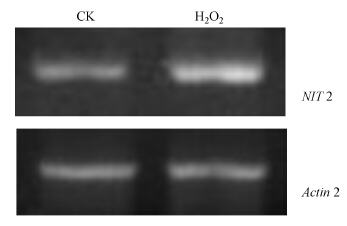 |
| 图 3 H2O2处理对拟南芥NIT2基因转录的影响 Fig.3 Transcriptional change of NIT2 gene in Arabidopsis plants exposed to H2O2 |
逆境胁迫往往引发植物细胞内ROS水平增高,ROS水平升高可能与胁迫生理中DNA甲基化特征改变和基因转录应答有关.本文采用亚硫酸氢盐修饰后测序技术对拟南芥NIT2基因启动子区的胞嘧啶甲基化进行了定量分析,发现H2O2胁迫组NIT2基因启动子区胞嘧啶的总甲基化水平基本不变,但其中不同位点胞嘧啶的甲基化特征发生了改变,出现甲基化水平增高或降低,并有几个位点胞嘧啶去 甲基化.该结果为H2O2影响植物基因组DNA甲基化修饰特征提供了直接证据,说明氧化胁迫是基因组DNA甲基化修饰特征改变的一个诱因.
腈水解酶是一类水解腈化合物中腈基功能团(R—CN)的酶,能催化吲哚乙腈转变为吲哚乙酸(IAA),在植物生长素合成过程中具有关键作用(Park et al., 2003).本研究中H2O2处理组拟南芥植株体内ROS增高、NIT2基因转录水平提高,表明胁迫诱发的ROS升高可能参与对NIT2基因转录的调控.逆境生理中NIT2基因的高表达有利于生长素的合成(Grsic-Rausch et al., 2000;Bao et al., 2002),生长素通过对多种途径的调节来调控植株的生理过程,提高植株的逆境适应能力.
植物DNA甲基化是调节基因功能的手段,它的生物学作用主要表现在基因表达调控和基因组防御两个方面.甲基化胞嘧啶通过直接或间接干扰转录活化因子与DNA的结合影响转录活动和相关基因的表达.拟南芥NIT2基因在甲基转移酶缺失突变体中表达增强,暗示该基因的表达可能受DNA甲基化修饰的调节;SO2胁迫后拟南芥地上组织中NIT2基因启动子区胞嘧啶甲基化特征和基因转录水平改变(李利红等,2012),与SO2胁迫生理中ROS水平升高同时发生,说明胁迫诱发植株组织中ROS的产生可能与基因组DNA胞嘧啶甲基化和基因转录有关;在本研究中,我们用外源H2O2处理诱发了拟南芥地上组织中NIT2基因启动子区胞嘧啶甲基化特征和基因转录水平的改变,证实了ROS水平升高对DNA表观修饰的影响及其与基因转录的关系.但比较SO2处理组(李利红等,2012)与本文H2O2处理组结果发现,SO2和H2O2处理组拟南芥叶组织中ROS升高均伴随着NIT2基因转录上调,处理组NIT2启动子区的胞嘧啶总甲基化水平与对照组相差不大,其中,CHG位点甲基化水平均升高,CHH位点中有6个位点胞嘧啶甲基化水平升高、8个位点甲基化水平降低,其中的5个CHH位点在SO2与H2O2组中甲基化水平具有一致的改变,包括升高、降低和去甲基化,但CG位点甲基化率在SO2处理组降低10.5%,而H2O2处理组中升高3.6%.由此可见,DNA甲基化特征与基因转录之间存在较为复杂的关系.由于表观遗传调控机制的复杂性,目前逆境生理中植物DNA甲基化修饰的研究还处于起步阶段,胁迫诱发甲基化特征的改变如何影响逆境生理中基因转录及植株对环境的适应能力还不清楚,NIT2基因启动子区胞嘧啶甲基化特征对植物逆境生理的调节作用有待进一步研究.至于ROS与DNA甲基化水平、基因转录之间的相互调控关系,不同位点甲基化改变的作用和影响,目前尚不清楚,而新的研究报告的不断出现,正逐步充实和发展着相关理论.
5 结论(Conclusions)H2O2胁迫能引发拟南芥NIT2基因启动子区胞嘧啶甲基化特征改变,基因转录上调,说明NIT2基因的转录应答和表观修饰参与了植株的逆境生理过程,胁迫诱发的胞内ROS增高是NIT2基因表达增强的诱因,可能在DNA甲基化特征改变中也具有某种作用.
| [1] | Bao F,Li J Y. 2002.Evidence that the auxin signaling pathway interacts with plant stress response[J].Acta Botanica Sinica,44(5): 532-536 |
| [2] | 程艳丽,宋纯鹏.2005.植物细胞H2O2的信号转导途径[J].中国科学C辑:生命科学,35(6): 480-489 |
| [3] | Chinnusamy V,Zhu J K.2009.Epigenetic regulation of stress responses in plants[J].Current Opinion in Plant Biology,12(2): 133-139 |
| [4] | Grsic-Rausch S,Kobelt P,Siemens J M,et al.2000.Expression and localization of nitrilase during symptom development of the clubroot disease in Arabidopsis[J].Plant Physiology,122(2): 369-378 |
| [5] | Henderson I R,Chan S R,Cao X,et al.2010.Accurate sodium bisulfite sequencing in plants[J].Epigenetics,5(1): 47-49 |
| [6] | Hetzl J,Foerster A M,Raidl G,et al.2007.CyMATE: a new tool for methylation analysis of plant genomic DNA after bisulphite sequencing[J].The Plant Journal,51(3): 526-536 |
| [7] | Jaenisch R,Bird A.2003.Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals[J].Nature,33(Suppl): 245-254 |
| [8] | 李利红,仪慧兰,王艺雯,等.2012.二氧化硫胁迫诱导拟南芥NIT2基因DNA甲基化修饰[J].农业环境科学学报,31(4): 685-690 |
| [9] | Lukens L N,Zhan S H.2007.The plant genome's methylation status and response to stress: implications for plant improvement[J].Current Opinion in Plant Biology,10(3): 317-322 |
| [10] | Mirouze M,Paszkowski J.2011.Epigenetic contribution to stress adaptation in plants[J].Current Opinion in Plant Biology,14(3): 267-274 |
| [11] | 彭海,席婷,张静,等.2011.胁迫条件下植物DNA甲基化的稳定性[J].中国农业科学,44 (12): 2431-2438 |
| [12] | Park W J,Kriechbaumer V,Möller A,et al.2003.The nitrilase ZmNIT2 converts indole-3-acetonitrile to indole-3-acetic acid[J].Plant Physiology,133(2): 794-802 |
| [13] | Vorwerk S,Biernachi S,Hillebrand H.2001.Enzymatic characterization of the recombinant Arabidopsis thaliana nitrilase subfamily encoded by the NIT2/NIT1/NIT3-gene cluster[J].Planta,212(4): 508-516 |
| [14] | Wang W S,Pan Y J,Zhao X Q,et al.2011.Drought-induced site-specific DNA methylation and its association with drought tolerance in rice (Oryza sativa L.) [J].Journal of Experimental Botany,62(6): 1951-1960 |
| [15] | 杨金兰,柳李旺,龚义勤,等.2007.镉胁迫下萝卜基因组DNA甲基化敏感扩增多态性分析[J].植物生理与分子生物学学报,33(3): 219-226 |
| [16] | Zhang X Y,Yazaki J,Sundaresan A,et al.2006.Genome-wide high-resolution mapping and functional analysis of DNA methylation in Arabidopsis[J].Cell,126(6): 1189-1201 |
 2015, Vol. 35
2015, Vol. 35


