2. 辽宁师范大学化学化工学院, 大连 116029;
3. 中国人民大学环境学院, 北京 100872
2. College of Chemistry and Chemical Engineering, Liaoning Normal University, Dalian 116029;
3. School of Environment and Natural Resources, Renmin University of China, Beijing 100872
微生物污染是饮用水最常见的健康风险之一.目前,饮用水中发现的病原微生物种类已超过140种(Reynolds et al., 2008),它们对人类健康造成了严重的威胁.世界卫生组织(WHO)的数据显示,每年有400万以上的儿童死于水传疾病(World Health Organization,2011).而我国在近10年间发生的271起饮用水突发污染公共事件中,微生物污染案例占据主导(王强等,2010),造成3万余人罹患疾病.因此,对水传病原微生物进行准确检测,是保障水质安全的必要手段.
目前,日常的水质微生物学检测主要采用基于培养的平皿计数法、多管发酵法(中华人民共和国卫生部,2006)进行.由于环境中大部分微生物处于有活性但无法培养(viable but non-culture,VBNC)的状态,这些传统方法往往会低估或者漏查病原微生物的数量或种类.另外,培养法虽然对设备和技术条件要求不高,但操作繁琐且耗时较长,在目前水源水和饮用水污染事故发生较为频繁的情况下,无法满足现场或应急检测快速定性的要求.
以定量PCR技术为代表的新型分子生物学技术可以通过标记有放射性、荧光和化学发光物质的探针与靶序列的杂交实现对水中可培养和不可培养的病原微生物的快速检测(Yang et al., 2007;Liu et al., 2008;Yang et al., 2012),但其设备和试剂成本较高,且标记物质对人体往往具有一定的毒性,本身具有一定的污染风险.纳米金颗粒(AuNPs)具有良好的生物相容性且无毒副作用,近年来被广泛应用于生物标记和检测(包华等,2009;Hua et al., 2012;Padmavathy et al., 2012;Alkilany et al., 2013).其中,普通PCR反应结合不修饰的纳米金显色法与定量PCR一样,不需要对扩增产物进行电泳即可确定结果,同时克服了设备和耗材成本高及试剂污染的缺点(James et al., 1998;谭碧生等,2002;Li et al., 2004;Kamaladasan et al., 2013),可以作为一种应急、定性的检测方法加以推广.因此,本文从纳米金溶液变色的最佳盐浓度、探针含量、PCR反应条件等方面进行探讨,并从纳米金制备、病原菌种类、检测特异性等方面进行考量,确定PCR反应结合不修饰的纳米金颗粒显色检测方法的最优检测条件.以期为现场或应急检测提供技术支持.
2 材料与方法(Materials and methods) 2.1 检测原理由图 1可知,纳米金胶体溶液(1)在制备条件下呈红色,在特定盐浓度作用下若无保护措施则会自聚合成颗粒物,使溶液呈紫色或因颗粒沉淀呈近于无色.提取水样中DNA进行PCR扩增反应后,向反应体系中加入适当浓度的特异性探针混合物(2)和(3),再经过一轮变性-退火过程,之后加入纳米金溶液和特定浓度的盐溶液观察颜色变化.若靶序列(4)存在,特异性探针会与其结合,纳米金颗粒在特定浓度的盐溶液下发生自聚合,溶液颜色呈紫色或近于无色;若样品中没有目标微生物,特异性探针无匹配的靶序列,则仍被纳米金颗粒表面吸附,形成保护作用(Li et al., 2004),使其在特定浓度的盐溶液中仍然保持游离状态,此时溶液保持红色不变.
 |
| 图 1 PCR-纳米金显色检测方法原理 Fig. 1 Schematic diagram for detection of DNA using unmodified AuNPs |
纳米金溶液的制备采用传统的柠檬酸还原法(Alkilany et al., 2013).本实验中,取48 mL超纯水+1 mL 1% 氯金酸溶液加热至90~100 ℃,回流,再加入0.0714 g柠檬酸三钠(用1 mL超纯水溶解),继续加热10~15 min后停止加热,冷却至室温,得到一定浓度实验用纳米金溶液.品质良好的纳米金溶液呈现均匀的红色,在3%的琼脂糖凝胶电泳下条带密实.图 2是实验用纳米金颗粒的扫描电镜图,粒径为15 nm左右.
 |
| 图 2 纳米金颗粒扫描电镜图 Fig. 2 Scanning electron microscope of the prepared AuNPs |
实验菌株为5种常见的水传病原细菌,菌株来源为广东省微生物研究所微生物菌种保藏中心.通过文献筛选合适的引物、运用Primer 3.0软件设计相应的探针.表 1列出了相应引物和探针的详细信息(Pirnay et al., 2000;Sen,2000;Ramesh et al., 2002;Ou et al., 2007;Sharma et al., 2010).
| 表 1 病原微生物种类及待扩增片段引物、探针 Table 1 Waterborne pathogen,primers and probes for target sequences |
取菌种培养平板,挑取菌种单克隆,溶解于50 μL裂解液(TaKaRa,Code No. 9164)中,80 ℃水浴15 min,轻微离心,取2 μL上清作为PCR反应模板.采用50 μL反应体系,对各靶病原微生物特异性DNA片段进行扩增,PCR条件为:95 ℃预变性5 min;95 ℃变性45 s,55.5 ℃退火45 s,72 ℃延伸45 s,进行35个循环反应;72 ℃延伸10 min.吸取扩增产物10 μL,加入共2 μL、10 μmol · L-1相应探针,进行杂交反应,条件为:95 ℃反应5 min,55.5 ℃反应3 min,40 ℃反应10 min.先后加入90 μL 纳米金溶液和35 μL 10 mmol · L-1的PBS,室温下肉眼观察颜色变化,另取100 μL 混合溶液加入96微孔板于酶标仪上测量450~750 nm波段下的吸光度值.
3 结果与讨论(Results and discussion) 3.1 引物及探针特异性实验中所有引物及探针序列均提交NCBI进行特异性分析,并最终优选出一套用于各待检病原微生物的特异性基因片段.同时,建立并优化各靶病原微生物特异性基因片段PCR扩增方法,最终达到所有反应可在同一退火条件(55.5 ℃)下完成.图 3为各实验菌株PCR扩增产物电泳结果.
 |
| 图 3 所选靶病原微生物的PCR扩增产物电泳结果(注:第1泳道为分子量标准,DNA maker DL2000;2,3泳道为耶尔森氏菌扩增产物;4,5泳道为志贺氏菌扩增产物;6,7泳道为铜绿假单胞菌扩增产物;8,9泳道为沙门氏菌扩增产物;10,11泳道为金黄色葡萄球菌扩增产物;每种细菌做2个平行) Fig. 3 Electrophoresis results of PCR amplification |
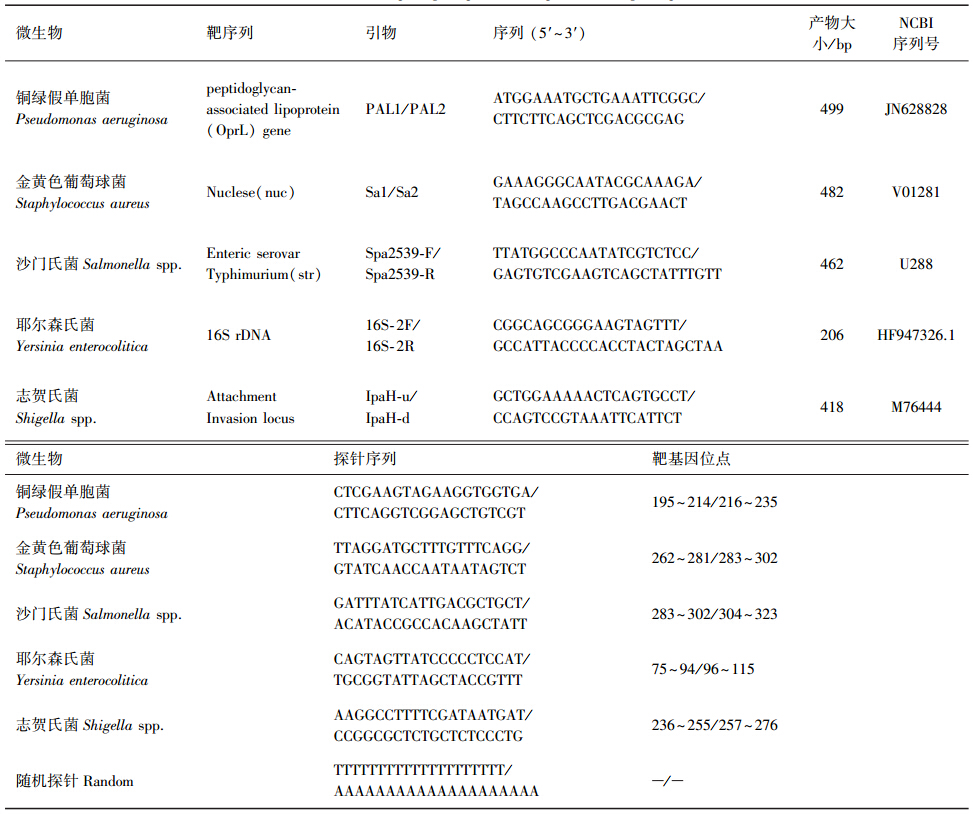
由于纳米金表面电荷非常敏感,在一定盐浓度的作用下表面电荷发生迁移,会造成其聚合并产生变色的结果.随盐浓度的增加,纳米金溶液颜色由红变紫,继续加大盐浓度会变黑直甚至无色(Li et al., 2004; Hussain et al., 2013).图 4中分别选取0~50 mmol · L-1(图中管壁上的数字即对应溶液盐浓度,单位为mmol · L-1)的PBS溶液进行纳米金变色实验,将其与纳米金溶液按照体积比1 ∶ 3(30 μL ∶ 90 μL)的比例混合.结果表明,2.5~12.5 mmol · L-1之间的终盐浓度均能使实验用纳米金溶液产生变色效果.本实验中,终浓度为2.5 mmol · L-1的PBS(图 4左起管4)是使实验用纳米金溶液变色的最小值.
 |
| 图 4 纳米金溶液变色的最低盐浓度 Fig. 4 The minimum salt concentration of nanogold discoloration |
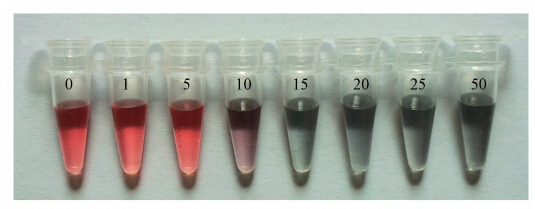
短链(探针)ss-DNA能够吸附在纳米金表面,使得特定浓度的盐溶液无法改变纳米金颗粒表面的电荷迁移,达到保护纳米金颗粒在变色的临界盐浓度作用下保持不变色的效果,且这一保护作用与探针序列无关(Li et al., 2004;Gao et al., 2001).图 5是加入不同浓度的短链(探针)ss-DNA后,纳米金溶液的变色情况.选取图 4中令纳米金溶液变色的临界实验条件(PBS终浓度2.5 mmol · L-1),向体系中添加浓度为10 μmol · L-1、不同体积、长度为20 bp短链ss-DNA(图 5管壁上的数字即为对应添加体积,单位为μL,C为不加PBS和ss-DNA的完全空白对照).结果发现,本实验中0.5 μL 浓度为10 μmol · L-1、长度为20 bp的ss-DNA是保护纳米金颗粒不变色的最小实验浓度(图 5左起管4).
 |
| 图 5 短链(探针)ss-DNA对纳米金溶液变色的影响 Fig. 5 Influence of nanogold discoloration after adding short probes |
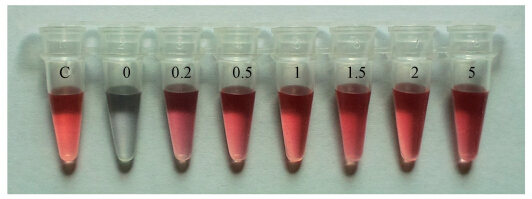
实验需确保长链(PCR产物)DNA对纳米金颗粒变色现象没有影响,本实验证实不高于10 μL、长度为200~500 bp的PCR产物对纳米金颗粒变色几乎没有影响.图 6是Pseudomonas aeruginosa、Staphylococcus aureus、Salmonella spp.、Shigella spp.、Yersinia enterocolitica 5种实验菌株的PCR扩增长链产物与纳米金溶液作用的结果.在图 5左起管5(即90 μL AuNPs+1 μL(10 μmol · L-1)ssDNA+PBS)的条件下,分别加入10 μL 5种不同菌株的PCR产物于实验组(图 6管壁标记Y、Z、S、P、J,分别对应Yersinia enterocolitica、Shigella spp.、Salmonella spp.、Pseudomonas aeruginosa和Staphylococcus aureus)与同体积的buffer于对照组(图 6管壁标记C),管壁标记B组为纳米金溶液混合终浓度为2.5 mmol · L-1 PBS的空白对照组.由图 6可知,实验组和对照组纳米金溶液颜色基本相同,区别于空白对照组的颜色.说明长链(PCR产物)DNA对纳米金溶液变色基本没有影响.
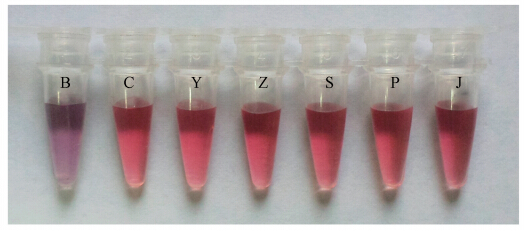 |
| 图 6 长链(PCR产物)DNA对纳米金溶液变色的影响 Fig. 6 Influence of nanogold discoloration after adding PCR products |
实验对5株实验菌株进行PCR-纳米金显色反应效果的验证,旨在得到肉眼就能定性病原微生物情况的方法.在普通PCR得到各实验菌株特异性片段扩增产物后,最终确定选取10 μL产物为符合本实验的最佳检测体积.将实验菌株的PCR产物混合20 pmol相应的探针(对照组:不互补探针,实验组:互补探针),再进行一轮变性-退火过程,最后加入90 μL纳米金溶液和一定的浓度盐溶液,观察各种情况下的混合体系变色情况.图 7是实验5种菌株的PCR-纳米金显色反应检测图,管壁标记Y、Z、S、P、J分别对应Yersinia enterocolitica、Shigella spp.、Salmonella spp.、Pseudomonas aeruginosa和Staphylococcus aureus 5株实验菌株.
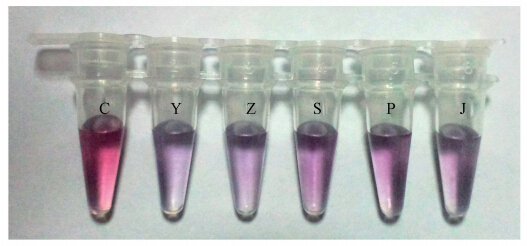 |
| 图 7 PCR-纳米金显色反应检测5株实验菌株 Fig. 7 Positive and negative denotes sample tubes containing PCR products DNA,PBS of Yersinia enterocolitica,Pseudomonas aeruginosa,Staphylococcus aureus,Salmonella spp.,Shigella spp.,respectively |
由图 7可知,对照组(C)由于不匹配短链ss-DNA的存在,纳米金颗粒受到保护,溶液保持一定的红色;实验组(Y、Z、S、P、J)由于短链ss-DNA的消耗,纳米金颗粒发生聚合,结果与对照组明显不同,呈现肉眼可见的颜色变紫或变浅.
图 8是该实验各混合体系对应的吸光度值.由图 8可知,不匹配的非特异性探针(Control)不会结合特定的PCR产物,游离的探针包裹纳米金颗粒形成保护,吸光度峰值保持530 nm左右不变,OD高达0.36;而匹配的特异性探针(图 8中5株实验用菌),由于探针的数量减少甚至消失,纳米金颗粒在一定的盐浓度下发生聚合,吸光度值下降,峰值范围约0.20~0.26,对应的溶液颜色变化为紫色或近于无色.
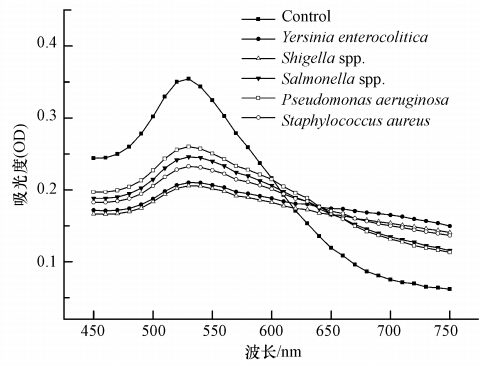 |
| 图 8 PCR-纳米金显色反应检测实验用菌对应的吸光度值 Fig. 8 UV-Vis spectra of the experimental bacterium |
PCR结合不修饰的纳米金检测法优势明显.首先,该法所需纳米金颗粒无需标记巯基(—SH)、磁珠或荧光物质等材料(GÖransson et al., 2010;Hua et al., 2012),降低了耗材成本;检测结果不需要银染等手段进行信号扩大(Taton et al., 2000),对仪器的需求低.此外,相对于化学发光、量子点荧光等检测手段(Liu et al., 2008),该法的检测灵敏度从μg级别提高到ng级别,只需PCR过程有特异性片段产生,就能通过肉眼可见的溶液颜色变化对相应病原微生物进行定性,适合条件相对不优越的现场检测,有较好的应用前景.
4 结论(Conclusions)1)在一定盐浓度的作用下,不修饰的纳米金颗粒发生自聚合,产生肉眼可见的颜色变化,而短链ss-DNA的存在能够在一定程度阻止该过程的发生.利用该原理,本实验初步验证:5 pmol 的短链ss-DNA是防止实验用纳米金颗粒变色的最小实验浓度.
2)相比于使用标记后的纳米金颗粒和其他检测方法,利用不标记的纳米金颗粒检测靶细菌的DNA,具有价格更加低廉、操作更加简便、结果可视化程度高等优点.
3)本实验验证对10 μL PCR产物(约100~200 ng)直接进行检测效果最明显,检测结果肉眼就能定性,并能同时检测5种常见的水传病原微生物情况.
| [1] | Alkilany A M, Lohse S E, Murphy C J. 2013. The gold standard: gold nanoparticle libraries to understand the nano-bio interface [J]. Accounts of Chemical Research, 46(3): 650-661 |
| [2] | 包华, 贾春平, 周忠良, 等. 2009. 基于纳米金探针和基因芯片的DNA 检测新方法[J]. 化学学报, 67(18): 2144-2148 |
| [3] | Gao D, Sheng Z, Han H. 2001. An ultrasensitive method for the detection of gene fragment from transgenics using label-free gold nanoparticle probe and dynamic light scattering[J]. Analytica Chimica Acta, 696(1/2): 1-5 |
| [4] | GÖransson J, De La Torre T Z G, Strmberg M, et al. 2010. Sensitive detection of bacterial DNA by magnetic nanoparticles [J]. Letters to Analytical Chemistry, 82(22): 9138-9140 |
| [5] | Hua D, Yi X, Liu Y H, et al. 2012. Gold nanoparticles with asymmetric polymerase chain reaction for colorimetric detection of DNA sequence [J]. Analytical Chemistry, 84(3): 1253-1258 |
| [6] | Hussain M M, Samir T M, Azzazy H M. 2013. Unmodified gold nanoparticles for direct and rapid detection of Mycobacterium tuberculosis complex [J]. Clinical Biochemistry, 46(7/8): 633-637 |
| [7] | James J S, Elghanian R, Mucic R C, et al. 1998. One-pot colorimetric differentiation of polynucleotides with single base imperfections using gold nanoparticle probes [J]. Journal of the American Chemical Society, 120(9): 1959-1964 |
| [8] | Kamaladasan K, Neo J L,Uttamchandani M. 2013. Direct visual detection of Salmonella genomic DNA using gold nanoparticles [J]. Molecular BioSystems, 9(4): 618-621 |
| [9] | Li H, Rothberg L J. 2004. R. Label-Free colorimetric detection of specific sequences in genomic DNA amplified by the polymerase chain reaction [J]. Journal of the American Chemical Society, 126(35): 10958-10961 |
| [10] | Liu Y J, Yao D J, Chang H Y, et al. 2008. Magnetic bead-based DNA detection with multi-layers quantum dots labeling for rapid detection of Escherichia coli O157: H7[J]. Biosensors and Bioelectronics, 24(4): 558-565 |
| [11] | Ou H Y, Cindy T S J, Thong K L, et al. 2007. Translational genomics to develop a salmonella enterica serovar paratyphi a multiplex polymerase chain reaction assay [J]. The Journal of Molecular Diagnostics, 9(5): 624-630 |
| [12] | Padmavathy B, Vinoth K R, Jaffar A B M. 2012. A direct detection of Escherichia coli genomic DNA using gold nanoprobes [J]. Journal of Nanobiotechnology, 10(1): 1-10 |
| [13] | Pirnay J P, De Vos D, Duinslaeger L, et al. 2000. Quantitation of Pseudomonas aeruginosa in wound biopsy samples: from bacterial culture to rapid 'real-time’ polymerase chain reaction[J]. Critical Care, 4(4): 255-261 |
| [14] | Ramesh A, Padmapriya B P, Chrashekar A, et al. 2002. Application of a convenient DNA extraction method and multiplex PCR for the direct detection of Staphylococcus aureus and Yersinia enterocolitica in milk samples[J]. Molecular and Cellular Probes, 16(4): 307-314 |
| [15] | Reynolds K A, Mena K D, Gerba C P. 2008. Risk of waterborne illness via drinking water in the United States[J]. Reviews of Environmental Contamination and Toxicology, 192: 117-158 |
| [16] | Sen K. 2000. Rapid identification of Yersinia enterocolitica in blood by the 5'nuclease PCR assay[J]. Journal of Clinical Microbiology, 38(5): 1953-1958 |
| [17] | Sharma A, Singh S K, Bajpai D. 2010. Phenotypic and genotypic characterization of Shigella spp. with reference to its virulence genes and antibiogram analysis from river Narmada[J]. Microbiological Research, 165(1): 33-42 |
| [18] | 谭碧生, 曹晓红, 莫志宏. 2002. 金纳米粒子的制备方法及在DNA检测中的应用[J]. 重庆大学学报, 26(4): 58-62 |
| [19] | Taton T A, Mirkin C A, Letsinger R L. 2000. Scanometric DNA array detection with nanoparticle probes[J]. Science, 289(5485): 1757-1760 |
| [20] | 王强,赵月朝,屈卫东,等. 2010. 1996—2006年我国饮用水污染突发公共卫生事件分析[J]. 环境与健康杂志, 27(4): 328-331 |
| [21] | WHO. 2011.Guidelines for Drinking-water Quality(4th ed)[M]. Geneva: World Health Organization |
| [22] | Yang C, Xie H, Li Y, et al. 2012. Direct and rapid quantum dots labelling of Escherichia coli cells[J]. Journal of Colloid and Interface Science, 393: 438-444 |
| [23] | Yang H, Qu L, Wimbrow A N, et al. 2007. Rapid detection of Listeria monocytogenes by nanoparticle-based immunomagnetic separation and real-time PCR [J]. International Journal of Food Microbiology, 118(2): 132-138 |
| [24] | 中华人民共和国卫生部. 2006. GB/T 5750.12—2006 生活饮用水标准检验方法 微生物指标[S].北京:中国标准出版社 |
 2014, Vol. 34
2014, Vol. 34