2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
脂肪酸结合蛋白(Fatty Acid Binding Proteins, FABPs)是动物细胞内一类调节脂肪代谢的蛋白质超家族。它作为脂肪分子伴侣参与脂肪介导的生物过程以及多种脂肪信号调节的系统代谢平衡[1]。FABPs蛋白家族进化保守,在果蝇、线虫、小鼠和人体中均有发现[2]。该蛋白家族在人体内组织或器官中分布有多种FABP蛋白亚型,表现出独特的组织特异性及细胞特异性[3]。其中,在脂肪细胞中大量分布的FABP4蛋白备受关注[4]。
FABP4蛋白,也称A-FABP或者aP2蛋白,主要分布于成熟的脂肪细胞和脂肪组织,也分布于巨噬细胞等[5]。该蛋白的表达量在脂肪细胞分化时增加,并且蛋白活性受长链脂肪酸、氧化低密度脂蛋白、过氧化物酶体增殖物激活受体γ、胰岛素的调节[6-7]。在细胞中,FABP4结合多种脂肪酸参与脂肪转运、降解和生成过程,同时在脂肪的细胞内信号通路、炎症反应和代谢调节等活动中扮演重要角色。研究表明,FABP4与脂肪酸代谢紊乱、二型糖尿病、动脉粥样硬化以及肥胖等疾病密切相关。FABP4蛋白抑制是二型糖尿病、脂肪肝和动脉粥样硬化等疾病的治疗方案[1, 8]。
近年来,人源FABP4蛋白与脂肪酸以及一系列小分子抑制剂的复合物晶体结构被陆续报道[9-12]。人源FABP4蛋白表现为典型的脂笼蛋白结构,其中10条依次互为反平行的β链折叠成扁型的桶状结构,位于β1-β2之间的螺旋-转角-螺旋结构域被认为是β桶的盖子,封闭该蛋白的顶端,而另一端β桶被短的α螺旋部分关闭。由此形成的空腔为脂肪酸等小分子配体结合位点。针对该位点的小分子抑制剂设计目前主要考虑两个方面:其一为由与脂肪酸带电荷羧基结合的氨基酸残基Arg106、Arg126和Tyr128组成的亲水位点;另一个为疏水残基Phe57为代表的一系列疏水残基组成的疏水位点[10]。由于FABP4蛋白对疏水配体有着天然的低选择性,容易与其他亚型交叉抑制而引发副作用。因此在已知的蛋白-配体结合模式以外,定义新的配体结合位点与相互作用模式,并开发相应的高亲和、高选择性抑制剂以避免副作用产生,将具有重要的现实意义。依托上海同步辐射装置(Shanghai Synchrotron Radiation Facility, SSRF)光源生物大分子晶体学实验站,开展FABP4蛋白-小分子片段复合物三维结构测定与筛选,能够为设计与发现高选择性抑制该蛋白生物功能的药物小分子提供关键理论依据。
阿司匹林也称乙酰水杨酸(Acetylsalicylic Acid, ASA)是一种经典非甾体抗炎药(Nonsteroidal Anti-inflammatory Drug, NSAID),目前被广泛应用于动脉粥样硬化的治疗中[13]。本文通过研究乙酰水杨酸及其水解物水杨酸(Salicylic Acid, SA)作为小分子片段与FABP4蛋白形成的复合物晶体结构,推测阿司匹林存在通过抑制FABP4蛋白生物功能来治疗动脉粥样硬化的一种可能分子机制。
1 材料与方法 1.1 FABP4蛋白表达纯化 1.1.1 FABP4蛋白表达将人源FABP4蛋白的cDNA重组到表达载体pET28a中,重组质粒转化大肠杆菌E.coli感受态细胞BL21(DE3)后过表达目标蛋白。挑取单克隆,加入到50 mL含有25 mg·mL-1卡那霉素的LB(Luria-Bertani)培养基,在转速220 r·min-1、37℃恒温箱振荡培养。过夜后将培养液转入800 mL含有25 mg·mL-1卡那霉素新鲜的LB培养基,37℃恒温箱振荡培养。当检测菌液的OD600nm值达到0.6-0.8时,加入0.2 mmol·L-1异丙基硫代半乳糖苷(Isopropylβ-D-Thiogalactoside, IPTG),20℃恒温诱导16h。低温高速(4℃,8 000 r·min-1)离心10 min收集菌体。
1.1.2 FABP4蛋白分离纯化收集的菌体加入20 mmol·L-1 NaH2PO4,pH=8.0,300 mmol·L-1 NaCl,5 %甘油的细胞裂解缓冲液中重悬。高速分散器将细胞悬浮液均匀分散,去除小的细胞团块。压力破碎细胞直至菌液澄清,温度设定为4℃,压力为100-120 MPa。获得细胞破碎液后离心管分装,4℃,15 000 r·min-1高速离心30min,收集上清液。
将蛋白离心上清液加入到含镍的金属螯合亲和层析色谱柱,含20 mmol·L-1咪唑的裂解缓冲液冲洗柱上杂蛋白。含200 mmol·L-1咪唑裂解缓冲液洗脱FABP4蛋白并收集。将收集的FABP4蛋白用截留分子量为3 kDa的超滤管(Millipore)进行浓缩。随后使用分子筛纯化上一步的蛋白。分子筛进样前,蛋白需10000r·min-1高速离心除去杂质和气泡。分子筛预装柱选用120mL HiLoad Superdex75 16/60 column(GE Healthcare),缓冲液为20 mmol·L-1 NaH2PO4,pH=7.5,50 mmol·L-1 NaCl,5%甘油。收集280 nm紫外吸收峰峰值处对应蛋白样品。SDS-PAGE(Sodium Dodecyl Sulphate-Polyacrylamide Gel Electrophoresis)检测显示蛋白样品纯度达95%以上,该蛋白进一步浓缩至10 mg·mL-1左右,用于蛋白结晶生长实验。
1.2 FABP4蛋白-小分子复合物晶体制备采用悬滴-气相扩散法制备单独蛋白晶体,两种结晶条件被选用为该蛋白的结晶母液,分别为1.6mol·L-1柠檬酸钠pH=6.5和20%聚乙二醇(PEG-10000),0.1 mol·L-1羟乙基哌嗪乙磺酸(4-(2-hydroxyerhyl)piperazine-1-erhaesulfonic acid, HEPES),pH=7.5[14]。用于结晶的蛋白样品浓度约为10 mg·mL-1,与结晶母液分别取2μL混合形成悬滴液。晶体板静置于18℃恒温室等待蛋白晶体生长。
蛋白配体复合物晶体通过利用小分子配体浸泡FABP4蛋白晶体实验获得。分别配制水杨酸和乙酰水杨酸配体浓度为10mmol·L-1的两种结晶母液,并将FABP4蛋白晶体在其中浸泡2h即得到蛋白-小分子复合物晶体。
1.3 晶体衍射数据收集及结构解析含有20%甘油的结晶母液作为晶体防冻液用于衍射数据收集前的晶体冷冻保存。将目标晶体转移到防冻液中静置10-20 s并快速转移液氮中保存。晶体衍射数据的收集在SSRF生物大分子线站BL17U1[15]进行,实验温度为100K,探测器为美国ADSC公司Quantum Q315r CCD。收集的FABP4蛋白晶体衍射数据用HKL2000软件包[16]和XDS[17]进行指标化、积分和整合处理。以PDB数据库中编码为2HNX的已知人源FABP4结构作为分子置换的搜索模型,用CCP4软件包[18]中的phaser-MR软件解析相位。采用PHENIX[19]进行结构模型修正,COOT[20]调图。PyMOL[21]制作三维结构图。
2 结果与讨论 2.1 蛋白表达纯化及复合物晶体制备FABP4蛋白在设计时含有6×His标签(anti-6×His tag monoclonal antibody),通过镍亲和层析柱可以初步纯化。为获得高度均一的高纯度蛋白,本实验进一步使用分子筛纯化蛋白。分子筛通过280 nm紫外吸收检测,洗脱体积在76 mL处出现过饱和的吸收峰,对应的相对分子质量为15 kDa,说明FABP4蛋白溶液状态下为单体存在。经过分子筛纯化步骤所得的蛋白经15%的SDS-PAGE检测,蛋白纯度已达到95%以上,适用于结晶实验。
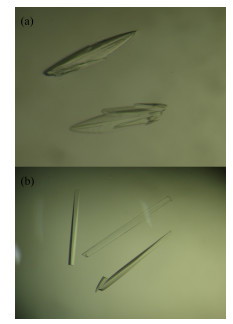
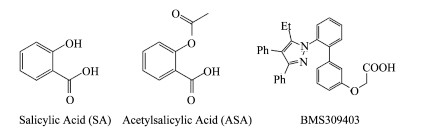
将纯化获得的蛋白在两种配方的结晶母液中进行晶体生长实验,这两种母液分别为1.6 mol·L-1柠檬酸钠pH=6.5和20% PEG-10000,0.1 mol·L-1 4-HEPES,pH=7.5。生长得到两种不同外观的蛋白晶体(图 1),而且这两种晶体的生长所需时间和所属分子空间群均不相同。在柠檬酸钠溶液中,生长3 d左右能够得到刀片状晶体(图 1(a)),其空间群为P212121。在HEPES/PEG结晶母液中,两周后能看到呈现棱柱状的晶体(图 1(b)),其所属空间群为P21212。然而由于高浓度的柠檬酸盐容易与小分子发生竞争,阻碍弱结合能力的小分子片段与FABP4的结合[12],最终利用HEPES/PEG结晶母液中生长的蛋白晶体与SA及ASA(图 2)结合,得到复合物晶体。利用这些复合物晶体采集到了较高分辨率的X-射线单晶衍射数据(约0.13nm),具体的晶体衍射数据处理结果统计参见表 1。
|
图 1 不同结晶条件下生长出的FABP4蛋白晶体 (a)柠檬酸钠溶液,(b) HEPES/PEG结晶母液 Figure 1 Two types of FABP4 crystals growing from two different mother liquors. (a) Sodium citrate solution, (b) HEPES/PEG solution |
|
图 2 水杨酸、乙酰水杨酸和FABP4抑制剂BMS309403的化学结构式 Figure 2 Chemical schemes of the ligands SA, ASA and the FABP4 inhibitor BMS309403. |
| 表 1 衍射数据采集与数据处理结果统计 Table 1 Statistics of crystal data collection and structure refinement. |
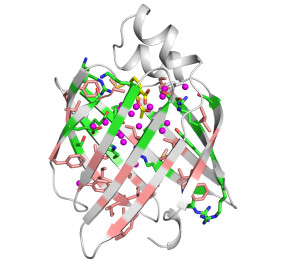
如图 3所示,人源FABP4蛋白表现为典型的脂笼蛋白结构。该蛋白主要由10条依次互为反平行的β链折叠成扁型的桶状结构,由图 3可以发现,组成“桶”内壁的氨基酸极性分布有明显的区域性。位于上部的残基大多为带电荷或极性侧链氨基酸,它们与螺旋-转角-螺旋结构域一起组成了完整的蛋白配体结合空腔。而“桶”的下半部则主要为非极性侧链氨基酸,它们通过疏水相互作用形成了桶状结构的底部。
|
图 3 FABP4配体结合空腔的结构水 Figure 3 Structural water molecules inside of the ligand binding cavity of FABP4. |
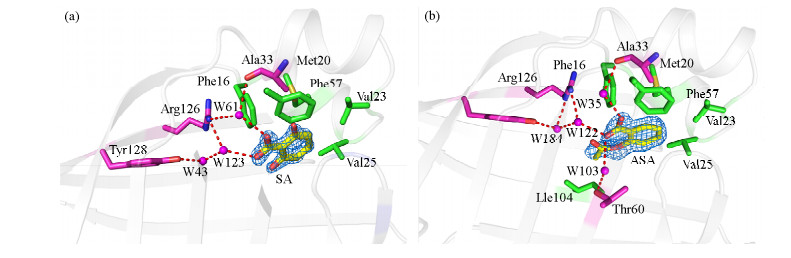
FABP4蛋白配体结合位点为一个两亲性的“口袋”。其中亲水端深入蛋白中部,疏水端靠近蛋白表面。位于该蛋白中部,序列保守的氨基酸残基Arg106、Arg126和Tyr128侧链组成亲水端位点。通过静电相互作用它们可以和脂肪酸等配体的羟基结合。而位于该蛋白螺旋-转角-螺旋结构域的残基Phe16、Met20、Val25、Ala33以及位于β3-β4之间loop上的Phe57、β5-β6之间loop上的Ala75等疏水残基则形成一个较为松散的疏水区与脂肪酸的疏水脂肪链相结合(图 4)。
|
图 4 水杨酸(a)及乙酰水杨酸(b)与FABP4配体结合位点 Figure 4 Ligand binding sites of FABP4 bound with SA (a) and ASA (b). |
复合物晶体结构显示FABP4蛋白内部存在21个坐标相对固定的结构水分子(图 3中小圆点标示),其中19个结构水分布在配体结合空腔内。通过氢键与组成空腔的极性残基侧链相互作用,这些网络化的结构水分子被稳定在配体结合位点。配体小分子的结合将部分有序的结构水置换出来是一个熵增加的过程,即结构水的存在与配体的结合存在相互竞争的关系。这一关系是影响抑制剂小分子结合FABP4的一个关键因素。
2.3 蛋白复合物的配体结合位点结构分析水杨酸及乙酰水杨酸的分子量较小(仅为经典抑制剂BMS309403的1/3),且亲水羧基与疏水苯环之间仅有一根碳-碳键相连(图 2)。已报道的FABP4-小分子复合物结构显示,配体结合空腔的亲水位点与疏水位点之间距离为3-4根碳-碳键的长度。因此FABP4的两个极性相反的结合位点对水杨酸及乙酰水杨酸小分子的结合存在竞争。
复合物晶体结构显示,在FABP4配体结合空腔中,SA(图 4(a))和ASA(图 4(b))两种小分子的电子密度图清晰可见。与已知小分子配体不同,SA及ASA的羧基与FABP4蛋白Arg126和Tyr128侧链之间分别需要通过一个及两个结构水分子的“中继”,形成较弱氢键相互作用。而在疏水结合区域,残基Phe16侧链苯环的CH几乎垂直指向水杨酸芳环平面,其中C-H-苯环中心的距离为0.37-0.40 nm之间(图 5),该结构所测定的几何距离参数表明残基Phe16侧链苯环的CH与水杨酸苯环π电子云之间存在C-H-π相互作用[22]。这一相互作用被认为是一种非经典的氢键相互作用,并发现广泛存在于蛋白质结构中[23]。同时,带正电荷的残基Arg126的侧链胍基团与Phe16侧链苯环几乎平行,平面之间的距离为0.33-0.40 nm,这一几何距离参数表明两者之间存在阳离子-π相互作用。它们之间的阳离子-π相互作用将使得Phe16侧链苯环上的CH氢原子的亲电性进一步增强,从而增强了Phe16-水杨酸苯环之间的C-H-π相互作用。
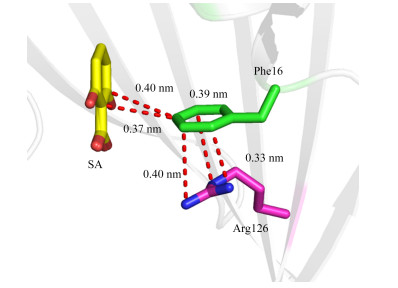
|
图 5 FABP4残基Arg126、Phe16与水杨酸苯环的几何距离关系 Figure 5 Geometrical distance relationship between Arg126, Phe16 residues of FABP4 and benzyl ring of SA. |
虽然水杨酸分子带电荷的羰基也能够与FABP4的亲水配体结合位点相互作用,破坏结构水的有序性造成熵增效应,Phe16-水杨酸苯环之间的C-H-π相互作用能够提供有效的焓增。以上两个因素合作的结果使得水杨酸小分子更应该结合在疏水结合位点,而不是通常认为的亲水结合位点,由此认为FABP4配体结合空腔的疏水位点提供结合能力贡献更大。本研究中的复合物晶体结构对这一猜想给出有力的实验证明。该结果可以为进一步设计高选择性的FABP4抑制剂提供新的途径。
3 结语本文利用经典药物阿司匹林(乙酰水杨酸)及其水解物水杨酸作为片段分子,成功地制备了脂肪酸结合蛋白FABP4与小分子片段的复合物晶体并解析了复合物晶体的结构。结构分析表明,FABP4疏水结合位点残基Phe16苯环C-H与水杨酸苯环平面之间存在较强的C-H-π非经典氢键相互作用,在乙酰水杨酸的竞争结合过程中比亲水位点的静电相互作用更具有优先性。这一发现为设计高选择性FABP4抑制剂提供了新的途径。
| [1] |
Furuhashi M, Hotamisligil G S. Fatty acid-binding proteins:role in metabolic diseases and potential as drug targets[J].
Nature Review Drug Discovery, 2008, 7 (6) : 489 –503.
DOI: 10.1038/nrd2589 ( 0) 0)
|
| [2] |
Zimmerman A W, Veerkamp H J. New insights into the structure and function of fatty acid-binding proteins[J].
Cellular and Molecular Life Sciences, 2002, 59 : 1096 –1116.
DOI: 10.1007/s00018-002-8490-y ( 0) 0)
|
| [3] |
Smathers R L, Petersen D R. The human fatty acid-binding protein family:evolutionary divergences and functions[J].
Human Genomics, 2010, 5 (3) : 170 –191.
( 0) 0)
|
| [4] |
Furuhashi M, Saitoh S, Shimamoto K, et al. Fatty Acid-Binding Protein 4(FABP4):pathophysiological insights and potent clinical biomarker of metabolic and cardiovascular diseases[J].
Clinical Medicine Insights:Cardiology, 2015, 8 (S3) : 23 –33.
DOI: 10.4137/cmc.s1067 ( 0) 0)
|
| [5] |
Makowski L, Boord J B, Maeda K, et al. Lack of macrophage fatty-acid-binding protein aP2 protects mice deficient in apolipoprotein E against atherosclerosis[J].
Nature Medicine, 2001, 7 (6) : 699 –705.
DOI: 10.1038/nm89076 ( 0) 0)
|
| [6] |
Ayers S D, Nedrow K L, Gillilan R E, et al. Continuous nucleocytoplasmic shuttling underlies transcriptional activation of PPARγby FABP4[J].
Biochemistry, 2007, 46 (23) : 6744 –6752.
DOI: 10.1021/bi700047a ( 0) 0)
|
| [7] |
Gan L, Liu Z, Cao W, et al. FABP4 reversed the regulation of leptin on mitochondrial fatty acid oxidation in mice adipocytes[J].
Scientific Reports, 2015, 5 : 13588 .
DOI: 10.1038/srep13588 ( 0) 0)
|
| [8] |
Furuhashi M, Tuncman G, Gorgun C Z, et al. Treatment of diabetes and atherosclerosis by inhibiting fatty-acid-binding protein aP2[J].
Nature, 2007, 447 (7147) : 959 –965.
DOI: 10.1038/nature05844 ( 0) 0)
|
| [9] |
Lehmann F, Haile S, Axen E, et al. Discovery of inhibitors of human adipocyte fatty acid-binding protein, a potential type 2 diabetes target[J].
Bioorganic & Medicinal Chemistry Letters, 2004, 14 (17) : 4445 –4448.
DOI: 10.1016/j.bmcl.2004.06.057 ( 0) 0)
|
| [10] |
Marr E, Tardie M, Carty M, et al. Expression, purification, crystallization and structure of human adipocyte lipid-binding protein (aP2)[J].
Acta Crystallographica Section F, 2006, 62 (11) : 1058 –1060.
DOI: 10.1107/S1744309106038656 ( 0) 0)
|
| [11] |
Cai H, Liu Q, Gao D, et al. Novel fatty acid binding protein 4(FABP4) inhibitors:virtual screening, synthesis and crystal structure determination[J].
European Journal of Medicinal Chemistry, 2015, 90 : 241 –250.
DOI: 10.1016/j.ejmech.2014.11.020 ( 0) 0)
|
| [12] |
Gonzalez J M, Fisher S Z. Structural analysis of ibuprofen binding to human adipocyte fatty-acid binding protein (FABP4)[J].
Acta Crystallographica Section F, 2015, 71 (2) : 163 –170.
DOI: 10.1107/S2053230X14027897 ( 0) 0)
|
| [13] |
杨钰, 于晓玲. 阿司匹林与动脉粥样硬化[J].
医学综述, 2006, 12 (19) : 1194 –1196.
YANG Yu, YU Xiaoling. Aspirin and atherosclerosis[J]. Medical Recapitulate, 2006, 12 (19) : 1194 –1196. (  0) 0)
|
| [14] |
Marr E, Tardie M, Carty M, et al. Expression, purification, crystallization and structure of human adipocyte lipid-binding protein (aP2)[J].
Acta Crystallographica Section F, 2006, 62 (Pt11) : 1058 –1060.
DOI: 10.1107/S1744309106038656 ( 0) 0)
|
| [15] |
Wang Q S, Yu F, Huang S, et al. The macromolecular crystallography beamline of SSRF[J].
Nuclear Science and Techniques, 2015, 26 (1) : 010102 .
DOI: 10.13538/j.1001-8042/nst.26.010102 ( 0) 0)
|
| [16] |
Otwinowski Z, Minor W. Processing of X-ray diffraction data collected in oscillation mode[J].
Macromolecular Crystallography, 1997, 276 : 307 –326.
DOI: 10.1016/S0076-6879(97)76066-X ( 0) 0)
|
| [17] |
Kabsch W. Integration, scaling, space-group assignment and post-refinement[J].
Acta Crystallographica Section D, 2010, 66 (Pt2) : 133 –144.
DOI: 10.1107/S0907444909047374 ( 0) 0)
|
| [18] |
Collaborative Computational Project N. The CCP4 suite:programs for protein crystallography[J].
Acta Crystallographica Section D, 1994, 50 (5) : 760 –763.
DOI: 10.1107/S0907444994003112 ( 0) 0)
|
| [19] |
Adams P D, Afonine P V, Bunkóczi G, et al. PHENIX:a comprehensive Python-based system for macromolecular structure solution[J].
Acta Crystallographica Section D, 2010, 66 (2) : 213 –221.
DOI: 10.1107/S0907444909052925 ( 0) 0)
|
| [20] |
Emsley P, Cowtan K. Coot:model-building tools for molecular graphics[J].
Acta Crystallographica Section D, 2004, 60 (12-1) : 2126 –2132.
DOI: 10.1107/S0907444904019158 ( 0) 0)
|
| [21] |
Delano W L. The PyMOL molecular graphic system[C]. Palo Alto, CA:Delano Scientific, 2002
( 0) 0)
|
| [22] |
Jennings W B, O'Connell N, Malone J F, et al. An evaluation of substituent effects on aromatic edge-to-face interactions and CF-[small pi] versus CH-[small pi] interactions using an imino torsion balance model[J].
Organic & Biomolecular Chemistry, 2013, 11 (32) : 5278 –5291.
DOI: 10.1039/C3OB40535D ( 0) 0)
|
| [23] |
Brandl M, Weiss M S, Jabs A, et al. C-h…π-interactions in proteins[J].
Journal of Molecular Biology, 2001, 307 (1) : 357 –377.
DOI: 10.1006/jmbi.2000.4473 ( 0) 0)
|


