Clinical distribution and drug-resistance gene analysis of carbapenem-resistant Pseudomonas aeruginosa in a tertiary hospital in Shanghai
-
摘要:
目的 了解上海某三甲医院耐碳青霉烯类铜绿假单胞菌(CRPA)的临床分布特点及耐药基因型,为指导临床抗菌药物的合理使用、降低细菌耐药性和控制院内感染提供科学依据。 方法 收集2019年1月1日至12月31日海军军医大学第一附属医院临床标本中分离出的连续不重复CRPA菌株(共94株),统计分析CRPA的科室分布、样本来源等。采用VITEK 2 Compact全自动微生物分析仪和纸片扩散法(Kirby-Bauer法)检测铜绿假单胞菌对13种抗菌药物的敏感性,采用PCR法扩增超广谱β-内酰胺酶耐药基因、碳青霉烯酶耐药基因、膜孔蛋白基因。采用基于k-mer算法的kSNP3软件对筛选菌株进行单核苷酸多态性(SNP)分析并构建进化树,基于PubMLST数据库中铜绿假单胞菌的分型方案进行多位点序列分型(MLST)分析。 结果 94株CRPA对碳青霉烯类抗生素亚胺培南(92.6%,87株)、美罗培南(87.2%,82株)的抗生素耐药率较高,对氨基糖苷类抗生素阿米卡星(10.6%,10株)、庆大霉素(20.2%,19株)、妥布霉素(20.2%,19株)的耐药率较低。科室分布排名前3位的是急救监护室(9.6%,9株)、脑血管外科(8.5%,8株)和呼吸科(8.5%,8株)。PCR检出外膜蛋白D2(OprD2)基因缺失菌株47株(50.0%),13株(13.8%)blaKPC阳性菌,4株(4.3%)blaVIM阳性菌,2株(2.1%)blaIMP阳性菌,1株(1.1%)blaNDM阳性菌,12株(12.8%)blaTEM阳性菌,4株(4.3%)blaPER阳性菌,2株(2.1%)blaGES阳性菌,blaOXA-48、blaBIC、blaSIM、blaVEB、blaSHV等基因未检出。MLST分析鉴定出36种不同的序列类型(ST),其中最常见的是ST463和ST274,并首次发现了2种新的ST(ST4023和ST4024)。 结论 临床分离的CRPA携带多种耐药基因,对多种临床常用抗菌药物同时耐药,其耐药主要与细菌携带blaKPC、blaVIM、blaTEM基因和缺失OprD2基因有关。临床应加强对CRPA的监测,合理使用抗菌药物,控制其在院内的传播。 -
关键词:
- 耐碳青霉烯类铜绿假单胞菌 /
- 耐药基因 /
- 耐药率 /
- 分子流行病学
Abstract:Objective To understand the clinical distribution characteristics and drug-resistance genotypes of carbapenem-resistant Pseudomonas aeruginosa (CRPA) in a tertiary hospital in Shanghai, so as to guide the rational use of antibiotics, reduce bacterial resistance and control nosocomial infections. Methods A total of 94 consecutive and non-redundant CRPA strains isolated from clinical specimens were collected in The First Affiliated Hospital of Naval Medical University between Jan. 1, and Dec. 31, 2019. The distribution of CRPA across departments and sample sources were analyzed. Antimicrobial susceptibility testing against 13 antibiotics was determined using the VITEK 2 Compact automated microbiology analyzer and the Kirby-Bauer disk diffusion method. Polymerase chain reaction (PCR) was employed to detect genes encoding extended-spectrum β-lactamase resistance gene, carbapenemase resistance gene, and porin resistance gene. Homology analysis of selected isolates was performed using kSNP3, a k-mer-based software, for single nucleotide polymorphism (SNP) analysis. An evolutionary tree was constructed to assess genetic relationships. Additionally, multilocus sequence typing (MLST) was performed using the Pseudomonas aeruginosa typing scheme from the PubMLST database. Results Among the 94 CRPA isolates, high resistance rates were observed for carbapenems, including imipenem (92.6%, 87 strains) and meropenem (87.2%, 82 strains). In contrast, low resistance rates were detected for aminoglycosides, such as amikacin (10.6%, 10 strains), gentamicin (20.2%, 19 strains), and tobramycin (20.2%, 19 strains). The top 3 departments in terms of isolate distribution were the Emergency Intensive Care Unit (9.6%, 9 strains), Department of Cerebrovascular Surgery (8.5%, 8 strains), and Department of Respiratory Medicine (8.5%, 8 strains). PCR analysis of 94 CRPA strains detected outer membrane protein D2 (OprD2) gene deletion in 47 (50.0%) strains, 13 (13.8%) strains were positive for blaKPC, 4 (4.3%) strains for blaVIM, 2 (2.1%) strains for blaIMP, 1 (1.1%) strain for blaNDM, 12 (12.8%) strains for blaTEM, 4 (4.3%) strains for blaPER, and 2 (2.1%) strains for blaGES, while blaOXA-48, blaBIC, blaSIM, blaVEB, and blaSHV were not detected. MLST identified 36 different sequence types (STs), with ST463 and ST274 being the most common, and 2 new ST (ST4023 and ST4024) were identified for the first time. Conclusion CRPA strains carry multiple resistance genes and exhibit concurrent resistance to several commonly used clinical antibiotics. The resistance is primarily associated with the presence of blaKPC, blaVIM and blaTEM genes and the deletion of OprD2 gene. Clinical monitoring of CRPA should be strengthened, and rational use of antimicrobial agents is essential to control its spread within the hospital. -
铜绿假单胞菌(Pseudomonas aeruginosa)是引发医院获得性感染的主要病原体之一,尤其在ICU和免疫力低下的患者中常见。近年来,碳青霉烯类抗生素的广泛使用导致耐碳青霉烯类铜绿假单胞菌(carbapenem-resistant Pseudomonas aeruginosa,CRPA),这类细菌对亚胺培南、美罗培南和多利培南等碳青霉烯类抗菌药物均表现出耐药性,已成为全球公共卫生领域的严峻挑战[1-2]。CRPA的耐药通常是多因素综合作用的结果,主要包括以下几方面:碳青霉烯酶基因的获得、膜孔蛋白外膜蛋白D(outer membrane protein D,OprD)的缺失或抑制、多药外排泵亚基A/B-外膜蛋白M(multidrug efflux pump subunits A/B-outer membrane protein M,mexAB-oprM)的过表达、内源性头孢菌素酶的过表达等[3],这些机制导致了CRPA对碳青霉烯类抗菌药物的高水平耐药性。此外,CRPA感染与高病死率密切相关,尤其是在免疫力低下的患者中,CRPA的耐药性进一步加剧了临床治疗的复杂性[4]。因此,明确CRPA的耐药基因型及其在医院内的临床分布,对指导临床合理使用抗菌药物和有效控制院内感染具有重要意义。
本研究分析了海军军医大学第一附属医院CRPA的临床分布特点及耐药基因型,旨在为临床抗菌药物的合理使用、降低细菌耐药性和控制院内感染提供科学依据。
1 材料和方法
1.1 菌株来源及鉴定
收集海军军医大学第一附属医院2019年1月1日至12月31日临床标本中分离出的连续不重复CRPA菌株,全部菌株使用基质辅助激光解吸电离飞行时间质谱仪(德国Bruker公司,型号:Microflex LT)鉴定。
1.2 质控菌株
铜绿假单胞菌(ATCC27853)购自上海市临床检验中心。
1.3 仪器与试剂
本研究中用于菌株分离和培养的血琼脂平板购自上海科玛嘉微生物技术有限公司,抗菌药物敏感性试验所用药敏纸片及MH(Mueller-Hinton)琼脂平板购自英国Oxoid公司。
PCR扩增所用的热循环仪为美国Bio-Rad公司生产,产物的电泳检测和图像采集分别采用北京六一生物科技有限公司生产的电泳仪和凝胶成像系统。
1.4 药物敏感试验
采用VITEK 2 Compact全自动微生物分析仪(法国bioMéieux公司)对菌株进行体外药物敏感试验,用纸片扩散法(Kirby-Bauer法)进行药物敏感补充试验,测定受试菌株对13种抗菌药物的敏感性,包括哌拉西林、头孢哌酮/舒巴坦、哌拉西林/他唑巴坦、头孢他啶、头孢吡肟、氨曲南、亚胺培南、美罗培南、阿米卡星、庆大霉素、妥布霉素、环丙沙星和左氧氟沙星。结果判定参照美国临床和实验室标准协会(Clinical and Laboratory Standards Institute,CLSI)2018年制定的标准[5]进行判读,以铜绿假单胞菌(ATCC27853)作为质控菌株。
1.5 耐药基因的检测
本研究共成功复苏94株CRPA,用于耐药基因的分子检测。采用PCR法扩增其所携带的超广谱β-内酰胺酶(extended-spectrum β-lactamase,ESBL)耐药基因(blaVEB、blaPER、blaTEM、blaSHV和blaGES)、碳青霉烯酶耐药基因(blaKPC、blaIMP、blaVIM、blaNDM、blaOXA-48、blaBIC和blaSIM)和OprD2。DNA采用煮沸法提取,PCR引物设计、反应体系及反应条件均参照文献[6-8]设置,引物序列见表 1。PCR扩增产物通过凝胶电泳检测,判定各目的基因的扩增结果。
表 1 相关耐药基因引物序列Table 1 Primer sequences of drug-resistance genesGene Sequence (5'-3') Reference blaKPC F: CGTCTAGTTCTGCTGTCTTG; R: CTTGTCATCCTTGTTAGGCG [6] blaIMP F: GGAATAGAGTGGCTTAAYTCTC; R: GGTTTAAYAAAACAACCACC [6] blaVIM F: GATGGTGTTTGGTCGCATA; R: CGAATGCGCAGCACCAG [6] blaNDM F: GGTTTGGCGATCTGGTTTTC; R: CGGAATGGCTCATCACGATC [6] blaOXA-48 F: GCGTGGTTAAGGATGAACAC; R: CATCAAGTTCAACCCAACCG [6] blaBIC F: TATGCAGCTCCTTTAAGGGC; R: TCATTGGCGGTGCCGTACAC [6] blaSIM F: TACAAGGGATTCGGCATCG; R: TAATGGCCTGTTCCCATGTG [6] blaVEB F: CGACTTCCATTTCCCGATGC; R: GGACTCTGCAACAAATACGC [7] blaPER F: ATGAATGTCATTATAAAAGC; R: AATTTGGGCTTAGGGCAGAA [7] blaTEM F: GAGTATTCAACATTTCCGTGTC; R: TAATCAGTGAGGCACCTATCTC [7] blaSHV F: AAGATCCACTATCGCCAGCAG; R: ATTCAGTTCCGTTTCCCAGCGG [7] blaGES F: ATGCGCTTCATTCACGCAC; R: CTATTTGTCCGTGCTAAGG [7] OprD2 F: GCGCATCTCCAAGACCATG; R: GCCACGCGATTTGACGGAG [8] blaKPC: Klebsiella pneumoniae carbapenemase, a class A β-lactamase; blaIMP: Imipenemase, a metallo-β-lactamase; blaVIM: Verona integron-encoded metallo-β-lactamase; blaNDM: New Delhi metallo-β-lactamase; blaOXA-48: OXA-48-type class D β-lactamase; blaBIC: Butirosin-inactivating carbapenemase, a class A β-lactamase; blaSIM: Seoul imipenemase, a metallo-β-lactamase; blaVEB: Vietnamese extended-spectrum β-lactamase; blaPER: Pseudomonas extended resistance β-lactamase; blaTEM: Temoniera β-lactamase; blaSHV: Sulfhydryl variable β-lactamase; blaGES: Guiana extended-spectrum β-lactamase; OprD2: Outer membrane protein D2; F: Forward; R: Reverse. 1.6 同源性分析
采用基于k-mer算法的kSNP3软件(https://sourceforge.net/projects/ksnp/)对筛选菌株进行单核苷酸多态性(single nucleotide polymorphism,SNP)分析并构建进化树。基于PubMLST数据库中铜绿假单胞菌的分型(https://pubmlst.org/organisms/pseudomonas-aeruginosa)方案进行多位点序列分型(multilocus sequence typing,MLST)分析,具体方法为:将菌株基因组组装序列提交至数据库,获得等位基因的编号及样本对应的序列类型(sequence type,ST),对于非100%匹配的等位基因,则为新发现的等位基因,将新发现的等位基因序列提交至该数据库,获得新的等位基因编号,同时申请获得新的ST。在PubMLST数据库上传ST信息。
1.7 统计学处理
应用WHONET 5.6软件进行菌株分布和耐药性分析,计数资料以例数或菌株数和百分数表示。
2 结果
2.1 菌株来源及科室分布
从2019年1月1日至12月31日的临床标本中分离出连续不重复CRPA菌株94株,其中73株分离自男性,21株分离自女性。患者来源于各个年龄段,以60岁以上老人居多(54.3%,51/94);标本来源以痰和肺泡灌洗液为主,占67.0%(63/94),其余来源包括无菌体液(7.4%,7/94)、分泌物拭子(7.4%,7/94)、引流液(6.4%,6/94)、血液(5.3%,5/94)和其他标本(6.4%,6/94);科室分布以急救监护室、脑血管外科和呼吸科为主,分别占9.6%(9/94)、8.5%(8/94)和8.5%(8/94);CRPA多分离自住院患者,约占88.3%(83/94)。
2.2 CRPA对抗菌药物的耐药率
94株CRPA对临床常用的13种抗菌药物的药敏试验结果如表 2所示,CRPA对碳青霉烯类抗生素亚胺培南、美罗培南的耐药率最高,分别为92.6%(87/94)和87.2%(82/94);对氨基糖苷类抗生素阿米卡星、庆大霉素、妥布霉素的耐药率最低,分别为10.6%(10/94)、20.2%(19/94)和20.2%(19/94)。
表 2 94株CRPA对抗菌药物的药敏情况Table 2 Antimicrobial susceptibility profiles of 94 CRPA strainsN=94, n(%) Antimicrobial agent Resistance rate Intermediate rate Sensitivity rate Piperacillin 33 (35.1) 19 (20.2) 42 (44.7) Cefoperazone-sulbactam 38 (40.4) 25 (26.6) 31 (33.0) Piperacillin-tazobactam 24 (25.5) 18 (19.2) 52 (55.3) Ceftazidime 29 (30.9) 24 (25.5) 41 (43.6) Cefepime 23 (24.5) 12 (12.8) 59 (62.8) Aztreonam 52 (55.3) 20 (21.3) 22 (23.4) Imipenem 87 (92.6) 3 (3.2) 4 (4.3) Meropenem 82 (87.2) 4 (4.3) 8 (8.5) Amikacin 10 (10.6) 0 84 (89.4) Gentamicin 19 (20.2) 4 (4.3) 71 (75.5) Tobramycin 19 (20.2) 0 75 (79.8) Ciprofloxacin 35 (37.2) 11 (11.7) 48 (51.1) Levofloxacin 36 (38.3) 8 (8.5) 50 (53.2) CRPA: Carbapenem-resistant Pseudomonas aeruginosa. 2.3 耐药基因检测结果
成功复苏94株CRPA进行PCR扩增+凝胶电泳。检出13株(13.8%)blaKPC阳性菌,4株(4.3%)blaVIM阳性菌,2株(2.1%)blaIMP阳性菌,1株(1.1%)blaNDM阳性菌,12株(12.8%)blaTEM阳性菌,4株(4.3%)blaPER阳性菌,2株(2.1%)blaGES阳性菌,blaOXA-48、blaBIC、blaSIM、blaVEB、blaSHV等基因未检出,OprD2基因缺失47株(50.0%)。
在本次分离的CRPA菌株中,各病区的耐药机制分布呈现以下特征:呼吸科病区分离的8株CRPA中,有5株检出OprD2基因缺失;心血管外科监护室分离的7株菌株均为blaKPC阳性,其中6株同时存在OprD2基因缺失;神经外科第一病区分离的7株菌株中,有6株为OprD2基因缺失,其中3株同时为blaKPC阳性;急诊监护室分离的9株菌株中,有5株为OprD2基因缺失;烧伤科监护室分离的6株菌株中,有4株为OprD2基因缺失。
2.4 检出不同耐药基因的CRPA菌株药敏情况对比
将碳青霉烯类基因检测结果与膜孔蛋白基因缺失的检测结果进行比对,使用雷达图直观展示耐药率、敏感率及中介率,可以看出只检出碳青霉烯类基因的CRPA耐药率最高,未检出任何基因的菌株对所有药物的敏感率最高。含碳青霉烯基因和膜孔蛋白基因缺失的CRPA菌株耐药谱最广,呈现对多种抗菌药物的广泛耐药特征。见图 1。
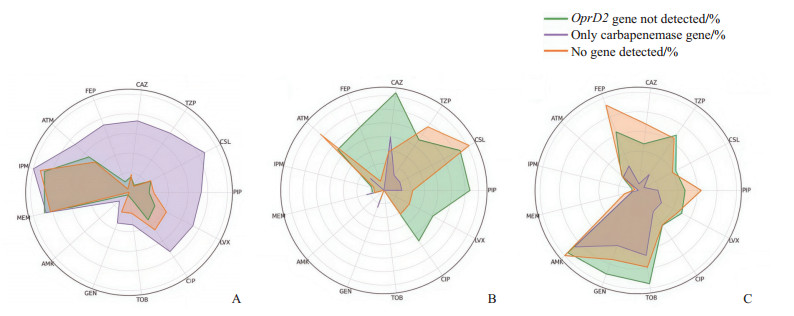 图 1 检出不同耐药基因的CRPA菌株药敏情况对比Fig. 1 Comparison of antimicrobial susceptibility profiles of CRPA strains with different resistance genes detectedA: Resistance rate; B: Intermediate rate; C: Sensitivity rate. CRPA: Carbapenem-resistant Pseudomonas aeruginosa; PIP: Piperacillin; LVX: Levofloxacin; CIP: Ciprofloxacin; TOB: Tobramycin; GEN: Gentamicin; AMK: Amikacin; MEM: Meropenem; IPM: Imipenem; ATM: Aztreonam; FEP: Cefepime; CAZ: Ceftazidime; TZP: Piperacillin-tazobactam; CSL: Cefoperazone-sulbactam; OprD2: Outer membrane protein D2.
图 1 检出不同耐药基因的CRPA菌株药敏情况对比Fig. 1 Comparison of antimicrobial susceptibility profiles of CRPA strains with different resistance genes detectedA: Resistance rate; B: Intermediate rate; C: Sensitivity rate. CRPA: Carbapenem-resistant Pseudomonas aeruginosa; PIP: Piperacillin; LVX: Levofloxacin; CIP: Ciprofloxacin; TOB: Tobramycin; GEN: Gentamicin; AMK: Amikacin; MEM: Meropenem; IPM: Imipenem; ATM: Aztreonam; FEP: Cefepime; CAZ: Ceftazidime; TZP: Piperacillin-tazobactam; CSL: Cefoperazone-sulbactam; OprD2: Outer membrane protein D2.2.5 SNP进化树分析及新MLST型别
2.5.1 SNP进化树分析
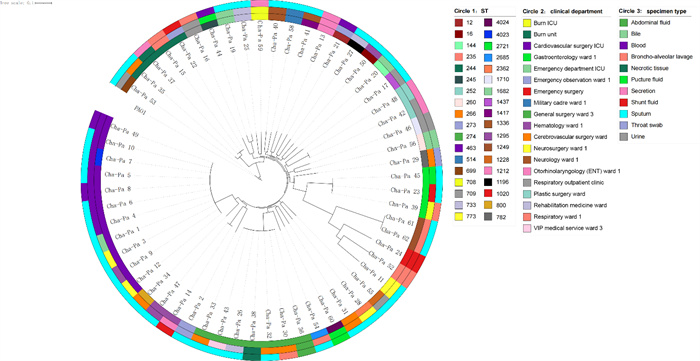
采用基于k-mer算法的kSNP3软件(https://sourceforge.net/projects/ksnp/)对筛选获得的产碳青霉烯酶及含有OprD2基因的62株铜绿假单胞菌进行SNP分析,并构建系统进化树。高通量测序获得菌株基因组序列,剔除3株测序失败菌株,以铜绿假单胞菌PAO 1(NC_002516)为参考基因组,分别将余下的59个样本与参考基因组进行比对,获得各个样本的比对结果,在此基础上提取出所有样本共有的SNP矩阵碱基串联后的fasta文件,基于这个文件,使用最大似然法构建进化树。见图 2。
2.5.2 新发现的MLST型别
本研究共鉴定出36种不同的ST(图 2),其中最常见的是ST463(16.9 %,10/59)和ST274(11.9 %,7/59)。ST463菌株均携带blaKPC-2和blaOXA-486基因,对哌拉西林、头孢哌酮/舒巴坦、哌拉西林/他唑巴坦、头孢他啶、头孢吡肟、氨曲南、亚胺培南、美罗培南、环丙沙星、左氧氟沙星等多种抗生素均呈现高水平耐药。而ST274菌株均携带blaOXA-486基因,对亚胺培南和美罗培南等碳青霉烯类抗菌药物呈现高水平耐药。同时,首次发现了2种新的ST:Cha-Pa 7号样本为ST4023,Cha-Pa 60号样本为ST4024。将对应菌株的等位基因信息和完整基因组信息上传至PubMLST数据库,获得新的ST编号,具体信息见表 3。
表 3 新发现的铜绿假单胞菌ST上传结果Table 3 Upload results of newly discovered ST in Pseudomonas aeruginosaSample ST acsA aroE guaA mutL nuoD ppsA trpE Cha-Pa 60 ST4024 17 24 37 5 3 38 25 Cha-Pa 7 ST4023 152 5 5 3 1 6 3 ST: Sequence type; acsA: Acetyl-CoA synthetase; aroE: Shikimate dehydrogenase; guaA: Guanosine synthase; mutL: DNA mismatch repair protein MutL; nuoD: NADH dehydrogenase subunit D; ppsA: Phosphoenolpyruvate synthase; trpE: Anthranilate synthase component Ⅰ. 3 讨论
铜绿假单胞菌是院内引起肺炎、尿路感染、血流感染的重要致病菌。在WHO 2024年公布的细菌重点病原体清单中,CRPA的优先级从2017年的“关键优先级”调整为“高优先级”,这一调整与全球范围内CRPA耐药性下降有关[9]。根据全国细菌耐药监测网的数据,我国近年来铜绿假单胞菌对碳青霉烯类抗菌药物的耐药率呈现逐年下降的趋势,与上述优先级调整相契合。然而全国细菌耐药监测网2022年的分析报告指出,2022年全国铜绿假单胞菌对碳青霉烯类抗菌药物的平均耐药率为16.6%,上海市为26.0%,居全国最高[10],表明仍需关注CRPA所带来的影响。
本研究结果显示,CRPA主要分布于急救监护室、脑血管外科和呼吸科的老年患者中,标本主要来源于呼吸道,这可能与老年人免疫力低下、病情重且常伴有基础疾病有关。老年患者免疫功能较弱,容易成为耐药菌的易感人群,尤其是那些接受过广谱抗生素治疗的患者,发生CRPA感染的风险更高[11]。同时,铜绿假单胞菌易定植于呼吸道,且痰标本相对其他标本来说易于留取,临床送检率高。这与既往文献报道[12]相似。
铜绿假单胞菌在抗生素的选择性压力下具有显著的获得新耐药机制的能力,有报道指出,在使用比阿培南治疗前后,铜绿假单胞菌在短短4 d时间内就发生了对碳青霉烯类抗生素耐药性的快速演变[3],表明某些抗菌剂的使用与耐药性的发展之间存在较强的相关性。铜绿假单胞菌对亚胺培南的耐药性与碳青霉烯类抗菌药物的使用密切相关,而铜绿假单胞菌对美罗培南的耐药性与第三代头孢菌素的使用也密切相关[13]。近年来,随着抗菌药物使用的增加,CRPA的耐药性迅速演变。本研究发现,短期内碳青霉烯类抗菌药物使用频繁的科室耐药率更高,提示抗菌药物的选择性压力可能是推动耐药性演化的重要因素。
根据本研究的统计数据,CRPA对亚胺培南(92.6%,87/94)和美罗培南(87.2%,82/94)的耐药率最高,并且存在对亚胺培南耐药但对美罗培南敏感的菌株,以及相反地对美罗培南耐药但对亚胺培南敏感的菌株,这与铜绿假单胞菌的耐药机制有关[14]。对亚胺培南耐药但对美罗培南敏感的表型,主要机制是oprD基因突变,该突变导致膜孔蛋白表达下调从而减少了对药物的摄取。而对美罗培南耐药但对亚胺培南敏感的表型则与MexAB-OprM的过表达有关,该泵主动将美罗培南输送至细菌细胞外,降低其细胞内浓度和抗菌活性。
本研究中,检出最多的碳青霉烯酶基因为blaKPC,且全部为blaKPC-2,检出率居第2位的是blaVIM,这与全球的流行病学研究结果[1]一致。有研究表明,blaKPC-2常通过质粒传播,并与毒力基因(如exoU)共存,这种基因组合不仅增强了菌株的致病性,还显著提高了其多重耐药能力[15]。ESBL耐药基因主要检出blaTEM,并且发现有blaVIM和blaTEM在同一分离株中共存的现象,这种共存表明存在多因素耐药机制,使这些菌株感染的治疗难度增加[16]。在复苏成功的94株CRPA中,有47株未检测到OprD2,这表明在这些临床分离株中观察到的耐药性主要是由染色体介导的OprD2丢失导致,而质粒介导产生碳青霉烯酶的机制是次要原因[17]。由blaKPC基因编码的碳青霉烯酶使细菌对碳青霉烯类抗生素产生高度耐药性,临床上限制了亚胺培南和美罗培南的使用。而OprD2基因的缺失则通过减少药物摄入进一步加剧耐药性。这些耐药机制的共存极大增加了CRPA感染的治疗难度,提示临床需针对性地优化治疗方案,如结合多种耐药机制的联合用药策略。
在传统流行病学分析中,脉冲场凝胶电泳作为同源性分析的金标准[18],已被广泛应用于细菌分型。然而,该方法对实验设备要求高,操作复杂,且失败率较高[19]。为了克服这些不足,本研究尝试通过SNP进化树结合MLST方法分析分离菌株的同源性[20]。由结果可见,SNP进化树能够进一步解析相同MLST的菌株,尤其是在相同病区分离的菌株中发现更高的一致性。这表明,SNP进化树不仅具有更高的分辨率和稳定性,同时在院内交叉感染防控方面具有重要的指导意义。
本研究对59株铜绿假单胞菌进行了MLST分析,结果显示出高克隆多样性,共鉴定出36种不同的ST,包括34种已知ST和2种新ST(ST4023和ST4024)。这2种新ST已提交至数据库,为全球科学家提供了新的数据资源,有助于不同地区研究人员比较菌株遗传背景,促进国际科研合作与公共健康监控。在已知的ST中,最常见的是ST463(16.9 %,10/59)和ST274(11.9 %,7/59)。ST463作为一种潜在的高风险克隆型,其流行性和多重耐药性已在多项研究中予以明确。有研究报道ST463菌株普遍携带blaKPC-2和blaOXA-486等耐药基因,并同时具有exoU等重要毒力基因,证实了其在临床感染中的高危潜力[15, 21]。此外,进一步研究发现,ST463菌株可携带包含blaIMP-45和blaAFM-1基因的转座子Tn6485f,展示了其基因组的复杂性和耐药基因获取能力[22]。本研究中,ST463菌株均携带blaKPC-2和blaOXA-486基因,并且对多种抗菌药物呈现高水平耐药,与上述文献的描述一致,表明ST463的耐药性不仅具有高度稳定性,还可能通过水平基因转移进一步扩散。同样,ST274菌株的区域分布和耐药特征也值得关注。有报道称,ST274菌株在中国南部具有较高分布率,其耐药性主要由blaOXA-486基因介导[23]。本研究数据进一步验证了这一发现,显示ST274菌株在携带blaOXA-486的同时,对碳青霉烯类抗生素呈现高水平耐药,提示其在区域流行病学中的重要性。本研究结合ST、耐药基因分布及药敏试验结果,对分离的铜绿假单胞菌的分子特征进行了系统分析,进一步揭示了不同克隆型菌株在耐药性传播中的关键作用。
由病区分布分析可见,心血管外科监护室检出的7株CRPA菌株均携带blaKPC基因,神经外科第一病区则以OprD2基因缺失菌株为主。此外,呼吸科、急诊监护室及烧伤科监护室亦出现OprD2基因缺失菌株的集中分布,提示本院CRPA的耐药机制可能存在科室特异性聚集与院内传播风险。对于这种情况,多模式综合防治策略是一个值得参考的方案,包括接触预防措施、主动监测培养、患者隔离或分组、手部卫生和环境清洁等。几乎所有实施这些策略的研究均报告了感染率的显著降低,表明这些策略在医疗保健环境中的有效性[24]。基于本研究结果,建议加强对CRPA的实时监测和耐药基因的定期检测、优化抗菌药物的使用策略。对于高危病区(如心血管外科监护室和神经外科第一病区)需严格执行隔离措施,并加强手卫生依从性,以减少CRPA在院内的传播风险。
-
图 1 检出不同耐药基因的CRPA菌株药敏情况对比
Fig. 1 Comparison of antimicrobial susceptibility profiles of CRPA strains with different resistance genes detected
A: Resistance rate; B: Intermediate rate; C: Sensitivity rate. CRPA: Carbapenem-resistant Pseudomonas aeruginosa; PIP: Piperacillin; LVX: Levofloxacin; CIP: Ciprofloxacin; TOB: Tobramycin; GEN: Gentamicin; AMK: Amikacin; MEM: Meropenem; IPM: Imipenem; ATM: Aztreonam; FEP: Cefepime; CAZ: Ceftazidime; TZP: Piperacillin-tazobactam; CSL: Cefoperazone-sulbactam; OprD2: Outer membrane protein D2.
表 1 相关耐药基因引物序列
Table 1 Primer sequences of drug-resistance genes
Gene Sequence (5'-3') Reference blaKPC F: CGTCTAGTTCTGCTGTCTTG; R: CTTGTCATCCTTGTTAGGCG [6] blaIMP F: GGAATAGAGTGGCTTAAYTCTC; R: GGTTTAAYAAAACAACCACC [6] blaVIM F: GATGGTGTTTGGTCGCATA; R: CGAATGCGCAGCACCAG [6] blaNDM F: GGTTTGGCGATCTGGTTTTC; R: CGGAATGGCTCATCACGATC [6] blaOXA-48 F: GCGTGGTTAAGGATGAACAC; R: CATCAAGTTCAACCCAACCG [6] blaBIC F: TATGCAGCTCCTTTAAGGGC; R: TCATTGGCGGTGCCGTACAC [6] blaSIM F: TACAAGGGATTCGGCATCG; R: TAATGGCCTGTTCCCATGTG [6] blaVEB F: CGACTTCCATTTCCCGATGC; R: GGACTCTGCAACAAATACGC [7] blaPER F: ATGAATGTCATTATAAAAGC; R: AATTTGGGCTTAGGGCAGAA [7] blaTEM F: GAGTATTCAACATTTCCGTGTC; R: TAATCAGTGAGGCACCTATCTC [7] blaSHV F: AAGATCCACTATCGCCAGCAG; R: ATTCAGTTCCGTTTCCCAGCGG [7] blaGES F: ATGCGCTTCATTCACGCAC; R: CTATTTGTCCGTGCTAAGG [7] OprD2 F: GCGCATCTCCAAGACCATG; R: GCCACGCGATTTGACGGAG [8] blaKPC: Klebsiella pneumoniae carbapenemase, a class A β-lactamase; blaIMP: Imipenemase, a metallo-β-lactamase; blaVIM: Verona integron-encoded metallo-β-lactamase; blaNDM: New Delhi metallo-β-lactamase; blaOXA-48: OXA-48-type class D β-lactamase; blaBIC: Butirosin-inactivating carbapenemase, a class A β-lactamase; blaSIM: Seoul imipenemase, a metallo-β-lactamase; blaVEB: Vietnamese extended-spectrum β-lactamase; blaPER: Pseudomonas extended resistance β-lactamase; blaTEM: Temoniera β-lactamase; blaSHV: Sulfhydryl variable β-lactamase; blaGES: Guiana extended-spectrum β-lactamase; OprD2: Outer membrane protein D2; F: Forward; R: Reverse. 表 2 94株CRPA对抗菌药物的药敏情况
Table 2 Antimicrobial susceptibility profiles of 94 CRPA strains
N=94, n(%) Antimicrobial agent Resistance rate Intermediate rate Sensitivity rate Piperacillin 33 (35.1) 19 (20.2) 42 (44.7) Cefoperazone-sulbactam 38 (40.4) 25 (26.6) 31 (33.0) Piperacillin-tazobactam 24 (25.5) 18 (19.2) 52 (55.3) Ceftazidime 29 (30.9) 24 (25.5) 41 (43.6) Cefepime 23 (24.5) 12 (12.8) 59 (62.8) Aztreonam 52 (55.3) 20 (21.3) 22 (23.4) Imipenem 87 (92.6) 3 (3.2) 4 (4.3) Meropenem 82 (87.2) 4 (4.3) 8 (8.5) Amikacin 10 (10.6) 0 84 (89.4) Gentamicin 19 (20.2) 4 (4.3) 71 (75.5) Tobramycin 19 (20.2) 0 75 (79.8) Ciprofloxacin 35 (37.2) 11 (11.7) 48 (51.1) Levofloxacin 36 (38.3) 8 (8.5) 50 (53.2) CRPA: Carbapenem-resistant Pseudomonas aeruginosa. 表 3 新发现的铜绿假单胞菌ST上传结果
Table 3 Upload results of newly discovered ST in Pseudomonas aeruginosa
Sample ST acsA aroE guaA mutL nuoD ppsA trpE Cha-Pa 60 ST4024 17 24 37 5 3 38 25 Cha-Pa 7 ST4023 152 5 5 3 1 6 3 ST: Sequence type; acsA: Acetyl-CoA synthetase; aroE: Shikimate dehydrogenase; guaA: Guanosine synthase; mutL: DNA mismatch repair protein MutL; nuoD: NADH dehydrogenase subunit D; ppsA: Phosphoenolpyruvate synthase; trpE: Anthranilate synthase component Ⅰ. -
[1] REYES J, KOMAROW L, CHEN L, et al. Global epidemiology and clinical outcomes of carbapenem-resistant Pseudomonas aeruginosa and associated carbapenemases (POP): a prospective cohort study[J]. Lancet Microbe, 2023, 4(3): e159-e170. DOI: 10.1016/S2666-5247(22)00329-9. [2] 丁丽, 陈佰义, 李敏, 等. 碳青霉烯类耐药革兰阴性菌联合药敏试验及报告专家共识[J]. 中国感染与化疗杂志, 2023, 23(1): 80-90. DOI: 10.16718/j.1009-7708.2023.01.013. [3] XU C, WANG D, ZHANG X, et al. Mechanisms for rapid evolution of carbapenem resistance in a clinical isolate of Pseudomonas aeruginosa[J]. Front Microbiol, 2020, 11: 1390. DOI: 10.3389/fmicb.2020.01390. [4] VIVO A, FITZPATRICK M A, SUDA K J, et al. Epidemiology and outcomes associated with carbapenem-resistant Acinetobacter baumannii and carbapenem-resistant Pseudomonas aeruginosa: a retrospective cohort study[J]. BMC Infect Dis, 2022, 22(1): 491. DOI: 10.1186/s12879-022-07436-w. [5] Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing. 28th ed. CLSI supplement M100-S28[S]. Wayne: CLSI, 2018. [6] POIREL L, WALSH T R, CUVILLIER V, et al. Multiplex PCR for detection of acquired carbapenemase genes[J]. Diagn Microbiol Infect Dis, 2011, 70(1): 119-123. DOI: 10.1016/j.diagmicrobio.2010.12.002. [7] SHAHCHERAGHI F, NIKBIN V S, FEIZABADI M M. Prevalence of ESBLs genes among multidrug-resistant isolates of Pseudomonas aeruginosa isolated from patients in Tehran[J]. Microb Drug Resist, 2009, 15(1): 37-39. DOI: 10.1089/mdr.2009.0880. [8] CAI S, CHEN Y, SONG D, et al. Study on the resistance mechanism via outer membrane protein OprD2 and metal β-lactamase expression in the cell wall of Pseudomonas aeruginosa[J]. Exp Ther Med, 2016, 12(5): 2869-2872. DOI: 10.3892/etm.2016.3690. [9] World Health Organization. 2024. WHO bacterial priority pathogens list[EB/OL].[2024-08-29]. https://www.who.int/news/item/17-05-2024-who-updates-list-of-drug-resistant-bacteria-most-threatening-to-human-health. [10] 全国细菌耐药监测网.2022年全国细菌耐药监测报告(简要版)[EB/OL].(2023-11-20)[2024-08-29].https://www.carss.cn/Report/Details?aId=917. [11] YUAN Q, GUO L, LI B, et al. Risk factors and outcomes of inpatients with carbapenem-resistant Pseudomonas aeruginosa bloodstream infections in China: a 9-year trend and multicenter cohort study[J]. Front Microbiol, 2023, 14: 1137811. DOI: 10.3389/fmicb.2023.1137811. [12] 宋皓月, 黄凯峰, 汤荣, 等. 2014-2017年上海市某医院铜绿假单胞菌不同耐药性菌株的临床分布[J]. 中国感染控制杂志, 2019, 18(12): 1093-1098. [13] ZOU Y M, MA Y, LIU J H, et al. Trends and correlation of antibacterial usage and bacterial resistance: time series analysis for antibacterial stewardship in a Chinese teaching hospital (2009-2013)[J]. Eur J Clin Microbiol Infect Dis, 2015, 34(4): 795-803. DOI: 10.1007/s10096-014-2293-6. [14] PRAGASAM A K, RAGHANIVEDHA M, ANANDAN S, et al. Characterization of Pseudomonas aeruginosa with discrepant carbapenem susceptibility profile[J]. Ann Clin Microbiol Antimicrob, 2016, 15: 12. DOI: 10.1186/s12941-016-0127-3. [15] HU Y, LIU C, WANG Q, et al. Emergence and expansion of a carbapenem-resistant Pseudomonas aeruginosa clone are associated with plasmid-borne blaKPC-2 and virulence-related genes[J]. mSystems, 2021, 6(3): e00154-21. DOI: 10.1128/mSystems.00154-21. [16] MUDDASSIR M, MUNIR S, RAZA A, et al. Epidemiology and high incidence of metallo-β-lactamase and AmpC-β-lactamases in nosocomial Pseudomonas aeruginosa[J]. Iran J Basic Med Sci, 2021, 24(10): 1373-1379. DOI: 10.22038/IJBMS.2021.57293.12748. [17] SHEN J, PAN Y, FANG Y. Role of the outer membrane protein OprD2 in carbapenem-resistance mechanisms of Pseudomonas aeruginosa[J]. PLoS One, 2015, 10(10): e0139995. DOI: 10.1371/journal.pone.0139995. [18] NEOH H M, TAN X E, SAPRI H F, et al. Pulsed-field gel electrophoresis (PFGE): a review of the "gold standard" for bacteria typing and current alternatives[J]. Infect Genet Evol, 2019, 74: 103935. DOI: 10.1016/j.meegid.2019.103935. [19] LOPEZ-CANOVAS L, MARTINEZ BENITEZ M B, HERRERA ISIDRON J A, et al. Pulsed field gel electrophoresis: past, present, and future[J]. Anal Biochem, 2019, 573: 17-29. DOI: 10.1016/j.ab.2019.02.020. [20] SALIPANTE S J, SENGUPTA D J, CUMMINGS L A, et al. Application of whole-genome sequencing for bacterial strain typing in molecular epidemiology[J]. J Clin Microbiol, 2015, 53(4): 1072-1079. DOI: 10.1128/JCM.03385-14. [21] HU Y, PENG W, WU Y, et al. A potential high-risk clone of Pseudomonas aeruginosa ST463[J]. Front Microbiol, 2021, 12: 670202. DOI: 10.3389/fmicb.2021.670202. [22] FANG Y, WANG N, WU Z, et al. An XDR Pseudomonas aeruginosa ST463 strain with an IncP-2 plasmid containing a novel transposon Tn6485f encoding blaIMP-45 and blaAFM-1 and a second plasmid with two copies of blaKPC-2[J]. Microbiol Spectr, 2023, 11(1): e0446222. DOI: 10.1128/spectrum.04462-22. [23] SUN L, GUO H, CHEN Y, et al. Genomic and phylogenetic analysis of a multidrug-resistant Pseudomonas aeruginosa ST463 strain co-carrying blaKPC-2, blaOXA-246, and blaOXA-486 in China[J]. J Glob Antimicrob Resist, 2023, 33: 301-303. DOI: 10.1016/j.jgar.2023.04.011. [24] TOMCZYK S, ZANICHELLI V, LINDSAY GRAYSON M, et al. Control of carbapenem-resistant Enterobacteriaceae, Acinetobacter baumannii, and Pseudomonas aeruginosa in healthcare facilities: a systematic review and reanalysis of quasi-experimental studies[J]. Clin Infect Dis, 2019, 68(5): 873-884. DOI: 10.1093/cid/ciy752.

 下载:
下载: