21世纪以来,数字技术的发展掀起了出版传播领域革命性的创新浪潮,对出版媒介的形态、出版内容的形式乃至出版观念都带来了前所未有的冲击和变革。在这个过程中,网络出版和数据出版等新兴出版形态应运而生,海量的科学数据不断被生成和发布,尤其是大数据概念的提出,使得科学研究变成以数据驱动为主的研究模式[1]。
科学数据的潜在价值也逐渐唤醒了出版领域对数据开放与共享的重视。2008年,BioMed Central出版社较早地进行了数据期刊的探索性实验,出版了开放获取期刊BMC Research Notes, 以数据论文作为其子栏目之一。2009年,由Copernicus Publications 出版的Earth System Science Data以发表数据论文为主要形式[2]。2014年5月,自然出版集团(Nature Publishing Group,NPG)推出了在线出版的开放获取数据期刊——Scientific Data,该杂志要求作者针对数据集进行详细描述,帮助科研人员发布、发现和重用研究数据[3]。Nature杂志在2014年10月申明,在其上发表的论文的计算机代码数据将可被访问,作者需要申明是否提供计算机代码以及所设置的访问权限[4]。面对数据出版的蓬勃发展,科学数据资源不仅被视为科学研究的成果,同样成为了支持更为复杂的创新研究所不可替代的资源存量,具有明显的可开发价值[5]。如何对不同出版平台上的科学数据进行管理、共享、集成和挖掘,让科学数据实现价值的最大化,帮助研究人员和科学家们更容易地将科学数据运用于自己的研究工作中,成为传统研究机构、出版机构以及广大研究人员共同关心的问题[6]。
相关学者已经对科学数据开放和出版展开了大量的研究。相关研究主要集中在对科研机构[7]、学术期刊[8]等的数据政策的调研与分析,对国内外科学数据开放和出版现状[9, 10]、特点[1]以及类型[11]的总结等方面。本文尝试从科学数据挖掘和利用的实践角度探讨如何致力于提高用户对开放科学数据的利用程度和效果。Nature Publishing Group(NPG)的ENCODE(the Encyclopedia of DNA Elements)项目在这个方面做了许多有益的尝试,开创了一种全新的出版模式,实现了在不同杂志之间进行主题线索的交织。因在出版创新方面的突出贡献,Nature ENCODE项目入围2013年ALPSP出版创新奖提名[12]。
本文在介绍ENCODE项目背景的基础上,以出版模式创新的角度,从Nature ENCODE创造的引导工具Nature ENCODE Explorer和提出的新概念Thread以及开发的分析工具ENCODE analysis virtual machine三个方面探讨项目方案,并以ENCODE Explorer展示界面为例介绍引导工具的效果与呈现,以期展现这种出版模式在科学数据挖掘和利用方面的创新之处,最后简要总结该出版模式的特点并展望其后续发展状况。
1 ENCODE项目背景与概况ENCODE是由美国国立人类基因组研究院(US National Human Genome Research Institute,NHGRI)在2003年9月组织并启动的跨国研究项目。ENCODE项目的一个主要目标就是去分析人类的基因组中占98.5%的所谓“垃圾”序列的功能。经过项目探索后发现,在人类基因组中,大约有80%的DNA至少从生物化学角度来看是有目的或者说是有功能的[13, 14]。2012年12月21日,ENCODE项目被Science评为年度十大科学突破之一[15]。
ENCODE项目的价值不仅仅在于对人类生命科学领域的重大贡献。ENCODE项目自2003年启动之后,吸引了来自美国、英国、西班牙、日本和新加坡五国的32个研究机构的442名科学家参与,项目于2012年9月基本结题,并将该项目的阶段性研究成果整理成30篇论文,发表于Nature(6篇)、Genome Research(6篇)和Genome Biology(18篇)三本杂志上[13]。项目组在研究过程中积累了丰富的数据资源,获得并分析了超过15万亿字节的原始数据。到项目结项时,共产生了1640个数据集,参与ENCODE项目研究的科研人员累计发表了400多篇论文,其中,有110多篇论文使用了ENCODE公布的研究数据[14]。
为了充分利用如此庞大的数据和文献资源,NHGRI早在2007年9月便投入了资金使ENCODE工程规模化,建立了数据协调中心(Data Coordinating Center,DCC)和数据分析中心(Data Analysis Center),前者用于追踪、存储和公布数据,而后者则用来对数据进行综合分析[16]。目前,所有ENCODE项目中产生的数据会被提交到数据协调中心进行质量控制并发布,发布后用户即可通过ENCODE项目注册的门户网站(http://www.encodeproject.org)免费获取到。
2 ENCODE的数据出版模式 2.1 The Nature ENCODE Explorer为了科研的需要,几乎所有的研究人员在面对大量信息和数据时,都会对搜集的研究论文、科研报告或书籍中最感兴趣的内容进行重点标记或者整理,从而在脑海中和书面上建立条理清晰的“信息图谱”。而NPG将这一过程“工具化”了,为了帮助他人浏览和分析ENCODE项目产生的30篇公开论文和大量数据,NPG充分运用数据可视化技术创造了一个互动的形象化引导工具——Nature ENCODE Explorer。NPG为ENCODE项目创建的网站(http://www.nature.com/encode/)通过Nature ENCODE Explorer把发表在不同期刊上的30篇相关论文有效整合在一起。Nature ENCODE Explorer是NPG与After the Flood公司合作完成的一个可视化项目,其作为一个互动的形象化的引导工具,展示了人类基因组相关论文中主题内容之间的关联,为使用者提供了一个与ENCODE项目组论文进行交互的方式。使用者在Nature ENCODE Explorer中既可以浏览单篇的论文,也可以通过Thread深入探究这些文章中的“原材料”。
Nature ENCODE Explorer的设计原则:①敏感的颜色层次。在对每个Thread的颜色进行编码的时候,对颜色与颜色之间做足够的区分。②重要的颜色被预留下来并用于关键功能元件。这个程序的关键在于——不仅仅需要做可视化设计,也要考虑可视化环境、可视化控制、与其他网站或应用程序的链接,并始终把用户视作界面设计过程的中心。
After the Flood公司是这样评价ENCODE计划的:“NPG在纸版科学可视化方面有丰富的经验,但这次是他们迄今为止最雄心勃勃的数字可视化项目。”[17]NPG对计划的科学性、完整性进行了测试,并希望系统能在浏览器和平板电脑上工作,所以需要非常强大、实用以及创新的可视化效果。如今,Nature ENCODE Explorer由于它的易用性获得了科学界的诸多赞誉。
2.2 Thread所谓“Thread”,即“线程”,也称为“主题线程”(Themed Thread),是ENCODE提出的一个科学信息出版的新概念,开辟了一种全新的数据挖掘程序。
当一个大型研究项目长时间内产生大量数据时,这些数据可能涵盖很多不同层次和主题的信息。ENCODE项目在准备发布其第一轮研究结果时就意识到,他们必须决定哪些信息在传统的研究论文范围内是最突出和最完整的。为了将这些包含在多个文件中的支离破碎的信息进行整合,他们建立了一套“Thread”。Thread是Nature ENCODE Explorer的核心,是一个探索多篇文章中的共同主题的方式,使得看似普通的研究报告在数字世界里以一种整合的形式呈现在读者面前,满足了研究人员希望能将每篇文章中最感兴趣的关注点进行拼贴汇总的需求。
ENCODE项目的作者除了完成论文的创作之外,还需要做一些额外的工作,即从每篇文章中挑选出他们认为能够引起不同研究领域内的科学家们极大兴趣的部分。这个提炼精华的步骤是研究人员们所希望看到的,同时也是科学信息发布的过程之一。于是,Nature的编辑们对项目发表的30篇文章中的相关段落和图表进行了整合汇总,提炼出了文章中 的13个生物主题,形成了13个“Thread”。需要说明的是,这13个Thread并不是含有ENCODE项目原始数据的生物主题,而是对文章中的内容进行的提炼汇总,每个Thread都是由一个特定主题的相关文章中的段落、数字和表格组成,其中包含有相关文章的链接,读者们能够很方便地在多篇文章之间切换,清楚地了解数据之间是如何联系起来的。
尽管Thread至今还没有一个标准的定义,也不能被数据库检索,但这种方式提供了一个从不同角度探索文章中信息的工具,力求使文章的价值最大化。与ENCODE项目的作者们共事的Nature杂志编辑Skipper曾说过:“研究者们喜欢线程的原因是其视觉吸引力和内容富有价值。未来的一个理想状态是能够自动生成任何主题的线程。”[18]
2.3 分析工具——虚拟计算机除了Thread的概念以外,ENCODE项目还同时引入了另一项科技创新技术——虚拟计算机(ENCODE analysis virtual machine),帮助用户对文章中的数据集进行重用和计算。在Nature发表的这30篇文章中,有一篇ENCODE 项目的概述文章中的“supplementary information(补充信息)”部分,包含了一套代码包,这套代码包提供与文章相关的分析方法的脚本和处理步骤。分析团队基于这套代码包建立了一个ENCODE分析虚拟机,其中每个分析程序都已经进行了运行测试,并且实现了一部分的实例提供给使用者,方便其决定是否或者如何建模分析。
ENCODE项目团队一直强调“透明度”。首先,虚拟计算机对使用者是免费提供的;其次,使用者可以随意运用相关软件对ENCODE的数据进行数据集计算。既可以进行数据的调阅、拼接、分析、整合和预测,也可以对文章中某些特定内容进一步分析研究,通过调整某些具体的参数,发现原作者没有发现的现象或规律。项目的成员还开发和训练了诸多软件工具,例如用于对多种数据类型进行综合分析的软件等。
这种看似复杂的多样性的分析方法对于从事研究工作的科学家们并不陌生,但也许会令缺少大数据处理经验的人感到困惑。然而,设计分析工具的目的不是提供易于使用的程序,或强大的工程解决方案,而是使得分析结果更具科学透明度。
另外需要说明的是,为了保证用于分析挖掘的科学数据的数量和质量,ENCODE制定了其数据发布政策,并定期更新。最新的ENCODE数据发布政策发布于2014年3月,以便研究者自由下载、分析和发布基于ENCODE数据的研究结果。首先,ENCODE项目组鼓励尽可能广泛地使用数据集,生产的所有数据在被释放到公共数据库之后,便可被不受限制地使用。其次,鼓励使用未发布的ENCODE数据的研究人员与该数据的提供者协商,合作发表出版物。当然这是非强制性的建议。再次,政策要求研究者在出版物或发言中使用ENCODE的数据集(发布的和未发布的)时,要以一定的形式援引ENCODE联盟,例如引用联盟最新的综合出版物,或者提及ENCODE数据协调中心(DCC),或数据集在GEO数据库里的唯一识别号,或者承认是由ENCODE联盟和实验室产生了特定的数据集。
为ENCODE项目提供数据分析支持的美国麻省理工学院(Massachusetts Institute of Technology,MIT)的生物信息学家Manolis Kellis就评价道:“ENCODE项目就是一个信息的宝库。”[14]有了Thread和虚拟计算机等工具的协助,弄清楚ENCODE项目产出的大量令人眼花缭乱的数据不再是难题,科学家们能够更容易将这些信息运用于自己的研究工作中,准确地发掘出研究论文中的有价值的信息,再一次推动科研工作的革新。这也是ENCODE项目的初衷。
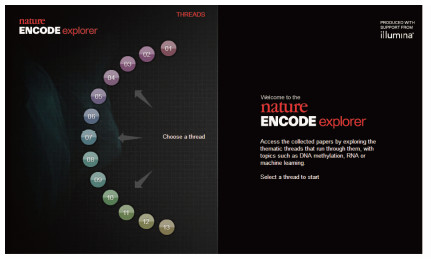
3 效果与呈现在Nature ENCODE的网站(http://www.nature.com/encode/)上,汇集了ENCODE计划的主要研究成果——同时发表在Nature,Genome Research和Genome Biology上的30篇相关论文和众多的综合分析。网站首页上方嵌入了Nature ENCODE Explorer的展示界面,简洁明了地提示用户需要做什么,即从13个Thread中任选其一,点击查看(参见图1)。
|
图 1 Nature ENCODE Explorer首页[19] |
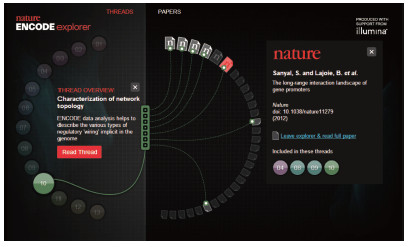
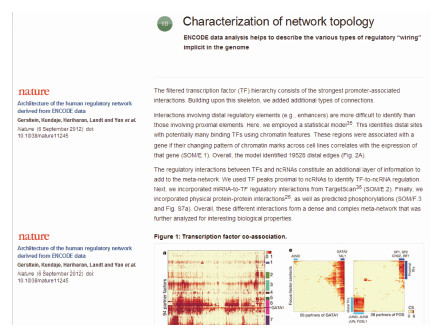
当一个Thread被触发时,会呈现对该Thread的概述(Thread Overview),并且与该Thread相关联的论文会高亮突出显示。任意选择一篇相关论文,界面右侧将会呈现这篇文章的基本信息,包括文章作者、题目、发表刊物、刊期,并提供一个阅读全文的链接(如图2所示)。值得一提的是,界面还会提示 用户该文章所属的其他主题,用户可随意点击其编号进行主题的切换,查看文章所属的其他主题。如前文所述,Nature ENCODE Explorer为用户汇总了13个特定的科学主题,用户点击“Read Thread”按钮,即可深入探究一个特定主题线程的详情,包含了从发表的ENCODE论文中提取的相关章节、图表等内容(如图3所示)。
|
图 2 Nature ENCODE Explorer的Thread[20] |
|
图 3 某个Thread的详情[21] |
ENCODE促发了一种新的出版模式的出现,这种出版模式在科学数据挖掘和利用方面的创新之处可以总结为以下两点:第一,项目创建的Nature ENCODE Explorer可以把不同期刊上相关论文的内容归类,基于可视化技术和数据挖掘技术,创造易于使用的可视化工具,对项目成果进行了有效整合;第二,ENCODE注重科学数据的重用,引入分析工具有效提升科学数据的再生价值。
然而在推广这种模式的过程中,也会面临一些障碍。首先,目前的文本挖掘工具还有些滞后,例如还不能充分提取相关的数据或条目。其次,整合不同期刊上的论文建立在不同出版商合作关系的基础之上,还可能受到论文的开放获取方式和相关版权协议的制约,如果出版商拒绝合作,“Thread”将难以组织而失去意义;再次,虚拟计算机的运行需要组织得当的、完全开放的数据,这也将是一项困难的工作。尽管面临这些实际问题,项目组在结题之后的几年中并没有停止成果产出,期间不断有科研论文问世,项目产生的数据集也在不断增加。
开放共享已经成为信息社会发展的必然趋势,伴随着大数据技术在出版领域的逐步渗透和数据开放步伐的加快,科学家们获得了比以往更多的可支配数据。对于科技期刊来说,如果能够克服政策和技术的障碍,允许对期刊论文(数据)进行重用,把不同出版平台上的论文(数据)以一定的标准整合在一起,由学界将各自研究领域中感兴趣的内容进行归类和整合,再配合数据可视化技术充分展现数据背后的精彩,将会营造一个更加开放、互动性更强、科研成果更丰富的学术环境,相信这将是科学家和研究人员们所期许的。
致谢: 感谢中国科学院文献情报中心编辑出版中心动态研究组以及马建华老师的精心指导。
 2015, Vol. 26
2015, Vol. 26 


 2)
2)