2. 鹏城实验室 广东 深圳 518055;
3. 北京大学 计算语言学教育部重点实验室 北京 100871
2. Peng Cheng Laboratory, Shenzhen 518055, China;
3. Institute of Computational Linguistics, Peking University, Beijing 100871, China
随着互联网技术的飞速发展,知识的存储和共享变得越来越便捷,随之而来的是知识总量的指数级增长,各领域的知识已不再是孤岛,而是在互联网的海洋中相互交融、交叉发展。自Google公司提出“Knowledge Graph”[1]后,这种可以绘制知识脉络、挖掘数据间的潜在关系、分析语义信息以及以图谱方式可视化地为用户提供知识信息的技术迅速引起了各领域的研究兴趣。知识图谱的前身是语义网[2],语义网致力于让计算机能够理解和处理文本中所表达的语义信息,从而支持网络环境下广泛有效的自动推理。而知识图谱作为知识载体,其最大的优势就是使知识可视化[3],让人们既能快速理清专业知识之间的逻辑脉络,也能把握住最关键的知识点,迅速找到自己需要的信息。
目前,医疗领域是知识图谱重要的应用领域之一。在医学知识图谱研究领域,SNOMED-CT[4-5]、IBM Watson Health[6]等,都致力于构建一套全面统一的医学术语系统,来对大部分临床信息进行统一的标准化描述。目前的医学知识本体库主要有医学概念知识库LinkBase[7]、TAMBIS本题库[8]等。一体化医学语言系统自动构建了医学知识库[9]。上海曙光医院构建了中医药知识图谱[10],利用自身优势,对大量的中医药数据和临床诊疗知识库进行了整合和利用。贾李蓉等[11]于2002年开始研制中医药学语言系统,目前已发展为包括13万多个概念、30余万术语以及100余万语义关系的大型语义网络。侯丽等[12]构建了基于本体的临床医学知识库,构建以知识节点为对象的疾病库、药物库、检查库三大医学知识库,并通过不同知识库中的知识节点建立关联。
建立一个结构科学、层次清晰、覆盖全面、高度关联的大规模、高质量中文医学知识图谱,离不开大规模医疗语料库的支撑[13-14]。在构建过程中,本研究首先对语料进行了分析,提出了初步的医学知识图谱描述体系,而后在医学专家的指导下进一步完善标注规范,并对多来源文本进行了多轮人工标注以及自动抽取工作。构建了一个覆盖面广、知识描述准确、结构完备的中文医学知识图谱,为智慧医疗领域的广泛应用奠定了专业知识基础。
1 知识图谱构建过程中文医学知识图谱的构建过程可分为两个阶段:一是描述体系设计;二是知识图谱构建。首先,我们调研了国内外权威的标准医学术语集,初步设计了概念分类体系,通过案例标注与分析,设计了关系分类体系,而后经过医学专家的评估,形成了医学知识图谱的描述体系。随后,通过从权威性、全面性和实用性等方面的考量,经过对比和整合,形成了多来源医学文本。对于这些非结构或半结构化的文本数据,采用了人工标注和自动提取两种方法相结合从中提取关系,其中自动提取使用了规则加tagging模型的方法。对于抽取出的实体及实体关系,进行人工审核评估,构建医学知识图谱知识本体,并完成多来源数据的知识融合,从而构建出专业性强、内容丰富的中文医学知识图谱。整体构建框架如图 1所示。
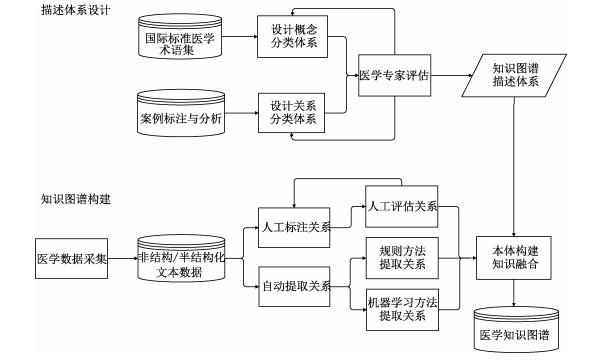
|
图 1 医学知识图谱构建框架 Fig. 1 Constructing framework of medical knowledge graph |
实体资源库的构建基于医学主题词表(medical subject headings,MeSH)、融合国际疾病分类[15](international classification of diseases,ICD-10)、解剖学治疗学及化学分类系统(anatomical therapeutic chemical,ATC)等医学术语为资源库中的实体。MeSH[16]是美国国立医学图书馆编制的医学主题词表,它是一部规范化、可扩充的动态性叙词表。ICD-10是国际疾病分类的第10个版本,是根据疾病的某些特征,按照规则将疾病分门别类,并用编码的方法来表示的系统。ATC是解剖学治疗学及化学分类系统,是世界卫生组织对药品的官方分类系统。实体资源库的构建不仅是本研究标注实体的依据,也是后续利用机器学习进行命名实体识别以及关系抽取的基础。
为保证标注质量以及标注进度,本研究选取了常见的106种疾病进行人工标注。通过分析多来源医疗文本结构信息,按照其不同的类型进行分类,分别从病因、并发症、诊断步骤、鉴别诊断、流行病学、病史和查体、监测、预防、预后等多个维度对疾病进行了描述。
在语料中经常会出现一句话里或一整段里没有提及该主题疾病的情况,这时就无法标注此种疾病的实体。为此,本研究对语料进行了预处理,以句号为特征,在每一句之前加上主题疾病的名字,并以@和原文做分割。如“患者可能有各种不典型症状,这些症状包括乏力、恶心、呕吐、腹痛以及晕厥。”预处理后为“非ST段抬高型心肌梗死@患者可能有各种不典型症状,这些症状包括乏力、恶心、呕吐、腹痛以及晕厥。”
2 知识图谱描述体系及标注规范本研究将实体分为12大类,分别为语义、疾病、部位、症状、检查、手术治疗、药物治疗、其他治疗、流行病学、预后、社会学和其他,并使用不同的参考标准界定每一类实体涵盖的范围。实体之间的关系包括:语义、疾病-疾病、疾病-部位、疾病-症状、疾病-检查、疾病-手术治疗、疾病-药物治疗、疾病-其他治疗、疾病-流行病学、疾病-预后、疾病-社会学、疾病-其他共12个类型关系。具体关系类型如表 1所示。
|
|
表 1 疾病类实体间常见关系类型 Tab. 1 Common types of relationships between disease-class entities |
命名实体标注的基本原则有:第一,不重叠标注,即同一段字符串不能标注为两个不同的实体;第二,不嵌套标注,即一个实体不能在另一个实体的内部;第三,实体要尽可能不含有标点符号及连接词(或、和、以及),主要目的是为了防止实体过长和实体嵌套。
将疾病类实体作为核心,以疾病为入口,标注疾病与其他类实体和属性值(字符串、数字)之间的关系类型。对于非结构化的文本,实体关系经常跨句出现甚至跨段出现,所以标注关系时,不仅仅局限于一个句子范围内的实体关系,还包括跨段句子范围内的关系。
3 知识图谱的构建 3.1 知识图谱构建过程为了提升标注效率,开发了面向医疗文本的实体及关系标注平台。对于医疗文本中出现的实体,选择实体标签,选中对应的文字,即可完成实体标注。在完成实体标注后,可以选择进行关系标注,再依次选择关系标签所对应的第一个实体和第二个实体,即可完成两个实体间的关系标注。标注平台可以实现实体标注和关系标注的切换,同时提供了文件管理功能,方便进行多轮标注工作。
第一阶段,在详细地分析了多来源医疗文本的特点之后,借鉴中文电子病历标注规范[17]的经验,本研究初步制定出了医学知识图谱描述体系结构,以此为基准,进行了3轮的试标注过程。试标注阶段主要目的在于收集标注中所发现的问题,经过和医学专家的讨论,进一步完善规范。与此同时,实体资源库的收集工作和标注平台的开发工作也在同步进行中。
第二阶段,为了保证标注过程的准确性和一致性,本研究采取了多轮标注的方法。一人标注完成后,另一人进行二次标注,两次标注不一致和不确定的地方要记录下来,留待讨论会讨论,经过医学专家和老师的确认后,再由一标人返回语料中进行修改,形成最终的三标版本。在此过程中,根据标注人员的反馈,本研究也在不断地优化、更新标注规范,使其更加贴合语料自身特点。整体构建流程图如图 2所示。
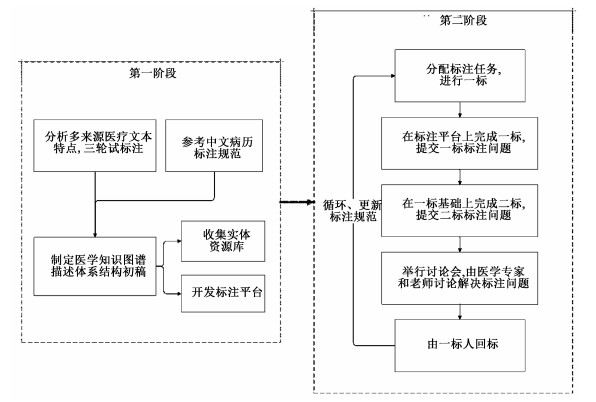
|
图 2 语料库构建流程图 Fig. 2 Flow chart of corpus construction |
知识图谱的描述形式为六元组,分别为实体1、关系、实体2,以及对每一元的约束或属性,表示为{ entity1,entity1_property,relation,relation_property,entity2,entity2_property},简记为{e1,e1_pro,rel,rel_pro,e2,e2_pro}。
CMeKG使用百度开源可视化库Echarts展示知识图谱,对于每一个实体,选择以该实体为主语的六元组进行显示,其中属性描述的三元可以为空,即六元组在实体和关系的属性都为空时会蜕化为三元组。如图 3所示,连接同一节点的相同颜色节点代表相同的语义关系,整体效果呈现为以查询实体为中心,具有语义关系的相关实体发散至四周的网状结构,每个关系边的权重相等。
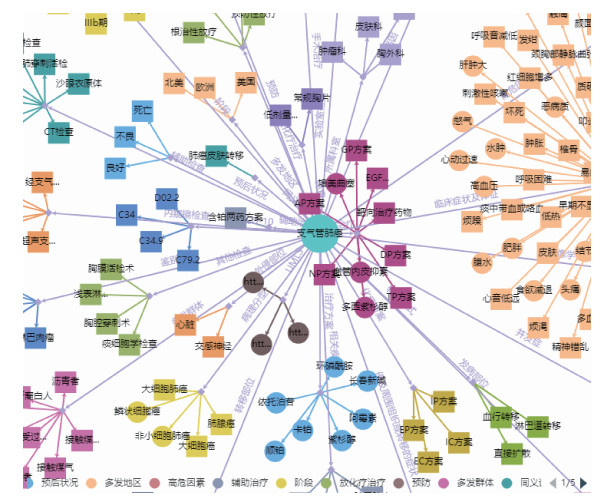
|
图 3 中文医学知识图谱展示界面 Fig. 3 Chinese medical knowledge graph display interface |
标注一致性用Kappa值[18]和F值[19]来表示。Kappa值在情感极性分类的语料标注中应用较广,但在实体识别中,若把未标注的文本作为反例的话,则反例数量巨大而难以统计。此种情况下F值接近于Kappa值,故可使用F值来对实体识别标注语料进行一致性评价[20]。具体做法是将三标者A1的标注结果作为标准答案,计算一标者A2的标注结果的精确度P和召回率R,进而计算F值,计算公式为
| $ P = \frac{{{A_1}{\rm{和}}{A_2}一致的标注结果总数}}{{{A_2}的标注总数}}, R = \frac{{{A_1}{\rm{和}}{A_2}一致的标注结果总数}}{{{A_2}的标注总数}}, F = \frac{{2 \times P \times R}}{{P + R}}。$ |
确定实体一致性时,只有当实体文本、实体类型标签和起止位置均相同时,才认为实体标注是一致的;确定关系一致性时,只有当实体对的两个实体、关系名称和起止位置均相同时,才认为关系标注是一致的。文献[21]指出,标注一致性达到80%以上时,可以认为语料的一致性是可信赖的。最终,本研究标注的命名实体识别一致率达到了87.3%,实体关系一致率达到了82.9%,说明本研究的标注结果是可信赖的。
共有两名医学专家和近二十名标注人员参与标注工作。共计完成标注3 029 448字、23 475种实体概念、32 530个实体关系三元组。去重后的实体数量如表 2所示,关系数量如表 3所示。
|
|
表 2 实体数量 Tab. 2 Number of entities |
|
|
表 3 关系数量 Tab. 3 Number of relationships |
以人工标注的106种疾病标注结果为基础,本研究还使用了规则加tagging模型对745种其他疾病进行了自动抽取工作,共抽取出140 224条实体关系,经过人工校对后,剔除57 676条错误信息,剩余82 548条关系,准确率为58.8%。因为医疗文本的非结构化信息比较复杂,所以自动抽取的准确率还有待进一步的提高。
中文医学知识图谱(http://cmekg.pcl.ac.cn)展示界面如图 3所示。图形化展示是中文医学知识图谱的重要部分,通过系统页面的首字母索引或者搜索框可以查询到各类疾病实体,并以疾病为中心放射性链接与之相关的各类实体及关系。
5 小结本文主要对中文医学知识图谱的标注进行了研究,具体从3个方面来进行展开:首先调研了国内外各类医学资源的整体情况,将其整合成多来源医疗文本作为标注文本;然后在分析语料库的结构基础上,和医学专家讨论制定出医学知识图谱描述体系,通过试标注给出初版标注规范;最后通过多轮迭代的方式标注医疗文本,并请医疗专家全程把控标注质量,以确保准确性,同时使用规则加机器学习的方法进行自动抽取,至2019年3月,构建了中文医学知识图谱CMeKG1.0版,包括6 310种疾病,20余万种实体概念,100余万个实体关系三元组。目前来看,机器标注的准确率还有待进一步提升,同时人工标注集和海量的医学文本相比规模还是比较小,未来将继续研究如何进一步提高自动标注的准确率,并且根据妇产科学、儿科学等临床医学主要学科进一步扩充标注疾病。
| [1] |
WANG C Y, GAO M, HE X F, et al. Challenges in Chinese knowledge graph construction[C]//31st IEEE International Conference on Data Engineering Workshops. Seoul, 2015.
(  0) 0) |
| [2] |
ZHANG X L, DU C L, LI P S, et al. Knowledge graph completion via local semantic contexts[M].Database systems for advanced applications. Cham: Springer International Publishing, 2016.
(  0) 0) |
| [3] |
刘则渊, 陈悦, 候海燕. 科学知识图谱方法与应用[M]. 北京: 人民出版社, 2007. LIU Z Y, CHEN Y, HOU H Y. Mapping of scientific knowledge: methods and applications[M]. Beijing: People's Publishing House, 2007. (  0) 0) |
| [4] |
牟冬梅, 张艳侠, 黄丽丽, 等. 基于SNOMED CT和FCA的医学领域本体构建研究[J]. 情报学报, 2013(6): 653-662. MU D M, ZHANG Y X, HUANG L L, et al. Constructing medical ontology based on SNOMED CT and FCA[J]. Journal of the China society for scientific and technical information, 2013(6): 653-662. DOI:10.3772/j.issn.1000-0135.2013.06.010 (  0) 0) |
| [5] |
AMARILLI A, GALÁRRAGA L, PREDA N, et al. Recent topics of research around the YAGO knowledge base[M]. Cham: Springer International Publishing, 2014.
(  0) 0) |
| [6] |
AUER S, BIZER C, KOBILAROV G, et al. DBpedia: a nucleus for a web of open data[M]. Berlin: Springer Berlin Heidelberg, 2007: 722-735.
(  0) 0) |
| [7] |
CEUSTERS W, MARTENS P, DHAEN C, et al. LinkFactory: an advanced formal ontology management system[J]. Proceedings of interactive tools for knowledge capture (KCAP 2001). Victoria B C, 2001: 75-204.
(  0) 0) |
| [8] |
STEVENS R, BAKER P, BECHHOFER S, et al. TAMBIS: transparent access to multiple bioinformatics information sources[J]. Bioinformatics, 2000, 16(2): 184-186. DOI:10.1093/bioinformatics/16.2.184 (  0) 0) |
| [9] |
NADKARNI P, CHEN R, BRANDT C. UMLS concept indexing for production databases: a feasibility study[J]. Journal of the American medical informatics association, 2001, 8(1): 80-91. DOI:10.1136/jamia.2001.0080080 (  0) 0) |
| [10] |
阮彤, 孙程琳, 王昊奋, 等. 中医药知识图谱构建与应用[J]. 医学信息学杂志, 2016, 37(4): 8-13. RUAN T, SUN C L, WANG H F, et al. Construction of traditional Chinese medicine knowledge graph and its application[J]. Journal of medical informatics, 2016, 37(4): 8-13. DOI:10.3969/j.issn.1673-6036.2016.04.002 (  0) 0) |
| [11] |
贾李蓉, 刘静, 于彤, 等. 中医药知识图谱构建[J]. 医学信息学杂志, 2015, 36(8): 51-53, 59. JIA L R, LIU J, YU T, et al. Construction of traditional Chinese medicine knowledge graph[J]. Journal of medical informatics, 2015, 36(8): 51-53, 59. DOI:10.3969/j.issn.1673-6036.2015.08.012 (  0) 0) |
| [12] |
侯丽, 钱庆, 黄利辉, 等. 基于本体的临床医学知识库系统构建探讨[J]. 医学信息学杂志, 2011, 32(4): 42-47. HOU L, QIAN Q, HUANG L H, et al. Discussion on clinical medicine knowledge base system construction based on ontology[J]. Journal of medical informatics, 2011, 32(4): 42-47. DOI:10.3969/j.issn.1673-6036.2011.04.011 (  0) 0) |
| [13] |
徐琳宏, 林鸿飞, 赵晶. 情感语料库的构建和分析[J]. 中文信息学报, 2008, 22(1): 116-122. XU L H, LIN H F, ZHAO J. Construction and analysis of emotional corpus[J]. Journal of Chinese information processing, 2008, 22(1): 116-122. DOI:10.3969/j.issn.1003-0077.2008.01.019 (  0) 0) |
| [14] |
张德政, 谢永红, 李曼, 等. 基于本体的中医知识图谱构建[J]. 情报工程, 2017, 3(1): 35-42. ZHANG D Z, XIE Y H, LI M, et al. Construction of knowledge graph of traditional Chinese medicine based on the ontology[J]. Technology intelligence engineering, 2017, 3(1): 35-42. (  0) 0) |
| [15] |
SUNDARARAJAN V, HENDERSON T, PERRY C, et al. New ICD-10 version of the Charlson comorbidity index predicted in-hospital mortality[J]. Journal of clinical epidemiology, 2004, 57(12): 1288-1294. DOI:10.1016/j.jclinepi.2004.03.012 (  0) 0) |
| [16] |
LIPSCOMB C E. Medical subject headings (MeSH)[J]. Bulletin of the medical library association, 2000, 88(3): 265-270. (  0) 0) |
| [17] |
杨锦锋, 于秋滨, 关毅, 等. 电子病历命名实体识别和实体关系抽取研究综述[J]. 自动化学报, 2014, 40(8): 1537-1562. YANG J F, YU Q B, GUAN Y, et al. An overview of research on electronic medical record oriented named entity recognition and entity relation extraction[J]. Acta automatica sinica, 2014, 40(8): 1537-1562. (  0) 0) |
| [18] |
JEAN C. Assessing agreement on classification tasks: the kappa statistic[J]. Computational linguistics, 1996, 22(2): 249-254. (  0) 0) |
| [19] |
HRIPCSAK G. Agreement, the F-measure, and reliability in information retrieval[J]. Journal of the American medical informatics association, 2005, 12(3): 296-298. DOI:10.1197/jamia.M1733 (  0) 0) |
| [20] |
OGREN P, SAVOVA G, CHUTE C. Constructing evaluation corpora for automated clinical named entity recognition[C]//Proceedings of the 12th World Congress on Health (Medical) Informatics. Marrakech, 2008: 28-30.
(  0) 0) |
| [21] |
ARTSTEIN R, POESIO M. Inter-coder agreement for computational linguistics[J]. Computational linguistics, 2008, 34(4): 555-596. DOI:10.1162/coli.07-034-R2 (  0) 0) |
 2020, Vol. 52
2020, Vol. 52


