2. 郑州大学 数字化影像技术研究中心 河南 郑州 450001;
3. 郑州大学 第一附属医院放射科 河南 郑州 450052;
4. 郑州大学 第五附属医院影像科 河南 郑州 450052
2. Digital Medical Image Research Center, Zhengzhou Universiyt, Zhengzhou 450001, China;
3. Department of Radiology, the First Affiliated Hospital of Zhengzhou University, Zhengzhou 450052, China;
4. Department of Radiology, the Fifth Affiliated Hospital of Zhengzhou University, Zhengzhou 450052, China
乳腺癌是女性发病率最高的肿瘤,早期精确诊断可使乳腺癌患者五年生存率从25%提高到99%[1]。基于乳腺X线摄影的乳腺定期筛查是发现乳腺肿瘤的重要手段[2]。放射科医生通常利用双侧头尾位和内外侧斜位四幅钼靶图像进行比较阅片[3]以提高诊断准确性。然而,由于放射科医生工作负担繁重和主观因素等影响,仍存在一定的漏诊和误诊现象。因此, 研究计算机辅助检测(computer-aided detection,CAD)方法提高医生诊断准确性,具有重要意义[4]。
近年来,基于深度学习的乳腺CAD研究有了较大进展[5-6]。文献[7]提出一种R-CNN乳腺肿块自动检测算法。通过多尺度深度置信网络和高斯混合模型筛选肿块感兴趣区域,并使用级联R-CNN网络提取图像特征,最后利用随机森林检测乳腺肿块,敏感性达到75%,平均每幅图像检测出4.8个假阳性区域。文献[8]在faster-RCNN基础上提出一种乳腺肿块检测算法,通过图像增强和分块预处理可使检测敏感性达到93%,但平均每幅图像检测出0.56个假阳性区域。文献[9]提出一种基于YOLO(you only look once)的乳腺肿块检测算法。通过调整角度扩充数据集,利用YOLO网络生成特征图,将特征图回归到原图像,预测肿块的位置和类别概率,敏感性可以达到93.2%,但特异性仅有78%。综上所述,深度学习在乳腺肿块检测中起着重要作用。但由于肿块与正常组织区分度低,成像时腺体重叠,当前基于单视图的深度学习乳腺肿块检测算法存在假阳性率较高的共性问题。
为了减少假阳性率,本文模拟医生在双视图上观察乳腺肿块诊断特征的方法,筛选并标注与肿块相似度较高的假阳性区域,在数据层扩充监督学习信息。然后基于YOLOv3框架设计适合双视图数据集的CNN网络:包括设计空间金字塔池化模块融合多尺度局部特征,增强微小肿块的特征表达能力;通过增加平衡参数和聚焦参数改进分类损失函数, 提升算法对肿块与假阳性的辨别能力。最后用大量的仿真实验验证了本文算法的有效性。
1 基于深度学习的乳腺肿块分析深度学习因具有特征自学习能力在目标检测领域呈现出突出潜力[10],代表算法包括faster-RCNN和YOLOv3目标检测方法。本节对比分析这两类算法对乳腺肿块检测的适用性。
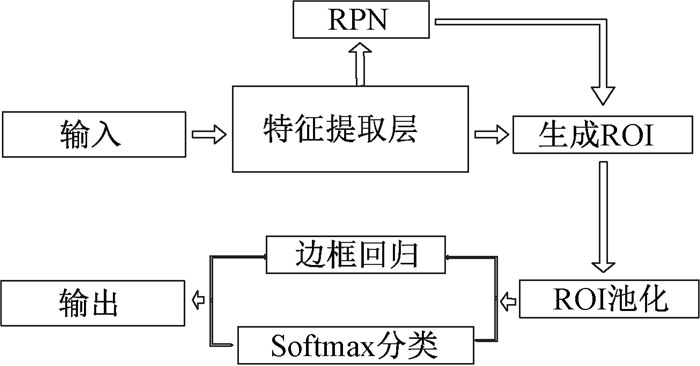
Faster-RCNN是一种基于候选框的目标检测算法,其算法结构如图 1所示,包括特征提取网络、候选框生成网络(region proposal network,RPN)、感兴趣区域(region of interest,ROI)生成、ROI池化层、Softmax分类器和边框回归几个主要步骤[11]。其特点是检测准确性较高,但计算复杂度较大。
|
图 1 Faster-RCNN算法结构 Fig. 1 The structure of faster-RCNN |
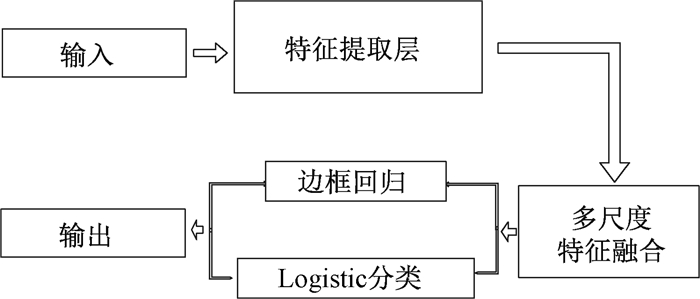
YOLOv3是一种基于回归的目标检测算法,其算法结构如图 2所示,包括特征提取网络、多尺度融合网络、Logistic分类和边框回归几个步骤[12]。其特点是将目标预测和边缘框回归整合到一个卷积神经网络,可以大幅降低检测时间。
|
图 2 YOLOv3算法结构 Fig. 2 The structure of YOLOv3 |
本文利用两种方法进行基于乳腺X线摄影图像的乳腺肿块检测对比实验。实验结果表明,faster-RCNN和YOLOv3算法都呈现出较高的乳腺肿块检测敏感性,但具有特异性偏低,检测假阳性率偏高的共性问题。因此,本文开展双视图信息融合的乳腺肿块检测算法,以期设计高正确率的深度学习乳腺肿块算法。
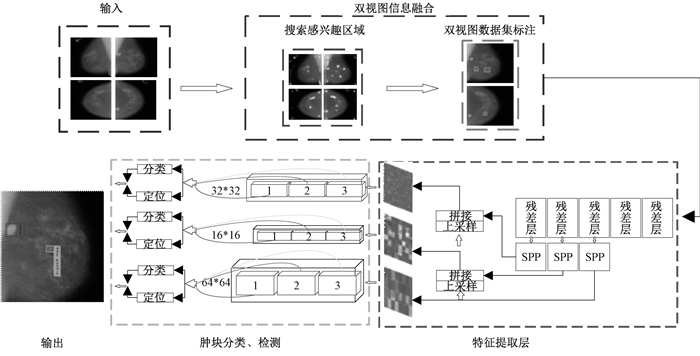
2 本文方法为了提高乳腺肿块检测敏感性并降低假阳性率,本文在YOLOv3算法框架基础上提出一种双视图信息融合的乳腺肿块自动检测方法。其算法结构如图 3所示,主要思路是分别在数据层和特征层进行双视图信息融合,并利用空间金字塔池化模块融合局部区域特征提升检测敏感性,设计类别损失函数,调节算法学习过程以提高检测特异性。
|
图 3 设计的网络结构 Fig. 3 Designed network architecture |
放射科医生通过在双视图上寻找对比白色高灰度值的感兴趣区域,以便精确定位肿块位置[3]。本节模拟医生的双视图阅片方法,利用双视图信息融合提高标注数据质量,包括建模搜索疑似肿块感兴趣区域和筛选并标注假阳性区域两个步骤。
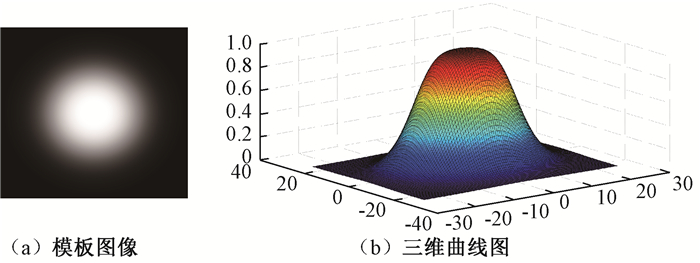
2.1.1 搜索感兴趣区域模板匹配作为一种基于区域分析的目标检测技术被广泛用于乳腺肿块检测[13]。由于多数乳腺肿块形状呈现出圆形或类圆形,且图像灰度值在感兴趣区域中心点较高、随着离中心点的距离增大而降低[3]。因此,本文建立如图 4(a)所示的双曲正割模板(Sech),计算公式为
| $ Sech\left( {x, y} \right) = \left( {2\alpha } \right)/({{\rm{e}}^{-\beta ({x^2} + {y^2})}} + {{\rm{e}}^{\beta ({x^2} + {y^2})}}), $ |
|
图 4 双曲正割模板 Fig. 4 Sech template |
式中:α=1、β=0.8为预设经验值;(x, y)表示模板上的像素点;Sech(x, y)表示该像素点灰度值。
Sech模板上的灰度值随像素点(x, y)的变化趋势如图 4(b)表示,可以较好地模拟乳腺X线摄影图像中乳腺类圆形肿块的灰度变化特征。利用归一化互相关函数逐像素计算模板图像与目标图像的相似度。图 5(a)、5(b)分别表示目标图像和生成的相似度图像。在图 5(b)上利用全局阈值取相似度系数较高的目标,获得如图 5(c)所示的疑似肿块感兴趣区域,图 5(d)展示疑似肿块感兴趣区域在原图上的位置。

|
图 5 模板匹配 Fig. 5 Template matching |
基于双曲正割模拟匹配法可以获得大量疑似肿块感兴趣区域,由于医生通常先在单视图上寻找确定如图 5(d)所示的疑似肿块感兴趣区域,然后再对比观察双视图信息及边缘信息,判断是否是真正的肿块病变。因此,本文通过在双视图上建立匹配模型,模拟医生去除假阳性区域的过程,在疑似肿块感兴趣区域中挑选出假阳性区域。
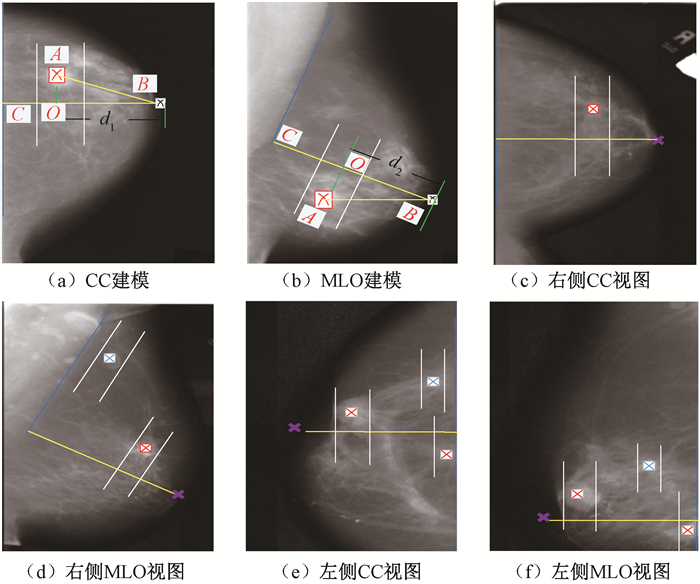
2.1.2 筛选假阳性区域匹配模型的建立过程如图 6(a)、6(b)所示:根据最小二乘直线拟合的方法拟合胸壁线,确定乳头位置B;过B作胸壁线的垂线交于点C,过ROI中心点A作BC的垂线交于点O,根据BO距离d1、d2确定ROI在双视图上的相对位置。考虑到成像时乳腺组织挤压产生的误差,设立一个以A为中心,宽度为W的条形匹配带,这里在DDSM数据库随机挑选300对头尾位、内外侧斜位视图乳腺图像,统计计算得到条形匹配带宽为83个像素,计算公式为w=2×[avg(|d1-d2|)+3×std(|d1-d2|)]。
|
图 6 筛选假阳性 Fig. 6 Selecting for false positives |
然后,根据放射科医生阅片标准标注假阳性:方法之一是将同侧不同视图对应条形匹配带内单独存在的类圆形区域标注为假阳性;方法之二是将异侧相同视图对应匹配带内同时存在的类圆形区域标注为假阳性[3]。根据DDSM公开数据库[14]提供的BI-RADS(breast image reporting and data system)标准标注肿块。标注结果如图 6(c)~6(f)所示,图中红色标记为肿块,蓝色标记为假阳性区域。
本节利用双曲正割模板和双视图匹配模型定位与肿块特征高度相似的假阳性区域,并通过增加标注先验信息,提高标注数据质量,为深度学习算法提供更多可辨识信息,以提升算法对肿块及假阳性的分辨能力。
2.2 多尺度局部特征融合YOLOv3算法框架的特征提取网络由5个残差模块组成,可以获得检测目标的多尺度全局特征。图 7展示了一个特征图可视化结果。观察图 7可以发现,图 7(a)所示浅层特征对乳腺组织几何细节信息表达能力强,适合检测微小肿块,图 7(b)所示深层特征语义信息表达能力强,适合区分肿块和假阳性区域。YOLOv3算法可以较好地融合多尺度全局特征,但其同一卷积层不同卷积核的特征输出之间缺乏关联。图 7(c)及图 7(d)分别表示同一卷积层多个卷积核特征输出,不同卷积核计算得到的特征图可以揭示图像不同视角的局部区域特征,对这些特征进行有效融合有助于提升模型细节特征表达能力。为此,本文提出分别在3个预测分支之前添加空间金字塔池化模块(spatial pyramid pooling, SPP)进行特征层的局部特征融合。

|
图 7 特征图可视化 Fig. 7 Feature map visualization |
如图 8所示,SPP模块由3个并行最大值池化分支构成,图 8(a)表示256×256×64维的输入特征图,图 8(b)~8(d)分别表示使用尺寸为5×5、9×9、13×13,步长为1的卷积核进行最大值池化得到的输出特征图,最后通过拼接操作得到如图 8(e)所示的256×256×256维特征图。对比图 8可以发现,尺寸不同的池化关注图像不同区域的特征,输出特征图经过多尺度和多维度的特征融合,能够为下一步的目标预测提供更丰富的局部特征信息,从而可以提高算法对肿块检测的敏感性。

|
图 8 SPP模块 Fig. 8 SPP block |
双视图数据集以肿块和假阳性为先验类别信息,有针对地设计类别损失函数以提升算法分类准确性。YOLOv3在预测阶段生成大量预测框,利用交叉熵损失函数衡量预测框中样本与真实样本之间的差异,通过最小化差异值来完成对模型的训练,交叉熵损失函数为:
| $ \begin{array}{c} CE({P_t}) = - {\rm{log}}({P_t});\\ {P_{\rm{t}}} = \left\{ {\begin{array}{*{20}{c}} {{P_i}, }&{{\rm{if}}\;{y_i} = 1, }\\ {1 - {P_i}, }&{{\rm{otherwise}},} \end{array}} \right. \end{array} $ |
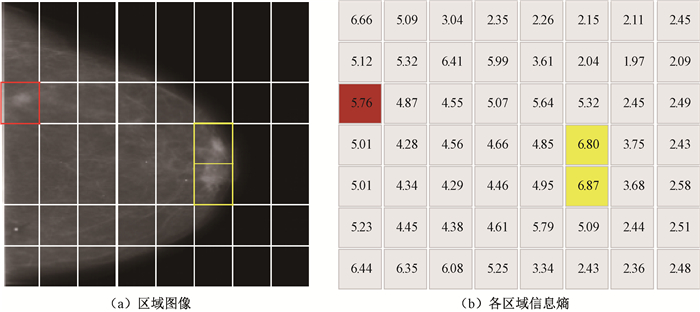
式中:Pi∈[0, 1]表示样本预测的类别概率;yi={+1, -1}表示肿块、正常区域类别信息。对图 9(a)所示的乳腺图像分区域分析发现,肿块和假阳性占比小,预测阶段肿块和假阳性所提供的有效梯度被大量负样本淹没,降低分类精度。进一步对各个子区域计算信息熵,图 9(b)中肿块和假阳性所在的红色、黄色区域信息熵较大,分类难度大,交叉熵损失函数难以捕获肿块与假阳性间的微小差异。
|
图 9 区域信息熵 Fig. 9 Regional information entropy |
针对上述问题,本文利用聚焦函数作为算法的分类损失函数,表达式为
| $ FL = \alpha {(1 - {P_t})^\gamma } \times CE({P_t})。$ |
当训练正常区域样本数量较多时,通过设置平衡权重α=0.5,降低正常区域样本所占权重,改善类别不均衡问题[15]。聚焦权重γ=2,当一个样本易分类时,Pt→1,调制系数(1-Pt)γ接近0,降低易分类样本损失值在总损失值中所占比例;当一个样本不易分类时,调制系数(1-Pt)γ接近1,增加难分类样本在总损失值所占比例,加强分类器对肿块和假阳性类别信息的学习,提高模型对肿块和假阳性的分辨能力。
3 实验与分析实验条件为:操作系统为Ubuntu16.04,深度学习框架为Darknet,CPU为Inter Xeon,GPU为NVIDIA Tesla K80。本文采用DDSM和MIAS公开数据集。从DDSM数据集中随机挑选2 276张乳腺X线摄影图像进行实验,训练集共有2 048张含有肿块的图像,测试集共有218张图像,其中含肿块图像151幅,正常图像67幅。为测试本文方法鲁棒性,使用MISA数据集测试训练好的模型。MIAS测试集包含85幅乳腺X线摄影图像,其中含肿块图像50幅,正常图像35幅。
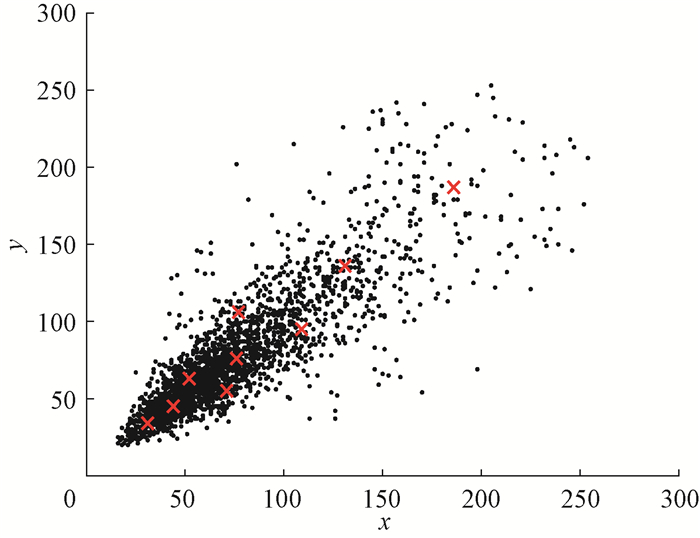
3.1 预设学习参数在模型训练过程中,通过学习真实框与先验框之间的尺寸偏移量,不断调整预测框的尺寸及位置,可以加速模型收敛并提高目标位置检测精度[16]。YOLOv3算法以COCO数据集为检测对象的9组预设先验框,其尺寸难以适应乳腺数据集的特点,为获得更准确的预设先验框,本文利用K-means算法在乳腺肿块数据集上聚类产生9种尺寸的先验框。如图 10所示,黑色点表示真实框的尺寸,红色点表示聚类产生的9种先验框,横、纵坐标分别表示真实框的长、宽。输入图像尺寸设置为512×512 pixel,迭代次数设置为40 000次,其中冲量系数与衰减系数分别配置为0.9与0.000 5,批次设置为4,初始学习率为0.001,学习率分别在迭代次数为20 000和25 000减小到之前的0.1倍。
|
图 10 先验框聚类 Fig. 10 Priors anchor clustering |
本文实验使用敏感性和特异性分别评价漏诊率和误诊率,使用平均每幅图像假阳性个数(FP/I)表示平均每幅图像过检肿块个数,使用P-R(precision recall)曲线综合评价模型性能。计算方法如表 1所示,其中:TP表示模型检测结果正确的肿块数量;FP表示模型误将正常区域检测为肿块数量;TN表示模型检测为正常区域的数量;FN表示模型检测正常区域,实际为肿块的数量。
|
|
表 1 评价方法 Tab. 1 Evaluation method |
本文分别在DDSM数据集上验证方法的有效性,在MISA数据集上验证鲁棒性,并与当前相关工作的文献方法进行对比。
不同方法在DDSM数据集上的测试结果如表 2所示。faster-RCNN模型敏感性达到91.4%,特异性达到55.2%,平均每幅图像假阳性0.15个。YOLOv3模型敏感性达到87.1%,特异性达到63.8%,平均每幅图像假阳性0.133个。两种模型具有相同特点:敏感性高,特异性低,假阳性率偏高。为验证双视图信息融合的有效性,本文以双视图标注数据作为YOLOv3模型训练集。实验结果表明,与原YOLOv3模型相比敏感性提升3.7%,特异性提升10.9%,平均每幅图像假阳性减少0.037个。本文方法在双视图的基础上利用空间金字塔池化模块和聚焦损失函数进一步提高模型性能,最终敏感性达到92.0%,特异性达到87.7%,平均每幅图像假阳性0.041个。与原YOLOv3模型相比敏感性提高4.9%,特异性提高23.9%,平均每幅图像假阳性降低0.092个。
|
|
表 2 不同算法在DDSM数据集上对比 Tab. 2 Comparison of different algorithms in DDSM |
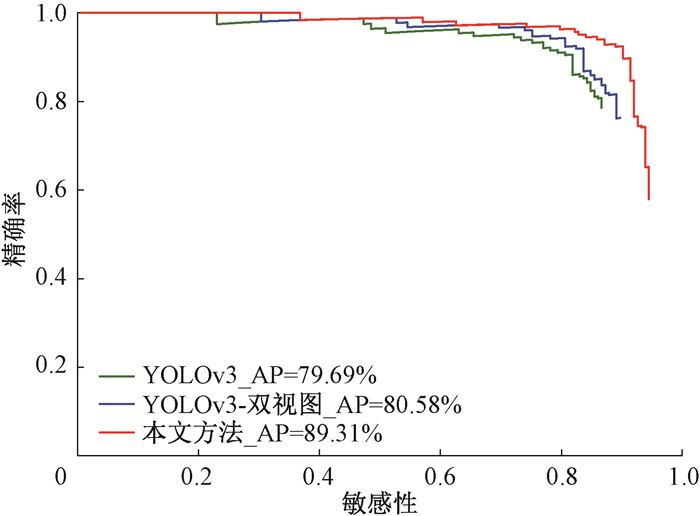
本文利用图 11所示的P-R曲线综合评价检测模型。图 11的横坐标和纵坐标分别表示检测敏感性和精确率,P-R曲线与坐标轴所围面积,表示乳腺肿块检测的平均精度。基于双视图信息融合的YOLOv3模型平均精度值达到80.58%,比原YOLOv3模型平均精度值提高0.89%。本文方法平均精度值达到89.31%,比YOLOv3模型平均精度值提高9.62%,模型检测性能显著提升。
|
图 11 P-R曲线 Fig. 11 Precision-recall curve |
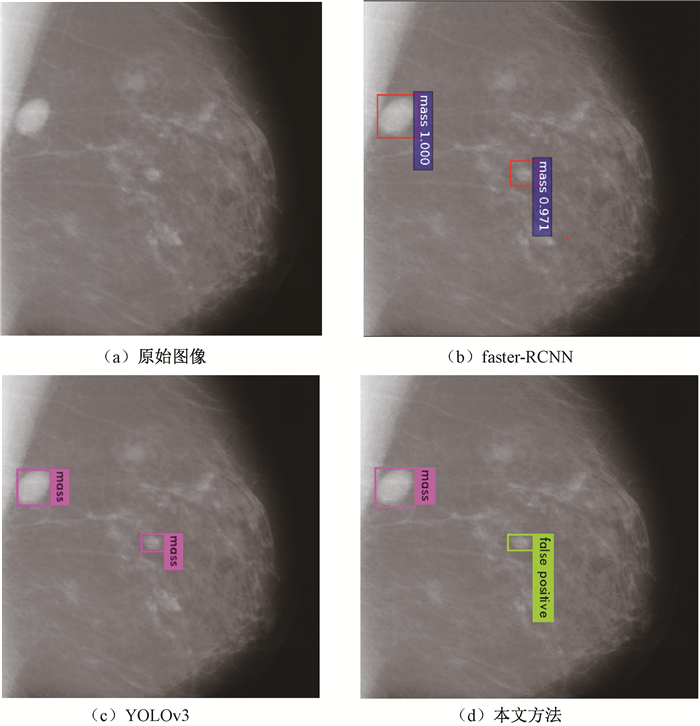
检测结果如图 12所示,图 12(a)表示原始图像,图 12(b)、12(c)分别表示faster-RCNN、YOLOv3算法检测结果,观察发现二者易将与肿块特征相似的正常组织误判为肿块。本文方法能更准确识别如图 12(d)所示的假阳性区域,降低了假阳性率。
|
图 12 结果可视化对比 Fig. 12 Visual comparision of results |
使用MISA数据集测试训练模型,验证其鲁棒性结果如表 3所示。本文提出的方法比YOLOv3敏感性提高10.5%,特异性提高16.4%,FP/I下降0.094。与在DDSM数据集上的验证结果具有一致的趋势,本文算法的各项性能指标均有明显提升,具有较好的鲁棒性。
|
|
表 3 不同算法在MISA数据集上对比 Tab. 3 Comparison of different algorithms in MISA |
将本文方法与当前主流的基于深度学习的乳腺肿块检测算法进行对比,结果如表 4所示。本文方法保证最高敏感性的同时,平均每幅图像假阳性个数最少。
|
|
表 4 基于深度学习的不同乳腺肿块检测方法对比 Tab. 4 Comparison with different methods of breast mass detection based on deep learning |
本文针对基于单视图的深度学习乳腺肿块检测算法假阳性率较高问题,提出了一种双视图信息融合的乳腺肿块检测方法。主要工作包括:1)提出双视图信息融合的方法,解决单视图肿块信息不完备的问题;2)设计金字塔池化模块,提取更丰富的乳腺图像局部特征;3)根据双视图数据集特点,设计聚焦损失函数,增强算法对肿块的分类能力。实验结果表明,本文算法与原始YOLOv3算法相比敏感性提高4.9%,特异性提高23.9%,平均每幅图像假阳性个数下降69.2%,各项性能指标均优于文献法,并在不同数据集上表现出良好的鲁棒性。下一步研究中,将利用医院采集的实际乳腺X线摄影图像,探索更适合临床应用的乳腺肿块自动检测方法。
| [1] |
HILLMAN B J, GOLDSMITH J C. The uncritical use of high-tech medical imaging[J]. New England journal of medicine, 2010, 363(1): 4-6. DOI:10.1056/NEJMp1003173 (  0) 0) |
| [2] |
LI Y F, CHEN H J, YANG Y Y, et al. Pectoral muscle segmentation in mammograms based on homogenous texture and intensity deviation[J]. Pattern recognition, 2013, 46(3): 681-691. DOI:10.1016/j.patcog.2012.09.021 (  0) 0) |
| [3] |
中国抗癌协会乳腺癌专业委员会. 中国抗癌协会乳腺癌诊治指南与规范(2019年版)[J]. 中国癌症杂志, 2019, 29(8): 609-679. CACA-CBCS. Chinese anti-cancer association:breast cancer diagnosis and treatment guidelines and norms[J]. China oncology, 2019, 29(8): 609-679. (  0) 0) |
| [4] |
CHU J H, MIN H, LIU L, et al. A novel computer aided breast mass detection scheme based on morphological enhancement and SLIC superpixel segmentation[J]. Medical physics, 2015, 42(7): 3859-3869. DOI:10.1118/1.4921612 (  0) 0) |
| [5] |
SPANHOL F A, OLIVEIRA L S, CAVALIN P R, et al. Deep features for breast cancer histopathological image classification[C]//IEEE International Conference on Systems, Man, and Cybernetics. Banff, 2017: 1868-1873.
(  0) 0) |
| [6] |
DHUNGEL N, CARNEIRO G, BRADLEY A P. The automated learning of deep features for breast mass classification from mammograms[M]. Cham: Springer International Publishing, 2016: 106-114.
(  0) 0) |
| [7] |
DHUNGEL N, CARNEIRO G, BRADLEY A P. Automated mass detection in mammograms using cascaded deep learning and random forests[C]//International Conference on Digital Image Computing: Techniques and Applications. Adelaide, 2015: 1-8.
(  0) 0) |
| [8] |
AKSELROD-BALLIN A, KARLINSKY L, HAZAN A, et al. Deep learning for automatic detection of abnormal findings in breast mammography[M]. Cham: Springer International Publishing, 2017: 321-329.
(  0) 0) |
| [9] |
AL-MASNI M A, AL-ANTARI M A, PARK J M, et al. Simultaneous detection and classification of breast masses in digital mammograms via a deep learning YOLO-based CAD system[J]. Computer methods and programs in biomedicine, 2018, 157: 85-94. DOI:10.1016/j.cmpb.2018.01.017 (  0) 0) |
| [10] |
于进勇, 丁鹏程, 王超. 卷积神经网络在目标检测中的应用综述[J]. 计算机科学, 2018, 45(A2): 17-26. YU J Y, DING P C, WANG C. Overview:application of convolution neural network in object detection[J]. Computer science, 2018, 45(A2): 17-26. (  0) 0) |
| [11] |
REN S Q, HE K M, GIRSHICK R, et al. Faster R-CNN: towards real-time object detection with region proposal networks[J]. IEEE transactions on pattern analysis and machine intelligence, 2017, 39(6): 1137-1149. DOI:10.1109/TPAMI.2016.2577031 (  0) 0) |
| [12] |
REDMON J, FARHADI A. YOLOv3: an incremental improvement[C]//Proceedings of IEEE Conference on Computer Vision and Pattern Recognition. Washington, 2018: 1-6.
(  0) 0) |
| [13] |
王俊茜, 徐勇, 孙利雷, 等.基于乳腺X线摄影的肿块检测综述[J/OL].自动化学报(2019-05-22)[2020-06-17].https://doi.org/10.16383/j.aas.c180662. WANG J Q, XU Y, SUN L L, et al. Survey of mass detection based on mammography[J/OL]. Acta automatica sinica (2019-05-22)[2020-06-17]. https://doi.org/10.16383/j.aas.c180662. (  0) 0) |
| [14] |
HEATH M, BOWYER K, KOPANS D, et al. Current status of the digital database for screening mammography[J]. Digital mammography, 1998, 457-460. (  0) 0) |
| [15] |
LIN T Y, GOYAL P, GIRSHICK R, et al. Focal loss for dense object detection[J]. IEEE transactions on pattern analysis and machine intelligence, 2020, 42(2): 318-327. DOI:10.1109/TPAMI.2018.2858826 (  0) 0) |
| [16] |
REDMON J, FARHADI A. YOLO9000: better, faster, stronger[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu, 2017: 6517-6525.
(  0) 0) |
| [17] |
LI Y F, ZHANG L L, CHEN H J, et al. Mass detection in mammograms by bilateral analysis using convolution neural network[J]. Computer methods and programs in biomedicine, 2020, 195: 105518. DOI:10.1016/j.cmpb.2020.105518 (  0) 0) |
| [18] |
BANDEIRA DINIZ J O, BANDEIRA DINIZ P H, AZEVEDO VALENTE T L, et al. Detection of mass regions in mammograms by bilateral analysis adapted to breast density using similarity indexes and convolutional neural networks[J]. Computer methods and programs in biomedicine, 2018, 156: 191-207. DOI:10.1016/j.cmpb.2018.01.007 (  0) 0) |
 2020, Vol. 52
2020, Vol. 52


