2. 中国科学院昆明动物研究所 遗传资源与进化国家重点实验室 云南 昆明 650223;
3. 中国科学院大学 昆明生命科学学院 云南 昆明 650500
2. State Key Laboratory of Genetic Resources and Evolution, Kunming Institute of Zoology, Chinese Academy of Sciences, Kunming 650223, China;
3. Kunming College of Life Science, University of Chinese Academy of Sciences, Kunming 650500, China
乳腺癌是我国妇女最常见的癌症,我国新发乳腺癌的病例占全球乳腺癌新发病例的12.2%[1].作为目前最大的癌症基因信息的数据库,TCGA收录了包括29种原位肿瘤类型,涉及基因组、转录组、蛋白质组及表观遗传等多层次的肿瘤数据,围绕TCGA数据库的研究提高了人们对肿瘤发病分子基础的科学认识,同时提高我们诊断、治疗和预防肿瘤的能力.这里我们结合临床病理特征、分子分型、生存分析、基因富集分析(GSEA)等生物信息手段,筛选影响乳腺癌进程的重要候选基因.在有丝分裂期间,染色体分离的保真度取决于对姐妹染色单体分离的精确控制,姐妹染色单体内聚的维持可以确保姐妹染色体正确分离[2-3].而有丝分裂的异常与肿瘤的发生存在密切关系[4],SGOL1可以维持着丝粒凝聚素从前期直至后期转变[5],是染色体稳定的守护者[6].已有研究发现在胰腺癌中SGOL1呈现过度表达[7-8].而在结肠癌中敲降SGOL1可以引起细胞周期阻滞在G2/M期,导致细胞凋亡以及造成染色体不稳定[3, 9].在肝癌细胞中SGOL1的异常表达可以导致有丝分裂延迟,最终导致细胞死亡[10].SGOL1是有丝分裂中必不可少的蛋白质,本文通过对TCGA数据库中乳腺癌数据集的分析,确定了7个与乳腺癌进程密切相关的基因.确定SGOL1作为实验功能验证的候选基因,进一步的实验结果显示SGOL1在癌组织中高表达,在MBA-MB231中敲降SGOL1能显著抑制细胞生长,细胞周期被阻滞在G2/M期,上述结果表明SGOL1能在乳腺癌进程中有重要作用.
1 材料与方法 1.1 TCGA数据下载登陆firehose(http://gdac.broadinstitute.org/)网站下载公开的乳腺癌(BRCA)转录组数据(RNASeqV2:https://wiki.nci.nih.gov/display/TCGA/RNASeq+Version+2)和肿瘤样本临床数据(数据版本:2016_01_28).其中:癌症样本1 093例;正常癌旁组织样本112例.
1.2 乳腺癌样本分子分型利用乳腺癌转录组数据集进行分子分型.首先通过Limma软件包的lowess-fit方法对基因的原始表达量进行归一化处理[11-12].然后使用Genefu软件包对乳腺癌样本进行PAM50分子分型[13-14].
1.3 基因表达与临床病理学参数的相关性分析使用survivalROC法将样本分为两组,基因表达边界值为3.255 207,低于该值为低表达组,高于该值为高表达组,结合TCGA的临床病理学参数(TNM分期)和乳腺癌分子分型结果,使用χ2检验检测基因表达与临床病理学参数的相关性.
1.4 生存曲线分析使用survivalROC法将样本分为两组,基因表达边界值为3.255 207,低于该值为低表达组,高于该值为高表达组,使用R语言survival软件包绘制总体生存曲线.
1.5 GSEA基因富集分析乳腺癌样本根据基因表达水平的高低分为两组.数据处理使用默认参数.利用FDR<0.25,P<0.01作为标准鉴定显著富集信号通路.
1.6 基因表达差异分析使用“RNASeqV2”数据中RSEM输出的reads count评估基因表达水平,采用Limma软件包中voom法[15]进行均一化处理,并用贝叶斯模型进行表达差异分析,筛选log FC>2的基因为表达差异基因.
1.7 细胞来源、主要试剂及仪器MDA-MB-231细胞购自中科院昆明动物所细胞库,siRNA购自广州瑞博公司,DMEM/F12培养基、胎牛血清(Gibco公司),Trizol总RNA提取试剂盒(Invitrogen公司),脂质体转染试剂盒Lipfectamine 2000,PrimeScriptTM RT Reagent反转录试剂盒(TaKaRa),Annexin V-FITC细胞凋亡检测试剂盒、FACSAria流式细胞仪(BD公司).
1.8 细胞培养及siRNA转染细胞使用DMEM/F12完全培养基(DMEM/F12+10%胎牛血清+100-U/ml青霉素/链霉素),在37 ℃,5%CO2培养箱中培养.待细胞贴壁生长,取对数生长期细胞,利用脂质体转染试剂盒Lipfectamine 2000进行转染处理.
1.9 RT-PCR采用Trizol法提取总RNA,使用PrimeScriptTM RT Reagent反转录试剂盒(TaKaRa)合成cDNA,PCR反应条件为:预变性94 ℃ 10 s;变性94 ℃ 5 s;退火/延伸60 ℃ 34 s;共40个循环;以β-Actin作为相对定量内参, 采用2-ΔΔCT法计算基因相对表达量[28].
1.10 细胞生长实验采用不同siRNA转染MDA-MB-231细胞,转染24 h后用胰蛋白酶将细胞消化并计数,按每孔2×105个细胞接种于6孔板,置于37 ℃培养箱培养.分别在细胞培养36 h、60 h、84 h时采用胰蛋白酶消化细胞并计数.
1.11 凋亡实验MDA-MB-231用不同siRNA处理72 h后,胰酶消化细胞,并用PBS洗两次,用染色缓冲液重悬细胞至每毫升1×106个细胞.取100 μl细胞(1×105),加入5 μl AnnexinV-FITC和5 μl PI室温下避光孵育15 min左右后,用流式细胞仪检测细胞凋亡.
1.12 周期实验MDA-MB-231用不同siRNA处理72 h后,采用无血清培养基饥饿处理24 h,之后采用完全培养基继续培养12 h,胰酶消化并收集细胞.加入1 ml预冷的70%乙醇中,吹打使细胞分散成单个细胞,4 ℃固定2 h.1 000 g离心3~5分钟,沉淀细胞.弃上清,用1 ml预冷PBS洗1遍.PI染色,每个样品用0.5 ml染色缓冲液重悬,加入25 μl PI染色液(20×)、10 μl RNase A(50×).37 ℃避光孵育30 min后,采用流式细胞仪检测细胞周期.
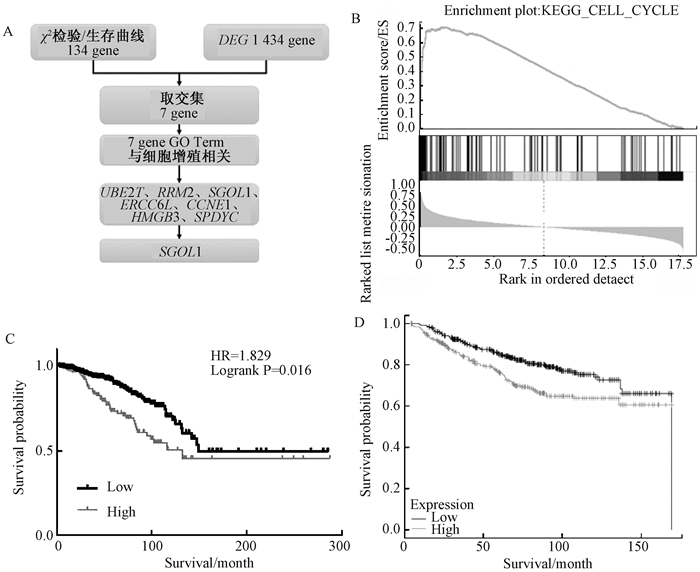
2 结果 2.1 筛选影响乳腺癌进程的候选基因为了筛选乳腺癌相关候选基因,我们第一步检测基因表达与乳腺癌亚型的相关性.目前基于乳腺癌分型的方法有两种:一种是依据临床数据中提供的相关免疫组织化学染色结果推断乳腺癌亚型;另一种是通过mRNA表达数据推断乳腺癌亚型.这里使用后者即PAM50分子分型的方法[13].结果将乳腺癌的样本分为Basal(197例)、Her2(362例)、Normal-like(323例)、LumA(110例)及LumB(101例)5类乳腺癌亚型.接着将样本根据单个基因的表达量,区别为高表达组和低表达组,通过χ2检验分析基因表达高低与乳腺癌亚型相关性,共计找出3 400个基因与乳腺癌亚型相关(P<=10-11),进一步对这些基因进行生存分析,发现其中134个基因与患者生存期存在显著相关(P<=10-3).为了进一步缩小范围,仅考虑在癌症相对于正常组织表达差异显著的基因.通过对TCGA表达差异分析共得到1 434个表达差异基因(log FC>2).最后结合在生存分析中显著的基因与表达差异分析中显著的基因取交集,共获得13个候选基因.通过基因功能注释(http://string-db.org)发现有7个基因与细胞周期、细胞增殖等过程相关.它们是UBE2T、RRM2、SGOL1、ERCC6L、CCNE1、HMGB3、SPDYC(图 1A).文献[17]提出UBE2在乳腺癌中高表达,通过抑制BRCA1的表达,促进肿瘤发展.RRM2在恶性肿瘤中呈现高表达状态,在非浸润型乳腺癌中,高表达能作为tamoxifen耐药性的标记[18].ERCC6L被发现在乳腺癌中高表达,且与恶性癌症密切相关,敲降ERCC6L能够导致细胞周期阻滞[19].CCNE1在HER2+乳腺癌中高表达[20].HMGB3在胃癌中高表达,敲降该基因能够有效降低细胞增殖和迁移[21].SPDYC并没有在癌症中有所报道,已有研究表明该基因与细胞周期调控相关[22].SGOL1在胰腺癌中高表达,在肺癌、结肠癌中低表达[7, 3],在乳腺癌中没有相关报道,通过GSEA分析发现SGOL1与细胞周期、有丝分裂等信号通路相关(图 1B).总体生存分析显示SGOL1高表达患者生存期显著缩短(图 1C),上述结果说明SGOL1可能是促癌基因.分析SGOL1的临床特征发现该基因与癌症大小(P=8.04e-06)、远端转移(P=2.84e-04)、癌症分级(P=3.84e-03)、癌症分子分型(P=2.20e-16)密切相关(表 1).因此选择SGOL1作为后续研究对象(表 1).
|
图 1 SGOL1与乳腺癌进程相关 Figure 1 SGOL1 is associated with breast cancer progression A表示TCGA数据库中筛选乳腺癌进程相关基因的流程;B表示GSEA分析显示SGOL1的差异表达与细胞周期过程相关;C、D:生存分析(左图数据来源于TCGA乳腺癌数据、右图数据来源于KM-plotter乳腺癌数据) |
|
|
表 1 SGOL1与病理学参数相关性 Table 1 Correlation between SGOL1 and pathological parameters |
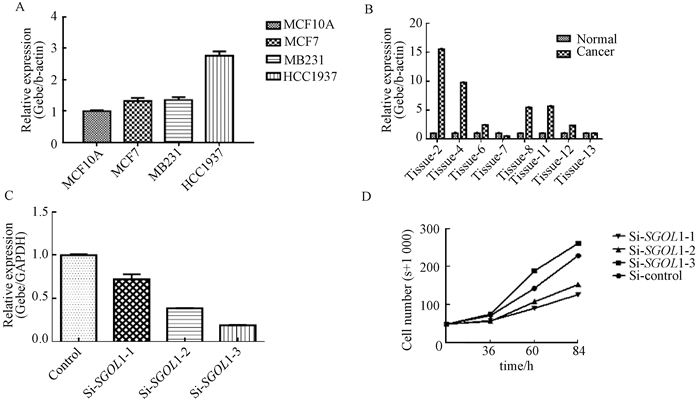
首先检测了SGOL1在人正常永生化的乳腺上皮细胞系(MCF10A)和人乳腺癌细胞系中的表达量,发现在SGOL1乳腺癌细胞系中表达偏高(图 2A).之后检测了癌组织以及癌旁正常组织中SGOL1的表达量,发现除了7号和13号样本中SGOL1的表达量略低于正常样本(图 2B),在其余样本中均发现SGOL1在癌组织中高表达.这些结果表明SGOL1在乳腺癌进程中很可能有重要作用.
|
图 2 SGOL1在乳腺癌中高表达 Figure 2 SGOL1 is highly expressed in breast cancer A表示qRT-PCR检测不同乳腺癌细胞系中SGOL1基因mRNA表达水平;B表示不同例乳腺癌组织与正常乳腺组织SGOL1基因mRNA相对表达量;C表示在MDA-MB-231乳腺癌细胞系中检测其敲降效率;D表示不同时间点检测敲降SGOL1基因对MDA-MB-231细胞系生长的影响 |
为了检验SGOL1与乳腺癌的关系,我们通过siRNA来敲降SGOL1(图 2C),设计了3对siRNA,结果显示si-SGOL1-2、si-SGOL1-3都有很好的敲降效果.
2.4 敲降SGOL1抑制细胞生长采用不同siRNA处理MDA-MB-231细胞后,我们发现与si-Control组相比较,除了si-SGOL1-1(无明显敲降效果),si-SGOL1-2和si-SGOL1-3后,细胞数目都明显低于si-Control组(图 2D).这些结果证明敲降SGOL1能够抑制MDA-MB-231细胞生长.
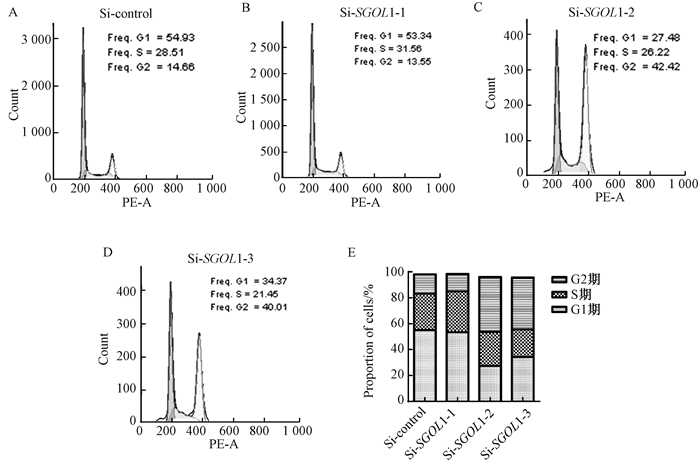
2.5 抑制G2/M细胞周期但不影响细胞凋亡.我们前期的实验结果发现敲降SGOL1能显著抑制细胞生长,因此我们开始探索敲降SGOL1后通过什么途径来影响细胞生长.敲降SGOL1后,首先检测了细胞周期的变化,实验结果发现,敲降SGOL1可使细胞G2/M期显着增加(图 3).
|
图 3 敲降SGOL1后用流式细胞仪检测细胞周期 Figure 3 Knock down SGOL1 and then use flow cytometry to detect cell cycle A表示对照组中细胞周期;B、C、D表示siRNA敲降SGOL1的细胞周期;E表示细胞周期的统计结果 |
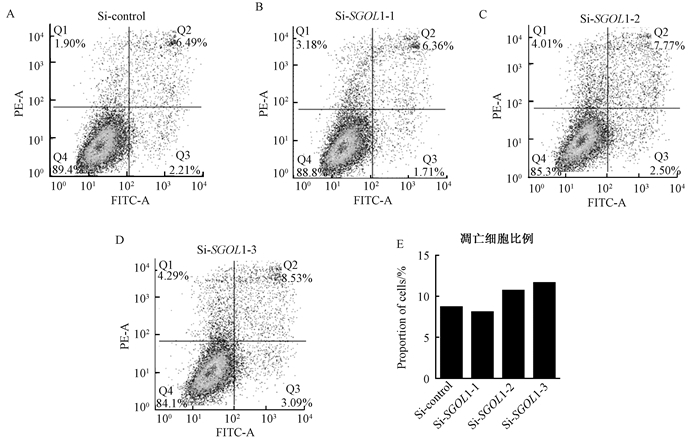
之后我们采用凋亡试剂盒来检测细胞凋亡,发现敲降SGOL1凋亡细胞虽然比control组略微有所上升(图 4),但并没有显著的差异.因此在乳腺癌中SGOL1很可能主要是通过影响细胞周期来影响细胞增殖的.
|
图 4 Annexin V/PI双染法检测敲降SGOL1对MDA-MB-231细胞凋亡的影响 Figure 4 Annexin V/PI double staining method to detect knockdown of SGOL1 on MDA-MB-231 cell apoptosis A表示对照组中细胞凋亡结果;B、C、D表示siRNA敲降SGOL1的细胞凋亡结果;E表示细胞凋亡的统计结果 |
由TCGA的临床数据统计可知SGOL1的表达与癌症大小、远端转移、癌症分级以及癌症分子分型密切相关.总体生存曲线显示该基因高表达患者相较于低表达患者生存期显著缩短.基因共表达网络分析显示,在乳腺癌中该基因所在的共表达模块可能与细胞周期相关,从而影响癌症的进程,GSEA分析可知SGOL1的表达差异与细胞周期和有丝分裂细胞通路相关.作为进化保守的蛋白质,SGOL1是有丝分裂过程中染色体准确分离所必需的.文献[23]表明哺乳动物细胞中敲降SGOL1导致染色体分离异常,引起遗传不稳定和肿瘤转化.文献[24]发现通过短肽竞争抑制SGOL1的功能能够促进癌症凋亡.结肠癌中该基因的下调使遗传不稳定进而影响癌症的发展过程,然而乳腺癌中SOGL1的表达水平却是上升的,该基因的高表达同样伴随大量的染色体不稳定现象[25-26].因此,SOGL1在乳腺癌症中控制染色体稳定性的分子机制可能不同于其他癌症类型.已有研究发现SGOL1对细胞凋亡和细胞周期都有影响[10],但是在乳腺癌中,我们发现SGOL1并不影响细胞的凋亡,但是能显著影响细胞周期.
乳腺癌是最常见的癌症之一,且近年来的发病人数逐年增加[27].近年来精准治疗作为癌症治疗的探索新方向,本项研究发现,乳腺癌中敲低SGOL1能够有效抑制癌细胞生长、致使细胞周期停滞在G2/M期,这能为癌症治疗提供了新的药物靶标.
| [1] |
FAN L, STRASSERWEIPPL K, LI J J, et al. Breast cancer in China[J]. Lancet oncology, 2014, 15(7): 279-289. DOI:10.1016/S1470-2045(13)70567-9 (  0) 0) |
| [2] |
WANG L H, MAYER B, STEMMANN O, et al. Centromere DNA decatenation depends on cohesin removal and is required for mammalian cell division[J]. Journal of cell science, 2010, 123(5): 806-813. DOI:10.1242/jcs.058255 (  0) 0) |
| [3] |
YAMADA H Y, YAO Y, WANG X, et al. Haploinsufficiency of SGO1 results in deregulated centrosome dynamics, enhanced chromosomal instability and colon tumorigenesis[J]. Cell cycle, 2012, 11(3): 479-488. DOI:10.4161/cc.11.3.18994 (  0) 0) |
| [4] |
FUNK L C, ZASADIL L M, WEAVER B A. Living in CIN: mitotic infidelity and its consequences for tumor promotion and suppression[J]. Developmental cell, 2016, 39(6): 638-652. DOI:10.1016/j.devcel.2016.10.023 (  0) 0) |
| [5] |
GIMENEZ-ABIAN J F, DIAZ-MARTINEZ L A, WIRTH K G, et al. Regulated separation of sister centromeres depends on the spindle assembly checkpoint but not on the anaphase promoting complex/cyclosome[J]. Cell cycle, 2005, 4(11): 1561-1575. DOI:10.4161/cc.4.11.2146 (  0) 0) |
| [6] |
DIAZ-MARTINEZ L A, YU H. Sgo1 as a guardian of chromosome stability[J]. Cell cycle, 2012, 11(4): 650-651. DOI:10.4161/cc.11.4.19361 (  0) 0) |
| [7] |
CUTTS R J, GADALETA E, HAHN S A, et al. The Pancreatic Expression database: 2011 update[J]. Nucleic acids research, 2011, 39: D1023-1028. DOI:10.1093/nar/gkq937 (  0) 0) |
| [8] |
ANDRE F, MICHIELS S, DESSEN P, et al. Exonic expression profiling of breast cancer and benign lesions: a retrospective analysis[J]. Lancet oncol, 2009, 10(4): 381-390. DOI:10.1016/S1470-2045(09)70024-5 (  0) 0) |
| [9] |
IWAIZUMI M, SHINMURA K, MORI H, et al. Human Sgo1 downregulation leads to chromosomal instability in colorectal cancer[J]. Gut, 2009, 58(2): 249-260. DOI:10.1136/gut.2008.149468 (  0) 0) |
| [10] |
WANG L H, YEN C J, LI T N, et al. Sgo1 is a potential therapeutic target for hepatocellular carcinoma[J]. Oncotarget, 2015, 6(4): 2023-2033. DOI:10.18632/oncotarget.v6i4 (  0) 0) |
| [11] |
KATZ Y, LI F, LAMBERT N J, et al. Musashi proteins are post-transcriptional regulators of the epithelial-luminal cell state[J]. Elife, 2014, 3: e03915. (  0) 0) |
| [12] |
RITCHIE M E, PHIPSON B, WU D, et al. Limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic acids research, 2015, 43(7): e47. DOI:10.1093/nar/gkv007 (  0) 0) |
| [13] |
FUMAGALLI D, BLANCHET-COHEN A, BROWN D, et al. Transfer of clinically relevant gene expression signatures in breast cancer: from Affymetrix microarray to Illumina RNA-sequencing technology[J]. BMC genomics, 2014, 15(1): 1008. DOI:10.1186/1471-2164-15-1008 (  0) 0) |
| [14] |
GENDOO D M, RATANASIRIGULCHAI N, SCHRODER M S, et al. Genefu: an R/Bioconductor package for computation of gene expression-based signatures in breast cancer[J]. Bioinformatics, 2016, 32(7): 1097-1099. DOI:10.1093/bioinformatics/btv693 (  0) 0) |
| [15] |
LAW C W, CHEN Y, SHI W, et al. Voom: precision weights unlock linear model analysis tools for RNA-seq read counts[J]. Genome biology, 2014, 15(2): R29. DOI:10.1186/gb-2014-15-2-r29 (  0) 0) |
| [16] |
HEAGERTY P J, LUMLEY T, PEPE M S. Time-dependent ROC curves for censored survival data and a diagnostic marker[J]. Biometrics, 2000, 56(2): 337-344. DOI:10.1111/j.0006-341X.2000.00337.x (  0) 0) |
| [17] |
UEKI T, PARK J H, NISHIDATE T, et al. Ubiquitination and downregulation of BRCA1 by ubiquitin-conjugating enzyme E2T overexpression in human breast cancer cells[J]. Cancer research, 2009, 69(22): 8752-8760. DOI:10.1158/0008-5472.CAN-09-1809 (  0) 0) |
| [18] |
PUTLURI N, MAITY S, KOMMAGANI R, et al. Pathway-centric integrative analysis identifies RRM2 as a prognostic marker in breast cancer associated with poor survival and tamoxifen resistance[J]. Neoplasia, 2014, 16(5): 390-402. DOI:10.1016/j.neo.2014.05.007 (  0) 0) |
| [19] |
PU S Y, YU Q, WU H, et al. ERCC6L, a DNA helicase, is involved in cell proliferation and associated with survival and progress in breast and kidney cancers[J]. Oncotarget, 2017, 8(26): 42116-42124. (  0) 0) |
| [20] |
LUHTALA S, STAFF S, TANNER M, et al. Cyclin E amplification, over-expression, and relapse-free survival in HER-2-positive primary breast cancer[J]. Tumour biology, 2016, 37(7): 9813-9823. DOI:10.1007/s13277-016-4870-z (  0) 0) |
| [21] |
GUO S, WANG Y, GAO Y, et al. Knockdown of high mobility group-box 3 (HMGB3) expression inhibits proliferation, reduces migration, and affects chemosensitivity in gastric cancer cells[J]. Medical science monitor international medical journal of experimental and clinical research, 2016, 22: 3951-3960. (  0) 0) |
| [22] |
CHENG A, SOLOMON M J. Speedy/Ringo C regulates S and G2 phase progression in human cells[J]. Cell cycle, 2008, 7(19): 3037-3047. DOI:10.4161/cc.7.19.6736 (  0) 0) |
| [23] |
WANG X, YANG Y, DAI W. Differential subcellular localizations of two human Sgo1 isoforms: implications in regulation of sister chromatid cohesion and microtubule dynamics[J]. Cell cycle, 2006, 5(6): 635-640. (  0) 0) |
| [24] |
YANG Y, WANG X, DAI W. Human Sgo1 is an excellent target for induction of apoptosis of transformed cells[J]. Cell cycle, 2006, 5(8): 896-901. DOI:10.4161/cc (  0) 0) |
| [25] |
TANNO Y, SUSUMU H, KAWAMURA M, et al. The inner centromere-shugoshin network prevents chromosomal instability[J]. Science, 2015, 349(6253): 1237-1240. DOI:10.1126/science.aaa2655 (  0) 0) |
| [26] |
RADFORD D M, FAIR K L, PHILLIPS N J, et al. Allelotyping of ductal carcinoma in situ of the breast: deletion of loci on 8p, 13q, 16q, 17p and 17q[J]. Cancer research, 1995, 55(15): 3399-3405. (  0) 0) |
| [27] |
ZUO T T, ZHENG R S, ZENG H M, et al. Female breast cancer incidence and mortality in China, 2013[J]. Thoracis cancer, 2017, 8(3): 214-218. DOI:10.1111/tca.2017.8.issue-3 (  0) 0) |
| [28] |
张芬. MicroRNA-124a对SCI继发性损伤的影响及相关机制研究[J]. 郑州大学学报(理学版), 2016, 48(1): 74-80. (  0) 0) |
 2017, Vol. 49
2017, Vol. 49


