磷是植物生长发育所必需的大量营养元素。虽然土壤中总磷的含量非常高,但大约 80% 的磷以有机态的形式存在,并且可溶性磷酸根离子极易被酸性土壤中的铁铝氧化物及石灰性土壤中的碳酸盐化合物吸附固定,而难以被植物吸收利用。因此,土壤中实际可溶性磷的浓度远远低于植物组织中的磷浓度[1]。植物 PHT1 磷转运蛋白家族是一类高亲和性的磷转运蛋白,它在植物磷吸收、转运、体内磷分配及再利用等过程中发挥了重要作用 [2–4]。近年来,随着研究的不断深入,全世界的研究人员相继发表了大量关于植物 PHT1 家族基因参与磷吸收转运过程及其调控途径等方面的研究报道,逐渐揭开了高等植物磷吸收转运及调控的奥秘。
1 植物 PHT1 家族基因的发现及特征从 20 世纪 90 年代开始,PHT1 家族基因逐渐被发现,它们是定位于细胞膜负责吸收和转运磷的蛋白。酿酒酵母 PHO84 是第一个被发现的高亲和磷转运子[5]。随后,研究人员以酵母 PHO84 的 EST (expressed sequence tag) 序列作为探针,首次在高等植物拟南芥中分离到 2 个磷转运子基因 AtPT1 和 AtPT2[6]。之后,几个研究团队几乎同时从拟南芥中分离出若干相同的高亲和磷转运子,分别命名为 PT 或 PHT。直到 2000 年,拟南芥全基因组测序工作完成,9 个 PHT1 家族成员被全部确定下来,并被统一命名为 PHT1;1-PHT1;9[7],但是在某些物种如大豆、番茄、小麦等中仍然沿用 PT 的命名方式[8–10]。截止目前,在高等植物中相继分离出大量的 PHT1 家族同源基因,物种包括拟南芥、水稻、玉米、大豆、大麦、小麦、番茄、土豆、辣椒、茄子、烟草、甘蓝型油菜、菊花等[2–4]。
已有研究表明,植物 PHT1 家族属于 MFS (major facilitator superfamily) 超家族的第九亚家族,不同物种 PHT1 家族蛋白的结构非常保守。一般情况,该家族蛋白的等电点为 7.6~9.2,分子量约为 57~61 kDa,含有 520~560 个氨基酸残基,通常具有 12 个亲脂跨膜结构域,主要由含 17~25 个氨基酸残基的 α 螺旋构成,在第六个与第七个跨膜区间普遍存在一个亲水环,约由 60 个氨基酸残基构成,最终形成“6 螺旋–亲水大环–6 螺旋”式的结构,其中 C-端、N-端和亲水大环都在质膜内部[11–15]。同时,该家族还具有 H2PO4–/nH+ 共运子、糖转运子和 MFS 通用转运子等的特征结构域和一段保守的氨基酸特征序列 GGDYPLSATIMSE[16–17]。
2 植物 PHT1 家族基因对磷的转运及亲和力研究表明,在多种类型的细胞中,无机磷的吸收是与质子共转运的过程,这一过程需要能量介导,并会导致膜外介质 pH 值的升高,而且磷吸收活性受质子载体效率的限制[18-19]。一般情况,转运1个磷酸根离子需要 2~4 个质子协同进入质膜,磷与质子共转运的同时会引起膜电位的变化,从而使硫酸根离子及少量的硝酸根离子和氯离子转运至膜内[20–22]。
PHT1 家族非常庞大,其成员对磷的转运特性存在较大差别。根据其动力学参数 Km 值,植物磷转运子可以分为高亲和力磷转运系统和低亲和力磷转运系统两类。但是对两种转运子的划分依据众说纷纭,究其原因在于对Km 值界限、转运子构象改变或个别氨基酸变异引起的 Km 值变化等界定不清晰。一般认为,高亲和 PHT1 基因受磷胁迫诱导,可将膜外低浓度磷转运到膜内,其 Km 值在几十毫摩尔级别;而低亲和 PHT1 基因是组成型表达,只有在磷充足供应时才将膜外的磷转运到膜内,其表达水平不受外界磷浓度影响,其 Km 值在几十毫摩尔到上百毫摩尔甚至更高级别[1, 23]。
目前对 PHT1 转运子的磷转运动力学研究主要利用酵母突变体 pho84/pho89 (酵母中主要的高亲和磷转运子) 进行酵母互补试验来测试。利用该技术检测的 PHT1 家族成员 Km 值变化较大,例如菊花 CmPT1 为 35.2 μmol/L[24],小麦 TaPHT1;4 为 35.3 μmol/L[25],大豆 GmPT1 和 GmPT2 分别为 6.65 和 6.63 mmol/L[13]。蒺藜状苜蓿 MtPT1、MtPT2、MtPT3 和 MtPT5 分别为 587 ± 60、641 ± 64、858 ± 91 和 13 ± 2 μmol/L [26],水稻 OsPHT1;8 为 21 μmol/L[27]。利用酵母互补试验测算植物 PHT1 转运子 Km 值有其弊端。该方法只能估算 PHT1 基因在酵母中 Km 的大致范围,而且不同研究得到的 Km 值可能存在较大差异,且与植物本体 PHT1 转运子 Km 值有明显差别[12]。因此,在酵母中的 Km 测值只能部分代表植物 PHT1 转运子的真实转运活力。为了更准确地测试各种植物 PHT1 转运子的转运效率,通常用几种方法验证同一 PHT1 基因的转运活性,如瓜蟾卵母细胞试验、植物悬浮细胞试验等[28]。
此外,PHT1 转运子序列变异可能会引起 Km 值的变化。例如,蒺藜苜蓿 MtPT5 与 MtPT1、MtPT2、MtPT3 有 84% 的氨基酸序列相似性,但表现出对磷酸盐不同的亲和性 (MtPT5 为高亲和性,MtPT1、MtPT2、MtPT3 为低亲和性),研究人员对这 4 个基因编码的氨基酸序列进行比较和建模发现,MtPT5 和其他 3 个基因编码的氨基酸序列存在 2 个片段的差异[26]。拟南芥 AtPHT1;1 转运子的 312 位络氨酸作为无机磷酸盐结合位点,将其突变为丙氨酸或者苯丙氨酸不会影响 PHT1;1 转运活力,而将其突变为天冬氨酸时,AtPHT1;1 编码蛋白的同源多聚体瓦解,由此可推测植物PHT1蛋白之间会通过形成复合物降低转运磷素效率进而调控体内磷平衡[29]。
3 植物 PHT1 家族基因的表达模式及功能高等植物 PHT1 家族成员众多,各成员间的时空表达模式有相似之处,但也存在较大差异。一般认为,PHT1 家族大多数成员受低磷信号调控且主要在根部表达。至今发现拟南芥除 PHT1;6 以外的 8 个成员至少在根中表达,其表达部位分布在主根皮层、中柱,根毛区表皮、皮层、中柱,侧根以及根冠等[3]。下面以拟南芥、水稻、大豆、茄科作物及其他高等植物为例简要综述各物种中 PHT1 家族成员的时空表达模式及功能。
3.1 拟南芥研究表明,拟南芥中的 PHT1 家族有 9 个成员[30],AtPHT1;1、AtPHT1;2 和 AtPHT1;3 对磷吸收的贡献相当高,而 AtPHT1;1 在长距离转运磷的过程中也发挥重要作用[31]。除 AtPHT1;6 主要在花器官中表达外,其余 8 个在根中表达的 PHT1 基因中,AtPHT1;1 和 AtPHT1;4 转录水平最高,且这 2 个基因都是高亲和转运子[3]。低磷培养下,PHT1;4 突变体对磷的吸收显著减少 40%,而在高磷培养下 AtPHT1;1/AtPHT1;4 双突变体对磷的吸收减少 75%,表明 AtPHT1;1 和 AtPHT1;4 无论在低磷还是高磷下对磷的吸收都起到重要作用[32–33]。AtPHT1;5 超表达会增加角果无机磷含量,提高一些负责清除多余磷相关基因的转录水平,而 AtPHT1;5 突变导致磷酸盐向地上部的分配减少。因此 AtPHT1;5 可能对无机磷从源到库的分配是至关重要的[34]。AtPHT1;8 和 AtPHT1;9 在根中受缺磷强烈诱导表达,研究表明这 2 个基因在长期低磷胁迫下发挥了重要作用[35]。通过对 AtPHT1;8 和 AtPHT1;9 突变体磷的吸收和积累量进行分析,发现它们都参与磷从根部向地上部运输。抑制AtPHT1;5 或 AtPHT1;9 的表达会引起其它磷响应基因转录水平的变化[34, 36]。
3.2 水稻研究表明,水稻 PHT1 家族成员共有 13 个[37]。在磷充足的条件下,OsPT1 超表达株系磷含量显著提高而 RNAi 干涉株系表型相反,研究表明高磷背景下 OsPT1 是吸收和转运磷的关键 PHT1 成员[38]。缺磷能显著诱导 OsPT2、OsPT4、OsPT8、OsPT9 和 OsPT10 等的表达,OsPT2 仅在主根和侧根的中柱中表达,而 OsPT6 在新生根的表皮细胞和皮层细胞中表达。Ai 等[39]研究表明 OsPT2 是低亲和磷转运子,主要负责转运和积累磷,而 OsPT6 对整个植株的磷吸收转运起到广泛作用。OsPT4 在根、叶、舌叶、雄蕊和颖果中都有表达,主要负责向根、茎和糙米中转运累积磷[40]。另有研究发现,OsPT4 在根和胚中的表达非常强,超表达或 knockout/knockdown OsPT4 会改变植株磷积累,同时影响胚的发育和种子的形成[41]。OsPT8 几乎在每个器官都有强烈表达,似乎是组成型转运子,但仍受缺磷诱导。在正常磷条件下,超表达 OsPT8 会使转基因植株表现出生长迟缓、植株矮小和叶片发黄等典型的磷中毒症状[27]。OsPT8 下调表达会导致老叶磷含量上升、新叶磷含量下降,在灌浆期胚乳和胚中的磷含量都急剧下降,表明该基因对磷素从源到库的再分配方面起到关键作用[42]。OsPT9 和 OsPT10 在根表皮、根毛及侧根中表达,无论在高磷还是低磷下,基因超表达体都会显著增强对磷的吸收[43]。
3.3 大豆研究人员通过对大豆的基因组数据库进行检索发现大豆有 14 个 PHT1 家族成员[44]。RT-PCR 分析表明大豆 GmPT1 和 GmPT2 等 2 个基因在幼苗根部和地上部表达,缺磷使 GmPT1 和 GmPT2 的表达发生轻微变化[13]。GmPT5 主要在根与新生成结瘤的连接处、幼嫩或成熟结瘤的丛枝中表达,意味着 GmPT5 可能负责将磷从根维管组织向结瘤中转运,尤其在缺磷胁迫下 GmPT5 转运活力更强,因此 GmPT5 可能是调控结瘤磷稳态的关键转运子[44]。GmPT7 在根部成熟的丛枝菌根 (arbuscular mycorrhiza,AM) 皮层细胞、根冠小柱细胞和无菌根组织的侧根原基细胞表达,在衰老叶片的维管束末端局部管胞细胞也有表达,主要负责把再活化的磷向种子转运[8]。研究表明,GmPT7、GmPT10 和 GmPT11 受 AM 诱导表达[45]。
3.4 茄科作物茄科作物 (Solanaceous species) 是仅次于禾本科和豆科作物的世界第三大类经济作物,共超过 200000 种茄科作物可与 AM 真菌形成共生菌根[46]。低磷会诱导番茄 LePT1 和 LePT2 两基因在根和叶中大量表达,在高磷条件下转入番茄 LePT1 或 LePT2 的水稻植株有效磷出现过量积累,导致植株矮小、分蘖数减少[47]。同时,也有研究表明 LePT1 和 LePT7 被检测到在所有组织中都有表达,低磷促使 LePT2 和 LePT6 主要在根中表达,而 LePT3、LePT4 和 LePT5 仅在低磷条件下受菌根菌强烈诱导表达,高磷条件下则不会被菌根诱导[9]。土豆与番茄 PHT1;1~PHT1;5 同源基因的表达模式相似:缺磷增强叶片和根部 PHT1;1 的表达,无磷特异诱导根部 PHT1;2 的表达,菌根能增强 PHT1;3 表达,并诱导 PHT1;4 和 PHT1;5 的表达[48]。PHT1 家族同源基因在辣椒、茄子和烟草中也具有相似的表达模式:PHT1;1 在根系中的表达量高于叶片,而 PHT1;2 只在根系中有表达;PHT1;1 和 PHT1;2 受缺磷信号诱导上调表达,但是接种菌根菌后 2 个基因的表达量均下降;在高磷供应下,被菌根菌侵染后植株中的 PHT1;2 在根系中表达增强;PHT1;3 被菌根诱导增强表达,PHT1;4 和 PHT1;5 为菌根特异性诱导表达[23, 49]。
3.5 其他高等植物小麦 TaPT2 受缺磷诱导表达,其表达水平与外界磷酸盐浓度密切相关且只在根中表达[10]。小麦 TaPHT1;4 同样在根中受低磷胁迫诱导表达,有趣的是 TaPHT1;4 白天表达量高,夜间表达量低[25]。大麦 HORvu;PHT1;1 只在根中表达且强烈受到缺磷诱导,而 HORvu;PHT1;6 在叶片和叶耳的维管束韧皮部表达[15]。HvPHT1;1 和 HvPHT1;2 的表达模式相似,受缺磷诱导,且都在次生根根毛的生毛细胞和节生根的中柱有高水平表达,叶片表达相对较低[50]。甘蓝型油菜 BnPHT1;4 主要在根部表达,在拟南芥中超表达 BnPHT1;4 能促进主根生长,抑制侧根生长并提高磷利用率[51]。Liu 等[24]研究发现菊花 CmPT1 受低磷诱导表达,在根、茎、叶中的表达量依次降低,超表达 CmPT1 使植物的株高、根体积、生物量、磷吸收量等增加。研究人员 2011 年首次对大叶杨的 12 个 PHT1 家族基因进行研究,发现低磷胁迫使大多数 PHT1 家族成员上调表达,尤其是 PHT1;9 和 PHT1;11,而 PHT1;10 仅在 AM 中强烈转录[52]。
4 植物 PHT1 家族基因的转录调控 4.1 介质无机磷浓度外界环境中的无机磷 (inorganic phosphate,Pi) 浓度是调控 PHT1 表达水平的重要因子之一,Pi 可以作为信号分子调控部分 PHT1 家族成员的转录水平 (详见本文第三部分)[3]。例如,Misson 等[33]利用 GUS 报告基因和原位杂交技术分析了不同无机磷水平下拟南芥磷转运子基因 Pht1;4 的转录水平,结果发现 AtPht1;4 在低磷浓度的介质中主要在根表皮、皮层和根冠中表达,该基因突变可以使植物磷吸收能力下降 40%。有意思的是,植物 PHT1 家族基因的表达水平不但受到介质磷浓度的调控,而且不同磷浓度对 PHT1 基因转录的调控程度不同。在外界无机磷浓度低于 10 μmol/L 的时候,基因的表达显著上升,而当外界无机磷浓度高于 250 μmol/L 的时候,基因的表达则维持在较低的水平,而且这种表达变化反应非常迅速[3, 33]。
4.2 启动子元件基因表达是由众多顺式作用元件和反式作用因子相互作用调控的。研究表明,在植物 PHT1 家族基因的启动子区存在一些特殊的元件,通过与其他基因相互作用调控 PHT1 家族基因的表达水平。分析发现在茄子和烟草 PHT1 家族同源基因的启动子区至少包括 MYCS (TTTCTTGTTCT) 和 P1BS (GNATATNC) 2 个顺式调控元件,负责激活菌根诱导 PHT1 转运子基因的转录,对 2 个元件的任意一个进行缺失或部分突变都会造成启动子活性显著减弱甚至全部丧失。因此,可以推测在双子叶植物中,丛枝菌根真菌 (arbuscular mycorrhizal fungi,AMF) 介导的无机磷吸收过程至少受 MYCS 和 P1BS 元件的调控[53]。甘蓝型油菜 BnPHT1;4 启动子区有 2 个 P1BS 元件和 2 个 W-box (TTGAC/T) 元件。研究表明,P1BS 元件是该基因被缺磷信号诱导表达的必需序列,而 W-box 对WRKY75 结合 BnPHT1;4 启动子是重要的[51]。此外,除了上述顺式作用元件外,研究人员还发现了另一类似于 P1BS 的元件,命名为 P1BS-like 元件,该元件同样可以调控 PHT1 家族基因的表达。例如,Schünmann 等[50]发现大麦 PHT1 家族基因的启动子区都包含 P1BS-like 元件,该元件能促进 HvPHT1;1 和 HvPHT1;2 等基因在低磷胁迫下表达上升。大麦 HvPHT1;1 启动子区包含 1 个 PHO-like motif 和 3 个与双子叶植物 P1BS 元件相似的 motifs,把启动子区的 3 个 P1BS 缩减为一个后会增加 HvPHT1;1 对低磷胁迫的响应[54]。
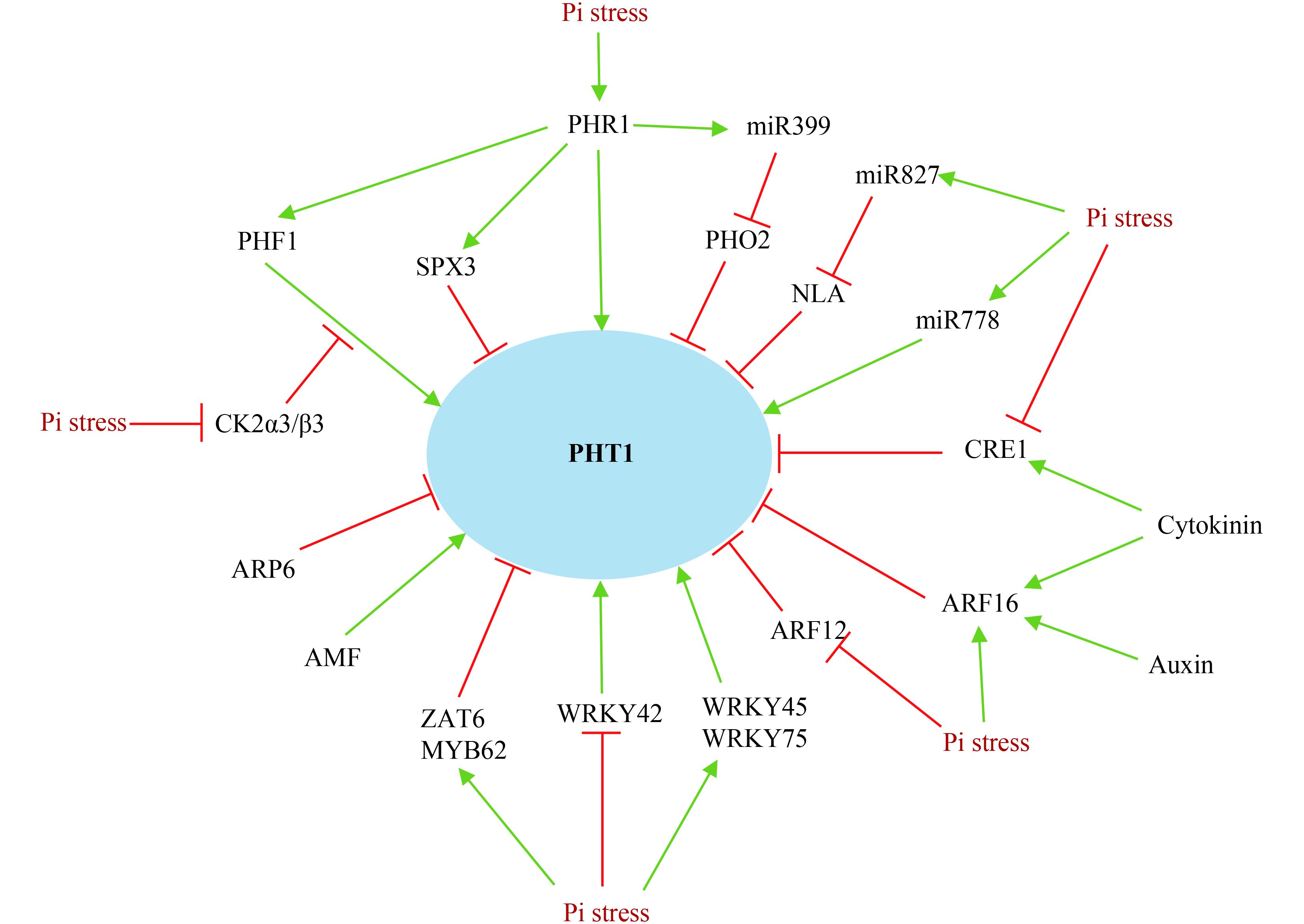
4.3 转录因子 (TF)PHR1 基因属于 MYB 超家族转录因子。该基因可以结合到磷胁迫诱导基因启动子区的 P1BS 元件上,进而调控基因的表达[3, 28, 55–57]。受 PHR1 基因调控的基因包括 PHT1 家族基因、PHO1、At4 和 miRNA399 等 (图 1)[28, 53, 55–57]。AtPHR1 是第一个在维管植物中分离的参与低磷胁迫转录调控的 MYB 超家族转录因子[57]。水稻中分离到 2 个 AtPHR1 同源基因 (OsPHR1 和 OsPHR2),其中 OsPHR2 的功能与 AtPHR1 类似。正常供磷条件下,超表达 OsPHR2 会使水稻地上部 OsPT1、OsPT5、OsPT7、OsPT9 和 OsPT12 等的表达上调[58]。同样,在油菜和拟南芥中超表达油菜 BnPHR1 同源基因会使高亲和磷转运子 BnPT2 及 ATPT2 的表达量显著升高[59]。对小麦 PHR1 同源基因 Ta-PHR1-A1 的研究结果显示该基因可以激活 Ta-PHT1.2 的表达[60]。然而 MYB 家族的另外一个转录因子 MYB62 与 PHR1 的作用正好相反,超表达该基因会显著抑制 Pht1;1 和 Pht1;4 的表达[61]。
WRKY 转录因子家族基因在调控 PHT1 转录中同样扮演了重要角色,该家族可以结合到基因启动子区的 W-box 元件上,调控基因的转录 (图 1)。拟南芥 WRKY75 受缺磷诱导表达,调节磷的吸收和根系的发育。抑制该基因的表达导致 Pht1;1 和 Pht1;4 的表达下降,进而影响植株磷吸收[62]。Wang 等[63]研究发现拟南芥 WRKY45 可以与 PHT1;1 启动子区的 2 个 W-box 结合,进而激活 PHT1;1 基因的转录,超表达 WRKY45 基因使得 PHT1;1 基因的表达显著上升,而抑制 WRKY45 基因的表达则出现相反的结果。此外,当磷供应充足时,拟南芥WRKY42 蛋白同样通过与 PHT1;1 启动子区结合正调控该基因的转录,在缺磷条件下因 WRKY42 蛋白的降解而导致调控解除[64]。
除此之外,其它转录因子基因在 PHT1 家族基因的转录调控中起作用。例如,拟南芥 ZAT6 基因是一个编码具有 C2H2 (cysteine-2/histidine-2) 锌指结构的转录因子,超表达 AtZAT6 可以抑制 Pht1;1 和 Pht1;4 的表达,减少转基因植株的磷吸收量[65]。
 |
|
图1
植物 PHT1 家族基因可能的调控途径
Fig. 1
Possible regulation pathway of plant PHT1 family genes
|
丛枝菌根 (arbuscular mycorrhiza,AM) 是植物根部和真菌互惠共生的结果,大多数维管开花植物都能形成菌根。一般认为营养交换是菌根共生的重要特征,AMF 在协助植物从土壤中吸收磷酸盐的同时从宿主植物中获得碳[66]。例如,有研究表明水稻吸收的磷酸盐 70% 来自共生菌根[67],而且不同种类的菌根真菌与宿主的结合程度强烈影响菌根对磷酸盐的吸收[2]。研究表明,AM 可以诱导植物根系中高亲和磷转运基因的表达 (图 1)[68]。目前在很多植物中均发现受菌根诱导表达的 PHT1 家族基因。例如水稻 OsPT11 和 OsPT13[69],玉米 ZmPT6[70],马铃薯 StPT3、StPT4 和 StPT5[71],大豆 GmPT10 和 GmPT11[45],番茄 LePT3、LePT4 和 LePT5[48, 72],苜蓿 MtPT4[73],矮牵牛花 PhPT4[74],白杨 PtPT8 及 PtPT10[52],栗 SiPHT1;8 和 SiPHT1;9[14],以及在其它植物中的同源基因等[75–77]。这些 PHT1 家族基因的表达水平会随着磷及菌根的存在与否而发生变化。有些磷转运子受菌根诱导表达,如 OsPT11、MtPT4 等,而有些磷转运子尤其是位于根系表皮中的磷转运子则受到菌根抑制表达,如 OsPHT1;1、MtPHT1;1 等。植物利用磷转运子基因通过根系表皮途径以及菌根途径进行磷的吸收,并保持很好的平衡[78]。同时,Xie 等[79]研究发现,在低磷条件下紫云英 AsPT1 和 AsPT4 基因在菌根皮层细胞被诱导表达,AsPT1 的超表达能提高菌根的生物量,而 AsPT1 的表达受到抑制时植株的丛枝会出现退化或死亡现象,Aspt4 沉默株系出现相同表型。这些结果表明 AM 共生体系依赖于 PHT1 家族基因 AsPT1 和 AsPT4 的表达。
5 调控植物 PHT1 家族成员转录的信号转导通路 5.1 PHOSPHATE TRANSPORTER TRAFFIC FACILITATOR 1 (PHF1)磷转运子的活性取决于其向质膜的转运过程。拟南芥 AtPHF1 基因编码的蛋白主要负责将 PHT1 转运蛋白从内质网运输至质膜,参与 PHT1 蛋白的细胞内运输调节 (图 1)。该基因主要在根系、花和衰老的叶片中表达。当 AtPHF1 发生突变时,内质网中的 AtPHT1;1 磷转运蛋白发生滞留,质膜上的 Pht1;1 磷转运蛋白减少,植株体内磷的累积下降。同时该基因的转录受 PHR1 基因的正调控[80]。水稻同源基因 OsPHF1 的功能与 AtPHF1 类似,当该基因发生突变时,磷转运子基因 OsPT2 和 OsPT8 在内质网上发生滞留,导致植物整体磷积累不足。但与 AtPHF1 基因不同的是 OsPHF1 基因的转录并不受 AtPHR1 同源基因 OsPHR2 的调控[81]。
5.2 磷酸化 (phosphorylation)研究表明,磷酸化在 PHT1 基因翻译后调控的过程中发挥了重要作用[3, 28]。Bayle 等[82]发现拟南芥 PHT1;1 从内质网的运出受它的第 514 位丝氨酸磷酸化水平的调节。低磷胁迫时,未被磷酸化的 PHT1 蛋白在 PHF1 的协助下,从内质网转移至细胞膜上,帮助植物吸收转运磷。而磷供应充足时,PHT1;1 蛋白的第 514 位丝氨酸位点发生磷酸化,导致 PHF1 无法与该蛋白结合,从而使得该蛋白从内质网向质膜的运输受阻,导致其滞留在内质网内。Chen 等[83]在水稻中的研究发现,CK2α3/β3 激酶在正常磷水平下可使 PT8 发生磷酸化,抑制 PT8 与 PHF1 的互作,进而影响 PT8 从内质网向质膜的运输。然而,磷胁迫会使 CK2α3/β3 激酶的 β3 亚基发生去磷酸化而降解,而使这种抑制作用解除进而促进磷的吸收,缓解植物的磷饥饿。因此,水稻 CK2α3/β3 激酶主要通过磷酸化 PHT1 蛋白负调控磷的吸收,而其自身的转录又受磷水平的调控 (图 1)。
5.3 含 SPX 结构域的蛋白家族很多含 SPX (SYG/Pho81/XPR1) 功能域的基因参与了植物磷信号的传导途径,表明该类基因在植物磷信号传递及磷动态平衡过程中发挥着重要的作用[28, 55–56]。拟南芥和水稻中分别发现了 4 个和 6 个 SPX 家族基因,命名为 AtSPX1-4 和 OsSPX1-6[84-85]。拟南芥 AtSPX1 和 AtSPX3 受缺磷强烈诱导表达。抑制 AtSPX3 的表达使地上部 Pht1;4 和 Pht1;5 的表达量显著上升,表明 AtSPX3 基因对 PHT1 家族基因起负调控的作用。同时AtSPX3 自身的转录水平受到 AtPHR1 基因的正调控 (图 1)[84]。水稻 OsSPX1 同样受低磷特异诱导,超表达 OsSPX1 会抑制 OsPT2、OsPT3、OsPT6 和 OsPT8 等 PHT1 家族基因的表达,而抑制 OsSPX1 的表达则会引起植株体内磷的超积累,说明 OsSPX1 负调控 PHT1 家族基因,进而影响磷的吸收。同时研究表明 OsSPX1 在磷调控网络中位于 OsPHR2 和 PHO2 的下游[85]。
近年来,研究人员发现了另外一类含 SPX 结构域的基因 NLA,该基因编码一个泛素 E3 连接酶,属于 SPX-RING (really interesting new gene) 蛋白家族,除了含有 SPX 结构域外还含有 RING 结构域[86]。研究表明,NLA 基因能促使 PHO2 编码的 E2 结合酶 UBC24 对质膜 PHT1 蛋白的泛素化,进而参与对 PHT1 家族基因的翻译后调控。当磷供应充足时,NLA 和 PHO2 (UBC24) 形成复合体对 PHT1;4 进行泛素化作用,促使细胞质膜上的 PHT1;4 蛋白通过内吞作用在液泡内进行。然而,NLA 和 PHO2 是 miR827 和 miR399 的下游靶基因,当磷缺乏时 NLA 和 PHO2 的转录本分别被 miR827 和 miR399 裂解,进而解除对 PHT1;4 蛋白的抑制,促进无机磷的吸收 (图 1)[86–87]。因此,NLA 和 PHO2 都是植物磷吸收的负调控因子。
5.4 MicroRNA非编码 RNA 虽然不能编码蛋白质,但是可以产生具有生物学功能的 microRNA,进而参与植物发育调控和逆境响应等重要过程。已有研究表明,microRNA 可以作为长距离信号参与植物低磷胁迫响应[28, 55–56]。目前研究较为清楚的是拟南芥 miR399,该基因受缺磷诱导表达。研究表明,miR399 能直接作用于 PHO2 基因的 5’-UTR 区,降解 PHO2 基因的 mRNA。PHO2 (UBC24) 基因编码一个 E2 泛素连接酶。在磷充足条件下该基因在植株根系中大量表达,并负调控高亲和磷转运子 Pht1;8 和 Pht1;9 的表达,抑制磷的吸收,而在缺磷的条件下,转录因子 PHR1 基因能激活 miRNA399 的转录,miR399 大量增加抑制了 PHO2 的表达,从而使植株启动高亲和磷转运系统,促进植株磷的吸收 (图 1)[56, 88–89]。
另一个报道与植物响应低磷胁迫信号途径相关的 microRNA 是 miRNA827。在缺磷的条件下,miR827 表达上升,降解 AtNLA 的 mRNA,从而激活植株磷的吸收以及磷从根系到地上部的转运 (如本文 5.3 所述)。最新的研究发现除了 miR399 和 miRNA827 外,miRNA778 也参与植物磷动态平衡过程。在低磷环境下,超表达 miR778 导致 miR399、Pht1;4 等基因的表达量上升,地上部无机磷和花青素的累积增加[90]。miR778 的靶基因 SUVH6 编码 H3K9 甲基转移酶,其表达水平受磷水平的调控,然而 SUVH6 参与植物响应低磷胁迫的机理仍需进一步研究。
5.5 植物激素植物激素如生长素 (auxin)、乙烯 (ethylene) 和细胞分裂素 (cytokinin) 等在植物响应低磷胁迫的过程中发挥了重要作用[55]。研究表明,生长素和细胞分裂素能抑制拟南芥高亲和磷转运子 AtPT1 的表达[91]。Wang 等[92]在水稻中克隆一个生长素响应因子基因 OsARF12,该基因发生突变时,参与水稻低磷胁迫调控的重要基因 OsPHR2 上调,其下游 PHT1 家族基因如 OsPT3、OsPT6、OsPT8 及 OsPT9 等的表达也显著增加,植株磷吸收增加,表明 OsARF12 作为负调控因子影响水稻磷的吸收和转运,而 OsARF12 的突变同样会影响缺磷条件下生长素的积累。OsARF16 基因是近年来在水稻中克隆的另外一个生长素响应因子。研究表明,该基因的表达同时受生长素和低磷胁迫的诱导。OsARF16 基因突变会影响生长素的极性运输,导致根系发育对生长素信号不敏感,以及对缺磷胁迫的响应能力减弱,表明 OsARF16 基因在生长素和磷胁迫信号感知的过程中发挥重要作用[93]。有趣的是,OsARF16 基因参与磷胁迫响应过程同时受细胞分裂素的调控。细胞分裂素会诱导OsARF16 的转录,抑制 PHT1 家族基因的表达,降低磷从根系向地上部的转运。当 OsARF16 基因发生突变时,OsPTs 基因的表达与野生型材料在磷胁迫条件下外源添加细胞分裂素相比显著增加,表明 OsARF16 基因参与了细胞分裂素介导的水稻磷转运和磷信号抑制过程[94]。此外,Talboys 等研究发现解淀粉芽孢杆菌 FZB42 (根际细菌) 能分泌生长素促进小麦根系生长,但强烈抑制 TaPHT1;8 和 TaPHT1.10 的表达,进而降低磷的吸收[95]。
细胞分裂素的主要功能是诱导植物细胞分裂。CRE1 基因编码一个组氨酸蛋白激酶,作为细胞分裂素受体参与激素信号感知过程。研究表明,细胞分裂素可以诱导该基因的表达,而低磷胁迫则会抑制其转录水平。当 CRE1 基因发生突变时,AtPT1 的转录水平上升[96]。
植株器官衰老期间,大分子物质程序性降解,使得衰老组织中的养分被再活化进而转运到新生组织中。研究表明,乙烯参与了牵牛花中核酸降解的调控过程,并且外源添加乙烯使花中 PhPht1;1 转录上升,表明乙烯可能在衰老期对磷的再移动发挥重要作用[97]。拟南芥 Pht1;5 基因主要在子叶、下胚轴、花蕾和老叶中表达,负责“源”和“库”器官间磷酸盐的移动。超表达 Pht1;5 基因会促进根毛的形成而抑制主根的生长。然而抑制转基因植株乙烯合成和信号感知时,该表型几乎恢复至野生型水平,表明乙烯信号与 PHT1 家族基因存在显著的互作关系[34]。
5.6 其他调控途径除上述信号调控途径参与 PHT1 家族的调控外,其他调控途径也有相关报道,包括染色质修饰、PHT1 家族基因互作,以及钙信号等。ARP6 基因编码一个核肌动蛋白相关蛋白 (nuclear actin-related protein),是 SWR1 染色质重构复合体的关键成分。该基因主要通过 H2A.Z 型组蛋白在染色质上的沉积来调控 PHT1 家族基因的转录[98]。
PHT1 家族基因间的互作也会影响基因的表达。例如,在拟南芥中敲除 AtPHT1;8 基因会改变 AtPHT1;5 和 AtPHT1;7 的转录本,AtPHT1;9 的突变会改变 AtPHT1;7 的转录本[36]。下调水稻 OsPT9 或 OsPT10 的表达并不引起植株无机磷含量的改变,但同时下调这 2 个基因的表达会引起无机磷含量显著降低,表明 OsPT9 和 OsPT10 对磷的吸收功能可能发生重叠[43]。
此外,研究表明钙离子及相关蛋白参与植物响应非生物胁迫的调节过程。破坏拟南芥液泡 Ca2+/H+ 转运子 CAX1 和 CAX3 基因会引起地上部磷酸盐等离子组成的显著变化。磷胁迫时,地上部 CAX1 基因的表达受到抑制,地上部 Ca2+ 稳态被打破,激发系统信号,由地上部传递至根系,进而对 PHT1 家族基因进行翻译后调控,影响根系磷转运系统的活性[99]。
6 总结与展望磷是植物生长发育所必需的大量营养元素,是植物体内核酸、磷脂和 ATP 的重要组成成分。土壤总磷含量较高,然而植物可利用的有效磷含量却很低。究其原因,主要是因为磷在土壤中容易被吸附固定,移动性很差,导致植物吸收磷不足[1]。农业上往往大量施用磷肥以满足植物生长需要,但是磷肥利用率偏低,且磷矿资源储量有限。为实现农业可持续发展的要求,通过分子生物学技术和遗传育种的办法改良作物磷吸收率和利用率,成为解决农业生产中磷缺乏胁迫的关键手段。目前,有关植物应对磷缺乏胁迫机制的研究很多,也克隆出一些与磷吸收利用相关的基因,但是结果表明植物对磷吸收利用的分子调控机理相当复杂,所以培育磷高效利用基因型作物任重而道远。
植物 PHT1 磷转运蛋白家族在植物磷吸收、转运、体内磷分配及再利用等过程中发挥了重要作用[3–4, 28, 55–56]。自 1996 年在高等植物中克隆出第一个磷转运基因以来,植物 PHT1 家族的克隆与功能研究不断推进并取得了长足的进展。这一研究领域已从模式植物、作物向其他高等植物中扩展,同时也进一步拓宽我们对高等植物 PHT1 家族的认识。已有研究表明,植物 PHT1 家族是一个庞大的家族,基因功能各异,基因表达调控机理复杂,涉及磷信号的感知、响应,转录水平的调控,以及功能基因蛋白 (PHT/PT) 活性的修饰等一系列过程 (图 1)。然而,对该家族蛋白的生化及结构生物学研究还待进一步深入。此外,对于一些基因组较复杂的多倍体物种如甘蓝型油菜、小麦、大麦及棉花等,关于 PHT1 家族与磷吸收转运的调控机理研究仍需要大量努力。然而,随着研究技术的不断成熟及新的生物技术的不断涌现,我们相信关于植物 PHT1 家族的认知必将推向一个新的高度。
| [1] | Schachtman D P, Reid R J, Ayling S M. Phosphorus uptake by plants: From soil to cell[J]. Plant Physiology, 1998, 116: 447–453. DOI:10.1104/pp.116.2.447 |
| [2] | Walder F, Brulé D, Koegel S, et al. Plant phosphorus acquisition in a common mycorrhizal network: regulation of phosphate transporter genes of the Pht1 family in sorghum and flax[J]. New Phytologist, 2015, 205(4): 1632–1645. DOI:10.1111/nph.13292 |
| [3] | Nussaume L, Kanno S, Javot H, et al. Phosphate import in plants: focus on the PHT1 transporters[J]. Frontiers in Plant Science, 2011, 2: 83. |
| [4] |
丁广大, 陈水森, 石磊, 等. 植物耐低磷胁迫的遗传调控机理研究进展[J].
植物营养与肥料学报, 2013, 19(3): 733–744.
Ding G D, Chen S S, Shi L, et al. Advances in genetic regulation mechanism of plant tolerance to phosphorus deficiency[J]. Journal of Plant Nutrition and Fertilizer, 2013, 19(3): 733–744. DOI:10.11674/zwyf.2013.0325 |
| [5] | Bun-Ya M, Nishimura M, Harashima S, Oshima Y. The PHO84 gene of Saccharomyces cerevisiae encodes an inorganic phosphate transporter [J]. Molecular and Cellular Biology, 1991, 11: 3229–3238. DOI:10.1128/MCB.11.6.3229 |
| [6] | Muchhal U S, Pardo J M, Raghothama K G. Phosphate transporters from the higher plant Arabidopsis thaliana [J]. Proceedings of the National Academy of Sciences, 1996, 93: 10519–10523. DOI:10.1073/pnas.93.19.10519 |
| [7] | Rausch C, Bucher M. Molecular mechanisms of phosphate transport in plants[J]. Planta, 2002, 216: 23–37. DOI:10.1007/s00425-002-0921-3 |
| [8] | Inoue Y, Kobae Y, Omoto E, et al. The soybean mycorrhiza-inducible phosphate transporter gene, GmPT7, also shows localized expression at the tips of vein endings of senescent leaves [J]. Plant and Cell Physiology, 2014, 55(12): 2102–2111. DOI:10.1093/pcp/pcu138 |
| [9] | Chen A Q, Chen X, Wang H M, et al. Genome-wide investigation and expression analysis suggest diverse roles and genetic redundancy of Pht1 family genes in response to Pi deficiency in tomato[J]. BMC Plant Biology, 2014, 14: 61. DOI:10.1186/1471-2229-14-61 |
| [10] | Guo C J, Guo L, Li X J, et al. TaPT2, a high-affinity phosphate transporter gene in wheat (Triticum aestivum L.), is crucial in plant Pi uptake under phosphorus deprivation [J]. Acta Physiologiae Plantarum, 2014, 36: 1373–1384. DOI:10.1007/s11738-014-1516-x |
| [11] | Poirier Y, Bucher M. Phosphate transport and homeostasis in Arabidopsis [J]. The Arabidopsis Book, 2002, : e0024. |
| [12] | Qin L, Guo Y, Chen L, et al. Functional characterization of 14 Pht1 family genes in yeast and their expressions in response to nutrient starvation in soybean[J]. PLoS One, 2012, 7(10): e47726. DOI:10.1371/journal.pone.0047726 |
| [13] | Wu Z, Zhao J, Gao R, et al. Molecular cloning, characterization and expression analysis of two members of the Pht1 family of phosphate transporters in Glycine max [J]. PLoS One, 2011, 6(6): e19752. DOI:10.1371/journal.pone.0019752 |
| [14] | Ceasar S A, Hodge A, Baker A, et al. Phosphate concentration and arbuscular mycorrhizal colonisation influence the growth, yield and expression of twelve PHT1 family phosphate transporters in foxtail millet (Setaria italica) [J]. PLoS One, 2014, 9(9): e108459. DOI:10.1371/journal.pone.0108459 |
| [15] | Anne L R, Daisy H, Janine M J, Frank W S. Characterization of two phosphate transporters from barley; evidence for diverse function and kinetic properties among members of the Pht1 family[J]. Plant Molecular Biology, 2003, 53: 27–36. DOI:10.1023/B:PLAN.0000009259.75314.15 |
| [16] |
李慧, 丛郁, 常有宏, 等. 豆梨磷转运蛋白质基因(PcPht1)的克隆, 表达及启动子分析
[J].
江苏农业学报, 2013, 29(4): 842–850.
Li H, Cong Y, Chang Y H, et al. Cloning, expression and promoter analysis of a phosphate transporter protein gene PcPht1 from Pyrus calleryana Dcne [J]. Jiangsu Journal of Agricultural Sciences, 2013, 29(4): 842–850. |
| [17] | Karandashov V, Bucher M. Symbiotic phosphate transport in arbuscular mycorrhizas[J]. Trends in Plant Science, 2005, 10(1): 22–29. DOI:10.1016/j.tplants.2004.12.003 |
| [18] | Ullrich C I, Novacky A J. Extra- and intracellular pH and membrane potential changes induced by K, Cl, H2PO4, and NO3- uptake and fusicoccin in root hairs of Limnobium stoloniferum [J]. Plant Physiology, 1990, 94: 1561–1567. DOI:10.1104/pp.94.4.1561 |
| [19] | Norihiro M, Satoru O, Yumiko S, et al. Overexpression of an Arabidopsis thaliana high-affinity phosphate transporter gene in tobacco cultured cells enhances cell growth under phosphate-limited conditions [J]. Plant Biology, 1997, 94: 7098–7102. |
| [20] | Ullrich-Eberius C I, Novacky A, Fischer E, Luttge U. Relationship between energy-dependent phosphate uptake and the electrical membrane potential in Lemnagibba G1 [J]. Plant Physiology, 1981, 67: 797–801. DOI:10.1104/pp.67.4.797 |
| [21] | Ullrich-Eberius C I, Novacky A, Van B A J E. Phosphate uptake in Lemnagibba G1: energetics and kinetics [J]. Planta, 1984, 161: 46–52. DOI:10.1007/BF00951459 |
| [22] | Preuss C P, Huang C Y, Gilliham M, et al. Channel-like characteristics of the low-affinity barley phosphate transporter PHT1;6 when expressed in Xenopus oocytes [J]. Plant Physiology, 2010, 152(3): 1431–1441. DOI:10.1104/pp.109.152009 |
| [23] |
陈爱群. 三种茄科作物Pht1家族磷转运蛋白基因的克隆及表达调控分析[D]. 南京: 南京农业大学博士学位论文, 2009.
Chen A Q. Isolation and analysis of expressional regulation of phosphate transporter genes in pht1 family from three solanaceous species[D]. Nanjing: PhD Dissertation of Nanjing Agricultural University, 2009. |
| [24] | Liu P, Chen S, Song A, et al. A putative high affinity phosphate transporter, CmPT1, enhances tolerance to Pi deficiency of chrysanthemum [J]. BMC Plant Biology, 2014, 14: 18. DOI:10.1186/1471-2229-14-18 |
| [25] | Liu X M, Zhao X L, Zhang L J, et al. TaPht1;4, a high-affinity phosphate transporter gene in wheat (Triticum aestivum), plays an important role in plant phosphate acquisition under phosphorus deprivation [J]. Functional Plant Biology, 2013, 40: 329–341. DOI:10.1071/FP12242 |
| [26] | Liu J Y, Versaw W K, Pumplin N, et al. Closely related members of the medicago truncatula PHT1 phosphate transporter gene family encode phosphate transporters with distinct biochemical activities [J]. The Journal of Biological Chemistry, 2008, 283(36): 24673–24681. DOI:10.1074/jbc.M802695200 |
| [27] |
贾宏昉. 水稻高亲和磷转运蛋白基因OsPhtl;8的功能研究[D]. 南京: 南京农业大学博士学位论文, 2011.
Jia H F. Functional analysis of a phosphate transporter gene OsPht1;8 in rice[D]. Nanjing: PhD Dissertation of Nanjing Agricultural University, 2011. |
| [28] | Baker A, Ceasar S A, Palmer A J, et al. Replace, reuse, recycle: improving the sustainable use of phosphorus by plants[J]. Journal of Experimental Botany, 2015, 66(12): 3523–3540. DOI:10.1093/jxb/erv210 |
| [29] | Fontenot E B, Ditusa S F, Kato N, et al. Increased phosphate transport of Arabidopsis thaliana Pht1;1 by site-directed mutagenesis of tyrosine 312 may be attributed to the disruption of homomeric interactions [J]. Plant, Cell and Environment, 2015, 38: 2012–2022. DOI:10.1111/pce.12522 |
| [30] | Mudge S R, Rae A L, Diatloff E, et al. Expression analysis suggests novel roles for members of the Pht1 family of phosphate transporters in Arabidopsis [J]. The Plant Journal, 2002, 31: 341–353. DOI:10.1046/j.1365-313X.2002.01356.x |
| [31] | Ayadi A, David P, Arrighi J F, et al. Reducing the genetic redundancy of Arabidopsis Phosphate transporter 1 transporters to study phosphate uptake and signaling [J]. Plant Physiology, 2015, 167: 1511–1526. DOI:10.1104/pp.114.252338 |
| [32] | Shin H S, H G R, Shin M J. Phosphate transport in Arabidopsis: Pht1;1 and Pht1;4 play a major role in phosphate acquisition from both low- and high-phosphate environments [J]. The Plant Journal, 2004, 39: 629–642. DOI:10.1111/tpj.2004.39.issue-4 |
| [33] | Misson J, Thibaud M C, Bechtold N, et al. Transcriptional regulation and functional properties of Arabidopsis Pht1;4, a high affinity transporter contributing greatly to phosphate uptake in phosphate deprived plants [J]. Plant Molecular Biology, 2004, 55: 727–741. DOI:10.1007/s11103-004-1965-5 |
| [34] | Nagarajan V K, Jain A, Poling M D, et al. Arabidopsis Pht1;5 mobilizes phosphate between source and sink organs and influences the interaction between phosphate homeostasis and ethylene signaling [J]. Plant Physiology, 2011, 156: 1149–1163. DOI:10.1104/pp.111.174805 |
| [35] | Remy E, Cabrito T R, Batista R A, et al. The Pht1;9 and Pht1;8 transporters mediate inorganic phosphate acquisition by the Arabidopsis thaliana root during phosphorus starvation [J]. New Phytologist, 2012, 195: 356–371. DOI:10.1111/j.1469-8137.2012.04167.x |
| [36] | Lapis-Gaza H R, Jost R, Finnegan P M. Arabidopsis phosphate transporter 1 genes PHT1;8 and PHT1;9 are involved in root-to-shoot translocation of orthophosphate [J]. BMC Plant Biology, 2014, 14(1): 334. DOI:10.1186/s12870-014-0334-z |
| [37] | Goff S A, Ricke D, Lan T H, et al. A draft sequence of the rice genome (Oryza sativa L. ssp japonica) [J]. Science, 2002, 296: 92–100. DOI:10.1126/science.1068275 |
| [38] | Sun S B, Gu M, Cao Y, et al. A constitutive expressed phosphate transporter, OsPht1;1, modulates phosphate uptake and translocation in phosphate-replete rice [J]. Plant Physiology, 2012, 159: 1571–1581. DOI:10.1104/pp.112.196345 |
| [39] | Ai P H, Sun S B, Zhao J N, et al. Two rice phosphate transporters, OsPht1;2 and OsPht1;6, have different functions and kinetic properties in uptake and translocation [J]. The Plant Journal, 2009, 57: 798–809. DOI:10.1111/tpj.2009.57.issue-5 |
| [40] | Ye Y, Yuan J, Chang X, et al. The phosphate transporter gene OsPht1;4 is involved in phosphate homeostasis in rice [J]. PLoS One, 2015, 10(5): e0126186. DOI:10.1371/journal.pone.0126186 |
| [41] | Zhang F, Sun Y F, Pei W X, et al. Involvement of OsPht1;4 in phosphate acquisition and mobilization facilitates embryo development in rice [J]. The Plant Journal, 2015, 82: 556–569. DOI:10.1111/tpj.12804 |
| [42] | Li Y T, Zhang J, Zhang X, et al. Phosphate transporter OsPht1;8 in rice plays an important role in phosphorus redistribution from source to sink organs and allocation between embryo and endosperm of seeds [J]. Plant Science, 2015, 230: 23–32. DOI:10.1016/j.plantsci.2014.10.001 |
| [43] | Wang X F, Wang Y F, Miguel A P, et al. Phosphate transporters OsPHT1;9 and OsPHT1;10 are involved in phosphate uptake in rice [J]. Plant, Cell and Environment, 2014, 37: 1159–1170. DOI:10.1111/pce.12224 |
| [44] | Qin L, Zhao J, Tian J, et al. The high-affinity phosphate transporter GmPT5 regulates phosphate transport to nodules and nodulation in soybean [J]. Plant Physiology, 2012, 159: 1634–1643. DOI:10.1104/pp.112.199786 |
| [45] | Tamura Y, Kobae Y, Mizuno T, et al. Identification and expression analysis of arbuscular mycorrhiza-inducible phosphate transporter genes of soybean[J]. Bioscience, Biotechnology, and Biochemistry, 2012, 76: 309–313. DOI:10.1271/bbb.110684 |
| [46] | Nagy R, Karandashov V, Chague V, et al. The characterization of novel mycorrhiza-specific phosphate transporters from Lycopersicon esculentum and Solanum tuberosum uncovers functional redundancy in symbiotic phosphate transport in solanaceous species [J]. The Plant Journal, 2005, 42: 236–250. DOI:10.1111/tpj.2005.42.issue-2 |
| [47] |
洪帅. 番茄磷转运蛋白基因LePT1和LePT2在水稻中的功能鉴定[D]. 南京: 南京农业大学硕士学位论文, 2011.
Hong S. Function identification of two tomato phosphater transporter LePT1 and LePT2 genes in rice[D]. Nanjing: MS Thesis of Nanjing Agricultural University, 2011. |
| [48] | Chen A Q, Hu J, Sun S, et al. Conservation and divergence of both phosphate- and mycorrhiza-regulated physiological responses and expression patterns of phosphate transporters in solanaceous species[J]. New Physiology, 2007, 173(4): 817–831. DOI:10.1111/nph.2007.173.issue-4 |
| [49] | Tan Z J, Hu Y, Lin Z. Expression of NtPT5 is correlated with the degree of colonization in tobacco roots inoculated with Glomus etunicatum [J]. Plant Molecular Biology Reporter, 2012, 30: 885–893. DOI:10.1007/s11105-011-0402-6 |
| [50] | Schünmann P H D, Richardson A E, Smith F W, Delhaize E. Characterization of promoter expression patterns derived from the Pht1 phosphate transporter genes of barley (Hordeum vulgare L.) [J]. Journal of Experimental Botany, 2004, 55(398): 855–865. DOI:10.1093/jxb/erh103 |
| [51] | Ren F, Zhao C Z, Liu C S, et al. A Brassica napus PHT1 phosphate transporter, BnPHT1;4, promotes phosphate uptake and affects roots architecture of transgenic Arabidopsis [J]. Plant Molecular Biology, 2014, 86: 595–607. DOI:10.1007/s11103-014-0249-y |
| [52] | Loth-Pereda V, Orsini E, Courty P E, et al. Structure and expression profile of the phosphate PHT1 transporter gene family in mycorrhizal Populus trichocarpa [J]. Plant Physiology, 2011, 156: 2141–2154. DOI:10.1104/pp.111.180646 |
| [53] | Chen A Q, Gu M, Sun S, et al. Identification of two conserved cis-acting elements, MYCS and P1BS, involved in the regulation of mycorrhiza-activated phosphate transporters in eudicot species[J]. New Phytologist, 2011, 189: 1157–1169. DOI:10.1111/nph.2011.189.issue-4 |
| [54] | Schünmann P H D, Richardson A E, Vickers C E, Delhaize E. Promoter analysis of the barley phosphate transporter gene identifies regions controlling root expression and responsiveness to phosphate deprivation[J]. Plant Physiology, 2004, 136: 4205–4214. DOI:10.1104/pp.104.045823 |
| [55] | Chiou T J, Lin S I. Signaling network in sensing phosphate availability in plants[J]. Annual Review of Plant Biology, 2011, 62: 185–206. DOI:10.1146/annurev-arplant-042110-103849 |
| [56] | López-Arredondo D L, Leyva-González M A, González-Morales S I, et al. Phosphate nutrition: improving low-phosphate tolerance in crops[J]. Annual Review of Plant Biology, 2014, 65: 95–123. DOI:10.1146/annurev-arplant-050213-035949 |
| [57] | Rubio V, Linhares F, Solano R, et al. A conserved MYB transcription factor involved in phosphate starvation signaling both in vascular plants and in unicellular algae[J]. Genes & Development, 2001, 15(16): 2122–2133. |
| [58] | Zhou J, Jiao F C, Wu Z, et al. OsPHR2 is involved in phosphate-starvation signaling and excessive phosphate accumulation in shoots of plants [J]. Plant Physiology, 2008, 146(4): 1673–1686. DOI:10.1104/pp.107.111443 |
| [59] | Ren F, Guo Q Q, Chang L L, et al. Brassica napus PHR1 gene encoding a MYB-like protein functions in response to phosphate starvation [J]. PLoS One, 2012, 7(8): e44005. DOI:10.1371/journal.pone.0044005 |
| [60] | Wang J, Sun J, Miao J, et al. A wheat phosphate starvation response regulator Ta-PHR1 is involved in phosphate signalling and increases grain yield in wheat [J]. Annals of Botany, 2013, 111: 1139–1153. DOI:10.1093/aob/mct080 |
| [61] | Devaiah B N, Madhuvanthi R, Karthikeyan A S, Raghothama K G. Phosphate starvation responses and gibberellic acid biosynthesis are regulated by the MYB62 transcription factor in Arabidopsis [J]. Molecular Plant, 2009, 2(1): 43–58. DOI:10.1093/mp/ssn081 |
| [62] | Devaiah B N, Karthikeyan A S, Raghothama K G. WRKY75 transcription factor is a modulator of phosphate acquisition and root development in Arabidopsis [J]. Plant Physiology, 2007, 143(4): 1789–1801. DOI:10.1104/pp.106.093971 |
| [63] | Wang H, Xu Q, Kong Y H, et al. Arabidopsis WRKY45 transcription factor activates PHOSPHATE TRANSPORTER1; 1 expression in response to phosphate starvation [J]. Plant Physiology, 2014, 164(4): 2020–2029. DOI:10.1104/pp.113.235077 |
| [64] | Su T, Xu Q, Zhang F C, et al. WRKY42 modulates phosphate homeostasis through regulating phosphate translocation and acquisition in Arabidopsis [J]. Plant Physiology, 2015, 167(4): 1579–1591. DOI:10.1104/pp.114.253799 |
| [65] | Devaiah B N, Nagarajan V K, Raghothama K G. Phosphate homeostasis and root development in Arabidopsis are synchronized by the zinc finger transcription factor ZAT6 [J]. Plant Physiology, 2007, 145(1): 147–159. DOI:10.1104/pp.107.101691 |
| [66] | Javot H, Penmetsa R V, Terzaghi N, et al. A Medicago truncatula phosphate transporter indispensable for the arbuscular mycorrhizal symbiosis[J]. Proceedings of the National Academy of Sciences, 2007, 104(5): 1720–1725. DOI:10.1073/pnas.0608136104 |
| [67] | Yang S Y, Grønlund M, Jakobsen I, et al. Nonredundant regulation of rice arbuscular mycorrhizal symbiosis by two members of the PHOSPHATE TRANSPORTER1 gene family [J]. The Plant Cell, 2012, 24(10): 4236–4251. DOI:10.1105/tpc.112.104901 |
| [68] | Smith S E, Jakobsen I, Grønlund M, et al. Roles of arbuscular mycorrhizas in plant phosphorus nutrition: interactions between pathways of phosphorus uptake in arbuscular mycorrhizal roots have important implications for understanding and manipulating plant phosphorus acquisition[J]. Plant Physiology, 2011, 156(3): 1050–1057. DOI:10.1104/pp.111.174581 |
| [69] | Paszkowski U, Kroken S, Roux C, et al. Rice phosphate transporters include an evolutionarily divergent gene specifically activated in arbuscular mycorrhizal symbiosis[J]. Proceedings of the National Academy of Sciences, 2002, 99: 13324–13329. DOI:10.1073/pnas.202474599 |
| [70] | Glassop D, Smith S E, Smith F W. Cereal phosphate transporters associated with the mycorrhizal pathway of phosphate uptake into roots[J]. Planta, 2005, 222(4): 688–698. DOI:10.1007/s00425-005-0015-0 |
| [71] | Rausch C, Daram P, Brunner S, et al. A phosphate transporter expressed in arbuscule-containing cells in potato[J]. Nature, 2001, 414: 462–466. DOI:10.1038/35106601 |
| [72] | Nagy R, Drissner D, Amrhein N, et al. Mycorrhizal phosphate uptake pathway in tomato is phosphorus-repressible and transcriptionally regulated[J]. New Phytology, 2009, 181(4): 950–959. DOI:10.1111/j.1469-8137.2008.02721.x |
| [73] | Harrison M J, Dewbre G R, Liu J. A phosphate transporter from Medicago truncatula involved in the acquisition of phosphate released by arbuscular mycorrhizal fungi [J]. Plant Cell, 2002, 14(10): 2413–2429. DOI:10.1105/tpc.004861 |
| [74] | Tan Z, Hu Y, Lin Z. PhPT4 is a mycorrhizal-phosphate transporter suppressed by lysophosphatidylcholine in petunia roots [J]. Plant Molecular Biology Reporter, 2012, 30: 1480–1487. DOI:10.1007/s11105-012-0467-x |
| [75] | Shu B, Xia R X, Wang P. Differential regulation of PHT1 phosphate transporters from trifoliate orange (Poncirus trifoliata L. Raf) seedlings [J]. Scientia Horticulturae, 2012, 146: 115–123. DOI:10.1016/j.scienta.2012.08.014 |
| [76] | Wegmüller S, Svistoonoff S, Reinhardt D, et al. A transgenic dTph1 insertional mutagenesis system for forward genetics in mycorrhizal phosphate transport of Petunia [J]. The Plant Journal, 2008, 54: 1115–1127. DOI:10.1111/j.1365-313X.2008.03474.x |
| [77] | Saia S, Rappa V, Ruisi P, et al. Soil inoculation with symbiotic microorganisms promotes plant growth and nutrient transporter genes expression in durum wheat[J]. Frontiers in Plant Science, 2015, 6. |
| [78] | Javot H, Pumplin N, Harrison M J. Phosphate in the arbuscular mycorrhizal symbiosis: transport properties and regulatory roles[J]. Plant Cell & Environment, 2007, 30: 310–322. |
| [79] | Xie X, Huang W, Liu F, et al. Functional analysis of the novel mycorrhiza-specific phosphate transporter AsPT1 and PHT1 family from Astragalus sinicus during the arbuscular mycorrhizal symbiosis [J]. New Phytologist, 2013, 198(3): 836–852. DOI:10.1111/nph.12188 |
| [80] | González E, Solano R, Rubio V, et al. Phosphate transporter traffic facilitator 1 is a plant-specific SEC12-related protein that enables the endoplasmic reticulum exit of a high-affinity phosphate transporter in Arabidopsis [J]. The Plant Cell, 2005, 17(12): 3500–3512. DOI:10.1105/tpc.105.036640 |
| [81] | Chen J, Liu Y, Ni J, et al. OsPHF1 regulates the plasma membrane localization of low-and high-affinity inorganic phosphate transporters and determines inorganic phosphate uptake and translocation in rice [J]. Plant Physiology, 2011, 157(1): 269–278. DOI:10.1104/pp.111.181669 |
| [82] | Bayle V, Arrighi J F, Creff A, et al. Arabidopsis thaliana high-affinity phosphate transporters exhibit multiple levels of posttranslational regulation [J]. The Plant Cell, 2011, 23(4): 1523–1535. DOI:10.1105/tpc.110.081067 |
| [83] | Chen J, Wang Y, Wang F, et al. The rice CK2 kinase regulates trafficking of phosphate transporters in response to phosphate levels[J]. The Plant Cell, 2015, 27(3): 711–723. DOI:10.1105/tpc.114.135335 |
| [84] | Duan K, Yi K, Dang L, et al. Characterization of a sub-family of Arabidopsis genes with the SPX domain reveals their diverse functions in plant tolerance to phosphorus starvation [J]. The Plant Journal, 2008, 54(6): 965–975. DOI:10.1111/j.1365-313X.2008.03460.x |
| [85] | Wang C, Ying S, Huang H, et al. Involvement of OsSPX1 in phosphate homeostasis in rice [J]. The Plant Journal, 2009, 57(5): 895–904. DOI:10.1111/tpj.2009.57.issue-5 |
| [86] | Lin W Y, Huang T K, Chiou T J. NITROGEN LIMITATION ADAPTATION, a target of microRNA827, mediates degradation of plasma membrane-localized phosphate transporters to maintain phosphate homeostasis in Arabidopsis [J]. The Plant Cell, 2013, 25(10): 4061–4074. DOI:10.1105/tpc.113.116012 |
| [87] | Park B S, Seo J S, Chua N H. NITROGEN LIMITATION ADAPTATION recruits PHOSPHATE2 to target the phosphate transporter PT2 for degradation during the regulation of Arabidopsis phosphate homeostasis [J]. The Plant Cell, 2014, 26(1): 454–464. DOI:10.1105/tpc.113.120311 |
| [88] | Bari R, Pant B D, Stitt M, et al. PHO2, microRNA399, and PHR1 define a phosphate-signaling pathway in plants[J]. Plant Physiology, 2006, 141(3): 988–999. DOI:10.1104/pp.106.079707 |
| [89] | Huang T K, Han C L, Lin S I, et al. Identification of downstream components of ubiquitin-conjugating enzyme PHOSPHATE2 by quantitative membrane proteomics in Arabidopsis roots [J]. The Plant Cell, 2013, 25(10): 4044–4060. DOI:10.1105/tpc.113.115998 |
| [90] | Wang L, ZengJ H Q, Song J, et al. miRNA778 and SUVH6 are involved in phosphate homeostasis in Arabidopsis [J]. Plant Science, 2015, 238: 273–285. DOI:10.1016/j.plantsci.2015.06.020 |
| [91] | Karthikeyan A S, Varadarajan D K, Mukatira U T, et al. Regulated expression of Arabidopsis phosphate transporters [J]. Plant Physiology, 2002, 130(1): 221–233. DOI:10.1104/pp.020007 |
| [92] | Wang S K, Zhang S N, Sun C D, et al. Auxin response factor (OsARF12), a novel regulator for phosphate homeostasis in rice (Oryza sativa) [J]. New Phytologist, 2014, 201(1): 91–103. DOI:10.1111/nph.12499 |
| [93] | Shen C, Wang S, Zhang S, et al. OsARF16, a transcription factor, is required for auxin and phosphate starvation response in rice (Oryza sativa L.) [J]. Plant, Cell & Environment, 2013, 36(3): 607–620. |
| [94] | Shen C, Yue R, Yang Y, et al. OsARF16 is involved in cytokinin-mediated inhibition of phosphate transport and phosphate signaling in rice (Oryza sativa L.) [J]. PLoS One, 2014, 9(11): e112906. DOI:10.1371/journal.pone.0112906 |
| [95] | Talboys P J, Owen D W, Healey J R, et al. Auxin secretion by Bacillus amyloliquefaciens FZB42 both stimulates root exudation and limits phosphorus uptake in Triticum aestivium [J]. BMC Plant Biology, 2014, 14(1): 1. DOI:10.1186/1471-2229-14-1 |
| [96] | Franco-Zorrilla J M, Martin A C, Solano R, et al. Mutations at CRE1 impair cytokinin-induced repression of phosphate starvation responses in Arabidopsis [J]. The Plant Journal, 2002, 32(3): 353–360. DOI:10.1046/j.1365-313X.2002.01431.x |
| [97] | Chapin L J, Jones M L. Ethylene regulates phosphorus remobilization and expression of a phosphate transporter (PhPT1) during petunia corolla senescence [J]. Journal of Experimental Botany, 2009, 60(7): 2179–2190. DOI:10.1093/jxb/erp092 |
| [98] | Smith A P, Jain A, Deal R B, et al. Histone H2A.Z regulates the expression of several classes of phosphate starvation response genes but not as a transcriptional activator[J]. Plant Physiology, 2010, 152(1): 217–225. DOI:10.1104/pp.109.145532 |
| [99] | Liu T Y, Aung K, Tseng C Y, et al. Vacuolar Ca2+/H+ transport activity is required for systemic phosphate homeostasis involving shoot-to-root signaling in Arabidopsis [J]. Plant Physiology, 2011, 156(3): 1176–1189. DOI:10.1104/pp.111.175257 |
 2017, Vol. 23
2017, Vol. 23  doi:
doi: 
