文章信息
- 基于两样本孟德尔随机化的摄入咖啡与前列腺癌因果关系研究
- Casual Association Between Coffee Intake and Prostate Cancer Based on Two-sample Mendel Randomization
- 肿瘤防治研究, 2024, 51(1): 49-54
- Cancer Research on Prevention and Treatment, 2024, 51(1): 49-54
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2024.23.0672
- 收稿日期: 2023-06-25
- 修回日期: 2023-08-29
前列腺癌是发达国家男性最常见的实体恶性肿瘤,是癌症相关死亡的主要原因之一,且发病率逐年增高。据研究统计,2022年美国新增268 490例前列腺癌患者,为新增人数最多的恶性肿瘤,而死亡人数居第二位,为34 500例[1]。前列腺癌风险因素目前尚未十分明确,一些研究显示肥胖、吸烟、年龄、种族、家族史、基因变异等可能与前列腺癌发生发展相关[2]。近年来,一些研究发现饮食及营养可能参与促进或抑制前列腺癌[3],其中咖啡受到越来越多研究者的关注[4]。
咖啡是世界上最受欢迎和消费最广泛的饮料之一,其对个人健康的微小影响都可能会对公共健康造成巨大影响。研究显示,咖啡含有咖啡因、绿原酸、天然二萜等多种活性成分,可通过下调胰岛素样生长因子1表达、减少氧化应激等多种机制影响癌症的发生发展[5-7]。一项队列研究发现,摄入咖啡可使前列腺癌的风险降低53%,同时细胞实验证实咖啡因可抑制前列腺癌细胞的增殖和转移[8]。英国一项前瞻性队列研究对6 017例男性进行随访,结果显示摄入咖啡可使前列腺癌风险降低55%(HR=0.45, 95%CI: 0.23~0.90, P < 0.01)[9]。然而也有研究并未发现饮用咖啡与前列腺癌存在关联。欧洲一项研究对142 196例男性进行14年的随访,发现摄入咖啡与前列腺癌不存在因果关系(HR=1.02, 95%CI: 0.94~1.09)[10]。这些均为观察性研究,较易受到混杂因素的影响,结论可能存在较大偏倚。鉴于咖啡在人类饮食中的广泛性,需要进一步明确摄入咖啡与前列腺癌的关联。
孟德尔随机化(Mendelian randomization, MR)分析是近年来流行病学十分常用的一种研究设计,通过引入经济学中的工具变量(instrumental variable, IV)的概念,将基因变异作为暴露因素的工具变量,研究暴露因素与疾病的因果关系,是一种采用遗传数据来评价可变化的非遗传暴露因素的因果效应的技术。相较于传统的观察性流行病学研究,MR基于亲代等位基因随机分配给子代的孟德尔遗传定律,相当于在人群中开展随机对照试验,还具备避免耗费大量人力物力以及伦理容易通过的优点。目前尚缺乏探索摄入咖啡与前列腺癌关联的孟德尔随机化研究。基于此,本研究拟采用两样本孟德尔随机化方法研究摄入咖啡与前列腺癌的因果关系。
孟德尔随机化研究中的工具变量必须满足三个核心条件[11]:工具变量与暴露因素强相关(关联性假设);工具变量与混杂因素不相关(独立性假设);工具变量只能通过暴露因素影响结局(排他性假设)。本研究拟筛选与前列腺癌相关的核苷酸多态性(single nucleotide polymorphism, SNP)位点作为工具变量,评估摄入咖啡与前列腺癌的关联。
1 资料与方法 1.1 数据来源本研究采用两样本孟德尔随机化方法研究摄入咖啡与前列腺癌的关联。本研究暴露变量为摄入咖啡,结局变量为前列腺癌,暴露与结局的全基因组关联研究(genome wide association study, GWAS)数据均来源于英国生物样本库数据库(https://gwas.mrcieu.ac.uk/)。
1.2 数据整理以P < 5×10-8为标准筛选出与暴露相关(摄入咖啡)的工具变量,然后根据r2=0.001和kb=10 000这两个参数筛掉10 MB范围内r2 > 0.001中P值较大的SNP,去除存在连锁不平衡的SNP,保证筛选出的工具变量互相独立。然后进行二次筛选,以P < 5×10-8为标准剔除与前列腺癌相关的IVs。最后,计算F统计量,F > 10则表明IVs不太可能存在弱工具变量偏倚。
1.3 统计学方法 1.3.1 多效性检验孟德尔随机化研究还需满足的一条假设是工具变量只能通过暴露因素影响结局,因此需排除工具变量通过其他途径对结局造成影响,需要进行多效性检验。MR-Egger回归效应模型通常用来评估多效性是否存在,回归模型中的截距与0不存在统计学差异时,表明IV不存在多效性,反之则存在。
1.3.2 因果效应估计计算筛选出的IV与摄入咖啡、IV与前列腺癌的Beta值和SE值,利用逆方差加权法[12](inverse-variance weighting, IVW)进行整合,评估摄入咖啡与前列腺癌的关联。另外,为了评估结果的稳定性,使用MR-Egger回归[13]、加权中位数[14](weighted median estimator, WME)法进行补充验证。
1.3.3 异质性检验孟德尔随机化的异质性检验是对基于单个遗传变量的工具变量估计的兼容性的统计评估。如果IV之间不存在异质性,则可使用固定效应模型对结果进行合并分析,反之需分析存在异质性的原因及进行处理,若无法消除则使用随机效应模型。本研究使用MR-Egger方法评估工具变量之间是否存在异质性,P > 0.05代表筛选出的IV之间不存在异质性。
1.3.4 敏感性分析为提高结果的稳健性,需要对IV筛选纳入过程中产生的随机误差进行分析并进行敏感性分析。留一法(leave-one-out)是孟德尔随机化研究中最常用于敏感性分析的方法,通过逐步剔除每个SNP,评估剩余SNP的Meta效应,观察剔除每个SNP前后结果是否存在统计学差异。如果剔除某个SNP后的MR估计结果与总结果的差异无统计学意义,则表明该SNP不会对结果造成非特异性影响,结果是稳定的[15]。
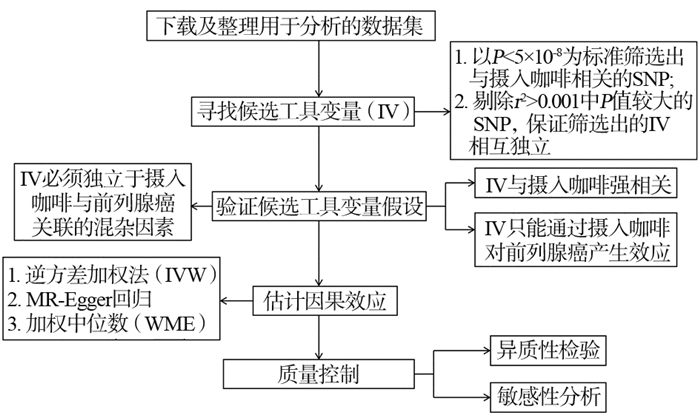
1.4 统计分析软件和技术路线本研究使用R4.2.2软件及“TwoSampleMR”软件包[16]进行统计分析,P < 0.05为差异有统计学意义。技术路线图见图 1。
|
| 图 1 孟德尔随机化研究流程图 Figure 1 Flowchart of Mendelian randomization study |
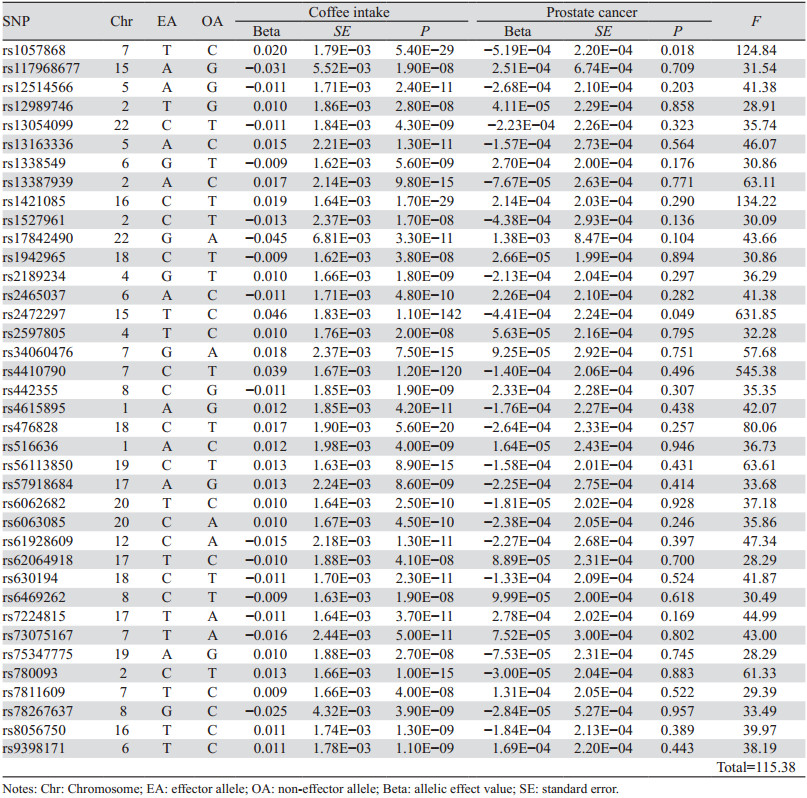
摄入咖啡的GWAS数据来源于一项2018年实施的GWAS研究,样本量为428 860例,包含9 851 867个SNP。前列腺癌的GWAS数据来源于一项2017年实施的研究,该研究样本量为337 199例,均为欧洲人,包含334 926例健康对照者和2 273例患者,10 894 596个SNP,见表 1。
从摄入咖啡的GWAS数据中以P < 5×10-8为标准筛选并进一步以r2=0.001和kb=10 000这两个参数去除存在连锁不平衡的SNP,得到40个与暴露相关的SNP。接着在前列腺癌GWAS数据中以P < 5×10-8为标准剔除与前列腺癌相关的SNP,最终筛选得到38个SNP。F统计量为115.38,表明工具变量不太可能存在弱工具变量偏倚,见表 2。
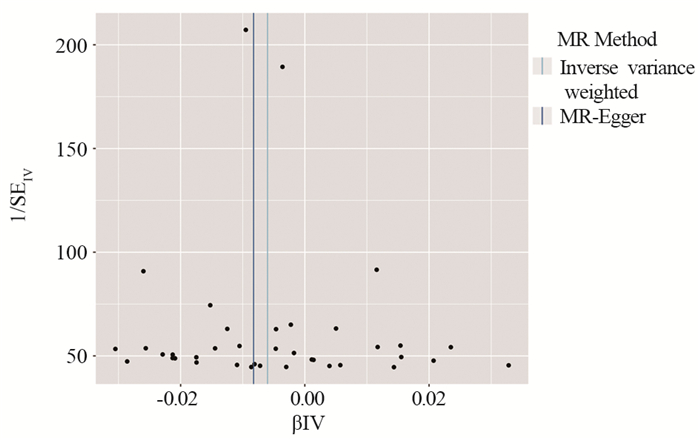
本研究通过MR-Egger回归验证筛选出的IV是否独立于咖啡-前列腺癌关联的混杂因素,结果显示Egger intercept=4.2E-5,P=0.581,说明研究结果未受基因多效性的显著影响。此外,漏斗图也显示本研究不太可能受到潜在偏倚的影响,见图 2。
|
| 图 2 孟德尔随机化结果漏斗图 Figure 2 Funnel plot of Mendelian randomization results |
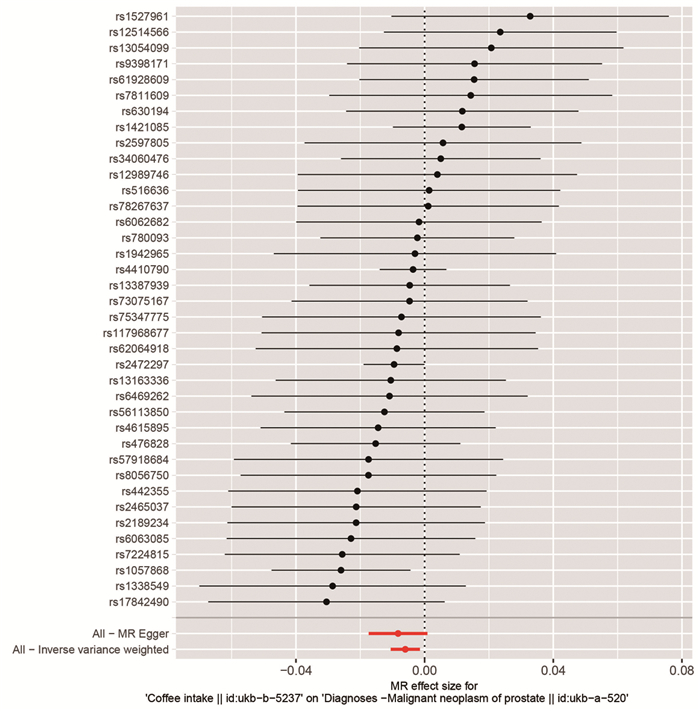
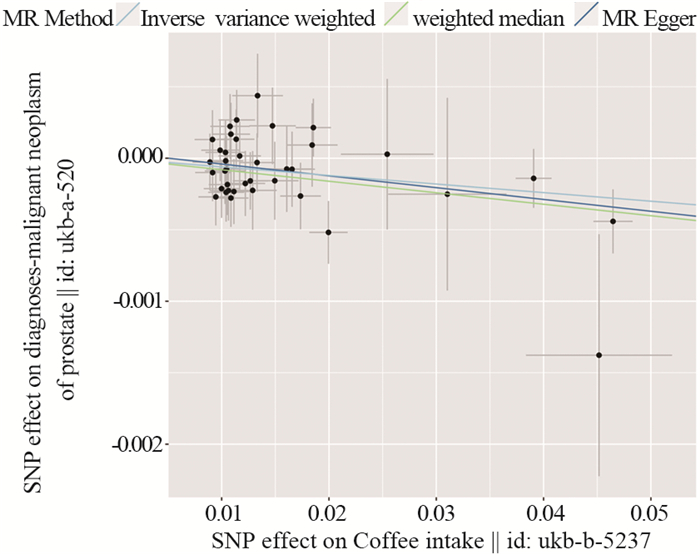
本研究最终纳入了38个SNP,每个SNP位点对前列腺癌的影响见图 3、4。逆方差加权法显示摄入咖啡可能降低前列腺癌风险,结果具有统计学意义(OR=0.994, 95%CI: 0.990~0.999, P=0.009)。采用加权中位数方法得到一致的结论(OR=0.991, 95%CI: 0.985~0.999, P=0.018),不过MR-Egger回归未发现两者存在关联(OR=0.992, 95%CI: 0.983~1.000, P=0.084)。
|
| 图 3 孟德尔随机化结果森林图 Figure 3 Forest plot of Mendelian randomization results |
|
| 图 4 孟德尔随机化结果散点图 Figure 4 Scatter plot of Mendelian randomization results |
由于暴露和结局数据来源于两个独立的队列研究,工具变量之间可能存在异质性。采用MR-Egger法进行异质性检验,得到的结果为Q=27.20, P=0.854,表明IV之间不存在异质性。
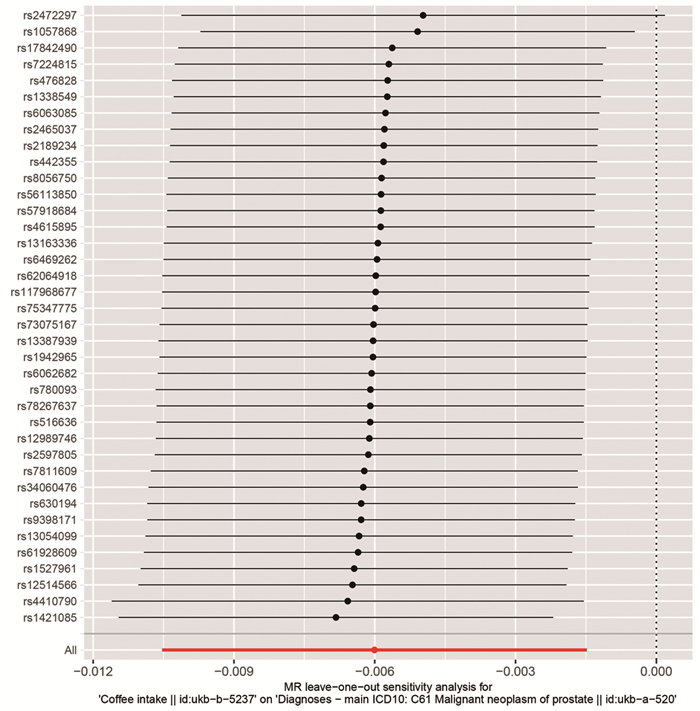
2.6 敏感性分析采用逐一剔除IV(留一法)对本研究结果进行敏感性分析,见图 5,剔除任意一个IV后,IVW方法计算余下IV的效应值与总IV效应值均无显著变化(P < 0.05),表明不存在对研究结果造成显著影响的IV,结论稳健,即摄入咖啡可能降低前列腺癌风险(OR: 0.994, 95%CI: 0.990~0.999, P=0.009)。
|
| 图 5 “留一法”敏感性分析结果 Figure 5 Sensitivity analysis results of the "leave-one-out" method |
探索前列腺癌的风险因素对于其防治具有重要意义。咖啡是人们日常生活中一种十分常见的饮品,本研究利用两样本孟德尔随机化方法评估摄入咖啡和前列腺癌的关联,结果显示摄入咖啡可能降低前列腺癌发病风险,异质性检验及敏感性分析进一步证实了结论的可靠性。
先前一些观察性研究支持本结论。日本学者对18 853名40~79岁的男性进行长达11年的随访,发现摄入咖啡可显著降低前列腺癌风险,且咖啡摄入量与前列腺癌风险呈负相关[17]。一项纳入多项队列研究的Meta分析结果证实摄入咖啡可降低前列腺癌风险(OR=0.91, 95%CI: 0.84~0.98)[18]。摄入咖啡降低前列腺癌发病风险的机制尚未十分明确,目前一些研究显示可能与以下机制有关:咖啡中的咖啡因具有较强的抗氧化能力,能减少活性氧产生,促进肿瘤细胞死亡及防止细胞基因突变[19];咖啡能够抑制细胞炎性反应,下调炎性反应因子诸如IL-6[20]、TNF-α[21]、CRP[22]的表达,减轻炎性反应因子对DNA的损伤及细胞上皮屏障的破坏[23];咖啡还可促进DNA修复蛋白O6-甲基鸟嘌呤-DNA甲基转移酶的表达,对受损的DNA进行修复,防止细胞发生癌变[24];另有研究发现咖啡可阻滞细胞周期,下调BCL-2表达,促进前列腺癌细胞凋亡[25];雄激素受体(androgen receptor, AR)是前列腺癌进展过程中一个至关重要的靶点,Iwamoto等[26]证实咖啡醇可显著下调AR表达,抑制上皮间质转化,抑制前列腺癌细胞侵袭及诱导凋亡。
本研究为摄入咖啡降低前列腺癌发病风险提供了更高等级的证据。首先,传统的观察性研究往往无法排除混杂因素以及受到回忆偏倚等诸多因素的影响,研究结论可信度较低。而孟德尔随机化方法基于等位基因随机分配给子代的原理,且需满足三个核心假设,类似于随机对照实验,因此能够最大程度避免受到混杂因素及选择偏倚、回忆偏倚的干扰。其次,基因变异先于疾病结局发生,不易产生反向因果关系。另外,孟德尔随机化研究是根据已经公开发布的GWAS汇总数据进行分析,样本量极大,显著增加了检验效能。
本研究仍然存在一些不足。第一,研究采用欧洲人群数据进行统计分析,结论可能无法适用于其他种族,尚需纳入更多国家及种族的人群进行研究。第二,研究获取的GWAS数据只包含了人群SNP等基本信息,缺乏咖啡摄入量、前列腺癌分期的详细信息,无法进行亚组分析。第三,研究虽然样本量较大,且经过严格的质量控制,但仍可能受到潜在未知的混杂因素影响研究结果,需更多独立的大样本研究来排除结论的偶然性。
综上所述,本研究通过两样本孟德尔随机化方法研究摄入咖啡与前列腺癌的因果关系,发现摄入咖啡可能降低前列腺癌发病风险。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
陈劲果:研究设计与实施、数据统计、论文构思与撰写
王之仕:文献检索与筛选
黄卫:研究设计指导、文章审阅及修改
| [1] |
Siegel RL, Miller KD, Fuchs HE, et al. Cancer statistics, 2022[J]. CA Cancer J Clin, 2022, 72(1): 7-33. DOI:10.3322/caac.21708 |
| [2] |
Pernar CH, Ebot EM, Wilson KM, et al. The Epidemiology of Prostate Cancer[J]. Cold Spring Harb Perspect Med, 2018, 8(12): a030361. DOI:10.1101/cshperspect.a030361 |
| [3] |
Matsushita M, Fujita K, Nonomura N. Influence of Diet and Nutrition on Prostate Cancer[J]. Int J Mol Sci, 2020, 21(4): 1447. DOI:10.3390/ijms21041447 |
| [4] |
Zuniga KB, Chan JM, Ryan CJ, et al. Diet and lifestyle considerations for patients with prostate cancer[J]. Urol Oncol, 2020, 38(3): 105-117. DOI:10.1016/j.urolonc.2019.06.018 |
| [5] |
Nieber K. The Impact of Coffee on Health[J]. Planta Med, 2017, 83(16): 1256-1263. DOI:10.1055/s-0043-115007 |
| [6] |
Pauwels E, Volterrani D. Coffee Consumption and Cancer Risk: An Assessment of the Health Implications Based on Recent Knowledge[J]. Med Princ Pract, 2021, 30(5): 401-411. DOI:10.1159/000516067 |
| [7] |
Discacciati A, Orsini N, Andersson SO, et al. Coffee consumption and risk of localized, advanced and fatal prostate cancer: a population-based prospective study[J]. Ann Oncol, 2013, 24(7): 1912-1918. DOI:10.1093/annonc/mdt105 |
| [8] |
Pounis G, Tabolacci C, Costanzo S, et al. Reduction by coffee consumption of prostate cancer risk: Evidence from the Moli-sani cohort and cellular models[J]. Int J Cancer, 2017, 141(1): 72-82. DOI:10.1002/ijc.30720 |
| [9] |
Shafique K, Mcloone P, Qureshi K, et al. Coffee consumption and prostate cancer risk: further evidence for inverse relationship[J]. Nutr J, 2012, 11: 42. DOI:10.1186/1475-2891-11-42 |
| [10] |
Sen A, Papadimitriou N, Lagiou P, et al. Coffee and tea consumption and risk of prostate cancer in the European Prospective Investigation into Cancer and Nutrition[J]. Int J Cancer, 2019, 144(2): 240-250. DOI:10.1002/ijc.31634 |
| [11] |
Greenland S. An introduction to instrumental variables for epidemiologists[J]. Int J Epidemiol, 2018, 47(1): 358. DOI:10.1093/ije/dyx275 |
| [12] |
Yavorska OO, Burgess S. MendelianRandomization: an R package for performing Mendelian randomization analyses using summarized data[J]. Int J Epidemiol, 2017, 46(6): 1734-1739. DOI:10.1093/ije/dyx034 |
| [13] |
Bowden J, Davey SG, Burgess S. Mendelian randomization with invalid instruments: effect estimation and bias detection through Egger regression[J]. Int J Epidemiol, 2015, 44(2): 512-525. DOI:10.1093/ije/dyv080 |
| [14] |
Bowden J, Davey SG, Haycock PC, et al. Consistent Estimation in Mendelian Randomization with Some Invalid Instruments Using a Weighted Median Estimator[J]. Genet Epidemiol, 2016, 40(4): 304-314. DOI:10.1002/gepi.21965 |
| [15] |
Hemani G, Zheng J, Elsworth B, et al. The MR-Base platform supports systematic causal inference across the human phenome[J]. Elife, 2018, 7: e34408. DOI:10.7554/eLife.34408 |
| [16] |
Hemani G, Tilling K, Davey SG. Orienting the causal relationship between imprecisely measured traits using GWAS summary data[J]. PLoS Genet, 2017, 13(11): e1007081. DOI:10.1371/journal.pgen.1007081 |
| [17] |
Li Q, Kakizaki M, Sugawara Y, et al. Coffee consumption and the risk of prostate cancer: the Ohsaki Cohort Study[J]. Br J Cancer, 2013, 108(11): 2381-2389. DOI:10.1038/bjc.2013.238 |
| [18] |
Chen X, Zhao Y, Tao Z, et al. Coffee consumption and risk of prostate cancer: a systematic review and meta-analysis[J]. BMJ Open, 2021, 11(2): e38902. |
| [19] |
Jasiewicz B, Sierakowska A, Jankowski W, et al. Antioxidant and cytotoxic activity of new di- and polyamine caffeine analogues[J]. Free Radic Res, 2018, 52(6): 724-736. DOI:10.1080/10715762.2018.1467561 |
| [20] |
Hang D, Kvaerner AS, Ma W, et al. Coffee consumption and plasma biomarkers of metabolic and inflammatory pathways in US health professionals[J]. Am J Clin Nutr, 2019, 109(3): 635-647. DOI:10.1093/ajcn/nqy295 |
| [21] |
Quarta S, Scoditti E, Carluccio MA, et al. Coffee Bioactive N-Methylpyridinium Attenuates Tumor Necrosis Factor (TNF)-alpha-Mediated Insulin Resistance and Inflammation in Human Adipocytes[J]. Biomolecules, 2021, 11(10): 1545. DOI:10.3390/biom11101545 |
| [22] |
Moua ED, Hu C, Day N, et al. Coffee Consumption and C-Reactive Protein Levels: A Systematic Review and Meta-Analysis[J]. Nutrients, 2020, 12(5): 1349. DOI:10.3390/nu12051349 |
| [23] |
Sfanos KS, Yegnasubramanian S, Nelson WG, et al. The inflammatory microenvironment and microbiome in prostate cancer development[J]. Nat Rev Urol, 2018, 15(1): 11-24. |
| [24] |
Bakuradze T, Lang R, Hofmann T, et al. Coffee consumption rapidly reduces background DNA strand breaks in healthy humans: Results of a short-term repeated uptake intervention study[J]. Mol Nutr Food Res, 2016, 60(3): 682-686. DOI:10.1002/mnfr.201500668 |
| [25] |
Lodise O, Patil K, Karshenboym I, et al. Inhibition of Prostate Cancer Cells by 4, 5-Dicaffeoylquinic Acid through Cell Cycle Arrest[J]. Prostate Cancer, 2019, 2019: 4520645. |
| [26] |
Iwamoto H, Izumi K, Natsagdorj A, et al. Coffee diterpenes kahweol acetate and cafestol synergistically inhibit the proliferation and migration of prostate cancer cells[J]. Prostate, 2019, 79(5): 468-479. DOI:10.1002/pros.23753 |
 2024, Vol. 51
2024, Vol. 51