文章信息
- 原发灶不明肿瘤的诊断技术现状及进展
- Current Status and Progress of Diagnostic Techniques for Cancer of Unknown Primary
- 肿瘤防治研究, 2023, 50(12): 1160-1164
- Cancer Research on Prevention and Treatment, 2023, 50(12): 1160-1164
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.23.0770
- 收稿日期: 2023-07-14
- 修回日期: 2023-09-26
2. 200032 上海,复旦大学上海医学院肿瘤学系;
3. 200032 上海,复旦大学生物医学研究院上海医学表观遗传学重点实验室;
4. 311100 杭州,杭州可帮基因科技有限公司;
5. 221004 徐州,徐州医科大学遗传学教研室,徐州市医学遗传与转化工程研究中心
2. Department of Oncology, Shanghai Medical College, Fudan University, Shanghai 200032, China;
3. Shanghai Key Laboratory of Medical Epigenetics, Institutes of Biomedical Sciences, Fudan University, Shanghai 200032, China;
4. Canhelp Genomics Co., Ltd., Hangzhou 311100, China;
5. Xuzhou Engineering Research Center of Medical Genetics and Transformation, Department of Genetics, Xuzhou Medical University, Xuzhou 221004, China
原发灶不明肿瘤(cancer of unknown primary, CUP)是一种转移性肿瘤,但无法确定其原发部位[1]。其发病率约占所有肿瘤的1%~2%[2],死亡率排名第4位[3]。无法确定原发部位的原因尚不清楚,可能与原发灶太小、生长缓慢,或者免疫系统破坏了微小的原发灶有关[4]。大多数CUP患者只接受局部治疗或经验性全身化疗,预后不良,平均总生存时间为6~9个月。针对CUP的诊断和治疗缺乏充分的循证医学依据[5]。近年来,随着分子生物学技术的进展和精准医疗的实施,国内外学者对该领域的关注逐渐增多[3, 6]。本文主要针对CUP的诊断技术现状及进展作一综述。
1 CUP的临床诊断诊断CUP的首要步骤是进行系统和完整的病史询问和体格检查,重点关注既往病史、活检或病灶切除史、退行性病变史和家族癌症病史[7]。体格检查应尽可能全面,包括泌尿、生殖系统和直肠的检查。男性患者需要进行前列腺检查,女性患者需要进行乳房和盆腔区域的检查[8]。
常用的肿瘤标志物(如CEA、AFP、CA125、CA19-9、CA72-4、CA15-3和PSA等)升高可能提示原发肿瘤的类型,但多数肿瘤标志物的敏感度和特异性较低。因此,肿瘤标志物通常用于监测治疗效果,在确定原发部位方面有限[1, 9]。
CT和MRI是临床上常用的影像学检查方法。胸腹部和盆腔CT是诊断CUP的首选影像学检查,而MRI对怀疑肝脏和乳腺来源的CUP有重要意义[8]。PET-CT检查在CUP的诊断中具有较高的敏感度、特异性和定位作用[10],可提高诊断准确性,曾是识别原发位置最有效的影像学工具。然而,当原发灶较小时,PET-CT对肿瘤溯源的作用有限。一项荟萃分析研究对2795例CUP患者进行了PET-CT检查,发现35%的患者治疗决策因此改变,其中22%的患者发现了原发灶,13%的患者发现了其他转移部位[11]。《中国肿瘤整合诊治指南(CACA):多原发和不明原发肿瘤(2023)》推荐有条件者尽早进行PET-CT检查[8]。
内镜的应用有限,除非存在相关临床症状、组织学或影像学提示可能存在肿瘤的情况,一般不建议将内镜作为常规检查手段。如果怀疑消化道肿瘤,可以选择进行胃肠镜等检查,并对可疑的病灶进行活检;如果怀疑头颈部肿瘤,则需要进行喉镜和鼻咽镜等检查[1, 9]。
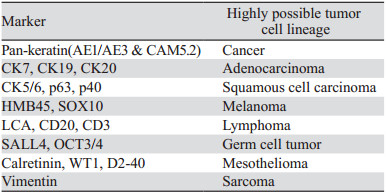
2 CUP的病理学诊断目前,组织病理学检查是对CUP进行诊断的主要方法。根据组织病理学特征,CUP通常可以分为高或中分化腺癌(约占50%)、低分化腺癌或未分化腺癌(约占30%)、鳞状细胞癌(约占15%)和未分化瘤(约占5%)等类型[3]。CUP最常见的部位为淋巴结,常见累及的脏器为骨、肺、肝和脑[12]。免疫组织化学(immunohistochemistry, IHC)是一种常用的定位分析方法,具有高敏感度、直接定位和准确性等特点,被广泛应用于确定肿瘤的谱系。对于CUP的IHC检查,主要包括以下步骤(表 1)[8]:首先确定肿瘤的大类,然后进一步缩小范围以确定肿瘤的亚型,最后通过形态学检查和检测特定IHC标志物来确认原发位点。然而,IHC结果可能受到多种因素的影响,包括活检取材不足、组织异质性、影响组织抗原性的因素以及观察者对结果解读的差异等。
近年来,随着分子生物学技术和生物信息学的快速发展,研究人员能够在短时间内对大量样品进行生物成分的分析,并全面准确地获取样品中的遗传信息。以往的研究表明,转移灶肿瘤细胞的基因表达谱与原发灶细胞更相似,而与转移部位的细胞有差异[13]。基于这一原理,研究人员开发了多种基因表达谱分析方法(gene expression profiling, GEP)用于肿瘤来源的追溯。GEP主要包括基于实时荧光定量PCR(real-time PCR, RT-PCR)和基因芯片两种分析技术。第一种分析技术以Ma等人开发的CancerTYPE ID为代表,该技术包含92个基因,并可以涵盖30种肿瘤类型。多中心临床研究验证结果表明,该方法在判别肿瘤类型方面的准确率达到了87%[14]。我们团队也利用RT-PCR技术研究了CUP的基因表达谱溯源诊断。我们使用支持向量机模型,确定了90个分子特征作为判断CUP原发灶的依据。在包含21种肿瘤的5 434例肿瘤样本中进行验证,验证集的准确率为94.6%[15]。在此基础上,复旦大学附属肿瘤医院牵头进行了一项多中心回顾性研究,包括323例原发性肿瘤和286例转移性肿瘤样本。该研究采用了90基因检测方法(Canhelp-Origin)对样本的组织起源进行判定,并与金标准病理诊断进行比较。总体准确率为90%,对低分化/未分化和鳞状细胞癌的判断准确率分别为90.0%和84.4%[15]。同时,90基因检测方法还能有效鉴别一些罕见肿瘤类型,如肉瘤样间皮瘤、胃肝样腺癌和黏液性肿瘤等。进一步研究评估该方法在真实世界中141例CUP患者辅助诊断的有效性和临床价值,结果显示82.3%的患者可以得到针对性的治疗支持[15]。此外,在多原发肿瘤、三阴性乳腺癌、脑转移性肿瘤和肝转移性肿瘤的原发灶溯源中,90基因检测同样具有良好的临床应用价值,总体准确性为92.0%~97.4%[16-19]。根据以上研究结果,国家药品监督管理局(national medical products administration, NMPA)已于2022年批准90基因检测方法用于对分化程度较差或疑似转移的实体瘤患者的组织溯源。MicroRNAs(miRNAs)在组织中具有高度特异性。除了上述基于mRNA表达谱的分析,肿瘤组织中miRNA的表达谱也可以有效预测肿瘤的原发部位。例如,Rosenfeld等研究通过分析48个miRNA的表达水平,成功识别出22种肿瘤的组织起源,该方法在预测CUP原发灶方面准确率为84%[13]。基于基因芯片技术的分析方法中,有一种被美国食品药品管理局(Food and Drug Administration, FDA)批准的检测方法称为Pathwork Tissue of Origin Test。该检测方法通过检测1 550个基因(冷冻组织)或2 000个基因(石蜡包埋组织)来鉴别15种癌症类型。一项多中心临床研究纳入了462例石蜡包埋组织样本,总体判读准确性为89%[14]。
3.2 高通量测序分析基因突变在肿瘤的发展过程中扮演重要角色。近年来,针对基因变异的靶向药物在实体肿瘤治疗中得到广泛应用,取得了突破性进展,如肺癌、黑色素瘤和乳腺癌等。随着高通量测序技术的进步,我们不断揭示了CUP特异性基因突变谱。有望针对这些特异基因突变进行靶向治疗,这将成为CUP患者的治疗选择之一。一项大型前瞻性研究对200例CUP患者进行了基因检测,并分析了潜在的靶向治疗位点以评估患者对相关靶向治疗的反应性。结果显示,在95%的标本中至少存在1个基因突变,其中85%的患者存在多个潜在的靶向治疗靶点[20]。这表明高通量测序分析有助于筛选CUP患者可能受益于靶向治疗的靶点,尤其是那些临床治疗方案有限、治疗效果较差的患者。另一项研究中,研究者收集了389例CUP患者的石蜡包埋组织样本,并进行了592个基因突变、52个融合基因和免疫标志物的分析。他们共检测出了70个致病或可能致病的基因突变。其中,最常见的突变基因是TP53(53.6%)、KRAS(21.7%)、ARID1A(13.5%)、PIK3CA(8.5%)和CDKN2A(8.1%)[21]。在一项比较靶向治疗/免疫治疗与铂类化疗在CUP患者中的疗效和安全性的Ⅱ期随机对照研究中(CUPISCO,Clinical Trials注册号:NCT03498521),通过分析303例CUP患者的基因变异谱,发现最常见的关键基因突变包括ERBB2(7.3%)、PIK3CA(6.3%)和NF1(5.6%)等,共有31.7%的患者能够匹配到特异性的治疗方案[22]。高通量测序分析不仅可以揭示CUP患者的靶向突变,还可用于鉴定肿瘤的组织起源。纪念斯隆-凯特琳癌症中心的研究团队开发了一种基于468个关键癌症特征基因的基因变异谱分类方法,可以区分出22种肿瘤类型,并为67.4%的CUP患者提供可能的组织起源[23]。另外,Schipper等基于全基因组测序构建了一个名为CUPPA的分类器,涵盖29种肿瘤类型。临床验证结果表明,该分类器可以指导68%的CUP患者进行诊断[24]。
程序性死亡配体1(programmed death-ligand 1, PD-L1)、肿瘤突变负荷(tumor mutation burden, TMB)和微卫星不稳定性(microsatellite instability, MSI)是常用的生物标志物,用于预测免疫治疗的疗效。一项针对303例CUP患者的研究分析了免疫治疗预测标志物。通过高通量测序检测TMB和MSI,并使用IHC方法检测PD-L1表达。在303例患者中,11.6%具有高肿瘤突变负荷(TMB-high),14%存在PD-L1过表达,1%具有高度微卫星不稳定性(MSI-high)[22]。另一项回顾性研究对389例CUP患者进行了分析,同样发现11.8%具有TMB-high,22%存在PD-L1过表达,1.8%具有MSI-high[21]。
3.3 表观遗传学分析表观遗传学是指在不改变DNA序列的情况下调控基因表达的遗传学变化,包括DNA甲基化、基因组印记和RNA编辑等。通过高通量测序的甲基化分析技术,研究人员对1600余例肿瘤患者的甲基化图谱进行分析,鉴定并识别出24种肿瘤特异性的甲基化改变模式[25]。根据该模式构建的EPICUP检测方法在肿瘤溯源判定上的预测准确率为87%,随之进行的特异性治疗可为患者带来总生存获益,患者的中位总生存时间提高了近8个月[26]。2018年,研究人员利用DNA甲基化数据和miRNA数据,分别对17种癌症建立了分类器,在独立测试集上的准确率可达到91.3% [27]。
3.4 液态活检液态活检是一种对患者体液样本中来源于肿瘤的细胞、核酸或蛋白等物质进行分析的技术,是肿瘤诊疗领域的新研究方向之一。已经发现、验证并尝试应用了许多基于液态活检的生物标志物于肿瘤预测、诊断、治疗和复发监测等领域[28]。研究者们在判断CUP的组织起源上也进行了基础性和转化性的研究[29]。通过对循环肿瘤细胞(circulating tumor cell, CTC)进行分选,并结合CK7和CK20等标志物的免疫荧光染色,可以辅助判定CUP的组织起源[30]。此外,通过对肿瘤来源的血小板(tumor-educated platelets, TEPs)进行转录组学测序,并结合人工智能分析,可以识别出6种不同起源的肿瘤类型。随后对228例肿瘤患者进行检测分析,其中位预测准确率为73%[31]。一种名为CancerSEEK的多组学检测方法,基于游离DNA(cell free DNA, cfDNA)和蛋白标志物,可用于早期筛查8种常见的肿瘤类型。在判断肿瘤患者肿瘤起源的过程中,综合前两位判定结果的中位准确率为83%[32]。一项针对442例CUP患者的研究表明,80%的患者循环肿瘤DNA(circulating tumor DNA, ctDNA)中可检测到基因变异,相关结果可用于指导靶向治疗[33]。2017年,Kang等利用ctDNA的检测数据开发了CancerLocator,该方法基于极大似然估计,可以估算ctDNA中来自肿瘤的DNA比例,并判断肿瘤原发位置。在测试集中,对6种癌症进行分类时准确率为73.5%[34]。圣杯(GRAIL)公司对6 689例人群的血浆cfDNA进行测序,并构建了包括50多种肿瘤类型的甲基化分类模型。该模型的总体敏感度为43.9%,特异性为99.3%[35]。
4 结语目前,CUP的诊断与治疗仍然是临床上的难题。由于缺乏循证医学依据,治疗主要基于经验性方法,导致多数患者预后不佳。CUP在不同国家的发病率稍有差异,因此本地区的流行病学特点可能对CUP的诊断和治疗提供帮助。我们团队通过对复旦大学附属肿瘤医院1 420例CUP患者的分析,首次揭示了我国CUP患者的临床病理特征[36]。在CUP的临床诊断中,应注重使用影像学、组织病理学和基因检测等多种检测方法来相互补充、指导和验证诊断。基因表达谱分析、高通量测序分析、表观遗传学分析和液态活检等原发灶判定方法的研究可为后续特异性治疗提供重要参考。虽然早期临床试验初步验证了分子检测在临床应用中的价值,但仍存在争议,需要进行更大规模的随机前瞻性临床试验来提供更有参考价值的证据。靶向治疗、免疫治疗等治疗手段为CUP治疗带来了新的选择,已经进行了一系列随机临床试验。未来,我们迫切需要更多有效的预测和监测标志物,以对CUP患者进行合理分层,并提供更具针对性的治疗选择,实现CUP精准化治疗,从而最终改善CUP患者的生存和生活质量。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
戚鹏:文献调研及论文撰写
孙益丰、徐清华:文献调研
周晓燕:论文设计及指导
| [1] |
Krämer A, Bochtler T, Pauli C, et al. Cancer of unknown primary: ESMO Clinical Practice Guideline for diagnosis, treatment and follow-up[J]. Ann Oncol, 2023, 34(3): 228-246. DOI:10.1016/j.annonc.2022.11.013 |
| [2] |
Rassy E, Pavlidis N. The currently declining incidence of cancer of unknown primary[J]. Cancer Epidemiol, 2019, 61: 139-141. DOI:10.1016/j.canep.2019.06.006 |
| [3] |
Rassy E, Pavlidis N. Progress in refining the clinical management of cancer of unknown primary in the molecular era[J]. Nat Rev Clin Oncol, 2020, 17(9): 541-554. DOI:10.1038/s41571-020-0359-1 |
| [4] |
Olivier T, Fernandez E, Labidi-Galy I, et al. Redefining cancer of unknown primary: Is precision medicine really shifting the paradigm?[J]. Cancer Treat Rev, 2021, 97: 102204. DOI:10.1016/j.ctrv.2021.102204 |
| [5] |
Rassy E, Assi T, Pavlidis N. Exploring the biological hallmarks of cancer of unknown primary: where do we stand today?[J]. Br J Cancer, 2020, 122(8): 1124-1132. DOI:10.1038/s41416-019-0723-z |
| [6] |
Rassy E, Parent P, Lefort F, et al. New rising entities in cancer of unknown primary: Is there a real therapeutic benefit?[J]. Crit Rev Oncol Hematol, 2020, 147: 102882. DOI:10.1016/j.critrevonc.2020.102882 |
| [7] |
Hemminki K, Sundquist K, Sundquist J, et al. Location of metastases in cancer of unknown primary are not random and signal familial clustering[J]. Sci Rep, 2016, 6: 22891. DOI:10.1038/srep22891 |
| [8] |
中国抗癌协会多原发和不明原发肿瘤专业委员会. 中国抗癌协会多原发和不明原发肿瘤诊治指南(2023年版)[J]. 中国癌症杂志, 2023, 33(4): 403-422. [Chinese Anti-Cancer Association Multiple and Unknown Primary Tumor Professional Committee. China Anti-Cancer Association guideline for diagnosis and treatment of cancer of multiple and unknown primaries (2023 edition)[J]. Zhongguo Ai Zheng Za Zhi, 2023, 33(4): 403-422.] |
| [9] |
Losa F, Fernández I, Etxaniz O, et al. SEOM-GECOD clinical guideline for unknown primary cancer (2021)[J]. Clin Transl Oncol, 2022, 24(4): 681-692. DOI:10.1007/s12094-022-02806-x |
| [10] |
Wolpert F, Weller M, Berghoff AS, et al. Diagnostic value of 18F-fluordesoxyglucose positron emission tomography for patients with brain metastasis from unknown primary site[J]. Eur J Cancer, 2018, 96: 64-72. DOI:10.1016/j.ejca.2018.03.010 |
| [11] |
Woo S, Becker AS, Do RKG, et al. Impact of 18F-Fluorodeoxyglucose positron emission tomography on management of cancer of unknown primary: systematic review and meta-analysis[J]. Eur J Cancer, 2021, 159: 60-77. DOI:10.1016/j.ejca.2021.09.031 |
| [12] |
Kolling S, Ventre F, Geuna E, et al. "Metastatic Cancer of Unknown Primary" or "Primary Metastatic Cancer"?[J]. Front Oncol, 2020, 9: 1546. DOI:10.3389/fonc.2019.01546 |
| [13] |
Conway AM, Mitchell C, Kilgour E, et al. Molecular characterisation and liquid biomarkers in Carcinoma of Unknown Primary (CUP): taking the "U" out of "CUP"[J]. Br J Cancer, 2019, 120(2): 141-153. DOI:10.1038/s41416-018-0332-2 |
| [14] |
Lee MS, Sanoff HK. Cancer of unknown primary[J]. BMJ, 2020, 371: m4050. |
| [15] |
Ye Q, Wang Q, Qi P, et al. Development and Clinical Validation of a 90-Gene Expression Assay for Identifying Tumor Tissue Origin[J]. J Mol Diagn, 2020, 22(9): 1139-1150. DOI:10.1016/j.jmoldx.2020.06.005 |
| [16] |
Wang Q, Xu M, Sun Y, et al. Gene Expression Profiling for Diagnosis of Triple-Negative Breast Cancer: A Multicenter, Retrospective Cohort Study[J]. Front Oncol, 2019, 9: 354. DOI:10.3389/fonc.2019.00354 |
| [17] |
Zheng Y, Ding Y, Wang Q, et al. 90-gene signature assay for tissue origin diagnosis of brain metastases[J]. J Transl Med, 2019, 17(1): 331. DOI:10.1186/s12967-019-2082-1 |
| [18] |
Zheng Y, Sun Y, Kuai Y, et al. Gene expression profiling for the diagnosis of multiple primary malignant tumors[J]. Cancer Cell Int, 2021, 21(1): 47. DOI:10.1186/s12935-021-01748-8 |
| [19] |
Wang Q, Li F, Jiang Q, et al. Gene Expression Profiling for Differential Diagnosis of Liver Metastases: A Multicenter, Retrospective Cohort Study[J]. Front Oncol, 2021, 11: 725988. DOI:10.3389/fonc.2021.725988 |
| [20] |
Ross JS, Wang K, Gay L, et al. Comprehensive Genomic Profiling of Carcinoma of Unknown Primary Site: New Routes to Targeted Therapies[J]. JAMA Oncol, 2015, 1(1): 40-49. DOI:10.1001/jamaoncol.2014.216 |
| [21] |
Gatalica Z, Xiu J, Swensen J, et al. Comprehensive analysis of cancers of unknown primary for the biomarkers of response to immune checkpoint blockade therapy[J]. Eur J Cancer, 2018, 94: 179-186. DOI:10.1016/j.ejca.2018.02.021 |
| [22] |
Ross JS, Sokol ES, Moch H, et al. Comprehensive Genomic Profiling of Carcinoma of Unknown Primary Origin: Retrospective Molecular Classification Considering the CUPISCO Study Design[J]. Oncologist, 2021, 26(3): e394-e402. DOI:10.1002/onco.13597 |
| [23] |
Penson A, Camacho N, Zheng Y, et al. Development of Genome-Derived Tumor Type Prediction to Inform Clinical Cancer Care[J]. JAMA Oncol, 2020, 6(1): 84. DOI:10.1001/jamaoncol.2019.3985 |
| [24] |
Schipper LJ, Samsom KG, Snaebjornsson P, et al. Complete genomic characterization in patients with cancer of unknown primary origin in routine diagnostics[J]. ESMO Open, 2022, 7(6): 100611. DOI:10.1016/j.esmoop.2022.100611 |
| [25] |
Fernandez AF, Assenov Y, Martin-Subero JI, et al. A DNA methylation fingerprint of 1628 human samples[J]. Genome Res, 2012, 22(2): 407-419. DOI:10.1101/gr.119867.110 |
| [26] |
Moran S, Martínez-Cardús A, Sayols S, et al. Epigenetic profiling to classify cancer of unknown primary: a multicentre, retrospective analysis[J]. Lancet Oncol, 2016, 17(10): 1386-1395. DOI:10.1016/S1470-2045(16)30297-2 |
| [27] |
Tang W, Wan S, Yang Z, et al. Tumor origin detection with tissue-specific miRNA and DNA methylation markers[J]. Bioinformatics, 2018, 34(3): 398-406. DOI:10.1093/bioinformatics/btx622 |
| [28] |
Im YR, Tsui DWY, Diaz LA Jr, et al. Next-Generation Liquid Biopsies: Embracing Data Science in Oncology[J]. Trends Cancer, 2021, 7(4): 283-292. DOI:10.1016/j.trecan.2020.11.001 |
| [29] |
El Rassy E, Khaled H, Pavlidis N. Liquid biopsy: a new diagnostic, predictive and prognostic window in cancers of unknown primary[J]. Eur J Cancer, 2018, 105: 28-32. DOI:10.1016/j.ejca.2018.09.035 |
| [30] |
Lu SH, Tsai WS, Chang YH, et al. Identifying cancer origin using circulating tumor cells[J]. Cancer Biol Ther, 2016, 17(4): 430-438. DOI:10.1080/15384047.2016.1141839 |
| [31] |
Best MG, Sol N, Kooi I, et al. RNA-Seq of Tumor-Educated Platelets Enables Blood-Based Pan-Cancer, Multiclass, and Molecular Pathway Cancer Diagnostics[J]. Cancer Cell, 2015, 28(5): 666-676. DOI:10.1016/j.ccell.2015.09.018 |
| [32] |
Cohen JD, Li L, Wang Y, et al. Detection and localization of surgically resectable cancers with a multi-analyte blood test[J]. Science, 2018, 359(6378): 926-930. DOI:10.1126/science.aar3247 |
| [33] |
Kato S, Krishnamurthy N, Banks KC, et al. Utility of Genomic Analysis In Circulating Tumor DNA from Patients with Carcinoma of Unknown Primary[J]. Cancer Res, 2017, 77(16): 4238-4246. DOI:10.1158/0008-5472.CAN-17-0628 |
| [34] |
Kang S, Li Q, Chen Q, et al. CancerLocator: non-invasive cancer diagnosis and tissue-of-origin prediction using methylation profiles of cell-free DNA[J]. Genome Biol, 2017, 18(1): 53. DOI:10.1186/s13059-017-1191-5 |
| [35] |
Liu MC, Oxnard GR, Klein EA, et al. Sensitive and specific multi-cancer detection and localization using methylation signatures in cell-free DNA[J]. Ann Oncol, 2020, 31(6): 745-759. DOI:10.1016/j.annonc.2020.02.011 |
| [36] |
Qi P, Sun Y, Liu X, et al. Clinicopathological, molecular and prognostic characteristics of cancer of unknown primary in China: An analysis of 1420 cases[J]. Cancer Med, 2023, 12(2): 1177-1188. DOI:10.1002/cam4.4973 |
 2023, Vol. 50
2023, Vol. 50