文章信息
- CRISPR/Cas9介导的KIFC1基因敲除对宫颈癌细胞增殖及凋亡的影响
- CRISPR/Cas9-mediated Knockout of KIFC1 Inhibits Proliferation and Induces Apoptosis of Cervical Cancer Cells
- 肿瘤防治研究, 2023, 50(11): 1051-1058
- Cancer Research on Prevention and Treatment, 2023, 50(11): 1051-1058
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.23.0611
- 收稿日期: 2023-06-08
- 修回日期: 2023-08-21
2. 350013 福州,国家卫健委非人灵长类生育调节技术评价重点实验室(福建省妇幼保健院);
3. 350001 福州,福建省妇幼保健院医学研究中心
2. NHC Key Laboratory of Technical Evaluation of Fertility Regulation for Non-human Primate (Fujian Maternity and Child Health Hospital), Fuzhou 350013, China;
3. Medical Research Center, Fujian Maternity and Child Health Hospital, Fuzhou 350001, China
驱动蛋白14家族成员C1(Kinesin family member C1, KIFC1, NP_002254)是一种从微管正极端向负极端运动的驱动蛋白,KIFC1可以促进微管间的交联、滑动和捆绑,在有丝分裂过程中参与纺锤体极体的聚集和组装,并在含有多个中心体的癌细胞中参与中心体的聚集。关于KIFC1的重要性尚存在争议,但在癌细胞中,KIFC1的过量表达是维持多个中心体肿瘤细胞正常分裂所必需的[1-2]。研究发现KIFC1在乳腺癌[3]、卵巢癌[4]、肝癌[5-6]、胃癌[7]、前列腺癌[8]、胰腺癌[9]等恶性肿瘤中特异性过表达,并可促进肿瘤细胞的增殖、迁移和上皮间质转化[4]。KIFC1通过调节PI3K/AKT信号通路调控子宫内膜癌进程,其表达量与子宫内膜癌的临床分期、病理类型及预后显著相关[10]。此外,KIFC1已被确定为三阴性乳腺癌患者预后不良的生物标志物之一[11]。KIFC1也可诱导多西他赛耐药,并与前列腺癌患者的不良预后有关[12]。敲低KIFC1可以抑制肝癌细胞增殖和侵袭,增加其对紫杉醇的敏感度[13]。因此,KIFC1作为肿瘤选择性治疗的靶点,值得进一步探讨。然而,关于KIFC1在宫颈癌发病机制中的作用研究较少,具体调控机制尚不清楚。
CRISPR/Cas9系统是目前最主流的基因编辑系统,具有效率高、成本低、操作简便等优点,广泛运用于基因敲除、基因敲入和多重基因编辑。本研究运用CRISPR/Cas9系统在人宫颈癌HeLa细胞中构建KIFC1基因敲除细胞系,探讨KIFC1表达缺失对细胞形态、细胞增殖、周期与凋亡的影响,为进一步研究KIFC1蛋白在宫颈癌发生发展中的作用提供细胞模型。
1 材料与方法 1.1 主要材料与试剂人宫颈癌细胞系HeLa(来源于ATCC细胞库CCL-2编号的细胞系)、CRISPR/Cas9系统质粒pSpCas9(BB)-2A-GFP(简称PX458)源于本实验室保存,DMEM培养基、胎牛血清和0.25%胰酶均购自美国Gibco公司,兔抗人KIFC1和鼠抗beta-actin单克隆抗体均购自英国Abcam公司,辣根过氧化物酶(HRP)标记二抗购自美国Cell Signaling Technology公司,TurboFect转染试剂(R0531)、T4 DNA连接酶(EL0016)和BbsⅠ酶(ER1011)均购自美国Thermo Scientific公司,感受态细胞DH5α(C502-03)、染料法qPCR预混液(Q711-02)、TA克隆试剂盒(C601-01)和Annexin V-FITC/PI凋亡检测试剂盒(A211-02)均购自南京诺唯赞公司,ExTaq酶(RR001Q)和反转录试剂盒(RR047A)均购自日本TaKaRa公司,PMSF(A610425)、RIPA缓冲液(C500005)、EdU细胞增殖检测试剂盒(E607204)和吖啶橙染色试剂盒(E607307)均购自上海生工公司,细胞周期与凋亡检测试剂盒(C1052)购自上海碧云天公司,细胞基因组DNA提取试剂盒(DP304)、无内毒素质粒小提中量试剂盒(DP118)、琼脂糖凝胶DNA回收试剂盒(DP219)、细胞总RNA提取试剂盒(DP451)均购自天根生化科技(北京)有限公司,引物合成和DNA测序由铂尚生物技术(上海)有限公司完成。
1.2 实验方法 1.2.1 靶向人KIFC1基因sgRNA的设计及表达载体的构建在美国国家生物技术信息中心官网(https://www.ncbi.nlm.nih.gov/)检索Homo sapiens KIFC1基因序列(NM_002263.4),将序列输入到在线sgRNA设计网站(http://chopchop.cbu.uib.no/),根据预测结果设计两对sgRNA序列(见表 1)和测序引物hKIFC1 target(见表 2)。sgRNA与BbsⅠ酶切后的PX458质粒连接,转化到大肠杆菌,筛选阳性克隆,提取质粒鉴定。
HeLa细胞在含10%胎牛血清的DMEM培养基中培养,待细胞密度达到70%时,按照转染试剂说明书分组转染,其中实验组为PX458-sgRNA1+PX458-sgRNA2,阴性对照组为PX458质粒,空白对照组为脂质体试剂。转染48和72 h后,使用倒置荧光显微镜(Nikon, Ts2R-FL)观察绿色荧光蛋白表达情况以确定转染效果。
1.2.3 KIFC1敲除单克隆HeLa细胞株的筛选及鉴定转染72 h后的HeLa细胞经胰酶消化吹打成单细胞悬液,流式细胞术分选收集绿色荧光蛋白表达阳性的细胞悬液。采用有限稀释法将单个细胞接种至96孔板中进行培养,待单孔细胞增殖至50%融合度时,传代并扩大培养获得单克隆细胞株。
提取细胞基因组DNA,进行PCR扩增。PCR扩增体系为:PCR程序为:95℃ 3 min;95℃ 15 s,55℃ 20 s,72℃ 30 s,33个循环;72℃ 10 min,4℃保温。胶回收PCR产物并进行TA克隆,挑选单克隆菌落进行菌落PCR验证及测序分析。最终以KIFC1敲除细胞株为实验组,野生型HeLa细胞为对照组进行后续实验操作。
1.2.4 Western blot检测KIFC1蛋白表达情况使用含1%PMSF的RIPA缓冲液裂解细胞,提取细胞总蛋白。制备好的蛋白样品进行SDS-PAGE,电转到PVDF膜上,5%脱脂奶粉封闭1 h,加入一抗4℃孵育过夜,HRP标记二抗孵育1 h,TBST洗涤3次,在膜上滴加显影液,放入显影系统(Bio-Rad,ChemiDoc MP)中成像。
1.2.5 RT-qPCR检测KIFC1基因转录水平提取细胞RNA,进行反转录反应。以cDNA为模板,采用SYBR Green染料法进行实时荧光定量PCR检测。qPCR反应体系为:2×ChamQ Universal SYBR qPCR Master Mix 10 μl,ddH2O上下游引物各1 μl,模板cDNA 1 μl,补ddH2O至20 μl。qPCR反应程序为:95℃ 30 s, 95℃ 10 s, 60℃ 30 s,40个循环;融解曲线95℃ 15 s,60℃ 60 s,95℃ 15 s。
1.2.6 免疫荧光法检测敲除KIFC1对细胞形态结构的影响将细胞接种于含12 mm爬片的24孔板中,待细胞贴壁生长后,吸弃培养基,PBS漂洗1次后,4%多聚甲醛固定20 min,PBS浸洗3次。0.25% Triton X-100/PBS透化10 min,PBS浸洗3次,用含3%BSA/PBST的封闭液室温孵育1 h,加入一抗4℃孵育过夜,二抗室温避光孵育1 h,两步分别用PBS浸洗3次去除多余抗体,DAPI与抗荧光淬灭剂等体积混匀封片,激光扫描共聚焦显微镜(Leica SP8)成像。
1.2.7 细胞生长曲线绘制及EdU染色细胞生长曲线绘制:各组细胞用胰酶消化成单细胞悬液并进行计数,调整细胞浓度为5×104/ml,接种至24孔板中正常培养。24 h后开始计数,连续计数8 d,绘制细胞生长曲线。
EdU染色:细胞爬片过夜培养后,按EdU染色试剂盒说明书进行染色,共聚焦显微镜(Leica SP8)获取图像,检测细胞增殖。
1.2.8 流式细胞术检测细胞周期收集各组细胞,使用预冷的PBS洗涤和重悬,缓慢加入预冷的75%乙醇,4℃固定过夜。次日,PBS洗涤,加入碘化丙啶(PI)37℃避光染色30 min,上流式细胞仪(BD LSRFortessa)检测,记录激发波长488 nm处的红色荧光。
1.2.9 流式细胞术及吖啶橙染色法检测细胞凋亡流式细胞术:细胞接种至6孔板中培养48 h后,胰酶消化收集细胞,按Annexin V-FITC/PI凋亡检测试剂盒说明书操作,1 h内上流式细胞仪(BD LSRFortessa)检测。
吖啶橙(AO)染色法:细胞消化后,PBS洗涤两次,按吖啶橙染色试剂盒说明书进行染色,荧光显微镜观察成像。
1.3 统计学方法所有实验均重复3次,采用GraphPad Prism 7.0和Microsoft Excels软件进行数据分析,结果以均值±标准差表示,两组间比较采用t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 KIFC1在HeLa细胞中的时空表达定位分析为评估KIFC1在HeLa细胞分裂中的功能,我们对KIFC1在细胞间期与分裂期的表达情况及亚细胞定位进行分析,利用免疫荧光技术对HeLa细胞的细胞核、微管及KIFC1蛋白进行荧光标记,测量荧光强度和相对细胞位置,见图 1A。结果显示,在细胞间期,KIFC1分散定位在细胞质中或大量转运进入细胞核,见图 1B。分裂期,尤其是在有丝分裂后期,KIFC1大量定位在中央纺锤体与星极纺锤体区,参与牵拉染色体的分离,见图 1C。
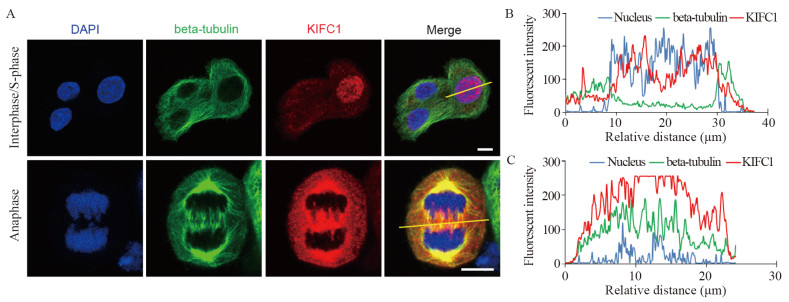
|
| A: immunofluorescence staining showed the expression of KIFC1 in HeLa cells, bars: 10μm; B: location of KIFC1 in the intercellular phase; C: location of KIFC1 during cell division. 图 1 KIFC1在HeLa细胞间期与分裂期的表达定位 Figure 1 Location of KIFC1 in HeLa cells during interphase and mitosis |
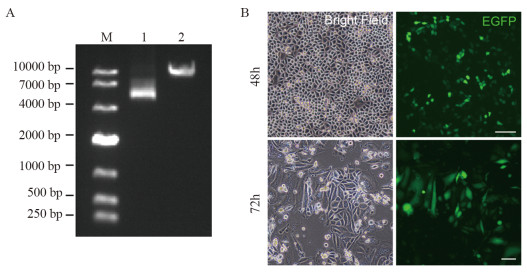
利用BbsⅠ酶对PX458质粒进行线性化处理,将合成的sgRNA序列与线性化PX458质粒连接,经转化、涂板、测序验证,构建sgRNA-Cas9重组载体,见图 2。该载体含有人U6启动子,带有Cas9和EGFP标签,并具有氨苄青霉素抗性。琼脂糖凝胶电泳结果显示,酶切成功的质粒在相应位置出现明显条带,见图 3A。
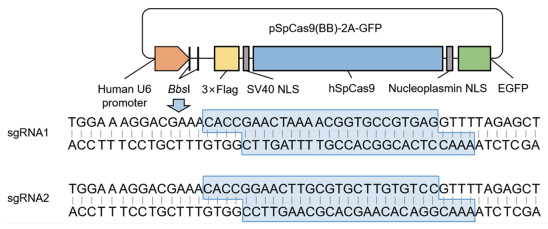
|
| 图 2 sgRNA-Cas9重组载体构建模式图 Figure 2 Schematic diagram of sgRNA-Cas9 recombinant vector construction |
|
| A: agarose gel electrophoresis of restriction enzyme-digested PX458 plasmid (M: DNA marker; 1: native undigested PX458 plasmid; 2. PX458 plasmid digested with BbsⅠrestriction enzyme); B: HeLa cells at 48h and 72h post transfection, bars: 50μm. 图 3 转染质粒后EGFP阳性细胞形态 Figure 3 EGFP positive cell morphology after plasmid transfection |
将PX458-sgRNA1和PX458-sgRNA2质粒共转染至HeLa细胞中,使用倒置荧光显微镜观察转染效果,转染48 h后可见多数细胞成功表达EGFP信号,72 h后大量EGFP阳性细胞表现出巨核或多细胞核的表型,见图 3B。
2.4 流式细胞术分选EGFP阳性细胞通过荧光流式细胞分选技术,收集EGFP强信号的细胞群,检测其转染效率为28.6%,见图 4A,收集该部分细胞群。经过荧光显微镜镜检,可以观察到所有分选后的细胞都携带绿色荧光,见图 4B。
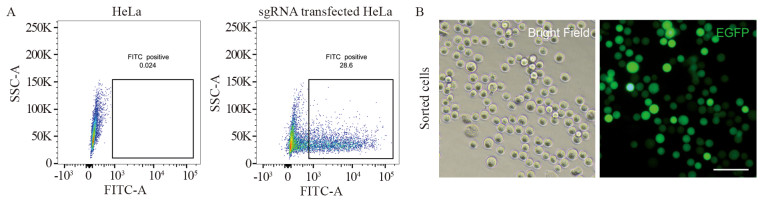
|
| A: flow cytometric sorting of EGFP-positive cells at 72h after transfection; B: fluorescence microscope images of sorted cells, bar: 50μm. 图 4 流式细胞术分选EGFP阳性HeLa细胞群 Figure 4 Flow cytometry sorting of EGFP-positive HeLa cells |
靶序列测序分析显示,KIFC1敲除细胞在386 bp和563 bp位点各缺失1个鸟嘌呤(G),在564 bp位点缺失1个胸腺嘧啶(T),见图 5A,这些碱基缺失可以造成KIFC1蛋白截断翻译。与对照HeLa细胞相比,敲除细胞株中KIFC1蛋白表达量和转录水平明显下降,见图 5B~D,与预期结果一致,证明HeLa细胞KIFC1基因敲除成功。
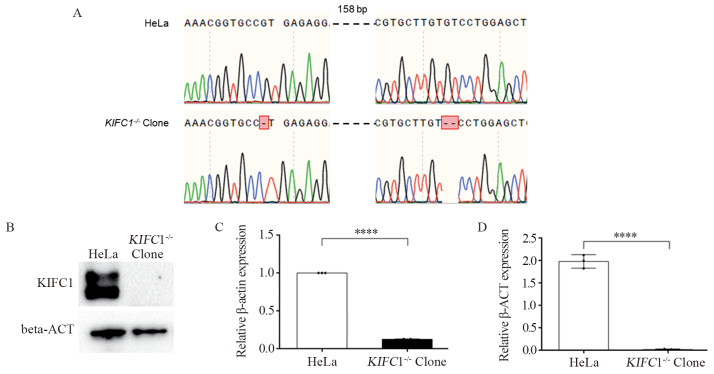
|
| A: nucleotide sequence alignment results of KIFC1 knockout cells and wild-type cells; B, C: Western blot and quantitative analyses of KIFC1 protein expression; D: RT-qPCR analysis of the transcriptional levels of KIFC1; ****: P < 0.0001. 图 5 敲除细胞KIFC1转录与蛋白表达水平验证 Figure 5 Verification of KIFC1 transcription and protein expression levels in knock-out cells |
在KIFC1敲除株中,大量细胞核表现出多核、微核、染色质异常包装和微管骨架装配错误,见图 6。细胞骨架异常延伸并过度组装,但细胞的贴壁能力明显变弱,细胞的增殖能力降低,部分细胞在生长过程中表现出明显的凋亡或死亡,见图 7A。
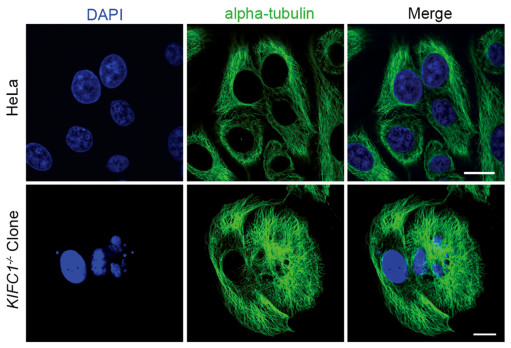
|
| KIFC1-/- cell exhibited abnormal microtubule assembly and multinclei, bars: 10μm. 图 6 KIFC1敲除细胞微管骨架形态分析 Figure 6 Morphological analysis of microtubule cytoskeleton in KIFC1 knockout cells |
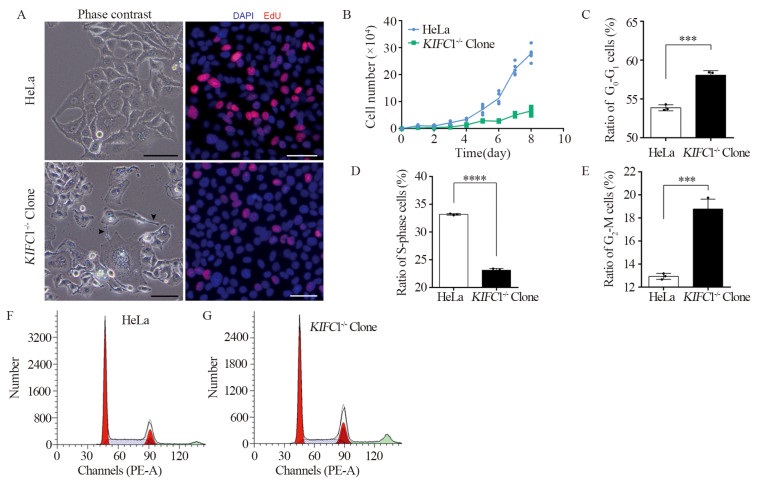
|
| A: cell proliferation showed by EdU staining, arrows: abnormal morphologies of cell, bars: 50μm; B: cell growth curves; C: proportion of cells in G0/G1 phase, ***: P < 0.001; D: proportion of cells in S phase, ****: P < 0.0001; E: proportion of cells in G2/M phase, ***: P < 0.001; F, G: flow cytometry cell cycle analysis of control groups and knockout cells. 图 7 KIFC1缺失对HeLa细胞形态、增殖及周期的影响 Figure 7 Effects of KIFC1 deletion on morphology, proliferation and cell cycle of HeLa cells |
EdU染色结果显示,与对照组相比,KIFC1敲除组中EdU标记的细胞明显减少,且DNA复制强阳性信号的细胞数量较少, 见图 7A。通过分析细胞生长曲线,KIFC1敲除组细胞生长速率显著低于对照组,见图 7B。以上结果均表明KIFC1敲除可使HeLa细胞增殖能力下降。
流式细胞术检测细胞周期分布结果显示,细胞周期调控发生显著的变化。其中,KIFC1敲除组G0/G1期和G2/M期细胞比例均显著高于对照组(P < 0.05),而S期细胞比例则明显低于对照组,提示细胞DNA合成受到抑制,见图 7C~G,进一步印证了KIFC1敲除导致HeLa细胞的增殖水平降低。
2.8 敲除KIFC1可促进HeLa细胞凋亡KIFC1敲除组细胞经吖啶橙染色后,通过荧光显微镜可观察到不同程度的细胞凋亡情况,凋亡细胞核呈致密浓染的黄绿色,而对照组细胞的细胞核表现为均匀的绿色荧光,见图 8A。
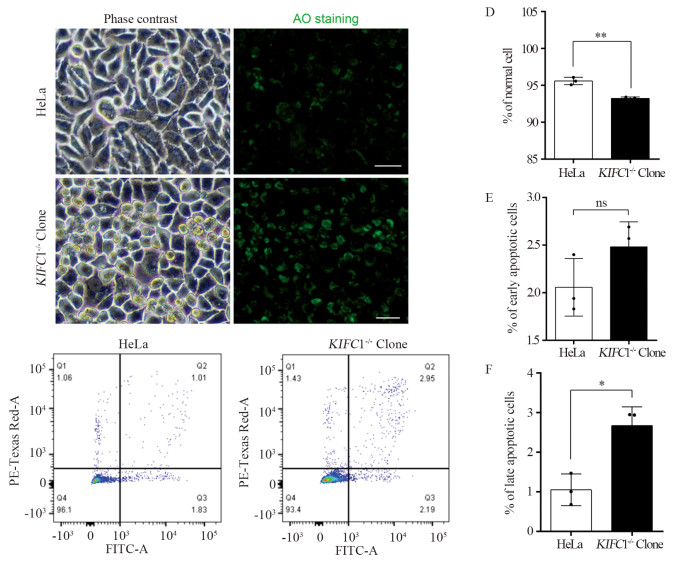
|
| A: detection of cell apoptosis by acridine orange staining, bars: 50μm; B, C: flow cytometry analysis of cell apoptosis; D: percentage of normal cells, **: P < 0.01; E: percentage of early apoptotic cells; F: percentage of late apoptotic cells, *: P < 0.05. 图 8 KIFC1缺失对HeLa细胞凋亡的影响 Figure 8 Effects of KIFC1 deletion on HeLa cell apoptosis |
通过Annexin/FITC共标的流式细胞术检测进一步发现,敲除KIFC1后正常细胞比例减少(P < 0.01; HeLa, 95.60±0.29%; KIFC1-/- Clone, 93.23±0.12%),见图 8B~D,早期凋亡细胞比例有所增加,见图 8E,晚期凋亡细胞比例显著增加(P < 0.05;HeLa,1.05±0.23%;KIFC1-/- Clone,2.67±0.28%),见图 8F。综上结果表明,敲除KIFC1可促进HeLa细胞凋亡。
3 讨论驱动蛋白是一类沿着微管运输分子的马达蛋白,在细胞器运输、细胞分裂、组织器官发育、信号转导等过程中发挥重要作用[14]。驱动蛋白的异常表达影响染色体的正确分布,引起有丝分裂过程中遗传物质变化和异常纺锤体形成,促进癌症的发生发展。近年来,驱动蛋白已经成为癌症药物开发的潜在靶点[15-18]。其中KIFC1是多种癌症诊断、治疗和预后的潜在生物标志物。KIFC1蛋白通过聚集中心体在癌细胞有丝分裂中发挥关键作用[19]。研究表明,KIFC1蛋白可直接结合CEP215,促进中心体与纺锤体极的连接,从而提高中心体聚集[20]。KIFC1还可能参与染色体排列、细胞周期中细胞器的运输,调节细胞周期G2-M期的进程。KIFC1的过表达驱动了控制有丝分裂检查点基因的过量表达,从而产生非整倍体,加速癌症发展的进程[21]。KIFC1蛋白参与多条肿瘤形成相关的信号通路。在子宫内膜癌中,KIFC1通过结合HMGA1促进c-myc的表达,从而加快c-myc介导的下游糖酵解基因的表达[22]。在肝癌中,KIFC1通过激活gankyrin/AKT/TWIST1通路,促进上皮间质转化和癌症转移[23]。KIFC1还可以通过Akt/GSK3β信号通路加速膀胱癌细胞增殖并诱导上皮间质转化[24]。在多种类型癌组织中,KIFC1的表达随着肿瘤发展级别的增加而增加,且KIFC1高表达易引起耐药性,与癌症患者的不良预后相关[25-28]。近期研究表明,DNA修复途径的ATM和ATR激酶诱导KIFC1-Ser26磷酸化,促进中心体聚集,导致耐药性和肿瘤复发[29]。这些研究揭示了KIFC1可能多方面参与癌症的发生发展过程。
目前KIFC1蛋白相关的研究通常使用基于RNAi机制的敲低细胞模型,我们利用CRISPR/Cas9技术在宫颈癌HeLa细胞中成功构建KIFC1敲除模型,可以更好地阐述KIFC1在宫颈癌中的作用机制。免疫荧光实验结果显示,在细胞间期,KIFC1大量定位在细胞核中,这与KIFC1尾部结构域的核定位序列(NLS)有关,KIFC1可与入核转运蛋白复合体中的Importin β结合,入核参与细胞周期的调控,并且其与核孔复合物NUP62相互作用可能介导核孔的重组[30]。在有丝分裂中后期,KIFC1与纺锤体共定位,表明KIFC1主要作用于纺锤体。在细胞分裂过程中,KIFC1参与微管交联、纺锤体两级形成、纺锤体组装和染色体分离等生命活动过程。之前的研究发现,KIFC1敲低可延长细胞的前中期,延缓细胞周期蛋白A的降解,进而破坏染色体凝集和排列,导致染色体错位、核型异常及微核的形成[31]。本研究观察到类似的结果,KIFC1基因的敲除可使细胞形态发生明显的改变,出现多核、微核等异常核结构,诱发微管骨架紊乱,还可以阻滞细胞周期,抑制DNA合成并诱导细胞凋亡,进而抑制HeLa细胞的增殖。这一结果提示作为宫颈癌治疗的潜在靶点,KIFC1具有广阔的发展前景。
综上所述,本研究以宫颈癌HeLa细胞系为研究模型,通过CRISPR/Cas9基因编辑技术敲除KIFC1基因,成功抑制KIFC1蛋白的表达。研究结果发现,KIFC1敲除可导致细胞骨架的异常装配,诱导细胞阻滞在G0/G1期、G2/M期,诱导HeLa细胞凋亡,从而减少细胞的增殖。未来,在KIFC1敲除细胞模型基础上可以结合临床实际问题如肿瘤治疗耐药性,深入研究KIFC1在宫颈癌中的潜在功能机制,为宫颈癌筛查、诊断和治疗提供新的思路。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
范晓静:实验设计及实施、数据整理、文章撰写
魏雅岚:实验设计及实施、数据统计与分析、文章审阅与修订
叶洲杰、朱丽萍:实验实施
王心睿:实验设计、经费支持、文章审阅与修订
| [1] |
Rath O, Kozielski F. Kinesins and cancer[J]. Nat Rev Cancer, 2012, 12(8): 527-539. DOI:10.1038/nrc3310 |
| [2] |
Firdous F, Raza HG, Chotana GA, et al. Centrosome clustering and chemotherapy[J]. Mini Rev Med Chem, 2023, 23(4): 429-451. DOI:10.2174/1389557522666220820113953 |
| [3] |
Wright N, Gong Z, Kittles R, et al. Kinesin family member C1 (KIFC1/HSET): a potential actionable biomarker of early stage breast tumorigenesis and progression of high-risk lesions[J]. J Pers Med, 2021, 11(12): 1361. DOI:10.3390/jpm11121361 |
| [4] |
Li JN, Diao HD, Guan X, et al. Kinesin Family Member C1 (KIFC1) Regulated by Centrosome Protein E (CENPE) Promotes Proliferation, Migration, and Epithelial-Mesenchymal Transition of Ovarian Cancers[J]. Med Sci Monit, 2020, 26: e927869. |
| [5] |
Li D, Yu T, Han JJ, et al. Prognosticvalue and immunological role of KIFC1 in hepatocellular carcinoma[J]. Front Mol Biosci, 2021, 8: 799651. |
| [6] |
Wang X, Wang M, Li XY, et al. KIFC1 promotes the proliferation of hepatocellular carcinoma in vitro and in vivo[J]. Oncol Lett, 2019, 18(6): 5739-5746. |
| [7] |
Jung J, Jeong H, Choi JW, et al. Increased expression levels of AURKA and KIFC1 are promising predictors of progression and poor survival associated with gastric cancer[J]. Pathol Res Pract, 2021, 224: 153524. DOI:10.1016/j.prp.2021.153524 |
| [8] |
Kostecka LG, Olseen A, Kang K, et al. High KIFC1 expression is associated with poor prognosis in prostate cancer[J]. Med Oncol, 2021, 38(5): 47. DOI:10.1007/s12032-021-01494-x |
| [9] |
Ishikawa A, Fujii H, Fukui T, et al. Expression of kinesin family member C1 in pancreatic ductal adenocarcinoma affects tumor progression and stemness[J]. Pathol Res Pract, 2023, 241: 154277. DOI:10.1016/j.prp.2022.154277 |
| [10] |
Zhou K, Zhao J, Qi L, et al. Kinesin Family Member C1 (KIFC1) Accelerates Proliferation and Invasion of Endometrial Cancer Cells Through Modulating the PI3K/AKT Signaling Pathway[J]. Technol Cancer Res Treat, 2020, 19: 1533033820964217. |
| [11] |
Li Y, Lu W, Chen D, et al. KIFC1 is a novel potential therapeutic target for breast cancer[J]. Cancer Biol Ther, 2015, 16(9): 1316-1322. DOI:10.1080/15384047.2015.1070980 |
| [12] |
Parvin A, Hao SL, Tan FQ, et al. Inhibition of kinesin motor protein KIFC1 by AZ82 induces multipolar mitosis and apoptosis in prostate cancer cell[J]. Gene, 2020, 760: 144989. DOI:10.1016/j.gene.2020.144989 |
| [13] |
Teng K, Wei S, Zhang C, et al. KIFC1 is activated by TCF-4 and promotes hepatocellular carcinoma pathogenesis by regulating HMGA1 transcriptional activity[J]. J Exp Clin Cancer Res, 2019, 38(1): 329. DOI:10.1186/s13046-019-1331-8 |
| [14] |
Konjikusic MJ, Gray RS, Wallingford JB. The developmental biology of kinesins[J]. Dev Biol, 2021, 469: 26-36. DOI:10.1016/j.ydbio.2020.09.009 |
| [15] |
Zhou Y, Yang LP, Xiong L, et al. KIF11 is upregulated in colorectal cancer and silencing of it impairs tumor growth and sensitizes colorectal cancer cells to oxaliplatin via p53/GSK3β signaling[J]. J Cancer, 2021, 12(12): 3741-3753. DOI:10.7150/jca.52103 |
| [16] |
Cohen-Shair Y, Mcfarland JM, Abdusamad M, et al. Aneuploidy renders cancer cells vulnerable to mitotic checkpoint inhibition[J]. Nature, 2021, 590(7846): 486-491. DOI:10.1038/s41586-020-03114-6 |
| [17] |
Sheng N, Xu YZ, Xi QH, et al. Overexpression of KIF2A is suppressed by miR-206 and associated with poor prognosis in ovarian cancer[J]. Cell Physiol Biochem, 2018, 50(2): 810-822. |
| [18] |
Mandal K, Pogoda K, Nandi S, et al. Role of a kinesin motor in cancer cell mechanics[J]. Nano Lett, 2019, 19(11): 7691-7702. DOI:10.1021/acs.nanolett.9b02592 |
| [19] |
Kleylein-Sohn J, Pöllinger B, Ohmer M, et al. Acentrosomal spindle organization renders cancer cells dependent on the kinesin HSET[J]. J Cell Sci, 2012, 125(Pt 22): 5391-5402. |
| [20] |
Chavali PL, Chandrasekaran G, Barr AR, et al. A CEP215-HSET complex links centrosomes with spindle poles and drives centrosome clustering in cancer[J]. Nat Commun, 2016, 7: 11005. DOI:10.1038/ncomms11005 |
| [21] |
Xiao YX, Yang WX. KIFC1: a promising chemotherapy target for cancer treatment[J]. Oncotarget, 2016, 7(30): 48656-48670. DOI:10.18632/oncotarget.8799 |
| [22] |
ZHOU K, LIN J, DAI M, et al. KIFC1 promotes aerobic glycolysis in endometrial cancer cells by regulating the c-myc pathway[J]. J Bioenerg Biomembr, 2021, 53(6): 703-713. DOI:10.1007/s10863-021-09924-1 |
| [23] |
Han J, Wang F, Lan Y, et al. KIFC1 regulated by miR-532-3p promotes epithelial-to-mesenchymal transition and metastasis of hepatocellular carcinoma via gankyrin/AKT signaling[J]. Oncogene, 2019, 38(3): 406-420. DOI:10.1038/s41388-018-0440-8 |
| [24] |
Xiao KH, Teng K, Ye YL, et al. Kinesin family member C1 accelerates bladder cancer cell proliferation and induces epithelial-mesenchymal transition via Akt/GSK3β signaling[J]. Cancer Sci, 2019, 110(9): 2822-2833. DOI:10.1111/cas.14126 |
| [25] |
Pawar S, Donthamsetty S, Pannu V, et al. KIFCI, a novel putative prognostic biomarker for ovarian adenocarcinomas: delineating protein interaction networks and signaling circuitries[J]. J Ovarian Res, 2014, 7: 53. DOI:10.1186/1757-2215-7-53 |
| [26] |
Wu J, Wang X, Yuan X, et al. Kinesin Family Member C1 Increases Temozolomide Resistance of Glioblastoma Through Promoting DNA Damage Repair[J]. Cell Transplant, 2021, 30: 963689721991466. |
| [27] |
Akabane S, Oue N, Sekino Y, et al. KIFC1 regulates ZWINT to promote tumor progression and spheroid formation in colorectal cancer[J]. Pathol Int, 2021, 71(7): 441-452. DOI:10.1111/pin.13098 |
| [28] |
Sekino Y, Pham QT, Kobatake K, et al. KIFC1 is associated with basal type, cisplatin resistance, PD-L1 expression and poor prognosis in bladder cancer[J]. J Clin Med, 2021, 10(21): 4837-4852. DOI:10.3390/jcm10214837 |
| [29] |
Fan G, Sun L, Meng L, et al. The ATM and ATR kinases regulate centrosome clustering and tumor recurrence by targeting KIFC1 phosphorylation[J]. Nat Commun, 2021, 12(1): 20. DOI:10.1038/s41467-020-20208-x |
| [30] |
Guo L, Mohd KS, Ren H, et al. Phosphorylation of importin-α1 by CDK1-cyclin B1 controls mitotic spindle assembly[J]. J Cell Sci, 2019, 132(18): jcs232314. |
| [31] |
Wei YL, Yang WX. Kinesin-14 motor protein KIFC1 participates in DNA synthesis and chromatin maintenance[J]. Cell Death Dis, 2019, 10(6): 402. DOI:10.1038/s41419-019-1619-9 |
 2023, Vol. 50
2023, Vol. 50




