文章信息
- 伪基因在肝癌中的研究进展
- Research Progress of Pseudogenes in Hepatocellular Carcinoma
- 肿瘤防治研究, 2023, 50(8): 813-819
- Cancer Research on Prevention and Treatment, 2023, 50(8): 813-819
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.23.0072
- 收稿日期: 2023-02-02
- 修回日期: 2023-03-09
肝癌为消化系统常见的恶性肿瘤,是全球癌症相关死亡率的主要原因之一。由于其隐匿的症状和早期转移的特征,大部分患者在确诊时已为晚期,导致疗效有限甚至治疗无效[1]。虽然近几年在手术切除、射频消融、药物使用等治疗手段方面不断取得进展,但肝癌患者的5年生存率依旧低于20%[2]。因此,确定潜在的患病机制和精确的诊断及预后标志物对肝癌患者的早期诊断与风险评估尤为重要。长链非编码RNA(long non-coding RNA, lncRNA)在一些生物学事件中起着重要作用,lncRNAs的失调与许多人类疾病有关,尤其是癌症[3]。根据lncRNAs对机体功能的差异将其分为不同组,包括转录超保守区(T-UCR)、增强子RNA(ERNA)、长基因间非编码RNA(lincRNAs)以及一组特殊的lncRNAs——伪基因[3]。长期以来,伪基因被描述为“基因化石”、“垃圾基因”等,从而反映它们的非功能状态[3]。近年来,随着许多大规模测序项目的完成,提供了大量丰富的功能基因组学数据,使伪基因逐渐走入人们的视野[4]。据统计,伪基因在人类基因组中大约有20 000个,其中6%是通过实验和计算方法确定的,并且研究还强调了伪基因在DNA、RNA和蛋白质水平上具有一系列功能作用[5]。
现如今,伪基因的活性已经被广泛研究,许多报告将其表达水平与疾病的进展联系起来,其中包括恶性肿瘤。伪基因可以被转录甚至翻译,在DNA、RNA和蛋白质水平上干扰关键蛋白编码基因的活性和功能,充当致癌基因和肿瘤抑制因子的调控因子[4]。在这篇综述中,我们讨论了伪基因在肝癌中的功能和临床相关研究,以及进展和未来的展望。
1 伪基因在肿瘤中的作用机制伪基因通过DNA、RNA与蛋白质水平在肿瘤的发生发展中发挥着重要作用。在DNA水平上,伪基因可以通过基因转换或同源重组相互作用并破坏其亲本基因活性[6],如伪基因PsiBRCA1与其下游乳腺癌和卵巢癌易感基因BRCA1之间的内含子2发生同源重组,使得BRCA1失去功能[7]。
一部分伪基因将其RNA产物作为蛋白质编码基因表达的调控因子,发挥着不同作用。研究表明,伪基因可以在包括癌症的多种组织和疾病中转录,伪基因转录本通过充当竞争性内源性RNA[8]、反义转录本[9]、内源性siRNA[10]干扰蛋白质编码基因的表达。在肝癌中,已经有相关伪基因衍生内源性siRNA的报道,如伪基因ψPPM1K含有反向重复序列,折叠为发夹RNA后,被加工为两个内源性siRNA,靶向抑制其亲本基因PPM1K与另一基因NEK8,分别导致线粒体活化改变和肝癌细胞增殖减少[11]。此外,由于伪基因与亲本基因的高度序列相似性,伪基因转录本也可以竞争RNA结合蛋白[12],竞争性内源性RNA(ceRNA)效应是伪基因最为公认的功能之一,并已在不同癌症中得到验证[8]。
最初伪基因被认为是含有过早终止密码子、插入缺失或移码突变的基因位点,这些突变会破坏其翻译能力。而近几年,一些研究强调了伪基因蛋白的存在,这些翻译产物与亲本蛋白相似,但在表达位点或病理生理条件上有所不同[13]。研究得出,干细胞标志物NANOG基因的伪基因之一NANOGP8,在胚胎干细胞的自我更新中起关键作用,可以编码一种在HepG2细胞(人肝癌细胞系)中被NANOG抗体检测到的蛋白质[14],促进肿瘤的发展。伪基因RNA的功能,见图 1。
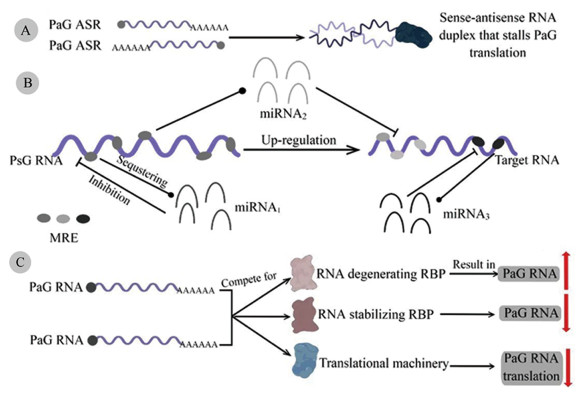
|
| A: parental sense mRNA of pseudogenes binding to parental sense mRNA slows down translation; B: pseudogene RNA as the target gene of ceRNA regulation; C: pseudogene RNA regulates RNA expression or protein translation through RNA-binding proteins or translation mechanisms. 图 1 伪基因RNA的功能[15] Figure 1 Functions of pseudogenic RNA[15] |
伪基因在肝癌中通过两种不同的过程来饰演特定的角色,一种过程为伪基因与其亲本基因通过共同作用调节肝癌细胞的生长;另一种过程为伪基因通过自身作用调节肝癌细胞的生长[16]。经测序项目得出,肝癌中有374个伪基因表达上调[17],它们通过以上两种过程促进肝癌的发生发展。
2.1 促进肝癌内部血管生成肿瘤内部血管的形成对肿瘤的生长具有重要作用。血管内皮生长因子A(vascular endothelial growth factor A, VEGFA)在血管生成的病理生理过程中起关键作用,许多肿瘤组织中显著升高[18]。缺氧诱导因子-1α(HIF-1α)是VEGFA的主要上游诱导因子,在肿瘤血管生成中起着关键作用[18]。据报道,伪基因UBE2CP3是一种癌基因[19],Lin等发现,UBE2CP3在肝癌组织中升高,且在高内皮血管(endothelial venule, EV)密度的组织中表达上调。过度表达UBE2CP3将激活ERK/HIF-1α信号通路,上调VEGFA的表达,促进人脐静脉内皮细胞增殖、迁移和血管形成,且在鸡胚绒毛尿囊膜(chickchorioallantoic membrane, CAM)血管生成实验和裸鼠致瘤性实验中均表明UBE2CP3表达上调了体内EV密度[20]。表明伪基因UBE2CP3通过上调表达VEGFA,促进肝癌内部血管的生成,从而促进肝癌的发生发展。
2.2 促进肝癌细胞增殖细胞恶性增殖是癌症发生发展的主要特征,其中细胞周期紊乱则是细胞恶性增殖的机制之一[21]。OCT4是一种转录因子,主要参与生殖细胞和胚胎干细胞的增殖和自我更新能力,促进多种肿瘤的进展[22]。POU5F1B是OCT4的伪基因之一,Pan等发现,POU5F1B在肝癌组织中显著上调,可以激活AKT通路,并且与Cyclin D1表达呈正相关,当POU5F1B过表达时,HepG2细胞的S期细胞百分比升高、G1/G0期细胞百分比降低[23],以上研究表明POU5F1B可以促进癌细胞增殖。在OCT4发挥促癌作用的同时,发现miR-145可以直接靶向作用于OCT4,抑制其在肝癌中的表达[24]。此外,OCT4-pg4为OCT4的另一伪基因,其表达与OCT4呈正相关,在肝癌中被异常激活。Wang等通过荧光素酶分析和基因敲除实验得出,OCT4-pg4作为ceRNA发挥作用,保护OCT4转录本不受miR-145抑制,从而促进癌细胞的增殖[25]。Huang等发现,伪基因PTTG3P在肝癌中表达增加,且能够加速G1/S转换,增加C-myc、Cyclin D1和p-Rb的表达来促进癌细胞的增殖[26]。
此外,ATF/cAMP反应元件结合蛋白家族中的ATF7,在细胞周期中可以促进Cyclin D1的分泌[27]。在肝癌中,miR-340-5p直接靶向ATF7抑制细胞增殖[28]。PCNAP1为PCNA的伪基因,He等对92例肝癌患者进行分析,得出PCNAP1在非感染性和HBV感染性患者的肝癌组织中均增加,且PCNAP1作为miR-340-5p的ceRNA,使ATF7表达升高,促进癌细胞的增殖[29]。
2.3 促进肝癌细胞侵袭转移侵袭和转移为肝癌细胞的生物学特征之一,对肝癌发生发展具有重要意义。Rac1为Rac亚家族的成员,在肿瘤组织中经常被激活并促进癌症转移[30]。AURKA为肝癌的促癌基因,且AURKAPS1为伪基因,两者呈正相关性,Li等研究发现,AURKAPS1在肝癌组织中显著升高,与肿瘤大小、TNM分期有关[31]。研究表明,AURKAPS1为一种竞争性内源性RNA(ceRNA),通过与miR-182、miR-155和miR-142的竞争性结合,增加RAC1的蛋白表达,激活PAK或ERK途径,进而促进肝癌细胞侵袭转移的能力[31]。
EMT在肿瘤中扮演着重要角色,为癌细胞侵袭转移的早期形式[32]。AKR1B10P1是与AKR1B10相关的亚型伪基因[33]。Wang等对87例肝癌患者的分析表明,AKR1B10与AKR1B10P1的表达在肝癌中均升高,两者之间呈现正相关,在其中35例患者中诊断出肝内转移,并且91.43%的肝转移患者AKR1B10P1表达相对较高,因此,AKR1B10P1被认为与肝癌转移有关[34]。同时,该研究表明,AKR1B10P1可以通过海绵作用吸附miR-138,两者相互作用稳定EMT诱导剂SOX4,使N-钙黏蛋白和波形蛋白上调,而E-钙黏蛋白下调,明显促进EMT和癌细胞的侵袭转移,小泛素相关修饰蛋白1(SUMO1)同样在肝癌中通过增强EMT促进癌细胞的侵袭转移。Zhou等发现,SUMO1P3为SUMO1的伪基因,在肝癌中表达显著增高,且敲除SUMO1P3可抑制癌细胞侵袭转移能力[35]。与此同时,Wu等通过双荧光素酶报告分析表明miR-320a为SUMO1P3潜在靶点,且SUMO1P3作为miR-320a的ceRNA,可以增强Wnt/β-catenin通路,进而促进癌细胞的生长和侵袭转移[36]。
PDK2属于丙酮酸脱氢酶激酶家族,可以下调线粒体丙酮酸脱氢酶复合物的活性,使乳酸的生成量升高,促进Warburg效应,增强癌细胞的侵袭转移能力[37]。在肝癌中miR-422a可以抑制PDK2的表达,Wei等发现,伪基因DUXAP8在肝癌中显著上调,与总存活期缩短、肿瘤体积增大显著相关。DUXAP8敲除实验表明,DUXAP8可以作为miR-422a的ceRNA,从而促进PDK2的表达,使EMT和癌细胞的侵袭转移能力增强[38]。
2.4 抑制肝癌细胞凋亡细胞凋亡又称为细胞程序性死亡,是抑制肿瘤细胞生长发展的主要机制之一。PDK1是一种丝氨酸色氨酸激酶,其在肝癌组织中的表达明显高于癌旁组织,且通过多种通路抑制肝癌细胞凋亡[39]。PDPK2P为PDK1的伪基因,PDPK2P表达与PDK1呈正相关。Pan等通过将肝癌细胞MHCC97L中伪基因PDPK2P过表达后发现,PDK1与p-AKT表达升高,caspase-3表达降低,PI3K表达无明显变化。因此得出,PDPK2P通过PDK1/AKT/Caspase-3通路抑制癌细胞凋亡[40]。另外伪基因SUMO1P3也可以通过下调caspase-3表达、上调Bcl-2表达,抑制癌细胞凋亡[41]。
PDIA3是一种伴侣蛋白,通过STAT3信号通路增强肝癌细胞的抗凋亡能力[42]。PDIA3P1为PDIA3的伪基因,两者之间呈正相关。研究表明,PDIA3P1作为miR-125a、miR-125b和miR-124的ceRNA,增强TRAF6的蛋白水平,导致NF-κB表达水平升高而使抑制癌细胞凋亡的能力增强[43]。
3 伪基因对肝癌细胞的抑制作用相比于对肝癌细胞具有促进作用的伪基因,另一部分具有抑制作用的伪基因数目有待进一步研究。经测序所得,肝癌中有34个伪基因表达下调[17],这些伪基因可以抑制肝癌细胞的生长和转移,进而抑制肝癌的发生发展。
3.1 抑制肝癌细胞增殖AKT信号通路是关于蛋白质合成、细胞生长和存活的中心调节器,AKT的激活与多种肿瘤的发病机制有关。AKT的失活可以抑制肝癌细胞的细胞周期进展和增殖[44]。DLG5是一种抑癌基因,在肝癌组织中低表达[45]。Dong等发现,伪基因TPTEP1在肝癌组织中表达降低。且据荧光素酶分析与过表达实验得出,miR-454-3p直接靶向抑制DLG5的表达,使AKT磷酸化增强。而TPTEP1作为miR-454-3p的ceRNA,增强DLG5的表达,减弱AKT磷酸化[46]。因此,伪基因TPTEP1通过miR-454-3p/DLG5轴抑制AKT信号通路,从而抑制肝癌细胞的增殖。同为在肝癌组织中低表达的伪基因WFDC21P,将其敲除后癌细胞增殖显著增强[47]。
PI3K/AKT通路是一种致癌途径,在癌症发展和肝癌发生中起着至关重要的作用[48]。PTEN是一种磷酸酶,可以消除PDK-1和AKT的磷酸化/活化,对PI3K/AKT通路进行抑制[48]。Chen等发现,PTEN与其伪基因PTENP1具有高度同源性,在肝癌组织中表达降低。且PTENP1表达抑制PI3K/AKT通路,增强GSK3b和p53的蛋白水平,同时负调节mTOR的磷酸化,降低Bcl-2和HIF-α的蛋白水平[49]。因此,PTENP1可以抑制癌细胞增殖,促进凋亡与自噬。
3.2 抑制肝癌细胞侵袭转移EMT为癌细胞转移侵袭的早期形式,其进展为癌症干细胞(cancer stem cell, CSC)表型的关键调节因子,有助于诱导其自我更新和分化具有固有特性和功能异质性的细胞,并产生转移性CSC,导致肿瘤的扩散、复发和转移[50]。ZEB1为EMT定义明确的驱动因素之一,研究表明其为癌基因,在多种癌症中异常表达[51]。Xiang等发现,伪基因UPAT在肝癌组织中表达明显降低且与ZEB1蛋白呈负相关,且ZEB1在转录水平上反向抑制UPAT表达,这可能是其导致肝癌中UPAT缺乏的部分原因。将UPTA过表达后,可观察到E-钙黏蛋白表达上调,ZEB1蛋白水平降低,还发现CSC相关标志物CD133和CD44以及CSC相关转录因子OCT4和SOX2的表达同时下调[52]。表明UPAT的过度表达会显著抑制肝癌细胞侵袭转移。研究表明,敲除伪基因WFDC21P导致E-钙黏蛋白表达被抑制,EMT增强进而使癌细胞侵袭转移能力被增强[47]。Wang等通过对108例肝癌患者的样本进行分析,得出伪基因AOC4P在肝癌组织中表达显著降低。将AOC4P过表达后,可抑制EMT显著降低癌细胞侵袭转移能力[53]。
4 伪基因对肝癌细胞的免疫作用近年来,随着基因和分子技术的不断进步,肿瘤微环境(tumor microenvironment, TME)在肿瘤表观遗传学、肿瘤分化、免疫逃逸和浸润转移中发挥着至关重要的作用。肿瘤可以通过TME为自己创造一系列有利条件,甚至可以逃避免疫循环。此外,TME中免疫反应紊乱在肝癌的发生和进展中起着关键作用[54]。近期研究表明,伪基因参与肿瘤免疫过程,包括抗原释放、抗原提呈、免疫细胞启动、T细胞激活和免疫细胞迁移,将肿瘤微环境转化为肿瘤免疫微环境[55],进而抑制肿瘤细胞的发生发展。
Ning等发现,在肝癌中Napsin B天冬氨酸肽酶伪基因(NAPSB)表达与免疫应答预测因子LAG-3、PD-1、PD-L1、CTLA-4的表达呈正相关,阻断PD-1与其配体PD-L1之间的相互作用[56],即免疫检查点阻断(immunologic checkpoint blockade, ICB)是目前最重要的免疫疗法[56]。表明高表达NAPSB可以增强免疫激活与免疫应答,抑制肝癌的发生发展。肝癌的免疫治疗已成为研究热点,我们热切期待着发现一些有前途的早期诊断与预后免疫相关伪基因标志物,并研究潜在的分子机制。
5 伪基因作为肝癌的预后标志物预测疾病进展和患者预期寿命对于临床具有重要意义,近几年内,发现许多有预后潜力的伪基因[57],且大多具有疾病特异性。在肝癌中,大多数有预后潜力的伪基因表达上调,只有少数与正常对照相比表达下调[58]。
表达上调的伪基因均表现出相同的特性,即均可以促进肝癌细胞侵袭转移和增殖的能力,从而促进肝癌的发生发展。RACGAP1P作为RACGAP1的伪基因,Wang等通过临床病理特征的相关性分析发现,与RACGAP1P低表达的患者相比,高表达患者的总生存期(overall survival, OS)和无病生存期(disease-free survival, DFS)均明显缩短,并且在早期复发的肝癌中RACGAP1P持续激活[59]。因此,RACGAP1P成为肝癌患者的一个预后因素。同时临床病理特征的相关性分析表明,伪基因UBE2CP3[20]、PTTG3P[26]、SUMO1P3[36]、DUXAP8[38]在肝癌组织中表达显著升高,且与肿瘤大小、TNM分期以及患者的低生存率呈正相关,提示这些伪基因高表达使肝癌患者预后不良。
相反,伪基因TPTEP1[46]、WFDC21P[47]、PTENP1[49]、UPTA[52]、AOC4P[53]、MT1DP[60]在肝癌组织中的表达下调,而它们的过表达可以促进癌细胞凋亡、抑制癌细胞增殖和侵袭转移,与肿瘤大小、TNM分期以及患者的低生存率呈负相关,均可以作为肝癌的预后因素。
6 伪基因作为肝癌的治疗靶点随着伪基因在肝癌内的功能被不断发掘,除了可以成为肝癌的预后标志物,伪基因也可以作为肝癌潜在的治疗靶点。化疗药物作为肝癌治疗的选择手段之一,对延长患者总生存期的效果较好[61],但伪基因PDIA3P1可以在使用DNA损伤性化疗药物(如阿霉素)治疗后表达上调,并且使肝癌细胞对化疗脱敏[49]。化疗药物5-Fu通常与其他药物联合用于肝癌的TACE治疗且效果较好,但肝癌细胞对5-Fu的耐药性成为治疗的一大障碍[62]。研究表明,PLEKHA8P1与其伪基因PLEKHA8P1均使肝癌细胞对5-Fu产生耐药性,而敲除PLEKHA8P1可使肝癌细胞对5-Fu的敏感度明显增强[63]。因此,伪基因PDIA3P、PLEKHA8P1可以作为肝癌相关化疗耐药研究的治疗靶点。
此外,甲胎蛋白(AFP)为肝癌的特异性标志物,HCC患者血清AFP与正常血清AFP浓度相比,患者血清AFP显著升高[64]。且在HepG2细胞中FOXA1与AFP启动子的反式激活增强,AFP高表达与FoxA1表达呈正相关[65]。研究发现,伪基因MT1DP可以通过抑制FoxA1表达而降低AFP在肝癌组织中的表达[60],可以作为抑制肝癌AFP生成一种治疗靶点。同时,伪基因INTS6P1可以作为诊断低表达AFP肝癌患者的前瞻性生物标志物,能够筛选出血清AFP水平低于20 ng/ml的HCC患者[66]。由此可见,伪基因INTS6P1可以作为诊断和筛查肝癌的血浆生物标志物。
7 结语从最初认为伪基因在体内没有功能,到现在越来越多的研究发现伪基因可以在多种生理病理层面上发挥重要作用,包括独立和对亲本基因的依赖调节。伪基因的异常表达与许多临床病理参数密切相关,如在临床应用中,PCNAP1表现出作为HBV感染性肝癌预后标志物和治疗靶点的潜力[35],且不断有可以调节肝癌细胞生长转移的伪基因被发现,增加对伪基因本身作为肝癌预后和治疗靶点潜力的期望,而目前我们对伪基因在肝癌中机制基础的认识和理解仍然有限。因此,伪基因在肝癌中的研究是一个有待探索的广阔领域,相信未来将会有越来越多关于伪基因的研究,从而深入对肝癌机制的理解。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
李凯欣:撰写论文
赵彦玉:整理文献与数据
陈彻、郭文杰、刘芳:修改论文
| [1] |
Anwanwan D, Singh SK, Singh S, et al. Challenges in liver cancer and possible treatment approaches[J]. Biochim Biophys Acta Rev Cancer, 2020, 1873(1): 188314. DOI:10.1016/j.bbcan.2019.188314 |
| [2] |
蔡秀军, 郑强, 蒋广宜. 肝癌外科治疗的现状和展望[J]. 中华外科杂志, 2019, 57(7): 494-499. [Cai XJ, Zheng Q, Jiang GY. Current status and prospect of surgical treatment of liver cancer[J]. Zhonghua Wai Ke Za Zhi, 2019, 57(7): 494-499.] |
| [3] |
Tan H, Zhang S, Zhang J, et al. Long non-coding RNAs in gastric cancer: New emerging biological functions and therapeutic implications[J]. Theranostics, 2020, 10(19): 8880-8902. DOI:10.7150/thno.47548 |
| [4] |
Sisu C. GENCODE Pseudogenes[J]. Methods Mol Biol, 2021, 2324: 67-82. |
| [5] |
Khan AA, Ali MS, Babar F, et al. Lack of CpG islands in human unitary pseudogenes and its implication[J]. Mamm Genome, 2021, 32(6): 443-447. DOI:10.1007/s00335-021-09893-1 |
| [6] |
Mehravar M, Ghaemimanesh F, Poursani EM. An Overview on the Complexity of OCT4: at the Level of DNA, RNA and Protein[J]. Stem Cell Rev Rep, 2021, 17(4): 1121-1136. DOI:10.1007/s12015-020-10098-3 |
| [7] |
Su L, Zhang J, Meng H, et al. Prevalence of BRCA1/2 large genomic rearrangements in Chinese women with sporadic triple-negative or familial breast cancer[J]. Clin Genet, 2018, 94(1): 165-169. DOI:10.1111/cge.13256 |
| [8] |
Lou W, Ding B, Fu P. Pseudogene-Derived lncRNAs and Their miRNA Sponging Mechanism in Human Cancer[J]. Front Cell Dev Biol, 2020, 8: 85. DOI:10.3389/fcell.2020.00085 |
| [9] |
Singh RK, Singh D, Yadava A, et al. Molecular fossils "pseudogenes" as functional signature in biological system[J]. Genes Genomics, 2020, 42(6): 619-630. DOI:10.1007/s13258-020-00935-7 |
| [10] |
Chan WL, Chang JG. Pseudogene-derived endogenous siRNAs and their function[J]. Methods Mol Biol, 2014, 1167: 227-239. |
| [11] |
Chan WL, Yuo CY, Yang WK, et al. Transcribed pseudogene ψPPM1K generates endogenous siRNA to suppress oncogenic cell growth in hepatocellular carcinoma[J]. Nucleic Acids Res, 2013, 41(6): 3734-3747. DOI:10.1093/nar/gkt047 |
| [12] |
Lim JQ, Lim ST, Ong CK. Misaligned sequencing reads from the GNAQ-pseudogene locus may yield GNAQ artefact variants[J]. Nat Commun, 2022, 13(1): 458. DOI:10.1038/s41467-022-28115-z |
| [13] |
Chiefari E, Arcidiacono B, Mirabelli M, et al. Methods to Study Protein-Binding to Pseudogene Transcripts[J]. Methods Mol Biol, 2021, 2324: 187-202. |
| [14] |
Thakur H, Mattoo AR. A Restriction Endonuclease-Based Assay to Distinguish NANOGP8 Retrogene from Parental NANOG[J]. Methods Mol Biol, 2021, 2324: 255-262. |
| [15] |
Xiao-Jie L, Ai-Mei G, Li-Juan J, et al. Pseudogene in cancer: real functions and promising signature[J]. J Med Genet, 2015, 52(1): 17-24. DOI:10.1136/jmedgenet-2014-102785 |
| [16] |
Liu J, Liu ZX, Wu QN, et al. Long noncoding RNA AGPG regulates PFKFB3-mediated tumor glycolytic reprogramming[J]. Nat Commun, 2020, 11(1): 1507. DOI:10.1038/s41467-020-15112-3 |
| [17] |
Yan L, Yue C, Xu Y, et al. Identification of Potential Diagnostic and Prognostic Pseudogenes in Hepatocellular Carcinoma Based on Pseudogene-miRNA-mRNA Competitive Network[J]. Med Sci Monit, 2020, 26: e921895. |
| [18] |
El Baba N, Farran M, Khalil EA, et al. The Role of Rho GTPases in VEGF Signaling in Cancer Cells[J]. Anal Cell Pathol (Amst), 2020, 2020: 2097214. |
| [19] |
Li D, She J, Hu X, et al. The ELF3-regulated lncRNA UBE2CP3 is over-stabilized by RNA-RNA interactions and drives gastric cancer metastasis via miR-138-5p/ITGA2 axis[J]. Oncogene, 2021, 40(35): 5403-5415. DOI:10.1038/s41388-021-01948-6 |
| [20] |
Lin J, Cao S, Wang Y, et al. Long non-coding RNA UBE2CP3 enhances HCC cell secretion of VEGFA and promotes angiogenesis by activating ERK1/2/HIF-1α/VEGFA signalling in hepatocellular carcinoma[J]. J Exp Clin Cancer Res, 2018, 37(1): 113. DOI:10.1186/s13046-018-0727-1 |
| [21] |
Leal-Esteban LC, Fajas L. Cell cycle regulators in cancer cell metabolism[J]. Biochim Biophys Acta Mol Basis Dis, 2020, 1866(5): 165715. DOI:10.1016/j.bbadis.2020.165715 |
| [22] |
Mohiuddin IS, Wei SJ, Kang MH. Role of OCT4 in cancer stem-like cells and chemotherapy resistance[J]. Biochim Biophys Acta Mol Basis Dis, 2020, 1866(4): 165432. DOI:10.1016/j.bbadis.2019.03.005 |
| [23] |
Pan Y, Zhan L, Chen L, et al. POU5F1B promotes hepatocellular carcinoma proliferation by activating AKT[J]. Biomed Pharmacother, 2018, 100: 374-380. DOI:10.1016/j.biopha.2018.02.023 |
| [24] |
Jia Y, Liu H, Zhuang Q, et al. Tumorigenicity of cancer stem-like cells derived from hepatocarcinoma is regulated by microRNA-145[J]. Oncol Rep, 2012, 27(6): 1865-1872. |
| [25] |
Wang L, Guo ZY, Zhang R, et al. Pseudogene OCT4-pg4 functions as a natural micro RNA sponge to regulate OCT4 expression by competing for miR-145 in hepatocellular carcinoma[J]. Carcinogenesis, 2013, 34(8): 1773-1781. DOI:10.1093/carcin/bgt139 |
| [26] |
Huang JL, Cao SW, Ou QS, et al. The long non-coding RNA PTTG3P promotes cell growth and metastasis via up-regulating PTTG1 and activating PI3K/AKT signaling in hepatocellular carcinoma[J]. Mol Cancer, 2018, 17(1): 93. DOI:10.1186/s12943-018-0841-x |
| [27] |
Lin J, Mao H, Ji Z, et al. Human epidermal growth factor receptor 2 inhibits activating transcription factor 7 to promote breast cancer cell migration by activating histone lysine demethylase 1[J]. J Cancer Res Ther, 2021, 17(5): 1192-1201. DOI:10.4103/jcrt.jcrt_649_21 |
| [28] |
Liu H, Li B, Qiao L, et al. miR-340-5p inhibits sheep adipocyte differentiation by targeting ATF7[J]. Anim Sci J, 2020, 91(1): e13462. DOI:10.1111/asj.13462 |
| [29] |
He M, Hu L, Bai P, et al. LncRNA PCNAP1 Promotes Hepatoma Cell Proliferation through Targeting miR-340-5p and is Associated with Patient Survival[J]. J Oncol, 2021, 2021: 6627173. |
| [30] |
Xu ZD, Hao T, Gan YH. RhoG/Rac1 signaling pathway involved in migration and invasion of salivary adenoid cystic carcinoma cells[J]. Oral Dis, 2020, 26(2): 302-312. DOI:10.1111/odi.13247 |
| [31] |
Li J, Guo W, Xue W, et al. Long noncoding RNA AURKAPS1 potentiates malignant hepatocellular carcinoma progression by regulating miR-142, miR-155 and miR-182[J]. Sci Rep, 2019, 9(1): 19645. DOI:10.1038/s41598-019-56036-3 |
| [32] |
Pastushenko I, Blanpain C. EMT Transition States during Tumor Progression and Metastasis[J]. Trends Cell Biol, 2019, 29(3): 212-226. DOI:10.1016/j.tcb.2018.12.001 |
| [33] |
Hao F, Fei X, Ren X, et al. Pseudogene AKR1B10P1 enhances tumorigenicity and regulates epithelial-mesenchymal transition in hepatocellular carcinoma via stabilizing SOX4[J]. J Cell Mol Med, 2020, 24(20): 11779-11790. DOI:10.1111/jcmm.15790 |
| [34] |
Wang N, Hao F, Ren J, et al. Positive feedback loop of AKR1B10P1/miR-138/SOX4 promotes cell growth in hepatocellular carcinoma cells[J]. Am J Transl Res, 2020, 12(9): 5465-5480. |
| [35] |
Zhou Y, He P, Xie X, et al. Knockdown of SUMO1P3 represses tumor growth and invasion and enhances radiosensitivity in hepatocellular carcinoma[J]. Mol Cell Biochem, 2019, 450(1-2): 125-134. DOI:10.1007/s11010-018-3379-8 |
| [36] |
Wu S, Chen S, Lin N, et al. Long non-coding RNA SUMO1P3 promotes hepatocellular carcinoma progression through activating Wnt/β-catenin signalling pathway by targeting miR-320a[J]. J Cell Mol Med, 2020, 24(5): 3108-3116. DOI:10.1111/jcmm.14977 |
| [37] |
Zhang Y, Chen M, Liu M, et al. Glycolysis-Related Genes Serve as Potential Prognostic Biomarkers in Clear Cell Renal Cell Carcinoma[J]. Oxid Med Cell Longev, 2021, 2021: 6699808. |
| [38] |
Wei F, Yang L, Jiang D, et al. Long noncoding RNA DUXAP8 contributes to the progression of hepatocellular carcinoma via regulating miR-422a/PDK2 axis[J]. Cancer Med, 2020, 9(7): 2480-2490. DOI:10.1002/cam4.2861 |
| [39] |
Bamodu OA, Chang HL, Ong JR, et al. Elevated PDK1 Expression Drives PI3K/AKT/MTOR Signaling Promotes Radiation-Resistant and Dedifferentiated Phenotype of Hepatocellular Carcinoma[J]. Cells, 2020, 9(3): 746. DOI:10.3390/cells9030746 |
| [40] |
Pan W, Li W, Zhao J, et al. lncRNA-PDPK2P promotes hepatocellular carcinoma progression through the PDK1/AKT/Caspase 3 pathway[J]. Mol Oncol, 2019, 13(10): 2246-2258. DOI:10.1002/1878-0261.12553 |
| [41] |
Hu S, Liu J, Feng S, et al. LncRNA SUMO1P3 acts as a prognostic biomarker and promotes hepatocellular carcinoma growth and metastasis[J]. Aging (Albany NY), 2021, 13(9): 12479-12492. |
| [42] |
Kondo R, Ishino K, Wada R, et al. Downregulation of protein disulfide isomerase A3 expression inhibits cell proliferation and induces apoptosis through STAT3 signaling in hepatocellular carcinoma[J]. Int J Oncol, 2019, 54(4): 1409-1421. |
| [43] |
Xie C, Zhang LZ, Chen ZL, et al. A hMTR4-PDIA3P1-miR-125/124-TRAF6 Regulatory Axis and Its Function in NF kappa B Signaling and Chemoresistance[J]. Hepatology, 2020, 71(5): 1660-1677. DOI:10.1002/hep.30931 |
| [44] |
Song M, Bode AM, Dong Z, et al. AKT as a Therapeutic Target for Cancer[J]. Cancer Res, 2019, 79(6): 1019-1031. DOI:10.1158/0008-5472.CAN-18-2738 |
| [45] |
Sarathi A, Palaniappan A. Novel significant stage-specific differentially expressed genes in hepatocellular carcinoma[J]. BMC Cancer, 2019, 19(1): 663. DOI:10.1186/s12885-019-5838-3 |
| [46] |
Dong Y, Wang Q, Sun J, et al. Long non-coding RNA TPTEP1 exerts inhibitory effects on hepatocellular carcinoma by impairing microRNA-454-3p-mediated DLG5 downregulation[J]. Dig Liver Dis, 2022, 54(2): 268-279. DOI:10.1016/j.dld.2021.04.014 |
| [47] |
Guan YF, Huang QL, Ai YL, et al. Nur77-activated lncRNA WFDC21P attenuates hepatocarcinogenesis via modulating glycolysis[J]. Oncogene, 2020, 39(11): 2408-2423. DOI:10.1038/s41388-020-1158-y |
| [48] |
Xie P, Peng Z, Chen Y, et al. Neddylation of PTEN regulates its nuclear import and promotes tumor development[J]. Cell Res, 2021, 31(3): 291-311. DOI:10.1038/s41422-020-00443-z |
| [49] |
Chen CL, Tseng YW, Wu JC, et al. Suppression of hepatocellular carcinoma by baculovirus-mediated expression of long non-coding RNA PTENP1 and MicroRNA regulation[J]. Biomaterials, 2015, 44: 71-81. DOI:10.1016/j.biomaterials.2014.12.023 |
| [50] |
Huang T, Song X, Xu D, et al. Stem cell programs in cancer initiation, progression, and therapy resistance[J]. Theranostics, 2020, 10(19): 8721-8743. DOI:10.7150/thno.41648 |
| [51] |
Wang J, He H, Jiang Q, et al. CBX6 Promotes HCC Metastasis Via Transcription Factors Snail/Zeb1-Mediated EMT Mechanism[J]. Onco Targets Ther, 2020, 13: 12489-12500. DOI:10.2147/OTT.S257363 |
| [52] |
Xiang L, Huang X, Wang S, et al. Deficiency of pseudogene UPAT leads to hepatocellular carcinoma progression and forms a positive feedback loop with ZEB1[J]. Cancer Sci, 2020, 111(11): 4102-4117. DOI:10.1111/cas.14620 |
| [53] |
Wang TH, Lin YS, Chen Y, et al. Long non-coding RNA AOC4P suppresses hepatocellular carcinoma metastasis by enhancing vimentin degradation and inhibiting epithelial-mesenchymal transition[J]. Oncotarget, 2015, 6(27): 23342-23357. DOI:10.18632/oncotarget.4344 |
| [54] |
Petitprez F, Meylan M, de Reyniès A, et al. The Tumor Microenvironment in the Response to Immune Checkpoint Blockade Therapies[J]. Front Immunol, 2020, 11: 784. DOI:10.3389/fimmu.2020.00784 |
| [55] |
Zhao R, Li B, Zhang S, et al. The N6-Methyladenosine-Modified Pseudogene HSPA7 Correlates With the Tumor Microenvironment and Predicts the Response to Immune Checkpoint Therapy in Glioblastoma[J]. Front Immunol, 2021, 12: 653711. DOI:10.3389/fimmu.2021.653711 |
| [56] |
Ning YM, Lin K, Liu XP, et al. NAPSB as a predictive marker for prognosis and therapy associated with an immuno-hot tumor microenvironment in hepatocellular carcinoma[J]. BMC Gastroenterol, 2022, 22(1): 392. DOI:10.1186/s12876-022-02475-8 |
| [57] |
Tang S, Zhuge Y. An immune-related pseudogene signature to improve prognosis prediction of endometrial carcinoma patients[J]. Biomed Eng Online, 2021, 20(1): 64. DOI:10.1186/s12938-021-00902-7 |
| [58] |
Sisu C. Pseudogenes as Biomarkers and Therapeutic Targets in Human Cancers[J]. Methods Mol Biol, 2021, 2324: 319-337. |
| [59] |
Wang MY, Chen DP, Qi B, et al. Pseudogene RACGAP1P activates RACGAP1/Rho/ERK signalling axis as a competing endogenous RNA to promote hepatocellular carcinoma early recurrence[J]. Cell Death Dis, 2019, 10(6): 426. |
| [60] |
Yu W, Qiao Y, Tang X, et al. Tumor suppressor long non-coding RNA, MT1DP is negatively regulated by YAP and Runx2 to inhibit FoxA1 in liver cancer cells[J]. Cell Signal, 2014, 26(12): 2961-2968. |
| [61] |
Shen Y, Zhang ZB, Wu SD, et al. Research on values of GDF-15 level in the diagnosis of primary liver cancer and evaluation of chemotherapeutic effect[J]. Eur Rev Med Pharmacol Sci, 2018, 22(12): 3749-3754. |
| [62] |
Li S, Liu XY, Pan Q, et al. Hepatitis C Virus-Induced FUT8 Causes 5-FU Drug Resistance in Human Hepatoma Huh7.5.1 Cells[J]. Viruses, 2019, 11(4): 378. |
| [63] |
Lee J, Hwang JH, Chun H, et al. PLEKHA8P1 Promotes Tumor Progression and Indicates Poor Prognosis of Liver Cancer[J]. Int J Mol Sci, 2021, 22(14): 7614. |
| [64] |
Wang X, Wang Q. Alpha-Fetoprotein and Hepatocellular Carcinoma Immunity[J]. Can J Gastroenterol Hepatol, 2018, 2018: 9049252. |
| [65] |
Wang S, Xiang C, Mou L, et al. Trans-acting non-synonymous variant of FOXA1 predisposes to hepatocellular carcinoma through modulating FOXA1-ERα transcriptional program and may have undergone natural selection[J]. Carcinogenesis, 2020, 41(2): 146-158. |
| [66] |
Lui KY, Peng HR, Lin JR, et al. Pseudogene integrator complex subunit 6 pseudogene 1 (INTS6P1) as a novel plasma-based biomarker for hepatocellular carcinoma screening[J]. Tumour Biol, 2016, 37(1): 1253-1260. |
 2023, Vol. 50
2023, Vol. 50


