文章信息
- 结直肠癌进展相关关键分子事件研究进展
- Research Progress of Key Molecular Events Related to Progression of Colorectal Cancer
- 肿瘤防治研究, 2023, 50(6): 609-615
- Cancer Research on Prevention and Treatment, 2023, 50(6): 609-615
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.22.1242
- 收稿日期: 2022-10-20
- 修回日期: 2022-11-30
2. 200433 上海,上海市医学生物防护重点实验室;
3. 200433 上海,生物安全防御教育部重点实验室;
4. 200092 上海,同济大学医学院
2. Shanghai Key Laboratory of Medical Bioprotection, Shanghai 200433, China;
3. Key Laboratory of Biological Defense, Ministry of Education, Shanghai 200433, China;
4. Tongji University School of Medicine, Shanghai 200092, China
近几年,结直肠癌(colorectal cancer, CRC)的发病率和病死率一直处于较高水平,CRC在所有癌症中的发病和死亡占比分别为10.0%和9.4%,排名分别位于第三位和第二位[1],是全世界癌症死亡的主要原因。预计到2030年,CRC的负担将增加60%,达到220多万新病例和110万癌症死亡病例[2]。CRC的发生发展与肿瘤微环境如炎性反应和遗传密切相关,与人口老龄化、饮食习惯、肥胖、缺乏体育锻炼和吸烟等危险因素相关。近一半的CRC患者死于CRC转移性疾病[3],确定未来是否发生恶性进展仍然是CRC预后预测方面的重点和难点。
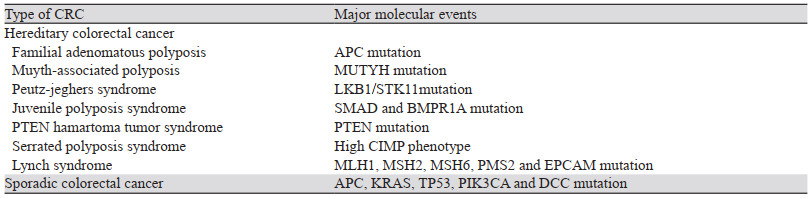
目前,对遗传性非息肉性CRC、家族腺瘤性息肉、林奇综合征(Lynch综合征)等遗传性的CRC相关突变基因已经有了较为精准的认识,见表 1。但散发性CRC在CRC中占比较高(约70%),尚无有效的预测标志物。CRC是高度异质性肿瘤,具有不同的分子亚型,驱动CRC恶变特性的分子机制尚未完全阐明。靶向分子抑制剂已经在许多癌症类型中获得成功,而对于CRC晚期或是转移患者,仍缺乏有效的分子标志物和表型靶点来预测预后或指导有效治疗策略。因此,对CRC进展相关分子事件的深入研究显得极为迫切。
家族性腺瘤性息肉病(familial adenomatous polyposis, FAP)引发CRC的病例占CRC总数比不到1%[4]。已有研究表明,FAP是染色体5q21上的APC基因突变引起的,并且以常染色体显性的方式遗传[5],但不是所有的FAP都能检测出APC突变[6],而APC的突变也呈现出高度异质性的点突变特征。尽管认为FAP中APC突变是遗传性的,是生殖系嵌合的结果,但基于二代测序结果发现,体细胞嵌合APC基因突变也可能是FAP的病因,并且出现体细胞新生突变现象[7]。
MUYTH相关性息肉病(MAP)是MUTYH突变导致的一种罕见息肉综合征,与FAP不同的是,它是一种常染色体隐性遗传疾病。MUTYH基因编码一种参与DNA氧化损伤修复的DNA糖基化酶,当这种损伤不能正常修复,会导致G: C在复制过程中被转换为T: A。MAP患者结肠息肉的风险较常人高,CRC风险也更高。
黑斑息肉综合征(Peutz-Jeghers syndrome, PJS)是一种罕见的常染色体显性综合征,其与整个胃肠道错构瘤性息肉病相关。PJS是由19号染色体上的一个常染色体显性遗传基因(LKB1/STK11)突变引起的,LKB1/STK11是一个肿瘤抑制基因,是细胞增殖、代谢、凋亡的一个重要调节基因,90%的PJS患者具有可识别的LKB1/STK11突变[8]。LKB1缺失导致代谢重编程赋予癌细胞生长或生存优势,PJS将增加结直肠癌风险[9]。
幼年性息肉病综合征(juvenile polyposis syndrome, JPS)为常染色体显性遗传性疾病,SMAD4和BMPR1A基因突变可明确诊断,特点是易在胃肠道(GI)发生错构瘤性息肉,特别是在胃、小肠、结肠和直肠,JPS中发生CRC的累积风险为39%~68%[10]。
PTEN错构瘤肿瘤综合征(PTEN hamartoma tumor syndrome, PHTS)是一种常染色体显性遗传疾病,与PTEN基因突变有关。PTEN作为一种重要的磷酸酶在抵消最关键的癌症PI3K/AKT信号通路中发挥着重要作用[11]。
锯齿状息肉病综合征(SPS)并没有发现种系突变,其发病被认为是高甲基化导致肿瘤抑制基因失活导致的,通常标记为高CIMP(CpG岛甲基化表型)。
Lynch综合征与DNA错配修复缺陷(dMMR)基因的常染色体显性突变关系密切,是最常见的遗传性癌症综合征,其发展的肿瘤通常呈现MSI和dMMR特征,它以四种不同MMR基因的常染色体显性突变为特征:MLH1、MSH2、MSH6和PMS2[12];另外,EPCAM突变可引起MSH2表达缺失,进而引发Lynch综合征。
1.2 散发性CRC分子突变事件与遗传性结直肠癌不同,散发性CRC通常被认为是点突变引起的,与遗传关联并不密切。点突变通常发生在不同基因上,因此散发性CRC具有高度异质性,遵循着正常细胞-腺瘤(息肉)-癌的转变过程。APC突变是关键的起始事件,然后触发非恶性腺瘤(息肉)的形成,腺瘤通常经过数年时间发展成癌,KRAS、TP53和DCC等基因也会继APC突变之后开始发生突变[13]。
2 转录后调控水平层面的表观遗传及分子网络的动态改变事件CRC的表观遗传变化、非编码RNA作为关键基因的重要调控因子已被证明在推动CRC发生发展中起到重要作用,科学家们对这些事件的探索只挖掘到了巨大调控网中的微小一角,我们仍需更多的证据将这些复杂分子事件串联在一起。
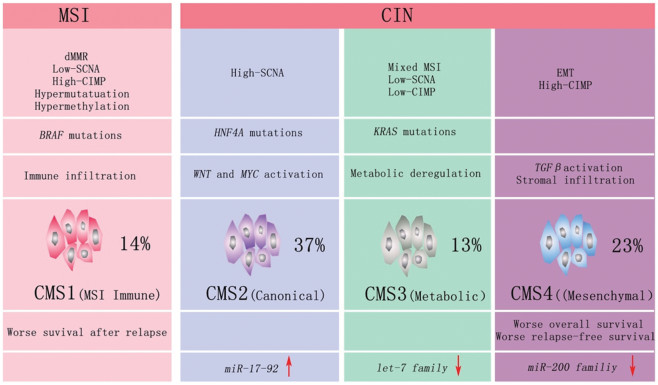
2.1 CRC共识分子事件CRC的高度异质性决定了其具有复杂多样的分子事件发生。为了对CRC进展的同质分子亚型作进一步研究,基于六种独立的基于转录组学的亚型分型系统,CRC亚型联盟(CRCSC)总结出CRC的四个共识分子亚型(CMS)[14]:CMS1、CMS2、CMS3、CMS4,见图 1。
|
| 图 1 结直肠癌4种共识分子亚型 Figure 1 Four consensus molecular subtypes of colorectal cancer |
总体上CMS1表现出MSI状态,而CMS2~4则显示出更高的染色体不稳定性(CIN)。除了CMS1和CMS3中受体酪氨酸激酶(RTK)和丝裂原活化蛋白激酶(MAPK)通路的基因普遍激活外,没有发现其他显著的关联[13]。在通路活性特征良好的细胞进行基因集富集分析方面,CMS1中广泛的免疫浸润相关基因表达增加,主要由TH1和细胞毒性T细胞组成,并强烈激活免疫逃避,这是MSI CRC的一个特点;CMS2肿瘤中上皮分化和WNT、MYC下游靶点的上调,与CRC癌变有关;CMS3肿瘤则与胃癌中描述的与“代谢”有关、基因组稳定的亚型有相似之处[15];在CMS4肿瘤中,上皮间充质转化(EMT)相关基因明显上调,并与TGFβ信号、血管生成、基质重塑通路和补体炎性反应系统的激活相关。CMS4样本具有与基质浸润相一致的基因表达谱;在CMS组与TCGA样本中蛋白组簇分析方面,CMS1肿瘤中免疫应答通路蛋白表达上调,而CMS4样本基质侵入、间充质细胞活化和补体通路中蛋白显著过表达[14]。
CRCSC还对CMS组中转录后调控的miRNA进行了相关性分析,不同CMS亚组miRNA的表达也有差异。例如CMS2肿瘤中与MYC相关miR-17-92表达上调[16],CMS3肿瘤中let-7 miR家族下调并伴随KRAS高表达,而与EMT调控相关的miR-200家族在CMS4中下调[17]。虽然,CMS的分层有助于我们更系统全面地了解CRC中分子事件的共性与特性,但目前仍然需要更多的临床及科研数据支撑和丰富这个体系。
2.2 环状RNA分子事件尽管CRCSC对CRC样本中分子驱动事件进行了共识分子亚型分析,让CRC的分子亚型分层更加清晰,对CMS组的miRNA富集也有研究,但对于转录后调控水平的环状RNA(circular RNA, circRNA)并没有纳入CMS分层中,因此,circRNA在CRC进化过程中的“神秘面纱”需要进一步揭开。
CRC进化发育(正常组织-腺瘤-癌)是一个动态漫长的过程,通常需要数十年时间,除了基因组突变等累积及细胞信号通路的激活,转录后表观调控机制(如cirRNA对于癌症进展)等研究也越来越被关注。近年来,随着对circRNA的研究深入,越来越多的证据证明,circRNA在癌症患者组织、血液等样本中具有特异性表达,其作为癌症诊断、预测、预后分子标志物或是治疗靶点具有巨大临床潜能。circRNA是一种高度稳定的分子,由pre-mRNA反向剪切而成,主要通过与不同的分子相互作用发挥不同功能。目前,对circRNA在CRC中的研究主要集中在以下几个方面:(1)作为miRNA海绵,吸附miRNA,进而与miRNA竞争性的调控下游基因的表达影响肿瘤的发生发展。这是目前对circRNA研究较多的机制。circPACRGL作为miR-142-3p/miR-506-3p的海绵,促进转化生长因子-β1(TGF-β1)的表达,通过miR-142-3p/miR-506-3p-TGF-β1轴促进CRC细胞增殖、迁移和侵袭[18]。(2)与RNA结合蛋白(RBP)结合发挥作用。N6-甲基腺苷(m6A)修饰的circNSUN2在结直肠癌伴肝转移(LM)患者的肿瘤组织和血清样本中上调,其通过形成circNSUN2/IGF2BP2/HMGA2胞质中RNA-蛋白三元复合物增强HMGA2 mRNA的稳定性,促进结直肠癌转移进展[19]。在血浆中可检测的circLPAR1与转录因子eIF3h结合,抑制METTL3-eIF3h相互作用,导致BRD4水平下降,显著抑制结直肠癌的发展[20]。(3)编码蛋白或肽段。有研究发现circFNDC3B可编码新蛋白circFNDC3B-218aa,而circFNDC3B-218aa通过降低Snail对结肠癌中FBP1产生抑制作用,从而抑制肿瘤进展和上皮-间质转化(EMT)[21]。
癌症进化发育过程中,细胞从开始具有“干性”,获得发育性基因表达程序,具有无限的自我更新能力而进化发育[22],进而癌变。有研究已经发现circ_001680通过调控miR-340靶基因BMI1促进CRC中癌症干细胞群体并诱导伊立替康治疗耐药的证据[23];circAGFG1通过海绵吸收miR-4262和miR-185-5p,调节YY1/CTNNB1轴来驱动CRC中细胞的转移和干性[24]。众多研究已证明circRNA在CRC过程中通过调控下游基因、调节细胞信号通路等途径发挥重要的表观调控作用,见表 2。这些研究对探索circRNA功能开启了新的方向。
随着高通量测序技术发展,circRNA在癌症组织的表达谱越来越全面,circRNA作为CRC诊断、预测、预后分子标志物或是治疗靶点的研究越来越丰富,但circRNA与驱动癌症突变基因的关系,我们仍然了解甚少。鉴于相关技术受限和样本收集难度,circRNA在单细胞水平时间及空间上的表达数据需要随着技术的进步而进一步扩充。circRNA在癌症中的生物发生、功能认识仍需深入研究。
3 CRC转移、耐药的调控机制 3.1 转移性结直肠癌分子机制有研究发现,约20%CRC患者初诊即被发现远端转移[33],转移性结直肠癌(mCRC)成为患者预后不良和主要死亡原因之一。APC、KRAS、TP53、PIK3CA等基因的突变在CRC的起始和早期发展中起着关键作用[34]。然而,因为CRC的高度异质性,细胞群的进化在转移过程中就变得更加复杂。一项CRC肝转移单细胞测序数据表明,来自肿瘤组织的细胞比正常组织细胞具有更多突变,来自MSI肿瘤的细胞比来自微卫星稳定(MSS)肿瘤的细胞具有更高的单核苷酸变异(SNV)数量;一些重要非同义的SNVs,如TP53、APC和SMAD4中的SNVs,在转移样本中的变异等位基因频率(VAF)增加;DNA错配修复相关基因如MLH1和CRC驱动基因如PIK3CA是共有的突变集,一些特异性出现在转移性肿瘤细胞克隆中的非同义突变基因如RFC1、XPO4、THEGL、SLC19A3、TBC1D4和KCNH5可能在转移瘤中具有特定功能[35],仍待进一步验证。mCRC转移样本(肝转移和肺转移)中的驱动基因也被证明具有异质性,除了著名的突变基因如APC和TP53可出现驱动突变外,新的候选突变基因ZFP36L2也被发现,并证明在细胞实验中能抑制CRC的转移[36]。在大量基因驱动突变事件(通常称为早期事件)的积累下,转移过程启动:原发性和转移性亚克隆均被发现,APC、TP53、KRAS、PIK3CA、TCF7L2和SMAD4的突变多为克隆突变,SMAD3、CDH11、CSMD1、EP300和ROBO1的突变多为亚克隆突变;在转移或耐药相关的基因中发现了具有异质性的亚克隆突变:KRAS、PTEN、PIK3CG[37]。有趣的是,与转移CRC肿瘤相比,有的原发肿瘤并没有发现TP53突变,而相应的原发肿瘤显示TP53调节因子共济失调突变基因(ATM)丝氨酸/苏氨酸激酶大量缺失[38],这揭示了单个基因在肿瘤转移中的动态进化事件。从CRC起始驱动突变开始逐步积累到转移的建立,在量变(突变积累)引起质变(转移)过程中的各种分子事件中,mCRC清晰的机制仍需要更多的证据来支撑。
3.2 靶向治疗相关的CRC耐药机制靶向治疗已经成为癌症治疗的重要手段,但肿瘤耐药克隆的出现,成为影响临床肿瘤患者治疗疗效及预后转归的一大棘手问题。抗表皮生长因子受体(EGFR)治疗已经成为治疗mCRC公认的手段之一。尽管野生型RAS肿瘤的患者可能从抗EGFR治疗中获益,但大多数患者最终会疾病进展,导致这一进展可能归因于持续的克隆进化,选择压力导致的继发性(获得性)治疗抵抗。RAS基因突变被认为是EGFR抑制剂治疗患者引起继发性耐药的一个重要机制,RAS以外的基因驱动变异也被证明对肿瘤生长和转归具有重要意义,可能在治疗相关的克隆多样化和获得性耐药中发挥作用。有研究表明已有TP53突变的肿瘤患者在抗EGFR治疗后,部分患者出现额外的TP53编码区突变,部分患者出现TP53、NRAS、SMAD4、IDH1和KRAS等基因的突发性突变[39]。
初期“一个基因,一种药物”大肠癌精准医疗模式出现,但单一药物对CRC靶向治疗的结果并不理想,例如EGFR抑制剂仅在KRAS野生型癌症患者中具有明显优势,而在KRAS突变的患者中,EGFR单抗治疗并不能很好地改善患者的总生存期[40]。在BRAFV600E突变的晚期CRC中使用单药EGFR抑制剂[41]或在KRAS突变疾病中使用MEK抑制剂[42]的治疗效果令人失望。在含有RAS或BRAF突变的肿瘤中,PIK3CA突变或PTEN缺失频繁共存,不仅KRAS突变对接受西妥昔单抗治疗后患者的预后有负面影响,BRAF、NRAS和PIK3CA外显子20突变对治疗低应答率也显著相关[43]。这些表明EGFR靶向治疗中导致耐药的未知分子事件增加了。然而,大多数CRC靶向药物实验尚缺乏特异性的生物标志物,未按照生物标志物对患者进行分层,也是制约精准治疗的瓶颈。美国病理协会联合其他机构推出了用于CRC分子生物学标志物研究指南,建立了针对CRC组织的分子生物标志物检测方案,以指导EGFR治疗和常规化疗[44]。
基于“一个基因,一种药物”的CRC精准医疗模式并不能普遍应用,“多基因、多药物”更为精密的联合医学模式应运而生。与使用FOLFIRI和FOLFOX加贝伐单抗治疗方案相比,FOLFOXIRI加贝伐单抗的应用,使mCRC患者具有更长的中位无进展生存期,中位总生存期从25.8个月提高到29.8个月[45]。BRAF、MEK和EGFR的联合抑制在BRAFV600突变的CRC临床试验中取得意想不到的疗效,实验组(三联治疗组和二联治疗组)较对照组具有更长的总生存期和更高的客观缓解率[46]。但靶向治疗下肿瘤进化驱动机制中的分子事件仍需进一步研究。
4 结语从最初的基因层面认识CRC,到现在众多导致CRC发生进展的基因变异事件慢慢的被挖掘,CRC的驱动基因变异事件被认识并有效用于指导早筛及临床治疗,有效地减少了CRC的发病率与死亡率,为患者带来了福音。CRC的分子标志物、治疗靶点、耐药机制在基因层面的研究已经取得了一定进展,但对于CRC进展中的转录后调控(如circRNA)方面的研究仍只是冰山一角。除了大量的circRNA在CRC进展中功能机制需要研究,其在CRC进化发育中扮演的具体角色及其与基因突变、MSI、免疫微环境、CMS分层事件的关系,需要进行归纳分层并尽早开发应用于临床。CRC尤其是mCRC驱动突变事件及转录后调控事件的复杂性仍然给治疗带来了巨大困难,空间转录组测序技术的出现可能有助于我们进一步了解这些复杂事件。研究CRC特异的相关分子事件,有助于我们对这些分子事件的作用进行更全面更精准的认知,从而推动CRC实际意义上的精准治疗和药物研发。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
周雄:文章撰写
胡明、蒋栋铭:文章撰写与作图
曹广文、谭晓㛃:文章指导与修改
| [1] |
周雄, 胡明, 李子帅, 等. 2020年全球及中国结直肠癌流行状况分析[J/OL]. 海军军医大学学报, 2022: 1-9. [Zhou X, Hu M, Li ZS, et al. Analysis of the epidemic situation of colorectal cancer in the world and China in 2020[J/OL]. Hai Jun Jun Yi Da Xue Xue Bao, 2022: 1-9.]
|
| [2] |
Arnold M, Sierra MS, Laversanne M, et al. Global patterns and trends in colorectal cancer incidence and mortality[J]. Gut, 2017, 66(4): 683-691. DOI:10.1136/gutjnl-2015-310912 |
| [3] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018[J]. CA Cancer J Clin, 2018, 68(1): 7-30. DOI:10.3322/caac.21442 |
| [4] |
Balmaña J, Balaguer F, Cervantes A, et al. Familial risk-colorectal cancer: ESMO Clinical Practice Guidelines[J]. Ann Oncol, 2013, 24(Suppl 6): vi73-vi80. |
| [5] |
Kidambi TD, Kohli DR, Samadder NJ, et al. Hereditary Polyposis Syndromesl[J]. Curr Treat Options Gastroentero, 2019, 17(4): 650-665. DOI:10.1007/s11938-019-00251-4 |
| [6] |
Grover S, Kastrinos F, Steyerberg EW, et al. Prevalence and phenotypes of APC and MUTYH mutations in patients with multiple colorectal adenomas[J]. JAMA, 2012, 308(5): 485-492. |
| [7] |
Kim B, Won D, Jang M, et al. Next-generation sequencing with comprehensive bioinformatics analysis facilitates somatic mosaic APC gene mutation detection in patients with familial adenomatous polyposis[J]. BMC Med Genomics, 2019, 12(1): 103. DOI:10.1186/s12920-019-0553-0 |
| [8] |
Spoto CPE, Gullo I, Carneiro F, et al. Hereditary gastrointestinal carcinomas and their precursors: An algorithm for genetic testing[J]. Semin Diagn Pathol, 2018, 35(3): 170-183. DOI:10.1053/j.semdp.2018.01.004 |
| [9] |
Zhang Y, Meng Q, Sun Q, et al. LKB1 deficiency-induced metabolic reprogramming in tumorigenesis and non-neoplastic diseases[J]. Mol Metab, 2021, 44: 101131. DOI:10.1016/j.molmet.2020.101131 |
| [10] |
Dal Buono A, Gaiani F, Poliani L, et al. Juvenile polyposis syndrome: An overview[J]. Best Pract Res Clin Gastroenterol, 2022, 58-59: 101799. DOI:10.1016/j.bpg.2022.101799 |
| [11] |
Yehia L, Keel E, Eng C. The Clinical Spectrum of PTEN Mutations[J]. Annu Rev Med, 2020, 71: 103-116. DOI:10.1146/annurev-med-052218-125823 |
| [12] |
Cerretelli G, Ager A, Arends MJ, et al. Molecular pathology of Lynch syndrome[J]. J Pathol, 2020, 250(5): 518-531. DOI:10.1002/path.5422 |
| [13] |
Vogelstein B, Papadopoulos N, Velculescu VE, et al. Cancer genome landscapes[J]. Science, 2013, 339(6127): 1546-1558. DOI:10.1126/science.1235122 |
| [14] |
Guinney J, Dienstmann R, Wang X, et al. The consensus molecular subtypes of colorectal cancer[J]. Nat Med, 2015, 21(11): 1350-1356. DOI:10.1038/nm.3967 |
| [15] |
Cancer Genome Atlas Research Network. Comprehensive molecular characterization of gastric adenocarcinoma[J]. Nature, 2014, 513(7517): 202-209. DOI:10.1038/nature13480 |
| [16] |
Li Y, Choi PS, Casey SC, et al. MYC through miR-17-92 suppresses specific target genes to maintain survival, autonomous proliferation, and a neoplastic state[J]. Cancer Cell, 2014, 26(2): 262-272. DOI:10.1016/j.ccr.2014.06.014 |
| [17] |
Cavallari I, Ciccarese F, Sharova E, et al. The miR-200 Family of microRNAs: Fine Tuners of Epithelial-Mesenchymal Transition and Circulating Cancer Biomarkers[J]. Cancers (Basel), 2021, 13(23): 5874. DOI:10.3390/cancers13235874 |
| [18] |
Shang A, Gu C, Wang W, et al. Exosomal circPACRGL promotes progression of colorectal cancer via the miR-142-3p/miR-506-3p-TGF-β1 axis[J]. Mol Cancer, 2020, 19(1): 117. DOI:10.1186/s12943-020-01235-0 |
| [19] |
Chen RX, Chen X, Xia LP, et al. N6-methyladenosine modification of circNSUN2 facilitates cytoplasmic export and stabilizes HMGA2 to promote colorectal liver metastasis[J]. Nat Commun, 2019, 10(1): 4695. DOI:10.1038/s41467-019-12651-2 |
| [20] |
Zheng R, Zhang K, Tan S, et al. Exosomal circLPAR1 functions in colorectal cancer diagnosis and tumorigenesis through suppressing BRD4 via METTL3-eIF3h interaction[J]. Mol Cancer, 2022, 21(1): 49. DOI:10.1186/s12943-021-01471-y |
| [21] |
Pan Z, Cai J, Lin J, et al. A novel protein encoded by circFNDC3B inhibits tumor progression and EMT through regulating Snail in colon cancer[J]. Mol Cancer, 2020, 19(1): 71. DOI:10.1186/s12943-020-01179-5 |
| [22] |
Liu W, Deng Y, Li Z, et al. Cancer Evo-Dev: A Theory of Inflammation-Induced Oncogenesis[J]. Front Immunol, 2021, 12: 768098. DOI:10.3389/fimmu.2021.768098 |
| [23] |
Jian X, He H, Zhu J, et al. Hsa_circ_001680 affects the proliferation and migration of CRC and mediates its chemoresistance by regulating BMI1 through miR-340[J]. Mol Cancer, 2020, 19(1): 20. DOI:10.1186/s12943-020-1134-8 |
| [24] |
Zhang L, Dong X, Yan B, et al. CircAGFG1 drives metastasis and stemness in colorectal cancer by modulating YY1/CTNNB1[J]. Cell Death Dis, 2020, 11(7): 542. DOI:10.1038/s41419-020-2707-6 |
| [25] |
Guo YX, Guo YY, Chen C, et al. Circ3823 contributes to growth, metastasis and angiogenesis of colorectal cancer: involvement of miR-30c-5p/TCF7 axis[J]. Mol Cancer, 2021, 20(1): 93. DOI:10.1186/s12943-021-01372-0 |
| [26] |
Liu X, Liu Y, Liu Z, et al. CircMYH9 drives colorectal cancer growth by regulating serine metabolism and redox homeostasis in a p53-dependent manner[J]. Mol Cancer, 2021, 20(1): 114. DOI:10.1186/s12943-021-01412-9 |
| [27] |
Yang H, Li X, Meng Q, et al. CircPTK2 (hsa_circ_0005273) as a novel therapeutic target for metastatic colorectal cancer[J]. Mol Cancer, 2020, 19(1): 13. DOI:10.1186/s12943-020-1139-3 |
| [28] |
Weng W, Wei Q, Toden S, et al. Circular RNA ciRS-7-A Promising Prognostic Biomarker and a Potential Therapeutic Target in Colorectal Cancer[J]. Clin Cancer Res, 2017, 23(14): 3918-3928. DOI:10.1158/1078-0432.CCR-16-2541 |
| [29] |
Liang ZX, Liu HS, Xiong L, et al. A novel NF-κB regulator encoded by circPLCE1 inhibits colorectal carcinoma progression by promoting RPS3 ubiquitin-dependent degradation[J]. Mol Cancer, 2021, 20(1): 103. DOI:10.1186/s12943-021-01404-9 |
| [30] |
Zheng L, Liang H, Zhang Q, et al. circPTEN1, a circular RNA generated from PTEN, suppresses cancer progression through inhibition of TGF-β/Smad signaling[J]. Mol Cancer, 2022, 21(1): 41. DOI:10.1186/s12943-022-01495-y |
| [31] |
Wang L, Zhou J, Zhang C, et al. A novel tumour suppressor protein encoded by circMAPK14 inhibits progression and metastasis of colorectal cancer by competitively binding to MKK6[J]. Clin Transl Med, 2021, 11(10): e613. |
| [32] |
Zhou J, Wang L, Sun Q, et al. Hsa_circ_0001666 suppresses the progression of colorectal cancer through the miR-576-5p/PCDH10 axis[J]. Clin Transl Med, 2021, 11(11): e565. |
| [33] |
Leufkens AM, van den Bosch MA, van Leeuwen MS, et al. Diagnostic accuracy of computed tomography for colon cancer staging: a systematic review[J]. Scand J Gastroenterol, 2011, 46(7-8): 887-894. DOI:10.3109/00365521.2011.574732 |
| [34] |
Cho SY, Chae J, Na D, et al. Unstable Genome and Transcriptome Dynamics during Tumor Metastasis Contribute to Therapeutic Heterogeneity in Colorectal Cancers[J]. Clin Cancer Res, 2019, 25(9): 2821-2834. DOI:10.1158/1078-0432.CCR-18-3460 |
| [35] |
Tang J, Tu K, Lu K, et al. Single-cell exome sequencing reveals multiple subclones in metastatic colorectal carcinoma[J]. Genome Med, 2021, 13(1): 148. DOI:10.1186/s13073-021-00962-3 |
| [36] |
Chen HN, Shu Y, Liao F, et al. Genomic evolution and diverse models of systemic metastases in colorectal cancer[J]. Gut, 2022, 71(2): 322-332. DOI:10.1136/gutjnl-2020-323703 |
| [37] |
Uchi R, Takahashi Y, Niida A, et al. Integrated Multiregional Analysis Proposing a New Model of Colorectal Cancer Evolution[J]. PLoS Genet, 2016, 12(2): e1005778. DOI:10.1371/journal.pgen.1005778 |
| [38] |
Ishaque N, Abba ML, Hauser C, et al. Whole genome sequencing puts forward hypotheses on metastasis evolution and therapy in colorectal cancer[J]. Nat Commun, 2018, 9(1): 4782. DOI:10.1038/s41467-018-07041-z |
| [39] |
Siena S, Sartore-Bianchi A, Garcia-Carbonero R, et al. Dynamic molecular analysis and clinical correlates of tumor evolution within a phaseⅡ trial of panitumumab-based therapy in metastatic colorectal cancer[J]. Ann Oncol, 2018, 29(1): 119-126. DOI:10.1093/annonc/mdx504 |
| [40] |
Douillard JY, Oliner KS, Siena S, et al. Panitumumab-FOLFOX4 treatment and RAS mutations in colorectal cancer[J]. N Engl J Med, 2013, 369(11): 1023-1034. DOI:10.1056/NEJMoa1305275 |
| [41] |
Tabernero J, Grothey A, Van Cutsem E, et al. Encorafenib Plus Cetuximab as a New Standard of Care for Previously Treated BRAF V600E-Mutant Metastatic Colorectal Cancer: Updated Survival Results and Subgroup Analyses from the BEACON Study[J]. J Clin Oncol, 2021, 39(4): 273-284. DOI:10.1200/JCO.20.02088 |
| [42] |
Zimmer L, Barlesi F, Martinez-Garcia M, et al. Phase I expansion and pharmacodynamic study of the oral MEK inhibitor RO4987655 (CH4987655) in selected patients with advanced cancer with RAS-RAF mutations[J]. Clin Cancer Res, 2014, 20(16): 4251-4261. DOI:10.1158/1078-0432.CCR-14-0341 |
| [43] |
De Roock W, Claes B, Bernasconi D, et al. Effects of KRAS, BRAF, NRAS, and PIK3CA mutationsonthe efficacy of cetuximab plus chemotherapy in chemotherapy-refractory metastatic colorectal cancer: a retrospective consortium analysis[J]. Lancet Oncol, 2010, 11(8): 753-762. DOI:10.1016/S1470-2045(10)70130-3 |
| [44] |
Sepulveda AR, Hamilton SR, Allegra CJ, et al. Molecular Biomarkers for the Evaluation of Colorectal Cancer: Guideline From the American Society for Clinical Pathology, College of American Pathologists, Association for Molecular Pathology, and American Society of Clinical Oncology[J]. J Mol Diagn, 2017, 19(2): 187-225. DOI:10.1016/j.jmoldx.2016.11.001 |
| [45] |
Cremolini C, Antoniotti C, Rossini D, et al. Upfront FOLFOXIRI plus bevacizumab and reintroduction after progression versus mFOLFOX6 plus bevacizumab followed by FOLFIRI plus bevacizumab in the treatment of patients with metastatic colorectal cancer (TRIBE2): a multicentre, open-label, phase 3, randomised, controlled trial[J]. Lancet Oncol, 2020, 21(4): 497-507. DOI:10.1016/S1470-2045(19)30862-9 |
| [46] |
Kopetz S, Grothey A, Yaeger R, et al. Encorafenib, Binimetinib, and Cetuximab in BRAF V600E-Mutated Colorectal Cancer[J]. N Engl J Med, 2019, 381(17): 1632-1643. DOI:10.1056/NEJMoa1908075 |
 2023, Vol. 50
2023, Vol. 50