文章信息
- 基于卷积神经网络的肿瘤影像诊断文献计量研究
- A Bibliometric Study of Oncology Imaging Diagnosis Based on Convolutional Neural Networks
- 肿瘤防治研究, 2023, 50(5): 512-517
- Cancer Research on Prevention and Treatment, 2023, 50(5): 512-517
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.22.1123
- 收稿日期: 2022-09-27
- 修回日期: 2022-11-16
2. 233030 蚌埠,蚌埠医学院护理学院
2. School of Nursing, Bengbu Medical College, Bengbu 233030, China
近年来,肿瘤的发病率和死亡率呈明显的上升趋势。据全球癌症统计数据显示,2020年全球有近1 930万新发癌症病例以及近1 000万例癌症死亡[1],癌症已经成为危害人类生命健康的重要疾病。随着医疗大数据时代的到来,使用深度学习的方法处理和分析影像数据已成为重要的研究趋势。特别是卷积神经网络算法可以帮助临床医生自动识别医学影像中的感兴趣区域。卷积神经网络属于深度学习模型,其架构专为图像分析而设计,卷积神经网络由多个构建块组成,包括卷积层、池化层和全连接层,通过反向传播算法自动地学习空间层次结构的特征[2]。被广泛应用于肿瘤的预测、诊断和解剖结构识别等研究[3]。
文献计量学是指通过运用数学和统计学的方法对知识载体进行定量分析的跨学科研究[4]。在肿瘤影像领域的应用主要集中于肺癌[5]、乳腺癌[6]、食管癌[7]等方面的可视化分析,而这些文献计量学研究只进行了某一类肿瘤的研究热点分析,所以无法把握肿瘤影像领域的研究趋势和热点。目前尚未发现关于卷积神经网络在肿瘤影像诊断中应用的有关文献计量分析,因此本研究基于SCI-E数据库,运用文献计量学的方法全面评估十年来该领域的研究现状和发展趋势,以助于研究人员掌握其发展脉络和研究热点。
1 资料与方法 1.1 文献来源及文献筛选标准文献来源于Web of Science Core Collection(WOSCC)的Science Citation Index Expanded(SCI-E)数据库,该数据库是国际公认的反映科学研究水准的数据库。纳入标准:文献类型为“Article”和“Review”,使用卷积神经网络对肿瘤进行检测、诊断和分割的研究,研究对象为人类,语言为English。排除标准:研究内容与卷积神经网络和肿瘤不相关或仅部分涉及,使用其他语言发表。
1.2 检索策略采用主题词检索。第一个检索式#1=(tumor*) OR(cancer*)OR(carcino*)OR (onco*);第二个检索式#2=(convolutional neural network) OR(CNN);第三个检索式#3=(medica* imag*) OR(CT)OR(MRI)OR(PET)OR(X-ray);第四个检索式#4=#1 AND #2 AND #3,表示检索到文献为同时满足前三个检索结果。检索时间范围:2012年1月1日——2022年4月30日。将检索到的文献由研究人员逐一阅读标题和摘要,根据纳入及排除标准进行筛选。
1.3 统计学方法将检索结果另存为带有完整记录和引用参考文献的纯文本文件。利用CiteSpace软件(5.8.R3)对相关文献进行可视化分析[8]。CiteSpace软件分析并制作纳入文献第一作者所在国家和机构、共被引作者、关键词以及突发性关键词的知识图谱。对意义相同而描述不一致的关键词进行标准化处理,例如将“convolutional neural network”合并为“CNN”。国家、机构和期刊用于研究文献的分布特征;作者共被引、关键词聚类以及突发性关键词用于分析当前的研究热点和发展趋势。时间跨度从2012年1月——2022年4月,时间切片为1年。阈值设置为Top N=50。可视化网络的剪裁方法设置为默认。CiteSpace以模块值(Q值)和平均轮廓值(S值)两个指标评价聚类结果。当Q > 3时代表划分的聚类有显著的差异性,当S > 5时表明聚类结果具有信服性[9]。
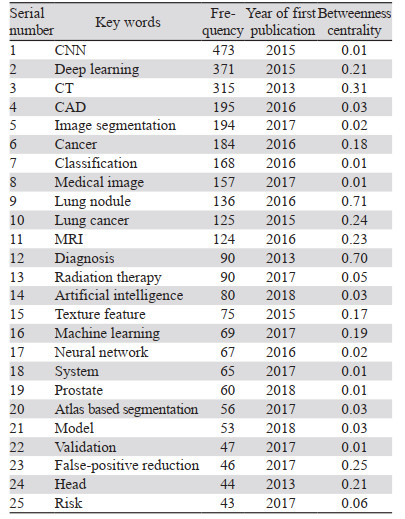
2 结果 2.1 发文量与研究分布 2.1.1 文献检索结果共检索到相关文献1 317篇,根据纳排标准,共纳入文献1 088篇,包括1021篇原创文献和67篇综述文献。从2012年至2022年公开发表的文献按时间顺序分布,从2017年开始该领域公开发表的文献呈现较快增长,并在2021年取得最多的发文量为362篇。虽然检索从2012年开始,但是第一篇相关文献出现在2013年,见图 1。
|
| 图 1 发文量随年度变化图 Figure 1 Change in annual number of published papers |
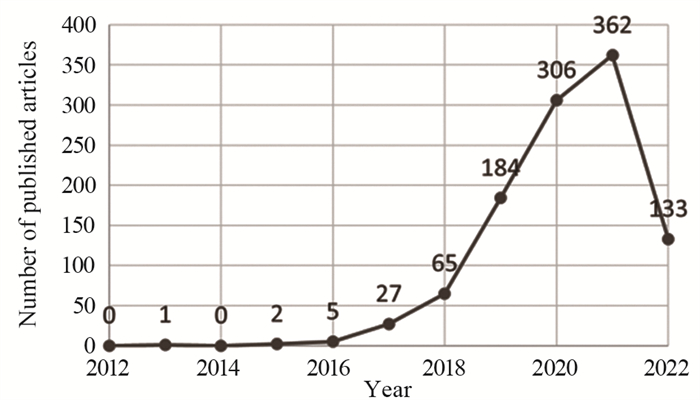
通过对发文国家进行可视化分析,得到发文国家共现网络图,图中不同的节点代表不同的元素,如第一作者发文的国家或机构,节点的大小反映了出版物的数量和频率,圆圈或连线的颜色对应不同的年份,节点之间连线的粗细代表协作、共现等关系的强弱。本图中清楚地显示出不同国家在卷积神经网络在肿瘤影像诊断领域的研究成果及影响力。文献出版数量前五的国家或地区是:中国425篇(39%)、美国299篇(27.5%)、印度70篇(6.4%)、韩国55篇(5.2%)和德国52篇(4.7%)。在可视化视图中,共有网络节点数64个和连接线87条,虽然有大量国家参与卷积神经网络在肿瘤影像诊断应用领域的研究,但仍以中、美等国为主,中美两国发文量占据发文总量的50%以上,见图 2。
|
| 图 2 发文国家共现网络分析图 Figure 2 Co-occurrence network analysis of countries |
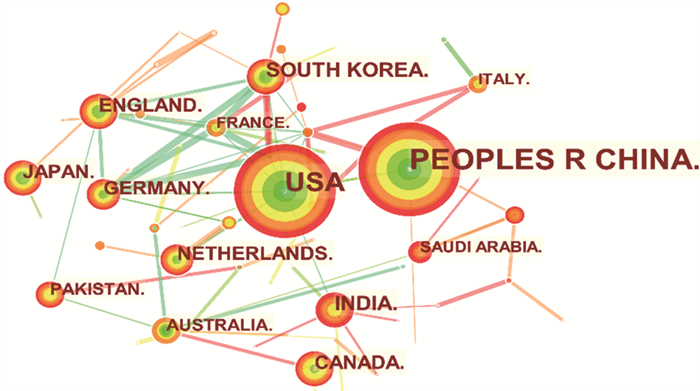
可视化分析显示相关的研究成果主要集中在中山大学39篇、上海交通大学29篇、中国科学院29篇、复旦大学21篇、哈佛大学医学院20篇。发文机构共现网络图共有网络节点数257个和连接数496条,见图 3。
|
| 图 3 发文机构共现网络分析图 Figure 3 Co-occurrence network analysis of institutions |
本研究对通过WOS数据库自带的文献统计工具进行期刊发文量统计。发文量前十名的期刊见图 4,发文量最多的期刊是Radiology Nuclear Medicine Medical Imaging(218篇),占总发文量的20%,远超排名第二的Engineering Biomedical(106篇)。

|
| 图 4 期刊发文量排名统计 Figure 4 Ranking of journal publications |
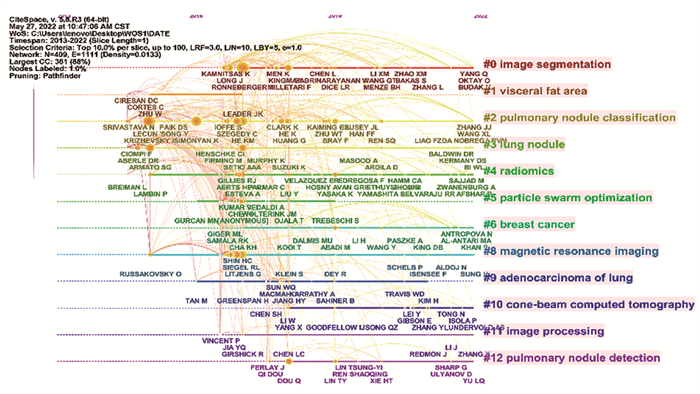
通过对作者共被引进行可视化分析,梳理研究学者重点关注的领域。作者共被引聚类结果模块值Q为0.68,轮廓值为0.86,共有节点数409个,连接线1 111条。通过对作者共被引聚类获得#0 image segmentation(2017-2022)、#1 visceral fat area(2015-2017)、#2 pulmonary nodule classification(2015-2022)、#3 lung nodule(2015-2022)、#4 radiomics(2015-2022)、#5 particle swarm optimization(2016-2019)、#6 breast cancer(2017-2022)、#8 magnetic resonance imaging(2015-2022)、#9 adenocarcinoma of lung(2016-2022)、# 10cone-beam computed tomography(2018-2022)、#11 image processing(2017-2022)和#12 pulmonary nodule detection(2018-2022)12个聚类,括号内代表的是聚类的起止年份,见图 5。
|
| 图 5 作者共被引时间线图可视化 Figure 5 Time plot of author co-citation |
CiteSpace软件提供了关键词的共现功能,从1 088篇文献中提取出423个关键词,对词频排名统计,同时计算每个关键词的中心度,其数值越大代表该关键词的重要性越大。本研究将频次40次以上的关键词作为高频词,共计得到25个高频关键词,高频关键词词频及中心性统计见表 1。在频率方面,“CNN”一词排名第一(473次),其次是“deep learning”,“CT”,“computer aided diagnosis”等,而中心性最大的是“lung nodule”,词频为136次。
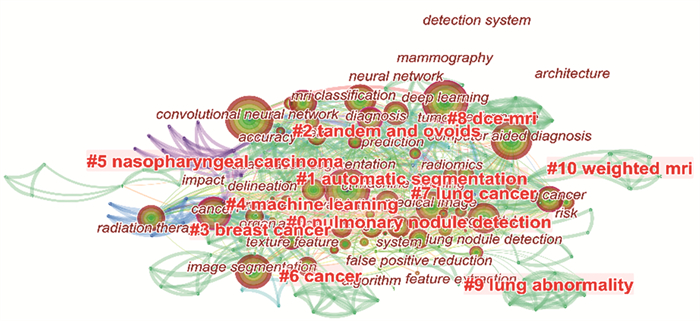
通过对关键词聚类获得#0 pulmonary nodule detection、#1 automatic segmentation、#2 tandem and ovoids、#3 breast cancer、#4 machine learning、#5 nasopharyngeal carcinoma、#6 cancer、#7 lung cancer、#8 dce-mri、#9 lung abnormality和#10 weighted mri 11个聚类,见图 6。关键词的聚类结果模块值Q为0.56,轮廓值为0.85。聚类图节点数共有237个,连接线1 204条。
|
| 图 6 关键词聚类分布可视化图 Figure 6 Visualization of keyword clustering distribution |
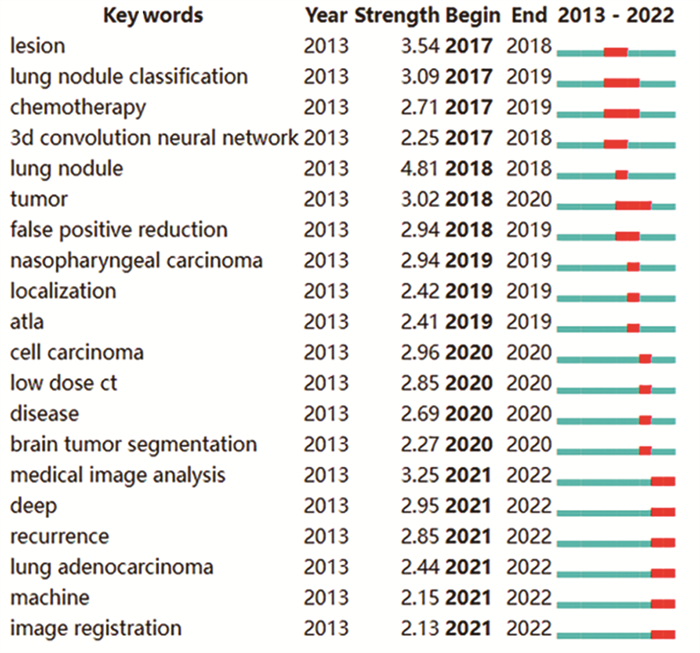
本研究通过使用CiteSpace软件对关键词进行突发性检测,以了解该领域的研究重点及未来的发展趋势。结果显示了突发性最强的前20个关键词,以及出现的时间段。绿线表示2013年至2022年的时间段,而每个突发性关键词的时间段由红线绘制,见图 7。
|
| 图 7 突发性关键词(前20名) Figure 7 Top 20 burst keywords (Top 20) |
本研究通过对十年来已经发表的文献进行了可视化分析,发现在2012年—2022年间卷积神经网络在肿瘤影像诊断应用领域的相关文献数量几乎呈指数级增长。在2021年达到高峰,数量为362篇,而后截至2022年4月发文量为133篇,预计未来一段时间增长的趋势将进一步保持,这表明越来越多的研究人员关注该领域。中国在该领域贡献了最多的文献量,5个主要研究机构贡献者中有4个是中国的医学研究机构,说明过去十年间中国在开展卷积神经网络的肿瘤影像诊断领域的研究工作中具有强大的核心竞争力。从期刊的分析来看,发文量排名前10的期刊,刊名中含有“计算机”、“医学影像”、“肿瘤”和“生物医学”等字样,说明该领域具有计算机科学、医学、工程学等多学科交叉的性质。
3.2 研究热点根据高频关键词和作者共被引时间线图可以发现,国际上关于卷积神经网络在肿瘤自动分割领域的研究较多,学者Ronneberger在作者共被引用排名最高(343次),其设计了一种像素级别的生物医学图像分割模型,实现了对肿瘤组织的分割[10]并被其他学者逐渐应用到肝脏[11]、肺结节[12]、胸部器官[13]等图像分割研究领域。关键词“lung nodule”中心度最大为0.71,表明肺结节的识别在卷积神经网络的肿瘤影像诊断应用领域占据着非常重要的地位[14]。
从对关键词聚类图谱分析发现,通过卷积神经网络研究乳腺癌的诊断和检测也是热点之一[15]。同时在“cancer”这一聚类词下,有关鼻咽癌[16]、皮肤癌[17]、前列腺癌[18]等肿瘤的诊断或鉴别诊断也都是当前研究的热点。
3.3 研究趋势根据突发性关键词可视化分析可知,肿瘤影像诊断领域卷积神经网络的应用在不同阶段呈现出不同的研究侧重点。在2021年之前,lesion(2017—2018)、lung nodule classification(2017—2019)、brain oncology segmentation(2020—2020)等突发性关键词表明该领域的研究主要集中于疾病分类、肺结节筛查和肿瘤分割等方面。从2021年开始,deep(2021—2022)、medical image analysis(2021—2022)、image registration(2021—2022)等突发性关键词表明研究核心向医学影像深度分析、多模态影像融合等方向发展。
通过对突发性关键词分析发现,“image registration”一词出现最晚,近年来研究学者更加关注多模态影像配准的方法学研究,传统的卷积神经网络模型的开发建立在一种模态影像的基础上,提供的信息往往有限,而利用MRI和PET、CT和PET、MRI和CT及MRI-T1和MRI-T2等多种方式实现多模态影像的融合[19],以提高模型识别的准确性。“medical image analysis”一词突发性最强,由于卷积神经网络模型存在过度拟合、可解释性和可重复性较差等局限性,所以通过将影像特征与基因组学特征相结合[20]、根据临床专家的先验知识指导模型结构和损失函数设计以及采用多中心机构的数据集训练验证模型[21]等方法,都是针对提高模型分析能力的优化和改进。所以提高卷积神经网络肿瘤影像模型的临床实用性和可解释性将是未来该领域研究的关键。
本研究通过绘制卷积神经网络的肿瘤影像诊断领域的文献知识图谱,全方位梳理了该领域的研究热点和发展趋势。同时也存在一定局限性,由于检索到的文献数量众多,本研究只对排名靠前的国家、机构、期刊、作者共被引和关键词进行了深入分析,但无法逐一分析具体文献,充分挖掘文献细节之间的关联,并且在作者共被引分析中存在只分析了第一作者和无法精准区分同名不同人的问题。以这项研究的结果为基础,未来该领域的计量学研究可侧重于重要研究学者和研究方向之间的内在关系。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
刘灵涛:设计研究方案、实施研究、撰写文章
刘玉文:指导分析数据
黄锦泉:实施研究、数据检索
张 楚:分析数据、解释数据
陈兴智:指导研究方案及撰写文章
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global Cancer Statistics 2020:GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
Yamashita R, Nishio M, Do RKG, et al. Convolutional neural networks: an overview and application in radiology[J]. Insights Imaging, 2018, 9(4): 611-629. DOI:10.1007/s13244-018-0639-9 |
| [3] |
Shen D, Wu G, Suk HI. Deep Learning in Medical Image Analysis[J]. Annu Rev Biomed Eng, 2017, 19(1): 221-248. DOI:10.1146/annurev-bioeng-071516-044442 |
| [4] |
周子番, 邱均平, 魏开洋. 从文献计量学到"五计学": 计量学方法的演化与发展[J]. 情报杂志, 2021, 40(10): 171-178. [Zhou ZF, Qiu JP, Wei KY. From Bibliometrics to "Five-Metrics": TheEvolution and Development of Bibliometrics Methods[J]. Qing Bao Za Zhi, 2021, 40(10): 171-178.] |
| [5] |
Liang H, Chen Z, Wei F, et al. Bibliometrics research on radiomics of lung cancer[J]. Transl Cancer Res, 2021, 10(8): 3757-3771. DOI:10.21037/tcr-21-1277 |
| [6] |
Zhang Y, Yu C, Zhao F, et al. Landscape of Artificial Intelligence in Breast Cancer (2000-2021): A Bibliometric Analysis[J]. FrontBiosci (Landmark Ed), 2022, 27(8): 224. DOI:10.31083/j.fbl2708224 |
| [7] |
Tu JX, Lin XT, Ye HQ, et al. Global research trends of artificial intelligence applied in esophageal carcinoma: A bibliometric analysis (2000-2022) via CiteSpace and VOSviewer[J]. Front Oncol, 2022, 12: 972357. DOI:10.3389/fonc.2022.972357 |
| [8] |
孙秋月, 孙力超, 张稚淳, 等. 基于CiteSpace的肿瘤干细胞研究态势的文献计量学分析[J]. 肿瘤防治研究, 2021, 48(9): 839-845. [Sun QY, Sun LC, Zhang ZC, et al. Trends in Research of Cancer Stem Cells: A Bibliometric Analysis Based on CiteSpace[J]. Zhong Liu Fang Zhi Yan Jiu, 2021, 48(9): 839-845.] |
| [9] |
陈悦, 陈超美, 刘则渊, 等. CiteSpace知识图谱的方法论功能[J]. 科学学研究, 2015, 33(2): 242-253. [Chen Y, Chen CM, Liu ZY, et al. The methodology function of CiteSpace mapping knowledge domains[J]. Ke Xue Xue Yan Jiu, 2015, 33(2): 242-253.] |
| [10] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional Networks for Biomedical Image Segmentation[C]//Navab N, Hornegger J, Wells WM, et al. Medical Image Computing and Computer-Assisted Intervention-MICCAI 2015. Cham: Springer International Publishing, 2015: 234-241.
|
| [11] |
Li X, Chen H, Qi X, et al. H-DenseUNet: Hybrid Densely Connected UNet for Liver and Oncology Segmentation From CT Volumes[J]. IEEE Trans Med Imaging, 2018, 37(12): 2663-2674. DOI:10.1109/TMI.2018.2845918 |
| [12] |
Wang S, Zhou M, Liu Z, et al. Central focused convolutional neural networks: Developing a data-driven model for lung nodule segmentation[J]. Med Image Anal, 2017, 40: 172-183. DOI:10.1016/j.media.2017.06.014 |
| [13] |
Dong X, Lei Y, Wang T, et al. Automatic multiorgan segmentation in thorax CT images using U-net-GAN[J]. Med Phys, 2019, 46(5): 2157-2168. DOI:10.1002/mp.13458 |
| [14] |
Zhang G, Yang Z, Gong L, et al. Classification of benign and malignant lung nodules from CT images based on hybrid features[J]. Phys Med Biol, 2019, 64(12): 125011. DOI:10.1088/1361-6560/ab2544 |
| [15] |
Heenaye-Mamode Khan M, Boodoo-Jahangeer N, Dullull W, et al. Multi-class classification of breast cancer abnormalities using Deep Convolutional Neural Network (CNN)[J]. PLoS One, 2021, 16(8): e0256500. DOI:10.1371/journal.pone.0256500 |
| [16] |
Wong LM, King AD, Ai QYH, et al. Convolutional neural network for discriminating nasopharyngeal carcinoma and benign hyperplasia on MRI[J]. Euro Radiol, 2021, 31(6): 3856-3863. DOI:10.1007/s00330-020-07451-y |
| [17] |
Esteva A, Kuprel B, Novoa RA, et al. Dermatologist-level classification of skin cancer with deep neural networks[J]. Nature, 2017, 542(7639): 115-118. DOI:10.1038/nature21056 |
| [18] |
Abdelmaksoud IR, Shalaby A, Mahmoud A, et al. Precise Identification of Prostate Cancer from DWI Using Transfer Learning[J]. Sensors (Basel), 2021, 21(11): 3664. DOI:10.3390/s21113664 |
| [19] |
Huang B, Yang F, Yin M, et al. A Review of Multimodal Medical Image Fusion Techniques[J]. Comput Math Methods Med, 2020, 2020: 8279342. |
| [20] |
Kirby J, Prior F, Petrick N, et al. Introduction to special issue on datasets hosted in The Cancer Imaging Archive (TCIA)[J]. Med Phys, 2020, 47(12): 6026-6028. DOI:10.1002/mp.14595 |
| [21] |
Zwirner K, Hilke FJ, Demidov G, et al. Radiogenomics in head and neck cancer: correlation of radiomic heterogeneity and somatic mutations in TP53, FAT1 and KMT2D[J]. Strahlenther Onkol, 2019, 195(9): 771-779. |
 2023, Vol. 50
2023, Vol. 50