文章信息
- AI细胞形态学联合DNA定量分析鉴别良恶性胸腹水的探讨
- AI Cytomorphology Combined with DNA-image Cytometry for Identifying Benign and Malignant Pleural Effusion and Ascites
- 肿瘤防治研究, 2023, 50(4): 390-396
- Cancer Research on Prevention and Treatment, 2023, 50(4): 390-396
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.22.0762
- 收稿日期: 2022-07-11
- 修回日期: 2022-09-02
2. 414000 岳阳,湖南师范大学附属岳阳医院肿瘤科
2. Department of Oncology, Yueyang People's Hospital, Hu'nan Normal University, Yueyang 414000, China
胸腹水是临床中常见的并发症,导致胸腹水的病因非常多,明确其良恶性对于临床医生判断疾病及预后至关重要。随着医学技术的发展,细胞形态学、分子诊断技术、免疫组织化学抗体等方法不断改善,已在临床中得到广泛应用。近年来出现了新的制片技术——液基细胞学(liquid-based cytologist technology, LCT)检测技术。研究表明[1-3]液基细胞学技术对细胞学检测的诊断价值高于传统细胞涂片法,但其仍由病理医生通过镜下诊断,这个过程繁琐并具有高度主观性,于是逐渐出现了无需病理医师阅片的DNA定量分析(DNA-image cytometry, DNA-ICM)鉴别良恶性胸腹水。国内外大量相关研究[4-8]表明DNA-ICM在鉴别良恶性胸腹水中有较高的临床应用价值,同时有研究[9]表明DNA定量分析异倍体的数目越高,其相关肿瘤的预后越差、中位生存期越短。20世纪90年代以来,人工智能(artificial intelligence, AI)分类系统开始对宫颈液基细胞进行多种分类与分级[10-11],利用该系统相似原理进行非妇科细胞学的检测也逐渐发展,包括甲状腺细胞[12]、尿液[13]、口腔上皮细胞[14-15]、胸腔积液[16],且具有较高的临床应用价值。良恶性胸腹水的准确鉴别有利于患者的早期诊断及早日获得临床最佳经济治疗,因此准确鉴别良恶性胸腹水对临床医师具有重要意义。
1 资料与方法 1.1 临床资料收集2021年5月—2022年3月在湖南师范大学附属岳阳医院住院患者的胸腹水标本,结合患者胸腹水细胞学、细胞蜡块、胸腹膜活检及免疫组织化学检测得出恶性胸腹水的标准结果,并查找相应原发病灶组织学诊断为恶性肿瘤;在排除恶性胸腹水后,根据临床资料及辅助检查得出良性胸腹水的标准结果。共收集360例胸腹水标本。恶性胸腹水105例,经原发病灶组织学病理统计,其中肺癌60例(肺腺癌51例,肺鳞癌5例,小细胞肺癌4例),卵巢腺癌22例,胃肠道肿瘤12例(结肠腺癌3例,胃腺腺癌3例,胆囊腺癌2例,直肠腺癌2例,胰腺腺癌1例,肝脏胆管细胞癌1例),乳腺癌11例。255例良性胸腹水,其中肺部感染56例,心力衰竭48例,结核性39例,低蛋白血症39例,肝硬化23例,肾衰竭21例,肺脓肿15例,其他综合原因11例,肝脓肿2例,结缔组织疾病1例。本研究获得湖南师范大学附属岳阳医院伦理委员会批准。
1.2 纳入与排除标准纳入标准:(1)入选病例一般资料完整,影像学检查提示胸腹腔积液且符合胸腹腔穿刺条件;(2)恶性胸腹腔积液诊断标准[17]:胸腹水细胞、细胞蜡块中找到恶性细胞、在胸腹膜活检组织中观察到恶性肿瘤的病理变化;(3)良性胸腹腔积液:根据恶性胸腹腔积液诊断标准[18]排除恶性病变。包括漏出液、感染性胸腔积液和结核性胸腹膜炎等。排除标准:(1)患者临床病史及诊疗资料不全以及无法进行胸腹水穿刺;(2)制片后胸腹水细胞数较少,DNA定量分析及人工智能细胞学分析系统无法得出结果;(3)无法明确标准诊断结果为良性或恶性的胸腹水标本。
1.3 操作方法LCT:常规抽取胸腹水标本2 h内1 500 r/min离心5 min(必要时加入抗凝剂4%枸橼酸钠以防凝固)→沉淀物加入细胞保存液涡旋振荡器振荡10 min→再次1 500 r/min,离心5 min→沉淀物加入带有黏附剂的制片仓→HE染色→晾干固定镜检,由两名病理医师镜检阅片判定结果。阳性结果:可疑癌细胞或癌细胞;阴性结果:未见癌细胞。
DNA-ICM:常规胸腹水标本2 h内1 500 r/min,离心5 min→沉淀物加入细胞保存液涡旋振荡器振荡10 min(必要时加入抗凝剂4%枸橼酸钠以防凝固)→再次1 500 r/min,离心5 min→沉淀物加入带有黏附剂的制片仓→Feulgen染色→晾干固定封片,运用全自动细胞DNA定量分析系统(湖南品信生物工程有限公司)扫描,得出DNA指数值(DNA index, DI)数量的峰值和面积图、异常及正常细胞的个数、各个细胞的DI值。DI=1为正常细胞;1 < DI < 2.5为正常增生或可疑癌细胞;DI≥2.5为癌细胞。结果判定:阳性:DI≥2.5(≥ 3个);阴性:DI值=1、1 < DI < 2.5或DI≥2.5(< 3个)。
AI:将上述液基细胞学染色制作的玻片放入吉米娜-玻片扫描影像分析系统(湖南品信生物工程有限公司)的玻片盒,按照系统操作步骤(具体操作请扫描本文OSID码),结果会提示阳性或阴性。
AI联合DNA-ICM辅助检测系统结果:DNA定量分析、人工智能细胞学检测的结果均为阴性则诊断为阴性结果,若两者均为阳性或其中一项为阳性结果,则由2位病理医师对结合扫描的细胞图像阅片进行审核,排除细胞重叠或杂质影响因素后可见异型细胞、可疑癌细胞、癌细胞定为阳性,未发现异型细胞为阴性。
1.4 统计学方法本研究采用SPSS25.0软件进行数据和统计分析。计算四种检测方法的敏感度、特异性、准确度、阳性预测值、阴性预测值、阳性似然比、阴性似然比、约登指数、受试者工作曲线并计算曲线下面积,采用卡方检验方法分别比较四种检测方法的诊断价值,P < 0.05表示差异有统计学意义,计算Kappa值进行一致性分析。
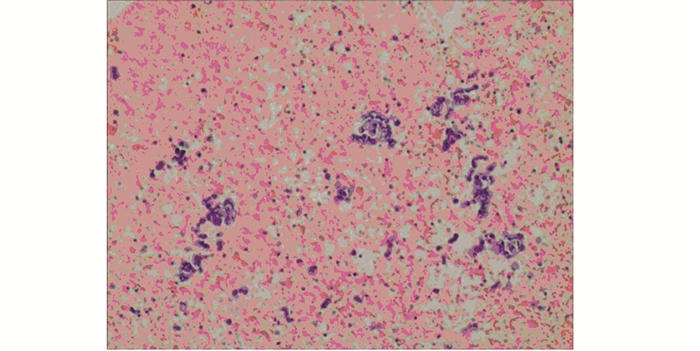
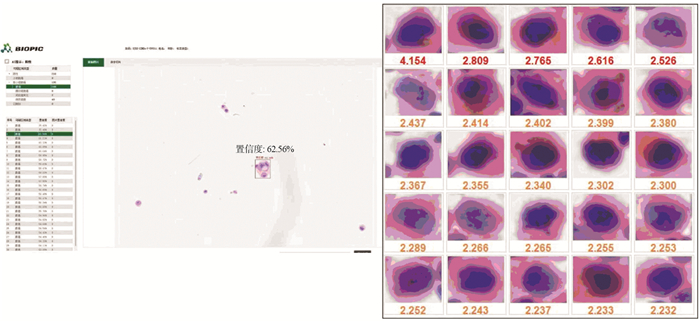
2 结果 2.1 四种诊断方法的检测结果105例恶性标本中LCT、DNA-ICM、AI、AI联合DNA-ICM辅助检测诊断阳性分别为81例、89例、97例、100例。255例良性标本中,四种检测方法诊断阳性分别为20例、31例、63例、15例。105例恶性标本中有8例经AI扫描为阴性,见图 1,并且该8例标本运用LCT检测均为阴性,见图 2。通过胸水标本离心获得浓缩沉淀细胞,加入固定液制成细胞块,所得细胞块经脱水、透明、浸蜡、包埋制成蜡块,再通过蜡块切片、HE染色、病理医师阅片得出癌性病变,病理类型为鳞癌3例、腺癌3例、小细胞癌2例,见图 3。另外,105例恶性标本中有19例LCT诊断为阴性,而通过AI联合DNA-ICM诊断为阳性,见图 4。

|
| 图 1 AI诊断阴性,胸水未发现异型细胞 Figure 1 AI diagnosis showed negative results. No heterotypic cells were found in pleural fluid |

|
| 图 2 LCT诊断阴性,胸水未发现异型细胞(HE ×100) Figure 2 LCT diagnosis showed negative results. No heterotypic cells were found in pleural fluid (HE ×100) |
|
| 图 3 胸水细胞蜡块技术示小细胞肺癌(HE ×100) Figure 3 Pleural fluid cell wax block technique showed the presence of small cell lung cancer (HE ×100) |
|
| 图 4 通过病理医师复核后AI联合DNA-ICM诊断该胸水结果为阳性 Figure 4 AI combined with DNA-ICM was used to diagnose pleural effusion, yielding positive results after being rechecked by the pathologist |
以入选标准中良恶性胸腹水的结果为标准,LCT、DNA-ICM、AI、AI联合DNA-ICM辅助诊断系统鉴别良恶性胸腹水的敏感度分别为77.14%、84.76%、92.38 %、95.23%;特异性分别为92.16%、83.92%、75.29%、94.12%;准确度分别为87.78%、84.17%、80.28%、94.44%;四种检测方法中病理医师通过AI联合DNA-ICM辅助诊断系统的敏感度、特异性、准确度最高,差异均具有统计学意义(均P < 0.05);三种单独检测方法比较,AI的敏感度最高,差异有统计学意义(P < 0.05);LCT的特异性和准确度最高,差异均具有统计学意义(均P < 0.05),见表 1。

|
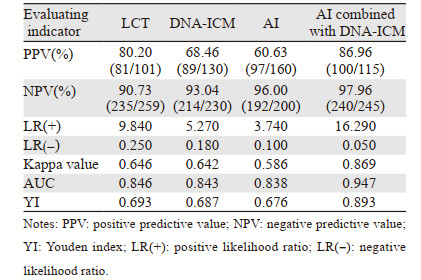
四种检测方法比较:AI联合DNA-ICM的阳性预测值86.96%、阴性预测值97.96%、阳性似然比16.29、阴性似然比为0.05、Kappa值为0.869、Youden指数为0.893、AUC为0.947均高于三种检测方法,见表 2、图 5。
|

|
| 图 5 四种检测方法对良恶性胸腹水鉴定的ROC曲线图 Figure 5 ROC curves of four detection methods for of identifying benign and malignant pleural effusion and ascites |
三种单独检测方法比较:LCT、DNA-ICM、AI检测的Kappa值分别为0.646、0.642、0.586;YI分别为0.693、0.687、0.676;AUC分别为0.846、0.843、0.838。阳性似然比分别为9.84、5.27、3.74;阴性似然比分别为0.25、0.18、0.10。阳性预测值分别为80.20%、68.46%、60.63%;阴性预测值分别为90.73%、93.04%、96.00%,三种检测方法中LCT检测方法的可靠性、真实性、诊断价值高,见表 2、图 5。
3 讨论LCT是目前鉴别胸腹水性质的主要方法之一,是一种新型的细胞学检查手段,与传统细胞学涂片法相比,该技术具有较高的细胞收集率、细胞结构清晰、分布均匀、厚度一致等优点[18]。本研究液基细胞学检测结果敏感度为77.14%,特异性为92.16%,准确度为87.78%,阳性预测值80.20%,阴性预测值90.73%。Wang团队[19]采用液基细胞检测法在70例胸腔积液的诊断敏感度为96.3%。国外一项多中心研究[20]运用液基细胞方法诊断2 721例恶性浆膜腔积液,其中包括2 505例胸腔积液和216例心包积液,显示88%的敏感度、100%的特异性、98%的准确度以及98%的阴性预测值。本研究结果与上述相关文献相比诊断价值较低,但同样具有较高的特异性及阴性预测值,可能与采取沉降式法或制片机的膜式吸附法不同制片技术及阅片差异所致,但液基细胞学技术在鉴别良恶性胸腹腔积液都具有相对较高的特异性而敏感度较低。
DNA-ICM的原理主要是通过Feulgen染色对核DNA进行成比例的染色,其染色与DNA的含量成正比,并测定细胞DNA含量值,即细胞核的积分光密度(integrated option density, IOD)来计算细胞核中DNA含量。DNA指数是被测细胞DNA在G0/G1期的IOD值与正常细胞DNA在G0/G1期IOD平均值的比值,DI为1表示该细胞为G1/G0的二倍体细胞,当有丝分裂开始时,变为四倍体(G2/M期)即DI=2[21-22]。诊断标准中1 < DI < 2.5则考虑为正常增生或可疑肿瘤细胞,DI≥2.5为肿瘤细胞。国外相关文献报道[23]DNA-ICM对70例胸腔积液标本进行检测,敏感度为88.89%,绘制其ROC曲线下面积为0.766。Meng等[4]用DNA-ICM对126例浆膜腔积液的样本进行测定,采用DI超过2.5的4个细胞是非整倍性的最佳截断值,其诊断恶性积液的敏感度为88.3%,特异性为100%。Liu等[5]利用DNA-ICM诊断良恶性胸腔积液的敏感度为85.00%、特异性为85.00%。本研究中,DNA-ICM鉴别良恶性胸腹水的敏感度为84.76%,与上述研究结果大致相同,本研究的特异性83.92%比Meng等[4]研究低,Meng等采用的是DI大于2.5的4个细胞即诊断为肿瘤细胞,而本研究的诊断标准为DI超过2.5大于3个细胞,导致特异性降低,同时要考虑到DNA定量分析受仪器、相机、成像原理、标本制片染色质量等多因素的影响。
AI的核心技术是全玻片数字扫描技术(whole slide imaging, WSI)及运用卷积神经网络(Convolutional Neural Networks, CNN)的深度学习算法[24]。Williams等[25]应用计算机技术自动将液基细胞涂片玻片转换为全方位高分辨率的全景图像,并采集病理切片的局部显微图像,可以大大减少病理医生的工作量并减少人为原因导致的误诊,降低患者病理切片的识别遗漏和丢失。CNN是AI在图像处理领域最突出的技术,CNN工作原理主要是先将计算机对WSI图片分为训练集和验证集,病理学家根据诊断标准对训练集上的图像进行标注,再由CNN通过学习标注过的数据和信息特征进行提取,开发一种算法,通过算法让AI对验证集自动作出标记并分析得出结论[26-27]。人工智能分类模型目前对宫颈液基细胞学报道略多,国内朱孝辉等[28]报道了AI分类系统对鉴别宫颈上皮内病变的敏感度为92.90%、特异性87.02%。Sanyal等[29]开发两种分类模型的综合评估在检测良恶性宫颈细胞学方面的敏感度为95.63%,特异性为79.85%,阴性预测值很高为99.19%,表明在筛查中具有潜在效用。
重叠的细胞、中性粒细胞和碎片导致的假阳性是评估过程中遇到的主要困难。目前国内对胸腹水的人工智能液基细胞学系统相关研究报道较少,2021年Li等[30]最新报道研究比较了深度学习和机器学习技术在鉴别良恶性胸腔积液的适用性,最终得出深度学习模型在训练、测试和验证集中的AUC分别为0.995、0.848、0.917。Su等[31]开发几种用于鉴别良恶性腹水的深度学习系统并比较了七个分类系统性能,最终获得一种最佳模型,该模型分类的准确率达96.80%,AUC达0.8851,此外,分类模型可进一步发展为具有更多类别而不仅仅是两个类别(良性和恶性),并且不需要重新训练细胞。Xie等[16]开发的深度学习算法在胸腔积液上评估,该方法的敏感度、特异性、准确度分别为87.50%、94.44%、91.67%,AUC为0.9526。本研究采用人工智能液基细胞学单独诊断良恶性胸腹水的敏感度、特异性、准确度分别为92.38%、75.29%、80.28%,敏感度与上述国内外研究相近,特异性较低的原因可能为人工智能模型训练集之间的图像识别、检测、分割、分析标注和诊断方法存在差异,考虑为:一是目前宫颈液基细胞学人工智能诊断系统的深度学习模型较胸腹水深度学习模型完善,宫颈细胞学图像中细胞核之间分离良好、细胞成分及种类较胸腹水少,其发展时期标注切片例数较多;二是深度学习的大部分模型需要进行标注,而胸腹水肿瘤细胞的异质性和复杂类型增加了人工智能标注的难度及不确定性,同时也决定了宫颈细胞与胸腹水细胞的分类诊断价值的巨大区别。
本研究中两名细胞病理医师根据人工智能细胞学及DNA定量分析扫描出的图像及相关结果进行阅片分析,最终得出敏感度、特异性、准确度分别为95.23%、94.12%、94.44%,表明病理医师结合人工智能细胞学与DNA-ICM检测结果较可靠并与标准结果的一致性好,其诊断价值较高。朱孝辉等[28]报道细胞病理医师根据AI辅助系统阅片得出的敏感度为99.34%、特异性为99.10%,AI最终有望部分替代细胞病理学医师的初筛工作,促进实现精确分类的最终目标。AI在临床中的全面实行尚需时日,究其原因:一是缺乏质量较高的病理切片,相比影像学方面,AI应用于病理领域面临着更大的挑战,需人工进行大量切片注释并且花费的成本较高;二是病理学人工智能系统泛化能力不足,某一医疗机构、区域来源、制作切片方式的不同导致所研发的产品存在差异,是否能识别其他地域的病理切片并作出准确诊断均是病理AI发展要考虑的环节。
综上,运用人工智能细胞学辅助诊断系统是数字病理学对细胞核DNA及细胞形态学的结合,同时在病理医师的复核阅片下其诊断敏感度得到提高,多重检测提高了特异性,能够显著提升甚至超越常规病理图像的诊断水平及范畴。若经济实力及设备允许,同时配合病理医师复核,可以挖掘更大的临床应用价值。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献:
江杨:实验操作,统计学分析,论文撰写
于惠芝:临床资料收集及病例分析
高亚:统计学分析,阅片判读
沈昱:实验设计及操作
毛敏:阅片判读,结果分析
刘崇梅:课题设计,论文修改
| [1] |
王应霞, 赵珍, 杨梅秋, 等. 液基细胞学检测在良恶性胸腹水鉴别诊断中的应用[J]. 昆明医科大学学报, 2016, 37(4): 45-48. [Wang YX, Zhao Z, Yang MQ, et al. Application of liquid based cytology in differential diagnosis of benign and malignant hydrothorax and ascites[J]. Kunming Yi Ke Da Xue Xue Bao, 2016, 37(4): 45-48.] |
| [2] |
Osaka R, Hayashi K, Onda T, et al. Evaluation of liquid based cytology for tongue squamous cell carcinoma: Comparison with conventional cytology[J]. Bull Tokyo Dent Coll, 2019, 60(1): 29-37. DOI:10.2209/tdcpublication.2018-0015 |
| [3] |
Ito K, Kimura R, Konishi H, et al. A comparison of liquid-based and conventional cytology using data for cervical cancer screening from the Japan Cancer Society[J]. Jpn J Clin Oncol, 2020, 50(2): 138-144. DOI:10.1093/jjco/hyz161 |
| [4] |
Meng Z, Shi J, Zhu C, et al. Automated quantification of DNA aneuploidy by image cytometry as an adjunct for the cytologic diagnosis of malignant effusion[J]. Anal Cell Pathol (Amst), 2013, 36(3-4): 107-115. DOI:10.1155/2013/501284 |
| [5] |
Liu C, Huang L, Zhang X, et al. Combination of DNA ploidy analysis and miR-21 or miR-24 in screening malignant pleural effusion[J]. Interact CardioVasc Thorac Surg, 2018, 26(3): 376-381. DOI:10.1093/icvts/ivx327 |
| [6] |
许娟. 细胞DNA倍体联合肿瘤标志物检测在良恶性胸腔积液、腹水鉴别中的应用价值[J]. 检验医学与临床, 2019, 16(23): 3469-3471. [Xu J. Application value of cell DNA ploidy combined with tumor marker detection in the identification of benign and malignant hydrothorax and ascites[J]. Jian Yan Yi Xue Yu Lin Chuang, 2019, 16(23): 3469-3471.] |
| [7] |
戴翠婷. DNA图像倍体分析细胞块脱落细胞涂片在胸腔积液良恶性诊断中的应用效果对比[J]. 基层医学论坛, 2020, 24(22): 3207-3208. [Dai CT. Comparison of application effect of DNA image ploidy analysis cell block exfoliated cell smear in the diagnosis of benign and malignant pleural effusion[J]. Ji Ceng Yi Xue Lun Tan, 2020, 24(22): 3207-3208.] |
| [8] |
张素霞, 周红, 宇小婷, 等. 全自动DNA定量分析检测对恶性胸腔积液诊断的价值[J]. 中华病理学杂志, 2015(9): 653-654. [Zhang SX, Zhou H, Yu XT, et al. The value of automatic DNA quantitative analysis in the diagnosis of malignant pleural effusion[J]. Zhonghua Bing Li Xue Za Zhi, 2015(9): 653-654.] |
| [9] |
Benlloch S, Martí-Ciriquián JL, Galbis-Caravajal JM, et al. Cell-free DNA concentration in pleural fluid and serum: quantitative approach and potential prognostic factor in patients with cancer and pleural effusions[J]. Clin Lung Cancer, 2006, 8(2): 140-145. DOI:10.3816/CLC.2006.n.043 |
| [10] |
Zhang L, Lu L, Nogues I, et al. DeepPap: deep convolutional networks for cervical cell classification[J]. IEEE J Biomed Health Inform, 2017, 21(6): 1633-1643. DOI:10.1109/JBHI.2017.2705583 |
| [11] |
李雪, 石中月, 杨志明, 等. 人工智能辅助分析在宫颈液基薄层细胞学检查中的应用价值[J]. 首都医科大学学报, 2020, 41(3): 360-363. [Li X, Shi ZY, Yang ZM, et al. Value about artificial intelligence-assisted liquid-based thin-layer cytology for cytologycervical cancer screening[J]. Shou Du Yi Ke Da Xue Xue Bao, 2020, 41(3): 360-363.] |
| [12] |
叶美华, 陈万远, 蔡博君, 等. 基于卷积神经网络的甲状腺液基细胞学病理辅助诊断模型的研究[J]. 中华病理学杂志, 2021, 50(4): 358-362. [Ye MH, Chen WY, Cai BJ, et al. A convolutional neural network based model for assisting pathological diagnoses on thyroid liquid-based cytology[J]. Zhonghua Bing Li Xue Za Zhi, 2021, 50(4): 358-362.] |
| [13] |
胡长爱, 杨大干, 叶章辉, 等. 基于智能数据和机器学习的尿液检验结果解释性报告[J]. 中华检验医学杂志, 2021, 44(6): 524-531. [Yang CA, Yang DG, Ye ZH, et al. Interpreting report of urinalysis based on intelligent data and machine learning[J]. Zhonghua Jian Yan Yi Xue Za Zhi, 2021, 44(6): 524-531.] |
| [14] |
Das N, Hussain E, Mahanta LB. Automated classification of cells into multiple classes in epithelial tissue of oral squamous cell carcinoma using transfer learning and convolutional neural network[J]. Neural Netw, 2020, 128: 47-60. DOI:10.1016/j.neunet.2020.05.003 |
| [15] |
Zhang H, Li W, Zhang H. An Image Recognition Framework for Oral Cancer Cells[J]. J Healthc Eng, 2021, 2021: 2449128. |
| [16] |
Xie X, Fu CC, Lv L, et al. Deep convolutional neural network-based classification of cancer cells on cytological pleural effusion images[J]. Mod Pathol, 2022, 35(5): 609-614. |
| [17] |
中国恶性胸腔积液诊断与治疗专家共识组. 恶性胸腔积液诊断与治疗专家共识[J]. 中华内科杂志, 2014, 53(3): 252-256. [Chinese expert consensus group on diagnosis and treatment of malignant pleural effusion. Expert consensus on diagnosis and treatment of malignant pleural effusion[J]. Zhonghua Nei Ke Za Zhi, 2014, 53(3): 252-256.] |
| [18] |
Jakhar D, Kaur I. Artificial intelligence, machine learning and deep learning: definitions and differences[J]. Clin Exp Dermatol, 2020, 45(1): 131-132. |
| [19] |
Wang S, Li D, Wang J, et al. A Comparative Study of Liquid-Based Cytology and DNA Image Cytometry in the Diagnosis of Serous Effusion[J]. Technol Cancer Res Treat, 2020, 19: 1533033820942298. |
| [20] |
Rossi ED, Bizzarro T, Schmitt F, et al. The role of liquid‐based cytology and ancillary techniques in pleural and pericardic effusions: an institutional experience[J]. Cancer Cytopathol, 2015, 123(4): 258-266. |
| [21] |
郭宇妮. 多抗体检测、细胞DNA定量分析在肺癌诊疗中的应用研究[D]. 温州医科大学, 2015. [Guo YN. The detection of Lung cancer makers and the apply of DNA body in the judge of disease nature[D]. Wenzhou Medical University, 2015.]
|
| [22] |
Liang Y, Yin Z, Liu H, et al. Weakly Supervised Deep Nuclei Segmentation with Sparsely Annotated Bounding Boxes for DNA Image Cytometry[J]. IEEE/ACM Transact Comput Biol Bioinform, 2021, PP.
|
| [23] |
钱同, 樊昱彤, 金玉, 等. 液基细胞学、肿瘤标志物SP70联合检测对肺腺癌转移胸腔积液的诊断价值[J]. 临床与病理杂志, 2020, 40(10): 2529-2533. [Qian T, Fan YT, Jin Y, et al. Value of tumor marker SP70 detection combined with liquid based cytology in diagnosing lung adenocarcinoma in pleural effusion[J]. Lin Chuang Yu Bing Li Za Zhi, 2020, 40(10): 2529-2533.] |
| [24] |
LeCun Y, Bengio Y, Hinton G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. |
| [25] |
Williams BJ, Bottoms D, Treanor D. Future-proofing pathology: the case for clinical adoption of digital pathology[J]. J Clin Pathol, 2017, 70(12): 1010-1018. |
| [26] |
马艳春, 刘永坚, 解庆, 等. 自动图像标注技术综述[J]. 计算机研究与发展, 2020, 57(11): 2348-2374. [Ma YC, Liu YJ, Xie Q, et al. ReView of Automatic Image Annotation Technology[J]. Ji Suan Ji Yan Jiu Yu Fa Zhan, 2020, 57(11): 2348-2374.] |
| [27] |
Ehteshami Bejnordi B, Mullooly M, Pfeiffer RM, et al. Using deep convolutional neural networks to identify and classify tumor-associated stroma in diagnostic breast biopsies[J]. Mod Pathol, 2018, 31(10): 1502-1512. |
| [28] |
朱孝辉, 李晓鸣, 张文丽, 等. 人工智能辅助诊断在宫颈液基薄层细胞学中的应用[J]. 中华病理学杂志, 2021, 50(4): 333-338. [Zhu XH, Li XM, Zhang WL, et al. Application of artificial intelligence-assisted diagnosis for cervical liquid‐based thin‐layer cytology[J]. Zhonghua Bing Li Xue Za Zhi, 2021, 50(4): 333-338.] |
| [29] |
Sanyal P, Barui S, Deb P, et al. Performance of a convolutional neural network in screening liquid based cervical cytology smears[J]. J Cytol, 2019, 36(3): 146-151. |
| [30] |
Li Y, Tian S, Huang Y, et al. Driverless artificial intelligence framework for the identification of malignant pleural effusion[J]. Transl Oncol, 2021, 14(1): 100896. |
| [31] |
Su F, Sun Y, Hu Y, et al. Development and validation of a deep learning system for ascites cytopathology interpretation[J]. Gastric Cancer, 2020, 23(6): 1041-1050. |
 2023, Vol. 50
2023, Vol. 50


