文章信息
- 胰腺癌中OASL的表达及其对癌细胞增殖和迁移的影响
- OASL Expression in Pancreatic Cancer and Its Effect on Proliferation and Migration of Pancreatic Cancer Cells
- 肿瘤防治研究, 2023, 50(1): 18-26
- Cancer Research on Prevention and Treatment, 2023, 50(1): 18-26
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2023.22.0741
- 收稿日期: 2022-07-05
- 修回日期: 2022-10-04
2. 637000 南充,川北医学院四川省医学影像重点实验室
2. Sichuan Key Laboratory of Medical Imaging, North Sichuan Medical College, Nanchong 637000, China
胰腺癌是一种临床症状隐匿、恶性程度高且死亡率高的消化道恶性肿瘤。据最新临床文献统计数据,目前胰腺癌仍然是恶性肿瘤里五年生存率最低的肿瘤,仅为10%[1]。胰腺癌的治疗方法仍以根治性手术切除为主,但是胰腺癌往往发现较晚,患者确诊时,因肿瘤发生远端转移或不可手术切除而丧失手术机会[2]。近年来在其他肿瘤治疗中取得良好效果的靶向治疗、免疫治疗等在胰腺癌的临床试验中效果仍不明显。因此,迫切需要寻求新的生物标志物和新的靶向位点,探究其在胰腺癌发生发展中的作用机制。
OASL是一种蛋白质编码基因,编码59kDa的蛋白质,故又称p59OASL。它位于12q24.31,有7个外显子,主要在骨髓、胃和肺中表达,与OAS1、OAS2和OAS3一起形成干扰素诱导的蛋白质保守家族。有报道指出,OASL基因可能是维持肺癌细胞对acRoots敏感度的决定性调节因子之一,并可能与耐药性的发展有关[3]。本研究在对TCGA数据库中OASL基因进行生物信息学分析基础上,对胰腺癌细胞株panc-1敲减和过表达OASL,探讨其对胰腺癌细胞生物学行为的影响及其相关分子机制。希望能为胰腺癌的临床诊治寻找到新的靶向标志物,优化诊治方案,延长患者的生存期。
1 材料与方法 1.1 实验材料人类胰腺癌panc-1细胞购自中国上海中科院细胞库。敲减panc-1细胞OASL基因的shRNA以及过表达OASL基因的慢病毒均购买于上海吉凯基因科技有限公司。DMEM基础培养基(货号:PM150210)、胎牛血清(货号:164210-50)均购自武汉普诺赛生命科技有限公司,胰酶(货号:SH30042.01)、双抗(货号:SV30010)购自美国Hyclone公司,Lipo8000TM转染试剂(货号:C0533)、嘌呤霉素(货号: ST551)、RIPA裂解液(货号:P0013B)、10% Tris-Gly预制胶(货号:P0455S)、PVDF膜(货号: FFP32)、兔抗人GAPDH多克隆抗体(货号: AF1186)、辣根过氧化物酶标记山羊抗兔IgG(货号:A0208)、MTT试剂(货号:ST316)均购自上海碧云天生物科技公司,伤口愈合插件(货号:80209)购自德国Ibidi公司,兔抗人OASL多克隆抗体(货号:821065)购自成都正能生物,逆转录试剂盒Fermentas(货号:K1622)、PowerUp SYBR Green预混液(货号:A25742)均购自美国Thermo Scientific公司,qPCR引物均购自北京擎科生物科技有限公司。GAPDH引物序列:正义:5’-GGAGCGAGATCCCTCCAAAAT-3’,反义:5’-GGCTGTTGTCATACTICICATGG-3’;OASL引物序列:正义:5’-CTGATGCAGGAACTGTATAGCAC-3’,反义:5’-CACAGCOTCTAGCACCTCTT-3’。
1.2 实验方法 1.2.1 生物信息学方法分析OASL与胰腺癌预后的关系本研究利用GEPIA数据库分析不同疾病状态(肿瘤或正常)下OASL基因的表达情况及生存分析。在数据库中选取Single Gene Analysis,输入基因名称:OASL,再分别点击Boxplots和Stage Plots,在Datasets Selection中选择PAAD;在Survival中选择Survival Plots,在Methods中分别选择Overall Survival和Disease Free Survival。
利用Kaplan-Meier Plotter数据库(http://kmplot.com/analysis/) 评估OASL的表达与胰腺癌患者总生存期(OS)和无复发生存期(RFS)的关系。在该平台中点击pan-cancer RNA-seq,Gene symbol中输入目的基因OASL,Split patients中选择Auto select best cutoff,Cancer中选择Pancreatic ductal adenocarcinoma,Survival分别选择OS和RFS。从TCGA数据库中检索胰腺癌患者的临床资料,用R语言对数据进行分析。调整后的P值< 0.05和|log2(倍数变化)| > 1被定义为阈值。
1.2.2 细胞培养、转染及分组细胞培养:人类胰腺癌panc-1细胞使用含10%FBS的DMEM培养基,放入37℃、5%CO2细胞培养箱中培养。
shRNA转染:当细胞生长状态良好,覆盖培养皿约90%面积时,吸去培养皿中的培养基,用PBS洗涤2次,胰蛋白酶消化细胞,再加入完全培养基配制成细胞悬液,以每孔3×105个细胞接种到6孔板内进行培养,培养24 h后,当每孔细胞密度达到70%~80%,弃掉培养基,每孔加入2 ml新鲜的完全培养基。取一个EP管,按照说明书以每孔加入125 μl Opti-MEM,再加入2.5 μg质粒,最后加入4 μl Lipo8000TM转染试剂(质粒用量(μg)和Lipo8000TM(μl)的用量比例为1:1.6)配置好混合液。每孔加入125 μl转染试剂—质粒混合物,轻轻混匀。放入37℃、5%CO2细胞培养箱中继续培养48 h,用荧光显微镜观察转染情况。加入含嘌呤霉素(Puromycin)的培养基继续培养,以获得稳定转染株细胞。
慢病毒转染:取对数生长期的panc-1细胞,以每孔1×105个细胞接种于6孔板中,放入37℃、5%CO2细胞培养箱中培养。当细胞汇合度达到20%~30%时,按照说明书推荐的MOI值(MOI=10),加入稀释为1×108 TU/ml浓度的病毒和HitransG P液。在培养箱中继续培养12~16 h后,更换完全培养基,转染72 h后在荧光显微镜下观察转染效率。加入含嘌呤霉素的培养基继续培养,以获得稳定转染株细胞。
实验分组:将shRNA转染所获得的OASL基因稳定低表达的panc-1细胞株命名为KD-OASL组,转染的阴性对照的panc-1细胞株命名为KD-NC组。将病毒转染所获得的OASL基因稳定高表达的panc-1细胞株命名为OE-OASL组,转染的阴性对照的panc-1细胞株命名为OE-NC组。
1.2.3 RT-qPCR实验取处于对数生长期的细胞,提取RNA。采用20 μl体系反转录。按顺序在EP管中加入cDNA模板3 μg、Oligo(dT)18 Primer 1 μl、Random Hexamer Primer 1 μl、RNA free ddH2O配满12 μl。混匀,1 000 r/min,短暂离心10 s,65℃孵育5 min,冰上冷却,1 000 r/min,离心10 s,再冰上冷却。按顺序加入5× Reaction Buffer 4 μl、RiboLock RNase Inhibitor 1 μl、10 mmol/L dNTP Mix 2 μl、ReverAidTM Reverse Transcriptase 1 μl。反转录条件:42℃ 60 min,75℃ 5min。使用10 μl体系进行RT-qPCR。每组加入cDNA 2 μl,PowerUpTM SYBRTM Green Master Mix 5 μl,Forward Primer 0.2 μl,Reverse Primer 0.2 μl,ddH2O 2.6 μl。反应条件为:50℃ 120 s,95℃ 120 s,95℃ 15 s,57℃ 15 s,72℃ 60 s,95℃ 10 s,65℃ 60 s,97℃ 1 s,37℃ 30 s,45个循环。以GAPDH为内参基因,转染效率用2-ΔΔCt法进行分析。
1.2.4 Western blot实验RIPA与蛋白酶抑制剂按照100:1的比例混匀,用于裂解细胞。取适量提取的蛋白,与5× SDS-PAGE蛋白上样缓冲液以4:1的体积比例混匀,60℃煮15 min。取10% Tris-Gly预制胶,在各泳道中加入25 μg蛋白进行电泳。用0.45 μm PVDF膜转膜后,将膜放入5%脱脂奶粉封闭液中封闭1 h。再加入稀释好的一抗,4℃缓慢摇动孵育过夜。加入稀释好的二抗,室温在摇床上缓慢摇动孵育1 h。将配好的化学发光液滴在PVDF膜上,在荧光成像仪内进行成像检测。Image J软件分析各条带灰度值。
1.2.5 MTT实验取对数生长期的各组细胞分别接种于96孔板中,每孔5×103个细胞,每组5个复孔。待细胞贴壁后,在24、48、72、96、120 h分别加入MTT试剂,每孔10 μl,置于培养箱孵育4 h,吸去上清液,小心不要吸掉孔底部形成的深紫色产物formazan,每孔再加入100 μl DMSO,将96孔板放在摇床上缓慢摇动30 min,待formazan结晶完全溶解后,于酶标仪595 nm处检测各组细胞的吸光度值。
1.2.6 平板克隆形成实验取对数生长期的各组细胞分别接种6孔板中,每孔300个细胞,每组3个复孔,每孔加入2 ml DMEM完全培养基,在37℃培养箱培养约10天,待长出肉眼可见的克隆,去掉培养基,PBS清洗2次,用4%的多聚甲醛对细胞固定15 min,吸弃4%的多聚甲醛,加入0.1%结晶紫染色15 min,洗去染液,晾干,拍照。使用Image J软件计数各组克隆数。
1.2.7 划痕实验取对数生长期的细胞分别接种于伤口愈合插件中,每孔3×104个细胞,37℃培养箱培养24 h,待细胞长至孔底90%以上面积时,取出小室插件,PBS清洗2次,加入无血清培养基,分别在0、12、24、36、48 h拍照,用Image J软件计算划痕面积。
1.2.8 Transwell迁移实验取对数生长期的细胞,用无血清培养基制备成浓度为每毫升1×105个细胞悬液,取出Transwell小室放入24孔板,向每个小室的上室加入200 μl重悬的细胞悬液,下室加入600 μl含10%FBS的完全培养基,37℃培养箱培养48 h,用PBS清洗小室2次,加入4%的多聚甲醛固定15 min,吸弃4%的多聚甲醛,再加入0.1%结晶紫染色15 min,洗去染液,用棉签擦去上室内的细胞,晾干后在显微镜下观察,拍照。使用Imaging J软件计数各组细胞数。
1.3 统计学方法One-way ANOVA分析OASL基因在胰腺癌肿瘤组织和癌旁正常组织中的表达差异;Log rank检验分析OASL基因表达与胰腺癌患者临床预后的相关性;t检验分析OASL基因差异表达;所有实验均独立重复三次。应用SPSS26.0软件进行统计分析,数据用x±s表示,两组计量资料之间比较采用t检验,三组及以上计量资料采用单因素方差分析。GraphPad Prism 9和Image J软件进行统计数据处理和绘图。P < 0.05为差异有统计学意义。
2 结果 2.1 OASL表达差异分析GEPIA数据库分析结果显示,与正常组织相比,OASL在肿瘤组织中高表达,见图 1A~B。并且,OASL基因在胰腺癌Ⅰ期表达量较低,而在Ⅱ~Ⅳ期中OASL基因表达显著升高,见图 1C,说明OASL的表达与胰腺癌的不同病理分期差异具有统计学意义(P=0.00262)。
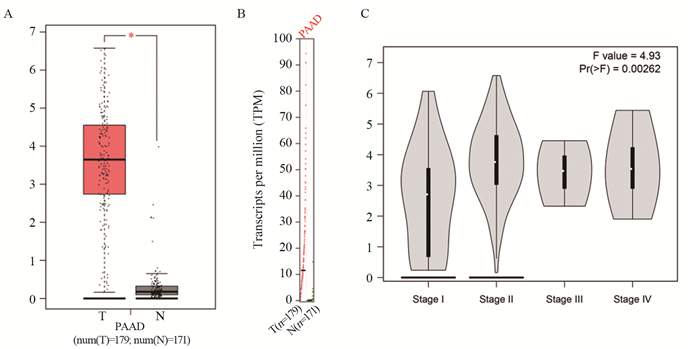
|
| *: P < 0.05. Expression levels(A) and transcript levels(B) of OASL in tumor and non-tumor tissues; C: expression levels of OASL in different stages. 图 1 OASL基因过表达后与肿瘤增殖、迁移和侵袭相关的蛋白表达 Figure 1 Expression of proteins related to tumor proliferation, migration, and invasion after the overexpression of OASL gene |
分析发现,OASL高表达的胰腺癌患者OS明显短于低表达患者(P < 0.05),而OASL表达量与患者的DFS无关(P=0.63),见图 2A~B。Kaplan-Meier plotter评估发现,OASL的表达与胰腺癌患者的OS和RFS均相关(P=0.001, P=0.03),见图 2C~D。
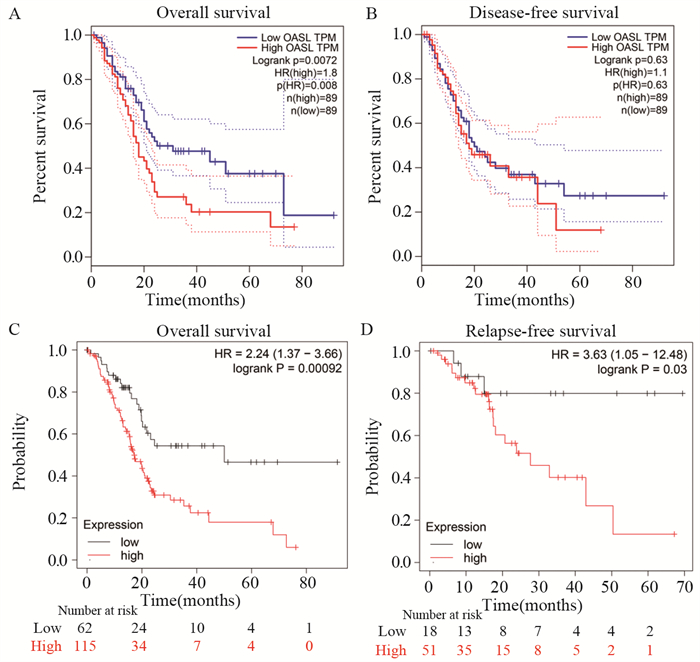
|
| 图 2 GEPIA(A, B)和Kaplan-Meier plotter(C, D)中胰腺癌患者OASL表达量与总生存期、无病生存期和无复发生存期的关系 Figure 2 Relationship between OASL expression and OS, DFS and RFS of patients with pancreatic cancer in GEPIA(A, B) and Kaplan-Meier plotter(C, D) database |
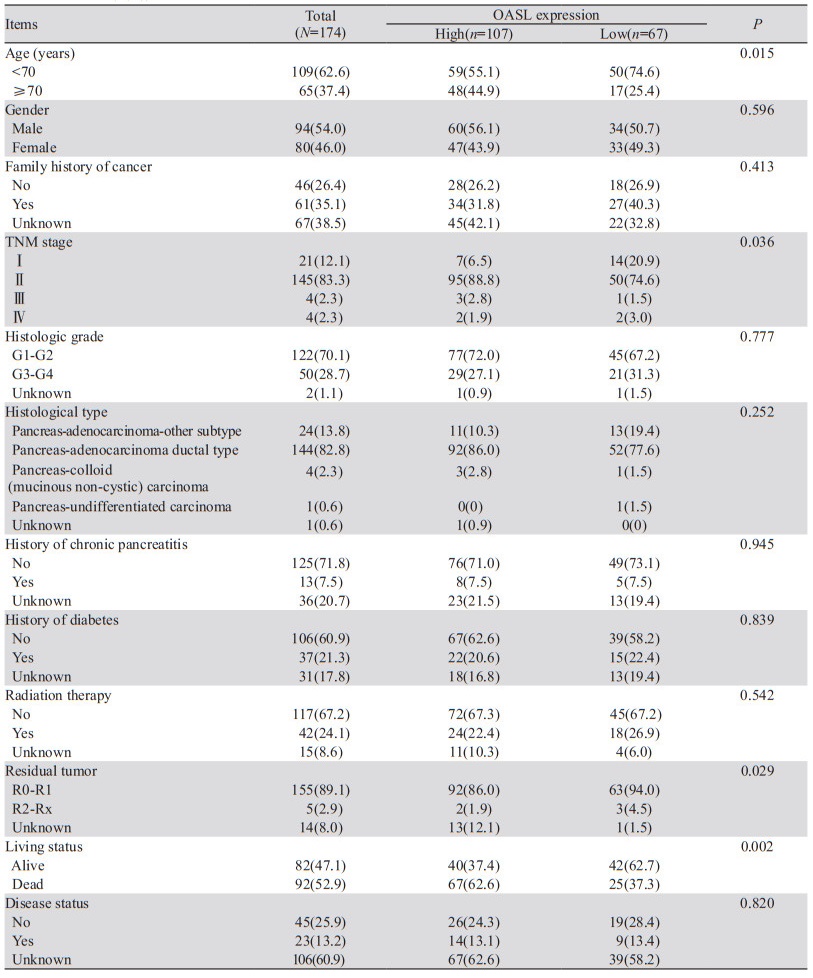
根据OS的最佳阈值将TCGA数据库中胰腺癌患者(n=174)分为高OASL表达组和低OASL表达组。结果显示,两组OASL的表达与患者年龄、TNM分期、残留肿瘤、生活状况等有关(P < 0.05),见表 1。
|
将用shRNA转染后的细胞用嘌呤霉素持续筛选,经过扩增后,在荧光显微镜下观察转染效率,结果发现,质粒转染效率均高于80%;通过慢病毒载体构建稳定高表达OASL的panc-1细胞,各组细胞的转染效率高于80%,见图 3A。
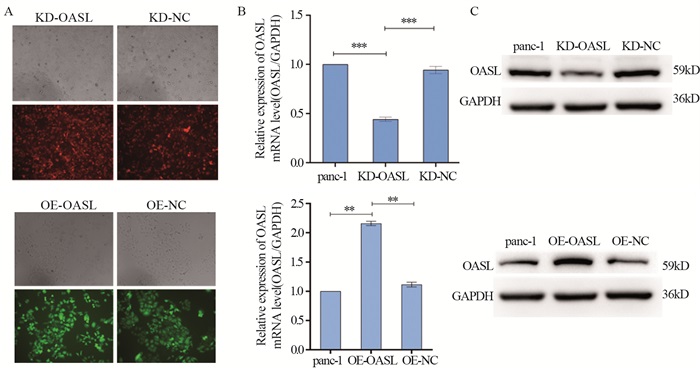
|
| **: P < 0.01, ***: P < 0.001. 图 3 细胞荧光(A)、RT-qPCR(B)和Western blot实验(C)检测OASL敲减和过表达效率 Figure 3 Knockdown and overexpression efficiency determined by cell-fluorescence(A), RT-qPCR(B), and Western blot analysis(C) |
与胰腺癌panc-1细胞和KD-NC细胞相比,KD-OASL细胞OASL基因在转录水平的表达显著降低(KD-OASL组vs. panc-1组,P=0.001; KD-OASL组vs. KD-NC组,P=0.001);与胰腺癌panc-1细胞和OE-NC细胞相比,OE-OASL细胞的OASL在分子水平的表达显著增加(OE-OASL组vs. panc-1组,P=0.006; OE-OASL组vs. OE-NC组,P=0.006),见图 3B。
2.6 Western blot检测转染细胞中OASL基因在蛋白水平的表达实验证明,在蛋白水平上KD-OASL细胞OASL的表达显著降低(KD-OASL组vs. panc-1组,P=0.009; KD-OASL组vs. KD-NC组,P=0.008),OE-OASL细胞的OASL表达明显增高(OE-OASL组vs. panc-1组,P=0.005; OE-OASL组vs. OE-NC组,P=0.006),见图 3C。
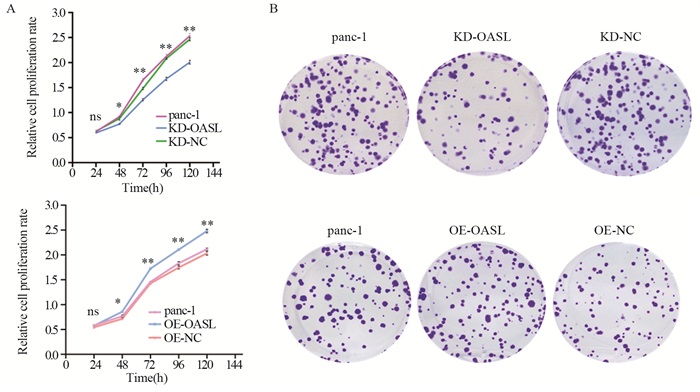
2.7 MTT实验检测OASL基因对细胞增殖的影响结果显示:KD-OASL细胞的增殖能力显著低于panc-1和KD-NC细胞,48 h时开始差异有统计学意义(KD-OASL组vs. panc-1组,P=0.031; KD-OASL组vs. KD-NC组,P=0.033),而OE-OASL细胞的增殖能力与panc-1细胞和OE-NC细胞相比明显增高(48 h时, OE-OASL组vs. panc-1组,P=0.045; OE-OASL组vs. OE-NC组,P=0.044),见图 4A。
|
| *: P < 0.05, **: P < 0.01, compared with panc-1 group or NC groups. 图 4 MTT法(A)和平板克隆法(B)检测KD-OASL和OE-OASL的增殖能力 Figure 4 Proliferation ability of KD-OASL and OE-OASL detected by MTT(A) and plate-cloning(B) assay |
与panc-1和KD-NC相比,KD-OASL的集落形成能力显著降低(KD-OASL组vs. panc-1组,P=0.006; KD-OASL组vs. KD-NC组,P=0.006),OE-OASL细胞集落形成能力显著提高(OE-OASL组vs. panc-1组,P=0.008; OE-OASL组vs. OE-NC组,P=0.007),见图 4B,结果与MTT实验一致。
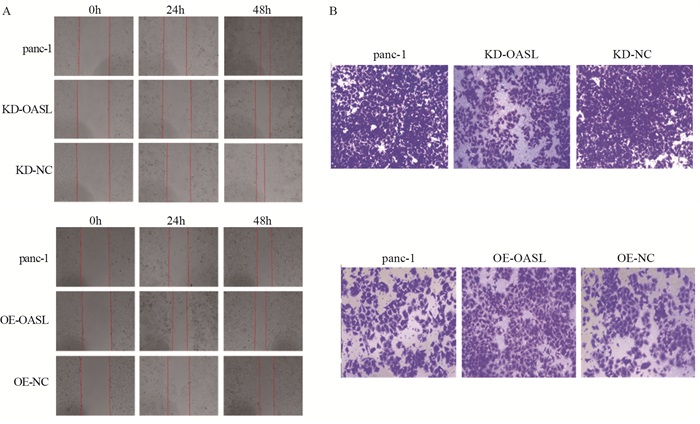
2.9 划痕实验检测OASL基因对胰腺癌细胞迁移能力的影响与panc-1细胞和KD-NC细胞相比,KD-OASL细胞的迁移能力被显著抑制(48 h时, KD-OASL组vs. panc-1组,P=0.004; KD-OASL组vs. KD-NC组,P=0.003),而OE-OASL细胞的迁移能力显著增强(48 h时, OE-OASL组vs. panc-1组,P=0.004; OE-OASL组vs. OE-NC组,P=0.003),见图 5A,说明OASL基因与panc-1细胞的迁移能力呈显著正相关性。
|
| 图 5 划痕(A)和Transwell迁移实验(B)检测KD-OASL和OE-OASL的迁移能力 Figure 5 Migration ability of KD-OASL and OE-OASL detected by scratch assay(A) and Transwell experiments(B) |
结果发现,KD-OASL细胞组穿过的细胞数明显少于panc-1细胞组和KD-NC细胞组(KD-OASL组vs. panc-1组,P=0.009; KD-OASL组vs. KD-NC组,P=0.010),而OE-OASL细胞组穿过的细胞数显著增加(OE-OASL组vs. panc-1组,P=0.006; OE-OASL组vs. OE-NC组,P=0.005),见图 5B。这说明OASL基因能影响胰腺癌panc-1细胞的迁移能力。
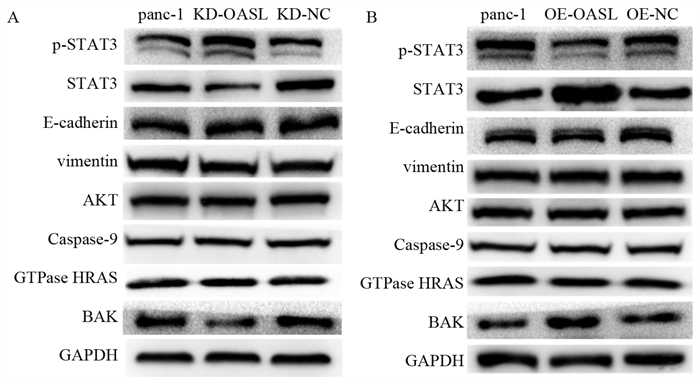
2.11 Western blot检测增殖、迁移相关蛋白的变化情况结果显示,在KD-OASL组中p-STAT3的表达显著提高(均P=0.003),而STAT3(KD-OASL组vs. panc-1组,P=0.019; KD-OASL组vs. KD-NC组,P=0.013)、BAK(均P=0.003)的表达水平显著降低,见图 6A,OE-OASL细胞组中却显示相反的结果,p-STAT3的表达被抑制(均P=0.004),STAT3(均P=0.019)和BAK(OE-OASL组vs. panc-1组,P=0.001; OE-OASL组vs. OE-NC组,P=0.002)表达水平明显增高,见图 6B。
|
| 图 6 OASL基因敲减(A)和过表达(B)后与肿瘤增殖、迁移和侵袭相关的蛋白表达 Figure 6 Expression of proteins related to tumor proliferation, migration, and invasion after knockdown(A) and overexpression(B) of OASL gene |
胰腺癌是消化道最常见的恶性肿瘤之一,诊断和治疗难度较大。其中约90%是起源于导管上皮的导管腺癌[4]。近年,虽然关于胰腺癌的治疗得到了广泛的发展,包括手术治疗、放射治疗、化疗以及基因治疗等。但因为缺乏特定的症状和早期标志物,85%的患者在确诊时已是局部晚期和(或)发生转移。患者术后5年生存率仅15%~20%[5-8]。常规化疗药物存在非特异性分布的局限性,导致生物利用度差、血液清除速度快、在体液中的溶解度相对较差[9-10]。因此,寻找具有胰腺癌高效性、特异性的生物标志物及治疗方法非常重要。
Zhang等[11]的研究指出OAS1在胰腺癌中高表达,而OAS1的高表达与患者预后不良有关。有报道指出,OASL基因可能是维持肺癌细胞对acRoots敏感度的决定性调节因子之一,并可能与耐药性的发展有关[4]。然而OASL基因表达与胰腺癌发生发展的关系鲜有报道。
KRAS作为胰腺癌突变最高的基因,涉及包括STAT3在内的多条信号通路影响胰腺癌的发生发展[12-16]。STAT3是信号转导及转录激活因子(signal transducer and activator of transcription, STAT)家族的一员,它以非活化的状态存在于细胞质中,该蛋白具有与酪氨酸磷酸化信号通路偶联的功能。该家族能与DNA结合,由STAT1、STAT2、STAT3、STAT4、STAT5a、STAT5b以及STAT6组成[17]。p-STAT3是活化的STAT3,与靶基因启动子上特异的DNA反应元件结合,影响肿瘤细胞增殖、凋亡及浸润,从而影响肿瘤的发展[18-19]。研究指出,STAT3与恶性肿瘤的发生发展有关,在肿瘤组织中STAT3被异常持续激活,从而促进肿瘤细胞的增殖、迁移和侵袭,抑制肿瘤细胞凋亡[20]。已有大量研究证实STAT3与胰腺癌、肺癌、肝癌、乳腺癌、卵巢癌等肿瘤的生长、转移、血管生成等过程有关[21]。BAK是位于STAT3下游的BCL-2家族的一员,具有促进细胞凋亡的作用,并且是目前发现的唯一一个定位在线粒体上的促凋亡蛋白成员[22]。BAK广泛分布于多种肿瘤中。有研究指出,BAK可能与多种肿瘤的发生、发展有关[23-25]。因此本研究希望通过探索OASL与STAT3信号通路的联系,尝试找到OASL基因参与胰腺癌细胞增殖和迁移改变的一些新的机制。
本研究利用质粒和慢病毒构建了OASL稳定低表达和过表达的胰腺癌panc-1细胞株,然后检测KD-OASL细胞株和OE-OASL细胞株的细胞增殖、克隆形成和迁移能力的变化,以及这些生物学行为变化相关的蛋白表达情况。结果显示,OASL敲减后细胞增殖减慢,克隆形成能力减弱,迁移能力降低,敲低株中STAT3、BAK的表达被抑制,而p-STAT3的表达水平显著提高;过表达OASL的效果相反,提示OASL可能通过STAT3信号通路影响细胞的生物学行为。本研究为探索OASL调控胰腺癌细胞生物学行为改变所涉及通路及具体作用机制提供了理论依据和数据支持,也为制定相应的治疗策略、改善患者预后奠定了基础。
作者贡献:
张人丹:实验操作、结果整理及文章撰写
赵春艳:课题思路指导
姚佳欣、胡先华:实验材料准备
母波:论文指导
| [1] |
Siegel RL, Miller KD, Fuchs HE, et al. Cancer statistics, 2022[J]. CA Cancer J Clin, 2022, 72(1): 7-33. DOI:10.3322/caac.21708 |
| [2] |
Mizrahi JD, Surana R, Valle JW, et al. Pancreatic cancer[J]. Lancet, 2020, 395(10242): 2008-2020. DOI:10.1016/S0140-6736(20)30974-0 |
| [3] |
Lv J, Wang L, Shen H, et al. Regulatory roles of OASL in lung cancer cell sensitivity to Actinidia chinensis Planch root extract (acRoots)[J]. Cell Biol Toxicol, 2018, 34(3): 207-218. DOI:10.1007/s10565-018-9422-4 |
| [4] |
Gu X, Zhou W, Han J. Factors Affecting the Readmission of Patients with Pancreatic Cancer after Surgery[J]. Appl Bionics Biomech, 2022, 2022: 6106914. |
| [5] |
Wu C, Li M, Meng H, et al. Analysis of status and countermeasures of cancer incidence and mortality in China[J]. Sci China Life Sci, 2019, 62(5): 640-647. DOI:10.1007/s11427-018-9461-5 |
| [6] |
Alessandro P, Patrick H, William H, et al. Characteristics of 10-Year Survivors of Pancreatic Ductal Adenocarcinoma[J]. JAMA Surg, 2015, 150(8): 701-710. DOI:10.1001/jamasurg.2015.0668 |
| [7] |
Kamisawa T, Wood LD, Itoi T, et al. Pancreatic cancer[J]. Lancet, 2016, 388(10039): 73-85. DOI:10.1016/S0140-6736(16)00141-0 |
| [8] |
Brown ZJ, Cloyd JM. Trends in the utilization of neoadjuvant therapy for pancreatic ductal adenocarcinoma[J]. J Surg Oncol, 2021, 123(6): 1432-1440. DOI:10.1002/jso.26384 |
| [9] |
Shukla A, Maiti P. Nanomedicine and versatile therapies for cancer treatment[J]. MedComm (2020), 2022, 3(3): e163. |
| [10] |
He Q, Yan R, Hou WT, et al. A pH-Responsive Zwitterionic Polyurethane Prodrug as Drug Delivery System for Enhanced Cancer Therapy[J]. Molecules, 2021, 26(17): 5274. DOI:10.3390/molecules26175274 |
| [11] |
Zhang C, Zou Y, Zhu Y, et al. Three Immune-Related Prognostic mRNAs as Therapeutic Targets for Pancreatic Cancer[J]. Front Med(Lausanne), 2021, 8: 649326. |
| [12] |
Sturm N, Ettrich TJ, Perkhofer L. The Impact of Biomarkers in Pancreatic Ductal Adenocarcinoma on Diagnosis, Surveillance and Therapy[J]. Cancers (Basel), 2022, 14(1): 217. DOI:10.3390/cancers14010217 |
| [13] |
Zheng B, Qu J, Ohuchida K, et al. LAMA4 upregulation is associated with high liver metastasis potential and poor survival outcome of Pancreatic Cancer[J]. Theranostics, 2020, 10(22): 10274-10289. DOI:10.7150/thno.47001 |
| [14] |
Lundy J, Harris M, Zalcberg J, et al. EUS-FNA Biopsies to Guide Precision Medicine in Pancreatic Cancer: Results of a Pilot Study to Identify KRAS Wild-Type Tumours for Targeted Therapy[J]. Front Oncol, 2021, 11: 770022. DOI:10.3389/fonc.2021.770022 |
| [15] |
Khan MA, Azim S, Zubair H, et al. Molecular Drivers of Pancreatic Cancer Pathogenesis: Looking Inward to Move Forward[J]. Int J Mol Sci, 2017, 18(4): 779. DOI:10.3390/ijms18040779 |
| [16] |
Ogawa S, Fukuda A, Matsumoto Y, et al. SETDB1 Inhibits p53-Mediated Apoptosis and Is Required for Formation of Pancreatic Ductal Adenocarcinomas in Mice[J]. Gastroenterology, 2020, 159(2): 682-696. DOI:10.1053/j.gastro.2020.04.047 |
| [17] |
Wang X, Liao X, Yu T, et al. Analysis of clinical significance and prospective molecular mechanism of main elements of the JAK/STAT pathway in hepatocellular carcinoma[J]. Int J Oncol, 2019, 55(4): 805-822. |
| [18] |
Shiah JV, Grandis JR, Johnson DE. Targeting STAT3 with Proteolysis Targeting Chimeras and Next-Generation Antisense Oligonucleotides[J]. Mol Cancer Ther, 2021, 20(2): 219-228. DOI:10.1158/1535-7163.MCT-20-0599 |
| [19] |
Du YE, Tu G, Yang G, et al. MiR-205/YAPI in Activated Fibroblasts of Breast Tumor Promotes VEGF-independent Angiogenesis through STAT3 Signaling[J]. Theranostics, 2017, 7(16): 3972-3988. DOI:10.7150/thno.18990 |
| [20] |
Laudisi F, Cherubini E, Monteleone G, et al. STAT3 Interactors as Potential Therapeutic Targets for Cancer Treatment[J]. Int J Mol Sci, 2018, 19(6): 1787. DOI:10.3390/ijms19061787 |
| [21] |
Zundler S, Neurath MF. Integrating Immunologic Signaling Networks: The JAK/STAT Pathway in Colitis and Colitis-Associated Cancer[J]. Vaccines (Basel), 2016, 4(1): 5. DOI:10.3390/vaccines4010005 |
| [22] |
Walensky LD. Targeting BAX to drug death directly[J]. Nat Chem Biol, 2019, 15(7): 657-665. DOI:10.1038/s41589-019-0306-6 |
| [23] |
Zhang G, Ma F, Li L, et al. Palbociclib triggers apoptosis in bladder cancer cells by Cdk2-induced Rad9-mediated reorganization of the Bak.Bcl-xl complex[J]. Biochem Pharmacol, 2019, 163: 133-141. DOI:10.1016/j.bcp.2019.02.017 |
| [24] |
Xin Z, Wu X, Ji T, et al. Bakuchiol: A newly discovered warrior against organ damage[J]. Pharmacol Res, 2019, 141: 208-213. DOI:10.1016/j.phrs.2019.01.001 |
| [25] |
Kuriyama S, Tsuji T, Sakuma T, et al. PLEKHN1 promotes apoptosis by enhancing Bax-Bak hetro-oligomerization through interaction with Bid in human colon cancer[J]. Cell Death Discov, 2018, 4: 11. |
 2023, Vol. 50
2023, Vol. 50


