文章信息
- 基于分子动力学模拟和实验评价探讨黄芪甲苷对HepG2细胞的作用机制
- Mechanism of Astragaloside Ⅳ on HepG2 Cells Based on Molecular Dynamics Simulation and Experimental Evaluation
- 肿瘤防治研究, 2022, 49(7): 655-661
- Cancer Research on Prevention and Treatment, 2022, 49(7): 655-661
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2022.21.1537
- 收稿日期: 2021-12-31
- 修回日期: 2022-04-18
2. 430000 武汉,武汉大学人民医院儿科;
3. 730000 兰州,兰州大学口腔医学院正畸科
2. Department of Pediatrics, Wuhan University People's Hospital, Wuhan 430000, China;
3. Department of Orthodontics, School of Stomatology, Lanzhou University, Lanzhou 730000, China
肝癌在人类癌症死因排名中占据第三[1],中医学将“肝癌”归属于“肝积”、“积聚”、“癥瘕”等范畴。《素问·刺法论》曰:“正气存内,邪不可干”。黄芪甲苷(astragaloside Ⅳ, AS-Ⅳ)为黄芪提取物中的一种,具有抑制氧化应激、减少炎性反应、促进内皮细胞成管、促进梗死区域血管再生、促进干细胞向血管内皮细胞分化的作用[2],同时也具有补益正气的效果。另有研究[3]表明,黄芪甲苷能诱导人结肠癌SW480细胞凋亡,但其抗癌机制尚有待深入研究。本研究以VEGF信号通路为切入点,探讨黄芪甲苷对HepG2细胞的作用及其可能机制。
1 材料与方法 1.1 材料与试剂人肝癌细胞株HepG2细胞(由兰州大学基础医学院提供)、黄芪甲苷(成都普瑞法科技开发有限公司,型号:84687-43-414)、胎牛血清(型号:C2800 0500)、100 u/ml青霉素G/100 μg/ml链霉素的DMEM完全培养基(型号:SH30023,美国赛默飞公司)、CCK-8(中国爱普拜公司,型号K1018)、TRIzol Servicebio®RT First Strand cDNA Synthesis Kit(武汉赛维尔公司,G3330)、2×SYBR Green qPCR Master Mix(武汉赛维尔公司, G3320)。
1.2 方法 1.2.1 黄芪甲苷靶点获取PubChem(https://pubchem.ncbi.nlm.nih.gov)[4]数据库检索Astragaloside Ⅳ,获取其2D结构,并在SwissTargetPrediction(http://www.swisstargetprediction.ch/)[5]、Genecards[6]及PharmMapper(http://www.lilab-ecust.cn/pharmmapper/)[7]数据库中检索Astragaloside Ⅳ,以Homo Sapiens为类别,去重后得到373个黄芪甲苷靶点。
1.2.2 肝细胞癌靶点基因及样本获取从TCGA[8]中下载肝细胞癌(hepatocellular carcinoma, HCC)相关的mRNA和表达量数据、转录组分析、基因表达量和RNA表达量数据(2020.08.02)。
1.2.3 黄芪甲苷与肝细胞癌“药物-疾病”共靶点在TCGA数据库中获取HCC患者的RNA-seq数据,以|log2FC| > 1,FDR < 0.05为分界标准,分析HCC组织与正常组织间的差异表达基因,构建差异基因热图;运用Venn Diagram,构建AS-Ⅳ作用靶点及HCC差异基因对应靶点交集,即为AS-Ⅳ作用于HCC的“药物-疾病”共靶点。
1.2.4 黄芪甲苷作用于肝细胞癌的蛋白互作网络分析在STRING(https://string-db.org/)[9]中检索“药物-疾病”共靶点,以Homo sapiens为种属,调整high confidence为0.4和0.7,得出蛋白互作网络图(protein-protein interaction network, PPI),分析出相关系数最大的基因,即为AS-Ⅳ作用于HCC的核心基因。
1.2.5 构建黄芪甲苷-肝细胞癌网络药理图将PPI蛋白相互作用的结果、节点和基因列表导入Cytoscape(Cytoscape_v3.7.0)[10],调整结合度,构建AS-Ⅳ作用于HCC的网络药理图。
1.2.6 模拟黄芪甲苷与核心基因的分子动力学作用选用PyRx0.8虚拟筛选工具作为对接程序,AutoDock Vina(Auto DockTools-1.5.6)作为对接引擎[7],从PubChem数据库下载黄芪甲苷结构,使用ChemBio3D(ΜLtra 14.0)软件构建分子,并通过MM2分子力学优化化合物构型(依次选择CalcuLations/MM2/Minimize Energy);然后从RCSB Protein Data Bank(https://www.rcsb.org)下载VEGFA蛋白的三维结构,并在蛋白与化合物中添加所有的氢原子、计算Gasteiger电荷、合并非极性氢原子;最后确定Vina分子对接的坐标和盒子大小,将参数exhaustiveness设置为20。AutoDock Vina进行半柔性对接,选取affinity最佳的构象,并使用PyMOL及Discovery studio进行作图。amber20软件包对分子对接获取的蛋白-小分子复合物进行分子动力学模拟。蛋白使用ff14SB力场参数,AS-Ⅳ小分子配体则使用gaff通用力场参数,并使用ANTECHAMBER模块计算其AM1-BCC原子电荷。分子动力学模拟工作流程共包括能量最小化、加热、平衡、生产动力学模拟等4步。首先,约束蛋白(和小分子)重原子,对水分子进行10 000步(含5000步最速下降法和5 000步共轭梯度法)能量最小化;随后,在50 ps时间内,将反应体系缓慢加热至300 K;加热完成后,在NPT系综下对体系进行50 ps的平衡。最后,将体系在NPT系综下进行50 ns的分子动力学模拟。每隔20 ps保存一次轨迹数据,并用CPPTRAJ模块进行相关分析。配体和蛋白的结合自由能计算则采用MMPBSA.py模块进行。结合自由能计算公式:ΔGbind=Gcomplex–Gprotein–Gligand,对蛋白与小分子配体的自由结合能进行计算。RMSD曲线代表了蛋白构象的波动情况。其中N是原子数,mi是原子i的质量,Xi是目标原子i的坐标矢量,Yi是参考原子i的坐标矢量,M是总质量。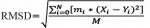
结合R语言,以P < 0.05为标准,以q < 0.05对P值过滤,对“药物-疾病”共靶点进行GO、KEGG富集分析,并将富集结果可视化。
1.2.8 黄芪甲苷含药培养基的制备将0.3 g黄芪甲苷溶解于1 ml DMSO中,用完全培养基配成30 μg/μl原液,经0.22 μm微孔滤膜滤过,置-20℃冰箱保存备用,实验时用完全培养基稀释至所需质量浓度的应用液(DMSO所占体积比小于1‰)。
1.2.9 细胞培养及分组人肝癌HepG2细胞保存在添加有胎牛血清和双抗的DMEM培养基中,置于37℃和5%CO2的湿化培养箱中培养;HepG2细胞分为对照组(DMEM组)与低(45 μg/ml)、中(90 μg/ml)、高(180 μg/ml)浓度AS-Ⅳ组(DMEM+AS-Ⅳ组)。
1.2.10 细胞存活率测定CCK-8法评估AS-Ⅳ对HepG2细胞生长影响,并确定AS-Ⅳ对HepG2细胞存活率影响的量效及时效值。
1.2.11 克隆形成实验取对数生长期的各组细胞,制备细胞悬液。将细胞悬液作梯度倍数稀释,分别接种细胞(500个)于含培养液的培养皿中,当培养14天时,培养皿中出现肉眼可见的克隆,终止培养。弃去上清液,4%多聚甲醛固定,结晶紫染色,采集数据。
1.2.12 细胞迁移实验细胞生长于含10%FBS DMEM培养基中,以每孔1×106个接种到6孔培养板中,生长24 h后,当单层细胞融合度达70%~80%,用新的200 μl枪头在单层培养细胞间划痕,弃培养液,PBS清洗,对照组加4 ml DMEM全培养基,实验组加等量体积的低、中、高浓度AS-Ⅳ,继续培养48 h,去培养液,采集数据。
1.2.13 细胞侵袭实验将HepG2细胞接种于上室,对照组加入无血清DMEM培养基,实验组分别加入含低、中、高浓度AS-Ⅳ的无血清DMEM培养基,下室均加入等量DMEM完全培养基,继续培养细胞48 h后,通过固定染色Transwell小室底部,并计算迁移至下室的细胞数。
1.2.14 流式细胞术对照组加DMEM完全培养基,实验组用低、中、高浓度AS-Ⅳ处理48 h后,收获HepG2细胞,用70%冰冷乙醇4℃固定24 h,PBS洗涤,再悬浮在1 ml PI染色液(40 μg/ml RNaseA和10 μg/ml PBS)中,室温黑暗中孵育30 min,测细胞周期占比;然后用FITC和PI染色液对用不同浓度AS-Ⅳ处理后的HepG2细胞进行双染,室温黑暗中孵育30 min,测细胞凋亡占比。
1.2.15 qRT-PCR根据产品说明书,TRIzol提取细胞总RNA,Servicebio®RT First Strand cDNA Synthesis Kit合成cDNA,2×SYBR Green qPCR Master Mix用于进行VEGFA、TGF-β1和GAPDH基因的定量检测。引物序列为:GAPDH:正义:GGAAGCTTGTCAT CAATGGAAATC,反义:TGATGACCCTTTTGGCTCCC;VEGFA:正义:GGAGGGCAGAATCATCACGA,反义:GATCATCTCCCTATGTGCTGG;TGF-β1:正义:CAGCAACAATTCCTGGCGATA,反义:GCTAAGGCGAAAG CCCTCAAT。结果处理ΔΔCT法:A=CTAS-Ⅳ干预后目的基因-CTAS-Ⅳ干预后内参基因,B=CT无AS-Ⅳ干预目的基因-CT无AS-Ⅳ干预内参基因,K=A-B,相对表达量=2-K。
1.3 统计学方法数据以(x±s)表示,多组和成对的比较采用GraphPad软件(9.0版)的单因素方差分析(ANOVA),两两比较方差齐时采用最小显著差异法t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 黄芪甲苷2D和3D结构模型建立将黄芪甲苷在PubChem BioAssay数据库(http://pubchem.ncbi.nlm.nih.gov/bioassay)对话框中进行检索获取其2D结构,将其2D结构式在软件ChemBio3D ΜLtra 14.0上进行导入,导出其3D结构式。
2.2 构建黄芪甲苷与肝细胞癌的“药物-疾病”共靶点分析AS-Ⅳ的靶点与肝细胞癌-正常组织中差异表达基因对应靶点的交集,并行差异分析,得出27个差异交集靶点:CSTN、HSP90AA1、VEGFA、CDK1、LGALS3、ADRA2B、ADRA1A、TYMS、SF3B3、SRC、F11、F7、ATP1A1、MMP1、ADAM17、SELP、GBA、EGLN3、TGFB1、PPARG、BAX、RAC1、CTNNB1、SMAD2、GSK3B、SIRT6、PPAT,即为AS-Ⅳ作用于HCC的“药物-疾病”共靶点。
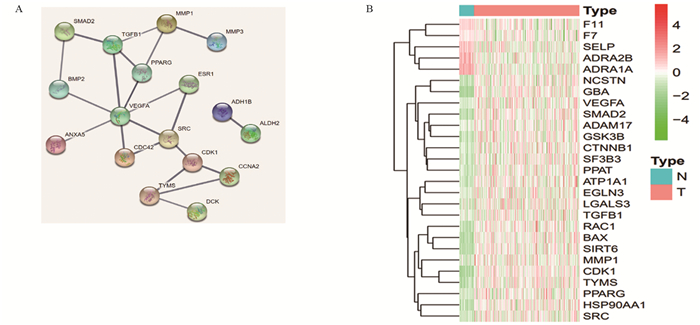
2.3 “药物-疾病”共靶点互作网络分析通过在STRING上构建PPI蛋白互作网络图, 筛选与“药物-疾病”共靶点中关联最密切且节点最多的靶点,标记为核心靶点,见图 1A,PPI蛋白网络互作图提示:AS-Ⅳ作用于HCC核心靶点为VEGFA。
|
| A: VEGFA was the core target of AS-Ⅳ in hepatocellular carcinoma; B: heat map of 27 differential genes. Red: up-regulation; Green: down-regulation. N: normal liver 图 1 药物-疾病共靶点蛋白互作网络图、差异基因热图 Figure 1 Drug-disease co-targeting PPI and heat map of differential genes |
根据“药物-疾病”共靶点在每个样本中表达情况,以FDR=0.05、log2FC=0为基准,构建27个差异基因热图,见图 1B,结果显示:核心基因VEGFA为下调基因。
2.5 “药物-疾病”共靶点互作网络药理图明确“药物-疾病”共靶点,并根据共靶点蛋白PPI互作网络及节点的多少,用degree进行区分节点,并用label注释,构建AS-Ⅳ作用于HCC的网络药理图,结果显示:靶点VEGFA的degree值最大,且节点最多。
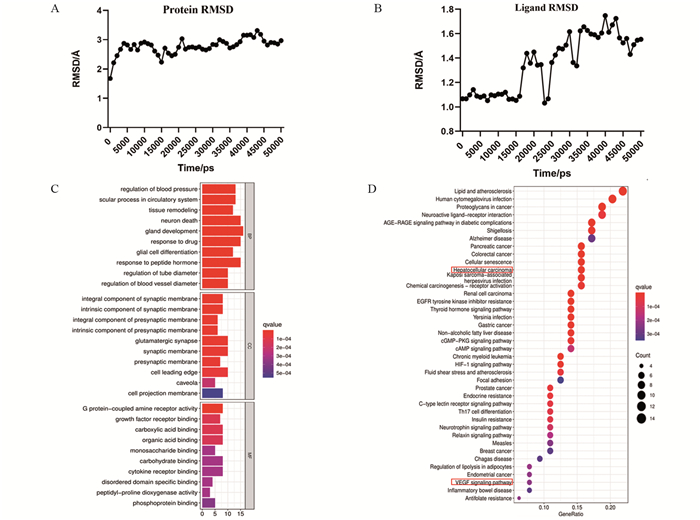
2.6 黄芪甲苷与核心蛋白的分子动力学模拟分析为了从分子水平阐明VEGFA蛋白与化合物AS-Ⅳ的作用模式,将化合物对接至蛋白,其对接打分为-6.8 kcal/mol,且与之结合的氨基酸有LYS107、LEU66、ASP63、LEU32、VAL33,另分子动力学模拟中,以分子对接构象为基准,比较分子对接分子自由结合能与分子动力学自由结合能示意图,结果显示:Pro70(Ⅴ)、Cyx68(Ⅴ)为其共同结合氨基酸。在模拟的初始阶段(0-5000 ps)构象存在一定变化,VEGFA蛋白的RMSD波动稍大(图 2A)。但5 000 ps以后,RMSD曲线趋于平稳,蛋白构象逐渐稳定,而AS-Ⅳ小分子的RMSD波动情况则与之相反(图 2B),说明AS-Ⅳ小分子配体与VEGFA蛋白结合后,动力学模拟过程中,AS-Ⅳ小分子RMSD存在波动,但分子对接与分子动力学模拟过程中具有共同自由结合能Pro70(Ⅴ)和Cyx68(Ⅴ),故分子动力学模拟进一步验证了分子对接的可行性。
|
| A: RMSD values of VEGFA protein; B: RMSD values of AS-Ⅳ ligands; C: functional analysis of differential genes; D: signaling pathway enrichment analysis. 图 2 蛋白VEGFA与AS-Ⅳ的分子动力学模拟RMSD值、差异基因GO、KEGG富集分析 Figure 2 Molecular dynamics simulation of protein VEGFA with AS-Ⅳ RMSD values, differential gene GO, KEGG enrichment analysis |
差异基因GO富集分析发现,前30种功能富集涉及血压调节、血管重构、血管直径的调节等,见图 2C;KEGG富集分析发现与肝细胞癌相关及与核心基因相关的信号通路,即Hepatocellular carcinoma(hsa05225)信号通路和VEGF(ko04370)信号通路,而与之相关的核心基因为TGF-β1和VEGFA,见图 2D。
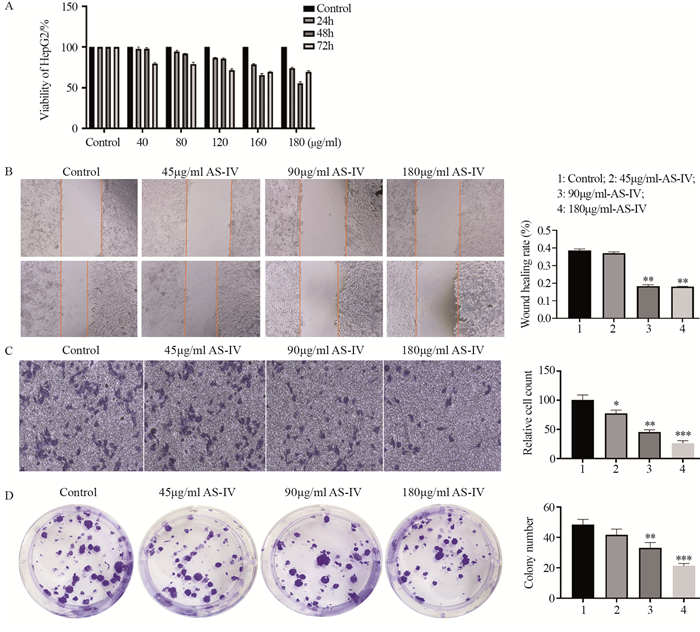
2.8 细胞增殖测定CCK-8法检测HepG2细胞的增殖能力,与对照组相比,当AS-Ⅳ浓度为180 μg/ml时,干预48 h,HepG2细胞增殖能力最弱;当AS-Ⅳ的浓度为180 μg/ml时,干预时间分别为24、48、72 h时,HepG2细胞存活率分别为84%、57.5%、72%(t=33.98, P=0.0000; t=39.28, P=0.0000; t=20.15, P=0.0000),见图 3A。
|
| A: the proliferation of HepG2 cells showed that the survival rate was the lowest when the concentration of AS-Ⅳ was maintained at 180 μg/ml and the duration of intervention was 48 h; B: the results of migration assay showed that high concentration of AS-Ⅳ had the strongest inhibitory effect on the migration ability of HepG2 cells compared with the control group; C: invasion assay experiments showed that high concentrations of AS-Ⅳ were most invasive when acting on HepG2 cells compared to the control group; D: the results of cloning assay showed that the highest proliferation inhibition rate of HepG2 cells was achieved by high concentration of AS-Ⅳ compared with the control group. AS-Ⅳ: Astragaloside Ⅳ; Compared with control group, *: P < 0.05, **: P < 0.01, ***: P < 0.001, compared with control group. 图 3 细胞增殖、迁移、侵袭、克隆检测 Figure 3 Cell proliferation, migration, invasion and cloning detection |
与对照组相比,低、中、高浓度AS-Ⅳ干预48 h时,迁移实验中,AS-Ⅳ处理组HepG2细胞的划痕愈合率分别为37%、19%、18%,提示其对肿瘤的迁移有抑制作用(t=2.793, P=0.0234; t=135.12, P=0.0000; t=48.27, P=0.0000),见图 3B;侵袭实验中,AS-Ⅳ处理组HepG2细胞侵袭相对抑制率分别为1.7%、27.1%、55.7%,(t=4.892, P=0.0012; t=12.80, P=0.0000; t=16.84, P=0.0000),见图 3C;克隆形成实验中,AS-Ⅳ处理组HepG2细胞的克隆形成率分别为4.8%、4.2%、2.1%(t=3.078, P=0.0217; t=7.416, P=0.0003; t=17.22, P=0.0000),见图 3D。
2.10 细胞凋亡与周期检测与对照组相比,低、中、高浓度AS-Ⅳ干预48 h时,HepG2细胞凋亡率分别为2.43%、15.58%、21.46%(t=0.9571, P=0.3927; t=38.44, P=0.0000; t=50.60, P=0.0000),见图 4A、C。细胞周期分布情况见图 4B、D,其中G1期占比分别为38.22%、44.9%、50.94%(t=0.3163, P=0.7676; t=7.954, P=0.0014; t=14.80, P=0.0001)。
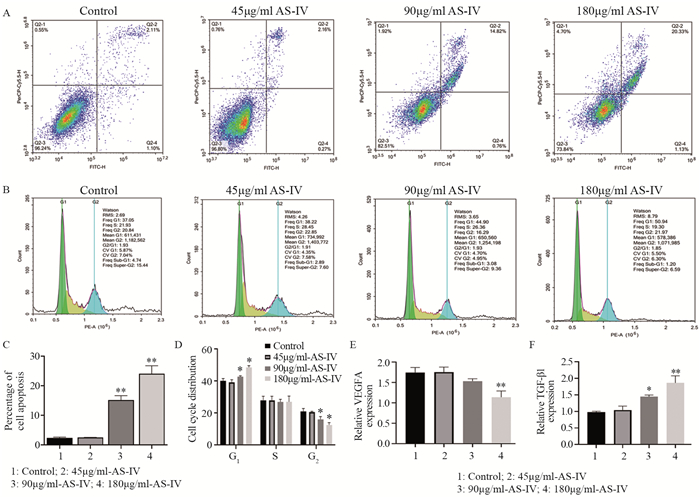
|
| Compared with control group, *: P < 0.05, **: P < 0.01, ***: P < 0.001, compared with control group. 图 4 AS-Ⅳ对HepG2凋亡、周期及核心基因表达的影响 Figure 4 Effect of AS-Ⅳ on apoptosis (A, C), cell cycle (B, D) and core gene expression (E, F) of HepG2 cells |
与对照组相比,低、中、高浓度AS-Ⅳ干预48 h时,VEGFA mRNA的相对表达量分别为1.72、1.5、1.12(t=0.09923, P=0.9257; t=2.685, P=0.0549; t=5.351, P=0.0059),见图 4E;基因TGF-β1 mRNA相对表达量为1.12、1.44、1.80(t=0.8347, P=0.4508; t=14.21, P=0.0001; t=7.154, P=0.0020),见图 4F。
3 讨论网络药理学与分子对接为阐明成分-靶点和靶点-疾病之间网络的分子机制和成分-蛋白自由结合能力提供了一个新的视角[11-12],这与中医药的“多成份”、“多靶点”和“多通路”的特点相适应;分子动力学模拟为实验前期提供了一个依据与可行性分析,同时也为阐述肝细胞癌的发病机制提供了可能;肝癌在中国的癌症发生率中占第四位,死亡率中占第三位[13],且肝细胞癌约占所有肝癌的90%[14],其病因与病毒性肝炎、黄曲霉毒素B1、酒精及遗传等因素相关,其发病机制多与P53、WNT/β-catenin、氧化应激、pI3K/AKT/MTOR通路及微生物群等相关[15],治疗以手术、化疗、放疗、生物免疫治疗等为主,但总体疗效较差。Hepatocellular carcinoma信号通路作为肝细胞癌主要信号通路之一,且与之相关的重要基因为TGF-β1,本研究通过数据挖掘及模拟,分析出AS-Ⅳ作用于肝细胞癌的核心基因为VEGFA,其过表达可以引发肿瘤细胞的增殖、转移和血管生成[16-17],VEGFA激活VEGFR2的磷酸化,在体外促进血管内皮细胞的成管、细胞迁移和侵袭[18];AS-Ⅳ作用于肝细胞癌的信号通路主要为VEGF信号通路及Hepatocellular carcinoma信号通路,而与之相关的核心基因为VEGFA与TGF-β1,所以AS-Ⅳ对肝细胞癌的靶向作用可能主要体现在对这两个基因的影响上。CCK-8实验、伤口愈合实验、克隆形成实验及细胞侵袭实验提示其有抑制HepG2细胞增殖和迁移、侵袭的能力,且能促进其凋亡,抑制其从G1期向G2期转移,也即抑制增殖所需蛋白的合成进而抑制其增殖;此外,AS-Ⅳ能抑制核心基因VEGFA表达和促进基因TGF-β1表达,其中基因VEGFA的过表达对肿瘤细胞的迁移、增殖、侵袭及血管的生存具有促进作用,同时基因TGF-β1能调节细胞增殖、生长、分化和运动,故其共同参与影响HepG2细胞的增殖。
综上,AS-Ⅳ对HepG2细胞增殖具有一定抑制作用,其机制可能与抑制HepG2细胞增殖、迁移、侵袭,并促进其凋亡有关,这个过程可能是通过促进基因TGF-β1 mRNA表达和抑制VEGF信号通路中基因VEGFA表达共同完成的。但本研究也存在一定的局限性,需进一步观察AS-Ⅳ对相关蛋白表达的影响以及体内实验进行验证。
作者贡献:
周志朋:实验设计与实施、文章撰写、基金项目申请
杨明珠:采集数据
蔡明钦:实验实施、分析、解释数据
薛娟娣:统计分析
吕晓云:文章审阅、研究经费支持、文章指导
| [1] |
Forner A, Reig M, Bruix J. Hepatocellular carcinoma[J]. Lancet, 2018, 391(10127): 1301-1314. DOI:10.1016/S0140-6736(18)30010-2 |
| [2] |
袁艳萍, 郑志娟, 李运伦. 黄芪甲苷改善心血管疾病中内皮机制的研究进展[J]. 中华中医药学刊, 2021, 39(8): 120-124. [Yuan YP, Zheng ZJ, Li YL. Research Advances on Mechanism of Astragaloside Ⅳ Improving Endothelium in Cardiovascular Diseases[J]. Zhonghua Zhong Yi Yao Xue Kan, 2021, 39(8): 120-124.] |
| [3] |
陈攀丽, 唐建荣, 张逸强. 黄芪甲苷对人结肠癌SW480细胞系增殖和凋亡的影响[J]. 肿瘤防治研究, 2019, 46(8): 702-706. [Chen PL, Tang JR, Zhang YQ. Regulatory Effect of Astragaloside A on Proliferation and Apoptosis of Human Colon Cancer SW480 Cell Line[J]. Zhong Liu Fang Zhi Yan Jiu, 2019, 46(8): 702-706. DOI:10.3971/j.issn.1000-8578.2019.18.186] |
| [4] |
Kim S, Chen J, Cheng T, et al. PubChem in 2021: new data content and improved web interfaces[J]. Nucleic Acids Res, 2021, 49(D1): D1388-D1395. DOI:10.1093/nar/gkaa971 |
| [5] |
Daina A, Michielin O, Zoete V. SwissTargetPrediction: updated data and new features for efficient prediction of protein targets of small molecules[J]. Nucl Acid Res, 2019, 47(W1): W357-W364. DOI:10.1093/nar/gkz382 |
| [6] |
Rappaport N, Fishilevich S, Nudel R, et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect[J]. Biomed Eng Online, 2017, 16(Suppl 1): 72. |
| [7] |
Tangyuenyongwatana P, Gritsanapan W. Identification of Potential Phytoestrogen of Selected Compounds from Zingiber cassumunar Roxb. Rhizomes Using PharmMapper and Molecular Docking[J]. Biointerface Res Applied Chem, 2021, 11(1): 7826-7834. |
| [8] |
Yavorski JM, Blanck G. TCGA: Increased oncoprotein coding region mutations correlate with a greater expression of apoptosis-effector genes and a positive outcome for stomach adenocarcinoma[J]. Cell Cycle, 2016, 15(16): 2157-2163. DOI:10.1080/15384101.2016.1195532 |
| [9] |
von Mering C, Jensen LJ, Snel B, et al. STRING: known and predicted protein-protein associations, integrated and transferred across organisms[J]. Nucleic Acids Res, 2005, 33(Database issue): D433-D437. |
| [10] |
Singhal A, Cao S, Churas C, et al. Multiscale community detection in Cytoscape[J]. PLoS Comput Biol, 2020, 16(10): e1008239. DOI:10.1371/journal.pcbi.1008239 |
| [11] |
Bai X, Tang Y, Li Q, et al. An Integrated Analysis of Network Pharmacology, Molecular Docking, and Experiment Validation to Explore the New Candidate Active Component and Mechanism of Cuscutae Semen-Mori Fructus Coupled-Herbs in Treating Oligoasthenozoospermia[J]. Drug Des Devel Ther, 2021, 15: 2059-2089. DOI:10.2147/DDDT.S307015 |
| [12] |
Li J, Fu A, Zhang L. An Overview of Scoring Functions Used for Protein-Ligand Interactions in Molecular Docking[J]. Interdiscip Sci, 2019, 11(2): 320-328. DOI:10.1007/s12539-019-00327-w |
| [13] |
Chen WQ, Zheng RS, Baade PD, et al. Cancer Statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [14] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [15] |
The Cancer Genome Atlas Research Network. Comprehensive and Integrative Genomic Characterization of Hepatocellular Carcinoma[J]. Cell, 2017, 169(7): 1327-1341. |
| [16] |
Lin JD, Cao SW, Wang Y, et al. Long non-coding RNA UBE2CP3 enhances HCC cell secretion of VEGFA and promotes angiogenesis by activating ERK1/2/HIF-1 alpha/VEGFA signalling in hepatocellular carcinoma[J]. J Exp Clin Cancer Res, 2018, 37(1): 113. DOI:10.1186/s13046-018-0727-1 |
| [17] |
Liu C, Xu DW, Xue B, et al. Upregulation of RUNX1 Suppresses Proliferation and Migration through Repressing VEGFA Expression in Hepatocellular Carcinoma[J]. Pathol Oncol Res, 2020, 26(2): 1301-1311. |
| [18] |
Yang WD, Li ZW, Qin R, et al. YY1 Promotes Endothelial Cell-Dependent Tumor Angiogenesis in Hepatocellular Carcinoma by Transcriptionally Activating VEGFA[J]. Front Oncol, 2019, 9: 1187. |
 2022, Vol. 49
2022, Vol. 49


