文章信息
- 干扰CHD1L基因对肝内胆管细胞癌细胞系增殖和侵袭的影响
- CHD1L Silencing Inhibits Proliferation and Invasion of Intrahepatic Cholangiocarci-noma Cell Lines
- 肿瘤防治研究, 2017, 44(8): 520-524
- Cancer Research on Prevention and Treatment, 2017, 44(8): 520-524
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2017.17.0014
- 收稿日期: 2017-01-06
- 修回日期: 2017-05-18
2. 330006 南昌,南昌大学第二附属医院,江西省分子医学重点实验室
2. Molecular Medicine Key Laboratory of Jiangxi Province, The Second Affiliated Hospital of Nanchang University, Nanchang 330006, China
染色质解旋酶DNA结合蛋白样-1样基因(chromodomain helicase/ATPase DNA binding protein1-like gene, CHD1L)是在肝癌中新发现的致癌基因,它属于Sucrose nonfermenting2(SNF-2)类家族成员,具有高度保守的解旋酶功能,在维持染色质稳定、转录调控和DNA修复中起重要作用[1-2]。近年来多篇文献报道CHD1L在恶性肿瘤中的侵袭和转移过程中扮演重要作用[3-5]。我们预实验研究提示CHD1L在肝内胆管细胞癌(ICC)组织中呈高表达。在此基础上,本研究拟先筛选在ICC细胞系中CHD1L呈高表达的细胞系,构建CHD1L-RNAi干扰目的基因的表达,采用CCK-8法、平板克隆集落形成、细胞划痕实验和Transwell迁移和侵袭等实验验证其对ICC细胞株的生物学功能的影响,并阐述其可能的分子机制,为进一步研究CHD1L在人ICC生物学特性及发展机制中提供理论和实验依据。
1 材料与方法 1.1 细胞系及试剂三种人ICC细胞系(RBE、HCCC-9810和HUCCT1)由江西省重点分子实验室保存,RPMI 1640培养液、胎牛血清(FBS)购自美国Gibco公司,TRIzol和LipofectamineTM 2000 Reagent购自美国Invitrogen公司,Transwell小室购自美国Millipore公司,Matrigel购自美国BD公司,反转录试剂盒、SYBR Green荧光定量PCR Mix、蛋白提取试剂盒、CCK-8试剂盒和HRP标记的山羊抗兔二抗IgG均购自北京全式金生物技术有限公司,兔抗人GAPDH多克隆抗体购自台湾Proteintech公司,兔抗人CHD1L单克隆抗体购自美国Abcam公司。
1.2 CHD1L-RNAi的构建和转染细胞培养于含10%FBS的RPMI 1640的完全培养液中,置于5%CO2培养箱中37℃贴壁培养,每隔2 d传代1次。hU6/GFP/Neo-CHD1L-RNAi1(TATTGGACATGCCACGAAA)、hU6/GFP/Neo-CHD1L-RNAi2(CAAGAGAAGGAGACTCATA)、hU6/GFP/Neo-CHD1L-RNAi3(ACAAACTCTTGCAGCCATT)由上海吉凯基因公司构建,并用hU6/GFP/Neo-CHD1L-shNC(TTCTCCGAACGTGTCACGT)作为阴性对照。将上述质粒转染至目的细胞后,利用Western blot法筛选出干扰效果最为理想的质粒。转染按照LipofectamineTM 2000 Reagent说明书操作。
1.3 Western blot法检测CHD1L蛋白的表达采用蛋白提取试剂盒提取细胞总蛋白,调节蛋白浓度。配制10%的分离胶合并5%的浓缩胶,取等量的蛋白上样。先80 V、后120 V电泳。根据目的胶的尺寸裁取相应的PVDF膜,250 mA转膜100 min。5%脱脂奶粉的TBST缓冲液室温封闭2 h,羊抗兔一抗(1:1 000)4℃孵育过夜,TBST洗涤3遍,每遍10 min。HRP标记的山羊抗兔二抗IgG(1:10 000)室温孵育1 h后,再次TBST洗涤3遍,每遍10 min。最后加入发光试剂,凝胶电泳成像系统进行读片分析。
1.4 qRT-PCR法检测CHD1L mRNA的表达先用TRIzol提取三种人ICC细胞系的RNA,PCR扩增仪(MJ Research Inc, 美国)反转录获得cDNA。CHD1L的上游引物:5’-GGGAAGACCTGCCAGATTTGCT-3’,下游引物:5’-ACGTGACTCCTGTTTCAGGTCTTG-3’;GAPDH的上游引物:5’-GTTGGAGGTCGGAGTCAACGGA-3’,下游引物:5’-GAGGGATCTCGCTCCTGGAGGA-3’(引物由上海生工股份有限公司合成)。qRT-PCR应用SYBR Green荧光定量PCR Mix试剂盒,在ABI 7500实时荧光定量PCR仪(Thermo Fisher Scientific,美国)上进行,具体步骤参照SYBR Green荧光定量PCR Mix说明书。
1.5 CCK-8法、平板克隆集落形成实验检测细胞增殖CCK-8法检测人ICC细胞的增殖能力:取对数生长期的CHD1L(未处理组)、CHD1L-NC(阴性对照组)和CHD1L-RNAi(实验组)制备成单细胞悬液,分别以每孔4×103个接种于96孔板中,继续培养,四周用PBS填满。分别以0、24、48、72、96 h 5个时间段检测,设置5个复孔,每孔加入10 μl CCK-8溶液,37℃培养箱培养2 h后酶标仪测定450 nm波长处各孔的吸光度,绘制生长曲线。平板克隆集落形成实验:在各组中,将含有1 000个细胞的2 ml的完全培养液加入6孔板中,在5%CO2、37℃培养箱中培养2周,PBS洗涤3次,再用4%的多聚甲醛固定20 min,0.1%结晶紫染色30 min,在显微镜下计算集落形成数量。
1.6 细胞划痕实验、Transwell迁移和侵袭实验检测细胞迁移和侵袭细胞划痕实验:取对数生长期的细胞用胰酶消化吹打并重悬为单细胞悬液后,细胞计数,将每毫升5×106个细胞接种至6孔板,置于5%CO2、37℃培养箱中培养,分成三组(同1.5方法),待细胞贴壁后,用100 μl的无菌枪头在6孔板中央划出2~3条划痕,洗去死细胞,分别于0、24 h拍照,计算细胞迁移率,细胞迁移率=(原始直径-当前直径)/原始直径×100%。Transwell迁移和侵袭实验:用RPMI 1640培养液将Matrigel稀释(1:6),取80 μl加入至Transwell上室,37℃放置过夜。采用上述分组,胰酶消化制备成每孔4×104个细胞悬液,接种至Transwell上室铺匀(其中Transwell迁移实验不用铺基质胶,余项步骤同Transwell侵袭实验),下室加入600 μl含20%FBS的RPMI 1640培养液,在5%CO2、37℃培养箱中培养24 h,取出Transwell小室,用棉签拭去聚碳酸酯膜表面的Matrigel胶,PBS冲洗,4%的多聚甲醛固定20 min,0.1%结晶紫染色30 min,PBS冲洗晾干,光学显微镜下观察并拍照。
1.7 统计学方法采用SPSS19.0统计软件进行数据分析,所获的全部数据以均数±标准差表示。采用单因素方差分析(ANOVA)和t检验,所有实验均重复3次,以P < 0.05为差异有统计学意义。
2 结果 2.1 采用Western blot和qRT-PCR筛选三种ICC细胞系中CHD1L蛋白和mRNA的表达Western blot结果显示,CHD1L和GAPDH分别在101 kD和36 kD有特异性条带,结果证实RBE细胞中CHD1L蛋白的表达量明显高于HCCC-9810和HUCCT1细胞(P=0.003, P=0.012),见图 1A。qRT-PCR结果亦显示,RBE细胞中CHD1L mRNA的表达量明显高于HCCC-9810和HUCCT1细胞(P=0.008, P=0.015),图 1B。

|
| 图 1 RBE、HCCC-9810及HUCCT1细胞系CHD1L蛋白(A)和mRNA(B)表达 Figure 1 CHD1L protein(A) and mRNA(B) expression in RBE, HCCC-9810 and HUCCT1 cell lines detected by Western blot and qRT-PCR |
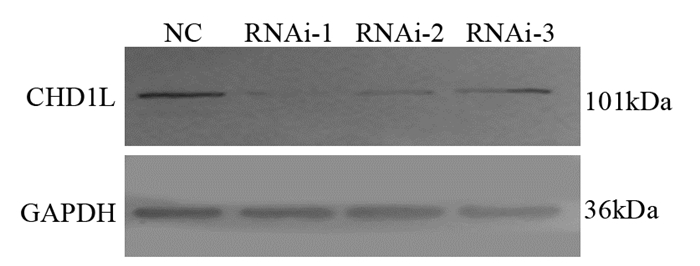
在LipofectamineTM 2000 Reagent转染试剂的介导下,能在荧光显微镜下见到绿色荧光,预实验结果显示在LipofectamineTM 2000转染试剂:质粒为1:2的情况下其转染效率最高、效果最好,转染率为(65.52±3.25)。由公司构建干扰组CHD1L-RNAi1、CHD1L-RNAi2、CHD1L-RNAi3和阴性对照组CHD1L-NC。将上述质粒转染入RBE细胞后检测CHD1L的蛋白表达,结果显示CHD1L-RNAi1的条带明显低于CHD1L-RNAi2和CHD1L-RNAi3,CHD1L-NC的条带明显高于其他条带,差异有统计学意义(P=0.003, P=0.008),见图 2。
|
| 图 2 Western blot检测各实验组细胞CHD1L蛋白表达情况 Figure 2 CHD1L expression in each group detected by Western blot |
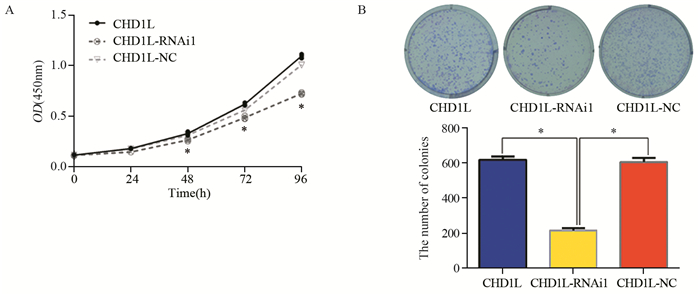
CCK-8结果显示CHD1L-RNAi1干扰后,RBE细胞在48、72、96 h增殖率明显下降,与未处理组和CHD1L-NC组对比差异有统计学意义(P < 0.05),而未处理组与CHD1L-NC组之间的差异无统计学意义(P > 0.05),见图 3A。平板克隆集落形成实验结果显示CHD1L-RNAi1干扰后,RBE细胞形成集落的数量(216.10±14.52)明显低于未处理组(618.67±20.26)和CHD1L-NC组(606.33±23.80),差异有统计学意义(P=0.002, P=0.001),见图 3B。上述结果均提示干扰CHD1L基因能够显著降低RBE细胞增殖能力。
|
| A: CCK-8 proliferation assays; B: plate clone formation assay; *: all P < 0.05 图 3 干扰CHD1L基因对RBE细胞增殖能力的影响 Figure 3 Effect of CHD1L silencing on proliferation ability of RBE cells |
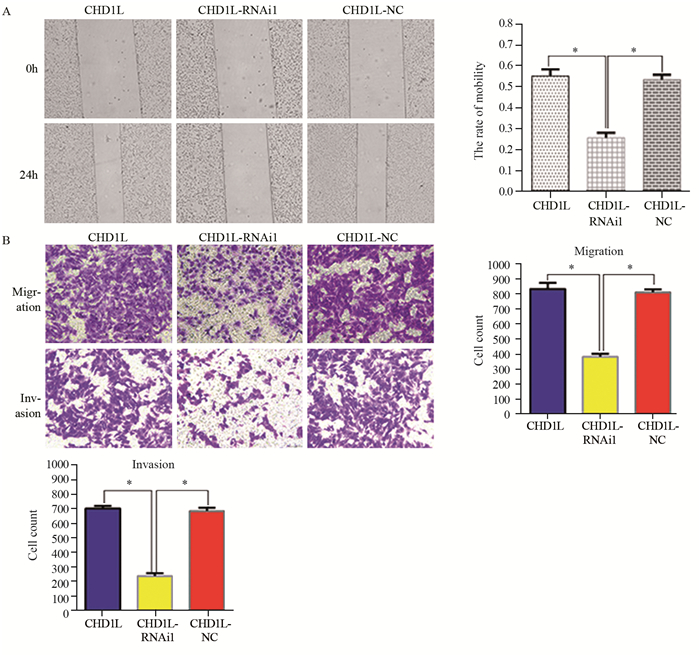
细胞划痕实验结果显示,与未处理组((5.5±3.2)%)和CHD1L-NC组((5.3±2.3)%)比较,干扰组CHD1L-RNAi1((2.55±2.4)%)中RBE细胞株的迁移率明显下降,差异有统计学意义(P=0.015, P=0.021),见图 4A。Transwell迁移和侵袭实验结果显示CHD1L-RNAi1组的穿膜细胞数量((381.35±21.13)、(236.65±20.03))与未处理组((833.33±40.50)、(702.33±18.04))、CHD1L-NC组((810.34±20.56)、(683.67±23.18))相比,细胞数量明显减少,差异有统计学意义(P=0.004, P=0.002),见图 4B。
|
| A: cell scratch assay; B: Transwell migration and invasion experiment; *: all P < 0.05 图 4 干扰CHD1L基因对RBE细胞体外迁移和侵袭能力的影响 Figure 4 Effect of CHD1L silencing on migration and invasion of RBE cells |
ICC是起源于肝内胆管上皮细胞的恶性肿瘤,具有起病隐匿、侵袭力强等特点。由于其早期诊断率低,易发生侵袭和转移,患者就诊时往往失去根治的机会,故预后极差[6]。因此,对于找到早期诊断识别可靠的肿瘤标志物、深入研究ICC发病机制、寻找新的治疗靶点等问题提出了迫切的需要。研究发现细胞的增殖、迁移和侵袭能力是肿瘤发生、发展的重要机制[7-8]。CHD1L是近几年新发现的一种位于染色体1q21致癌基因[9],该基因属于SNF-2类家族成员,具有高度保守的解旋酶功能,主要通过细胞增殖、调节G1/S周期和抑制凋亡等机制致癌。
CHD1L最初在肝癌中被发现其具有促进肿瘤发生、发展的作用[10-11],研究逐渐发现CHD1L的高表达与乳腺癌、胃癌、鼻咽癌等多种恶性肿瘤的发生、发展密切相关[12-14]。Sun等[4]研究表明,通过采用免疫组织化学和蛋白印迹检测CHD1L在胶质瘤组织和细胞株中都呈高表达,干扰其表达,胶质瘤细胞的侵袭能力下降。He等[15]对248例非小细胞型肺癌患者研究,发现CHD1L的过表达与肺癌的转移密切相关,并有可能成为肺腺癌的预后标志物。我们预实验通过对120例ICC组织和40例正常组织采用免疫组织化学分析,发现CHD1L在ICC中表达明显高于正常组织。但是,CHD1L基因能否促进ICC的增殖、侵袭和转移尚未研究。
本研究结果显示RBE细胞中CHD1L蛋白和mRNA的表达量明显高于HCCC-9810和HUCCT1细胞,因此干扰RBE细胞中的CHD1L表达能得到最理想的实验结果。将三种干扰CHD1L-RNAi质粒转染RBE细胞后,与CHD1L-NC组对比,结果显示三种干扰质粒均呈现不同程度的下降,其中干扰效果最好的质粒是CHD1L-RNAi1。CCK-8法和平板克隆集落形成实验结果表明干扰CHD1L基因能够显著降低RBE细胞增殖能力。细胞划痕实验和Transwell迁移实验结果显示,转染CHD1L-RNAi1后,能明显抑制RBE细胞的迁移能力。Transwell侵袭实验结果亦显示相同的结果,干扰CHD1L后,RBE细胞的侵袭能力亦受到明显的抑制作用。综上所述,CHD1L能促进ICC细胞的的增殖、迁移和侵袭,使癌细胞活性升高,从而增加ICC的恶性程度。Chen等[10]通过研究发现,CHD1L能够介导ARHGEF9的上调,诱导丝状伪足形成,促进上皮间质转化的形成,从而促进肝癌的侵袭和转移。干扰CHD1L的表达,能有效地抑制肝癌细胞的侵袭和转移能力。Mu等[3]运用免疫组织化学检测乳腺癌组织,发现CHD1L过表达与基质金属蛋白酶(MMP-2和MMP-9)表达呈正相关,通过体内外功能实验进一步证实,CHD1L能介导PI3K/Akt/ARK5/mTOR/MMP信号通路促进乳腺癌细胞增殖和侵袭。因此,下一步我们将深入探讨CHD1L参与信号通路的关键蛋白(如MMP-2和MMP-9)影响癌细胞的增殖能力和信号通路的关键步骤EMT影响细胞的侵袭能力,从而阐明CHD1L参与ICC细胞增殖和侵袭的具体分子机制。
综上所述,本研究结果表明CHD1L可促进ICC细胞体外的增殖、迁移和侵袭,具有广泛的生物学效应。今后需进一步对CHD1L在ICC的表达水平与患者的临床特征和病理资料进行分级分析,探讨CHD1L表达水平与ICC患者预后之间的关系。但是CHD1L能否成为ICC靶向治疗的靶点,及其在ICC中的重要作用及参与癌症的网络调控尚需更深入的研究。
| [1] | Ahel D, Hořejší Z, Wiechens N, et al. Poly(ADP-ribose)-dependent regulation of DNA repair by the chromatin remodeling enzyme ALC1[J]. Science, 2009, 325(5945): 1240–3. DOI:10.1126/science.1177321 |
| [2] | Liu M, Chen L, Ma NF, et al. CHD1L Promotes Lineage Reversion of Hepatocellular Carcinoma Through Opening Chromatin for Key Developmental Transcription Factors[J]. Hepatology, 2016, 63(5): 1544–59. DOI:10.1002/hep.v63.5 |
| [3] | Mu QJ, Li HL, Yao Y, et al. Chromodomain helicase/ATPase DNAbinding protein 1-like gene (CHD1L) expression and implications for invasion and metastasis of breast cancer[J]. PLoS One, 2015, 10(11): e0143030. DOI:10.1371/journal.pone.0143030 |
| [4] | Sun J, Zhang L, Zhao H, et al. CHD1L regulates cell cycle, apoptosis, and migration in Glioma[J]. Cell Mol Neurobiol, 2016, 36(4): 565–76. DOI:10.1007/s10571-015-0237-z |
| [5] | Li Y, Chen L, Chan TH, et al. SPOCK1 is regulated by CHD1L and blocks apoptosis and promotes HCC cell invasiveness and metastasis in mice[J]. Gastroenterology, 2013, 144(1): 179–91. DOI:10.1053/j.gastro.2012.09.042 |
| [6] | Siegel RL, Miller KD, Jemal A. Cancer statistics, 2016[J]. CA Cancer J Clin, 2016, 66(1): 7–30. DOI:10.3322/caac.21332 |
| [7] | Sulpice L, Rayar M, Desille M, et al. Molecular profiling of stroma identifies osteopontin as an independent predictor of poor prognosis in intrahepatic cholangioc-arcinoma[J]. Hepatology, 2013, 58(6): 1992–2000. DOI:10.1002/hep.26577 |
| [8] | Shu YJ, Weng H, Ye YY, et al. SPOCK1 as a potential cancer prognostic marker promotes the proliferation and metastasis of gallbladder cancer cells by activating the PI3K/AKT pathway[J]. Mol Cancer, 2015, 14: 12. DOI:10.1186/s12943-014-0276-y |
| [9] | Ma NF, Hu L, Fung JM, et al. Isolation and characterization of a novel oncogene, amplified in liver cancer 1, within a commonly amplified region at 1q21 in hepatocellular carcinoma[J]. Hepatology, 2008, 47(2): 503–10. |
| [10] | Chen L, Chan TH, Yuan YF, et al. CHD1L promotes hepatocellular carcinoma progression and metastasis in mice and is associated with these processes in human patients[J]. J Clin Invest, 2010, 120(4): 1178–91. DOI:10.1172/JCI40665 |
| [11] | Chan TH, Chen L, Liu M, et al. Translationally controlled tumor protein induces mitotic defects and chromosome missegregation in hepatocellular carcinoma development[J]. Hepatology, 2012, 55(2): 491–505. DOI:10.1002/hep.24709 |
| [12] | Wu J, Zong Y, Fei X, et al. Presence of CHD1L over-expression is associated with aggressive tumor biology and is a novel prognostic biomarker for patient survival in human breast cancer[J]. PLoS One, 2014, 9(8): e98673. DOI:10.1371/journal.pone.0098673 |
| [13] | Su Z, Zhao J, Xian G, et al. CHD1L is a novel independent prognostic factor for gastric cancer[J]. Clin Transl Oncol, 2014, 16(8): 702–7. DOI:10.1007/s12094-013-1136-8 |
| [14] | Su FR, Ding JH, Bo L, et al. Chromodomain helicase/ATPase DNA binding protein 1-like protein expression predicts poor prognosis in nasopharyngeal carcinoma[J]. Exp Ther Med, 2014, 8(6): 1745–50. DOI:10.3892/etm.2014.2017 |
| [15] | He LR, Ma NF, Chen JW, et al. Overexpression of CHD1L is positively associated with metastasis of lung adenocarcinoma and predicts patients poor survival[J]. Oncotarget, 2015, 6(31): 31181–90. DOI:10.18632/oncotarget.v6i31 |
 2017, Vol. 44
2017, Vol. 44


