文章信息
- LncRNA KCNQ1OT1在卵巢癌组织中的表达及其临床意义
- Expression and Clinical Significance of LncRNA KCNQ1OT1 in Ovarian Cancer Tissues
- 肿瘤防治研究, 2017, 44(5): 329-333
- Cancer Research on Prevention and Treatment, 2017, 44(5): 329-333
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2017.05.004
- 收稿日期: 2016-11-23
- 修回日期: 2017-02-14
卵巢恶性肿瘤具有起病隐匿、转移性强、预后较差等特点,是女性生殖系统常见的恶性肿瘤之一,死亡率位居妇科恶性肿瘤之首,发病率居女性生殖器恶性肿瘤的第二位,仅次于宫颈癌。其发病率为22.9%,并以每年1%的速度递增。上皮性卵巢癌(epithelial ovarian cancer, EOC),占卵巢癌病例的85%~90%。卵巢癌高发年龄在40~70岁间,患者5年生存率为30%[1-2]。由于临床症状不明显,患者在出现严重腹痛或消化道症状时才就诊,初诊患者中有2/3已属晚期,其中超过70%已有盆腔转移或淋巴转移[3]。越来越多的证据表明长链非编码RNA(LncRNA)是一种新兴的癌症生物学功能调节剂,可以作为潜在的癌症诊断、预后和靶向治疗的生物标志物[4-5]。近年研究证明,长链非编码RNA KCNQ1重叠转录物1(LncRNA KCNQ1OT1)在多种恶性肿瘤中表达,参与调节肿瘤的发生、发展[6-8],然而,其在卵巢癌中的作用目前尚不明确。本研究旨在研究LncRNA KCNQ1OT1在卵巢癌组织中的表达及其临床意义。
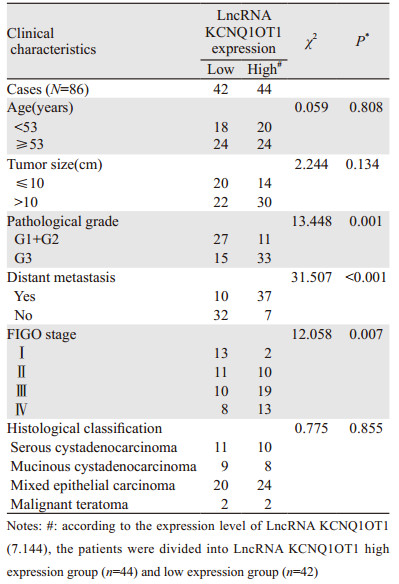
1 资料与方法 1.1 研究对象收集2012年1月至2015年12月期间在绵阳市中心医院妇产科就诊的86例卵巢癌患者的组织标本,患者年龄30~78岁,中位年龄53岁,53岁及以上者48例,53岁以下者38例。Ⅰ期15例,Ⅱ期21例,Ⅲ期29例,Ⅳ期21例。同时收集40例年龄相当的正常卵巢组织(来源于子宫肌瘤或子宫腺肌症行附件切除的患者)作为对照。术中切取卵巢癌及正常卵巢的部分组织立即置液氮中,用于提取RNA。患者一般资料包括年龄、FIGO分期、肿瘤分级及远处转移等临床特征见表 1。所有患者病例资料完整,术前均未行放化疗。本研究通过本院伦理委员会批准,并且所有患者均签署知情同意书。
 |
正常卵巢上皮细胞株HOSE 6.3及上皮性卵巢癌细胞株OVCAR-3均购自美国ATCC,细胞置于含10%胎牛血清的RPMI 1640培养液中,5%CO2、37℃饱和湿度的无菌恒温箱内培养。根据不同细胞的生长情况定期换液和传代。
1.3 细胞转染细胞转至6孔板中培养,待细胞生长到60%融合时,采用LipofectamineTM 2000和OPTI-MEMⅠ(Invitrogen)将Si-KCNQ1OT1(100 pmol)及Si-NC(阴性对照)分别转入细胞,转染后48 h,分别提取细胞总RNA备用。
1.4 Real-time PCR分析LncRNA KCNQ1OT1的表达水平采用TRIzol法提取组织及细胞中总RNA,反转录反应参照AMV反转录试剂盒说明,在20 μl体系中加2 μg总RNA进行cDNA的合成。Real-time PCR采用2×SYBR Green PCR Master Mix,取适量cDNA作为模板,引物浓度0.4 μmol/L,15 μl体系进行扩增,每个待测样本设置3个平行样,根据目标基因设计合成相应上下游引物进行PCR扩增,以GAPDH作为内参照。KCNQ1OT1上游引物:5’-CCCAGAAATCCACACCTCGG-3’;下游引物:5’-TCCTCAGTGAGCAGATGGAGA-3’;P53上游引物:5′-TACATGGGCCGAGGCAAGATAA-3’;下游引物:5’-ATAGCCCAGGGAAGTGAAGGTGTC-3′。PCR反应在定量PCR反应仪上进行。三次独立实验后得到的数据运用公式RQ=2-ΔΔCt的方法进行分析。
1.5 细胞转染按照每孔1×107个细胞数量均匀铺于6孔板内,铺板后约14~18 h时细胞的融合度达到60%~80%,换用无抗生素含10%血清的培养液换液。取出若干个无菌的EP管,分别加入200 μl的转染缓冲液和4 μl的脂质体,2 μg的pcDNA3.0-KCNQ1OT1/pcDNA3.0,吹打混匀,静置10 min后,分别加入6孔板内,转染后(4~6)h换用含双抗含血清的培养液继续培养。转染后48 h收取细胞提取RNA备用。siKCNQ1OT1上游引物:5’-TCGAGGGTACCGGATTCCCAGGC-3’;下游引物:5’-CGCAGGACACCCCAACTACAGCC-3’。
1.6 细胞增殖活性的检测收集对数期细胞,用细胞培养液调整悬液浓度至每毫升1×105个细胞,96孔板每孔加入100 μl细胞悬液(细胞数量为1×104个),5%CO2、37℃孵育,参照脂质体Lipofectamine 2000说明书分别转染各实验组和空质粒对照组。转染24、48、72、96 h后,应用细胞计数仪进行细胞计数。应用CCK-8法测定细胞增殖活性及化疗敏感度的变化。
1.7 Matrigel侵袭实验将Matrigel胶在4℃下过夜溶解,用无血清DMEM/F12培养液稀释。每孔100 μl均匀涂布于预冷的Transwell小室(孔径8 nm)滤膜上表面,将24孔培养板置于37℃孵育2 h,使Matrigel胶充分聚合呈凝胶状模拟体内细胞外基质。以含血清培养液作为趋化因子。取各组细胞100 μ1(1×105个细胞,用无血清培养液配制)加入上室内,在下室内加入600 μl的含血清培养液,作为趋化小细胞肺癌细胞迀移的动力,将Transwell小室放置于细胞培养箱内,5%CO2、37℃孵育48 h;用棉棒擦去微孔滤膜上室面的Matrigel胶及未穿越的细胞,0.4%结晶紫染色,显微镜下计数穿越的细胞数,以穿越细胞相对数目表示。
1.8 统计学方法采用SPSS13.0统计软件进行数据分析。计量资料采用t检验或One-way ANOVA进行分析。LncRNA KCNQ1OT1与患者各临床病理参数之间的关系采用Chi-Square检验;采用Kaplan-Meier法评估LncRNA KCNQ1OT1对患者生存时间的影响,采用Cox比例风险模型分析影响卵巢癌预后的因素,P < 0.05为差异有统计学意义。
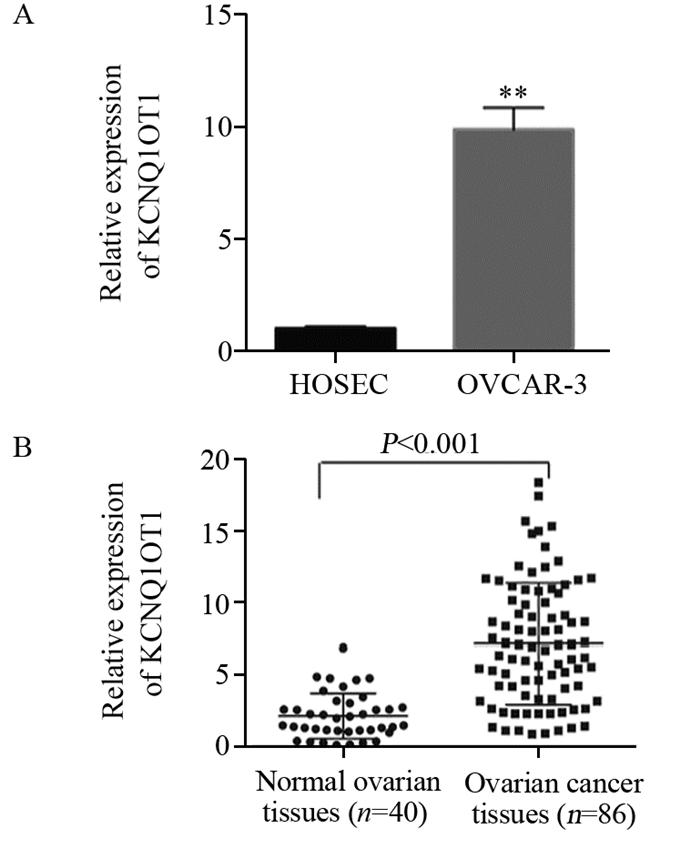
2 结果 2.1 LncRNA KCNQ1OT1在卵巢癌细胞及组织标本中的表达qRT-PCR法检测LncRNA KCNQ1OT1在正常卵巢上皮细胞株HOSEC及上皮性卵巢癌细胞株OVCAR-3及组织中的表达,结果发现LncRNA KCNQ1OT1在上皮性卵巢癌细胞株OVCAR-3中的表达较正常卵巢上皮细胞株HOSEC明显增高,差异有统计学意义(t=9.504, P < 0.001), 见图 1A。LncRNA KCNQ1OT1在86例卵巢癌组织中的平均表达水平为(7.144±0.445),与40例非肿瘤组织(1.476±0.250)比较,差异有统计学意义(t=7.266, P < 0.001),见图 1B。
|
| 图 1 qRT-PCR法检测LncRNA KCNQ1OT1在卵巢癌细胞 (A) 及卵巢癌组织 (B) 中的表达 Figure 1 The expression of LncRNA KCNQ10T1 were measured in ovarian cancer cells (A) and ovarian cancer tissues (B) by qRT-PCR |
根据LncRNA KCNQ1OT1平均表达水平(7.144),将患者分为LncRNA KCNQ1OT1高表达组44例及低表达组42例,分析LncRNA KCNQ1OT1的表达与患者临床病理特征的关系,结果发现LncRNA KCNQ1OT1的表达与患者的年龄、组织学分类及肿瘤大小无关,差异无统计学意义(均P > 0.05);而与肿瘤病理分级、FIGO分期、远处转移及LncRNA KCNQ1OT1的表达相关,差异有统计学意义(均P < 0.05),见表 1。
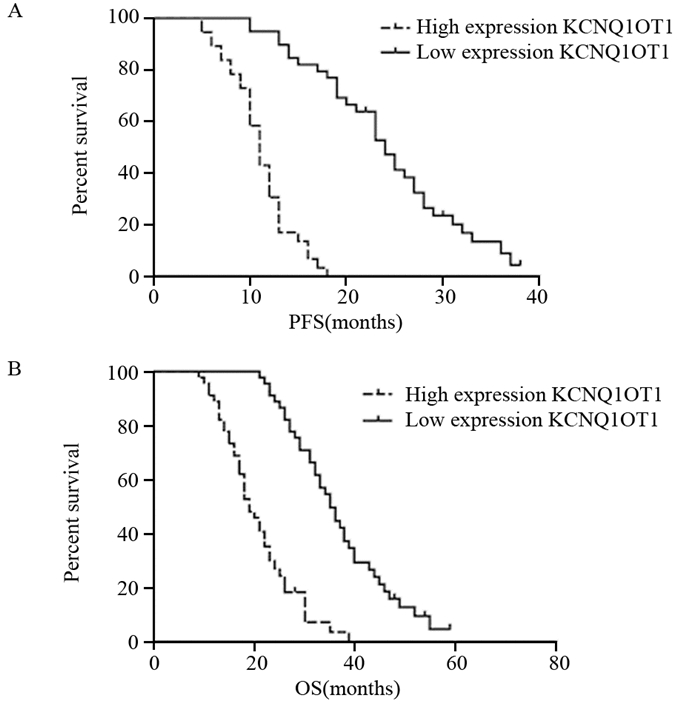
2.3 LncRNA KCNQ1OT1的表达与卵巢癌患者生存分析采用Kaplan-Meier法估计LncRNA KCNQ1OT1的表达与患者生存时间的关系,结果发现高表达LncRNA KCNQ1OT1的患者无进展生存时间为(11.00±4.02)月,较低表达者(24.00±2.82)月缩短,差异有统计学意义(χ2=48.24, P < 0.001),见图 2A;低表达LncRNA KCNQ1OT1的患者总生存时间为(35.00±5.37)月,较高表达者(19.00±6.75)月明显延长,差异有统计学意义(χ2=46.83, P < 0.001),见图 2B。
|
| 图 2 LncRNA KCNQ1OT1的表达与卵巢癌患者无进展生存时间及总生存时间的关系 Figure 2 The relationship of LncRNA KCNQ1OT1 expression with progression free survival and overall survival of ovarian patients |
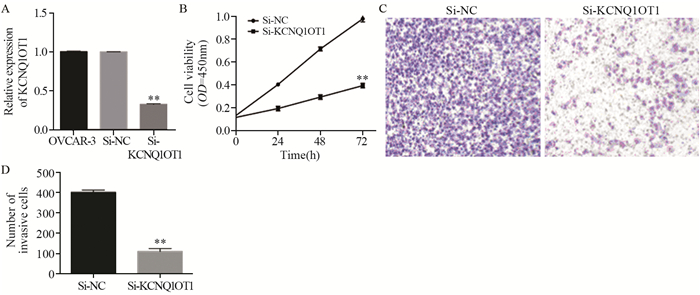
SKOV3细胞转染si-KCNQ1OT1或si-NC后,qRT-PCR检测KCNQ1OT1的表达,结果提示与si-NC组比较,细胞转染si-KCNQ1OT1后KCNQ1OT1 mRNA水平明显降低,差异有统计学意义(F=6.890, P=0.008)。CCK8检测细胞增殖的变化,结果提示转染si-KCNQ1OT1后细胞的增殖降低,侵袭力减弱(t=4.550, P=0.009),见图 3。
|
| A: qRT-PCR revealed that KCNQ1OT1 was efficiently knocked down by treatment with si-KCNQ1OT1 in SKOV3 cells; B: SKOV3 cells transfected with si-KCNQ1OT1 displayed significantly lower proliferation ability, compared with those transfected with si-NC; C: Transwell assay was used to detect the invasion ability of SKOV3 cells transfected with si-NC and si-KCNQ1OT1; D: SKOV3 cells transfected with si-KCNQ1OT1 displayed significantly lower invasion ability, compared with those transfected with si-NC; **: P < 0.01, compared with si-NC 图 3 下调SKOV3细胞中LncRNA KCNQ1OT1的表达抑制细胞的侵袭和增殖 Figure 3 Knockdown of LncRNA KCNQ1OT1 inhibited invasion and proliferation of SKOV3 cells |
卵巢癌患者病死率居妇科恶性肿瘤之首,因其特殊生理结构,并且缺乏针对性的早期筛查方法,多数患者发现时已是晚期[9]。因此寻找早期筛查、诊断及预后标志物已成为目前卵巢癌研究的热点。随着研究的深入,20世纪90年代后学者发现了具有基因调控能力的长链非编码RNA,即LncRNA。这些RNA可以特异性地调控基因的表达或转录过程,研究较清楚的有H19和Xist等[10-11]。20世纪90年代,随着全基因组测序的发展,尤其是基因芯片和二代测序的产生,人们开始相信基因组实际上是“普遍转录”的[12-13]。研究发现人基因组中高达70%~90%的序列都存在转录,在功能基因间的间隔区中,存在有大量的非编码转录本。但是当时认为,这些转录大多没有功能,属于“转录噪声”,即非编码RNA实际上不执行任何功能。近年来研究发现,越来越多的非编码RNA被证明具有特殊而重要的功能,与肿瘤的形成、浸润、转移等生物学过程相关,有望成为新的肿瘤标志物和治疗靶点,在肿瘤诊断和治疗方面显示出良好的临床应用前景[9, 14-15]。研究发现LncRNA GAPLINC通过靶向作用于SNAI2促进结直肠癌细胞的侵袭[16]。上调LncRNA NEAT1的表达会促进膀胱癌的进展[17]。LncRNA CRNDE的上调与结直肠癌预后差相关,其表达与IRX5 mRNA的表达呈正相关关系[18];MALAT1是第一个在肺癌中被研究的LncRNA,有研究提出它可以作为早期肺腺癌中患者的一个独立预后标志物[19]。此外研究还发现,在非小细胞肺癌(NSCLC)样本中HOTAIR高表达的患者预后相对较差[20];CCAT2在NSCLC组织中显著高表达,可能是一个肺腺癌特异的LncRNA[21]。GAS6-AS1可能是NSCLC患者一个独立预后标志物[22]。以上研究结果提示LncRNA在多种肿瘤中表达,与肿瘤的发生发展相关。然而关于LncRNA在卵巢癌中的功能研究,目前国内外相关报道较少。已有研究发现HOTAIR rs920778的多态性与卵巢癌的易感性及不良预后相关[23]。下调LncRNA NR_026689能够抑制卵巢癌细胞HO-8910PM的增殖和侵袭,增加其凋亡[24]。LncRNA MALAT1在卵巢癌组织中上调,能促进卵巢癌细胞SKOV3的增殖和侵袭[25]。LncRNA C17orf91作为卵巢癌癌基因与患者的预后相关[26]。过表达LncRNA PVT1通过调节凋亡信号通路促进卵巢癌细胞对顺铂耐药[27]。
课题组前期通过基因芯片发现LncRNA KCNQ1OT1卵巢癌细胞株SKOV3中明显增高,但其在卵巢癌中的表达及具体的作用机制目前国内外尚未见相关报道。近年来研究发现KCNQ1OT1在多种肿瘤中表达[28-29]。KCNQ1OT1是一个位于KCNQ1位点的长链非编码RNA基因[6, 8, 30]。本研究通过实时荧光定量PCR法检测卵巢癌组织、癌旁组织及细胞中LncRNA KCNQ1OT1表达,分析其与临床病理特征和预后的关系。同时,通过转染siRNA下调LncRNA KCNQ1OT1的表达,检测细胞增殖和侵袭的变化。结果发现卵巢癌组织及细胞株中LncRNA KCNQ1OT1的表达水平显著高于非肿瘤组织和正常卵巢上皮细胞。高表达LncRNA KCNQ1OT1的患者的总生存期和无进展生存时间较低表达者缩短。LncRNA KCNQ1OT1的表达与FIGO分期、肿瘤分级和远处转移相关。此外,下调LncRNA KCNQ1OT1的表达能够明显抑制细胞的增殖和侵袭。以上研究结果提示LncRNA KCNQ1OT1参与调节卵巢癌的发生、发展,可能作为卵巢癌患者预后的一个潜在生物标志物和治疗靶点。但由于临床病例较少,其具体作用机制尚需扩大样本量及实验室进一步研究。
| [1] | Chai Y, Liu J, Zhang Z, et al. HuR-regulated lncRNA NEAT1 stability in tumorigenesis and progression of ovarian cancer[J]. Cancer Med, 2016, 5(7): 1588–98. DOI:10.1002/cam4.2016.5.issue-7 |
| [2] | Chen Q, Liu X, Xu L, et al. Long non-coding RNA BACE1-AS is a novel target for anisomycin-mediated suppression of ovarian cancer stem cell proliferation and invasion[J]. Oncol Rep, 2016, 35(4): 1916–24. |
| [3] | Ren P, Yang XQ, Zhai XL, et al. Overexpression of Rab27B is correlated with distant metastasis and poor prognosis in ovarian cancer[J]. Oncol Lett, 2016, 12(2): 1539–45. |
| [4] | Hong HH, Hou LK, Pan X, et al. Long non-coding RNA UCA1 is a predictive biomarker of cancer[J]. Oncotarget, 2016, 7(28): 44442–7. |
| [5] | Hu L, Chen SH, Lv QL, et al. Clinical Significance of Long Non-Coding RNA CASC8 rs10505477 Polymorphism in Lung Cancer Susceptibility, Platinum-Based Chemotherapy Response, and Toxicity[J]. Int J Environ Res Public Health, 2016, 13(6): pii:E545. DOI:10.3390/ijerph13060545 |
| [6] | Nakano S, Murakami K, Meguro M, et al. Expression profile of LIT1/KCNQ1OT1 and epigenetic status at the KvDMR1 in colorectal cancers[J]. Cancer Sci, 2006, 97(11): 1147–54. DOI:10.1111/cas.2006.97.issue-11 |
| [7] | Soejima H, Nakagawachi T, Zhao W, et al. Silencing of imprinted CDKN1C gene expression is associated with loss of CpG and histone H3 lysine 9 methylation at DMR-LIT1 in esophageal cancer[J]. Oncogene, 2004, 23(25): 4380–8. DOI:10.1038/sj.onc.1207576 |
| [8] | Wan J, Huang M, Zhao H, et al. A novel tetranucleotide repeat polymorphism within KCNQ1OT1 confers risk for hepatocellular carcinoma[J]. DNA Cell Biol, 2013, 32(11): 628–34. DOI:10.1089/dna.2013.2118 |
| [9] | Meryet-Figuiere M, Lambert B, Gauduchon P, et al. An overview of long non-coding RNAs in ovarian cancers[J]. Oncotarget, 2016, 7(28): 44719–34. |
| [10] | Araujo ES, Vasques LR, Stabellini R, et al. Stability of XIST repression in relation to genomic imprinting following global genome demethylation in a human cell line[J]. Braz J Med Biol Res, 2014, 47(12): 1029–35. DOI:10.1590/1414-431X20144058 |
| [11] | Wang SH, Ma F, Tang ZH, et al. Long non-coding RNA H19 regulates FOXM1 expression by competitively binding endogenous miR-342-3p in gallbladder cancer[J]. J Exp Clin Cancer Res, 2016, 35(1): 160. DOI:10.1186/s13046-016-0436-6 |
| [12] | Hu L, Lv QL, Chen SH, et al. Up-Regulation of Long Non-Coding RNA AB073614 Predicts a Poor Prognosis in Patients with Glioma[J]. Int J Environ Res Public Health, 2016, 13(4): 433. DOI:10.3390/ijerph13040433 |
| [13] | Huang S, Qing C, Huang Z, et al. The long non-coding RNA CCAT2 is up-regulated in ovarian cancer and associated with poor prognosis[J]. Diagn Pathol, 2016, 11(1): 49. DOI:10.1186/s13000-016-0499-x |
| [14] | Jiang P, Wu X, Wang X, et al. NEAT1 upregulates EGCG-induced CTR1 to enhance cisplatin sensitivity in lung cancer cells[J]. Diagn Pathol, 2016, 7(28): 43337–51. |
| [15] | Ozes AR, Miller DF, Ozes ON, et al. NF-kappaB-HOTAIR axis links DNA damage response, chemoresistance and cellular senescence in ovarian cancer[J]. Oncogene, 2016, 35(41): 5350–61. DOI:10.1038/onc.2016.75 |
| [16] | Yang P, Chen T, Xu Z, et al. Long noncoding RNA GAPLINC promotes invasion in colorectal cancer by targeting SNAI2 through binding with PSF and NONO[J]. Oncotarget, 2016, 7(27): 42194. |
| [17] | XianGuo C, ZongYao H, Jun Z, et al. Promoting progression and clinicopathological significance of NEAT1 over-expression in bladder cancer[J]. Oncotarget, 2016. |
| [18] | Liu T, Zhang X, Yang YM, et al. Increased expression of the long noncoding RNA CRNDE-h indicates a poor prognosis in colorectal cancer, and is positively correlated with IRX5 mRNA expression[J]. Onco Targets Ther, 2016, 9: 1437–48. |
| [19] | Huang NS, Chi YY, Xue JY, et al. Long non-coding RNA metastasis associated in lung adenocarcinoma transcript 1(MALAT1) interacts with estrogen receptor and predicted poor survival in breast cancer[J]. Oncotarget, 2016, 7(25): 37957–65. |
| [20] | Zhou C, Ye L, Jiang C, et al. Long noncoding RNA HOTAIR, a hypoxia-inducible factor-1alpha activated driver of malignancy, enhances hypoxic cancer cell proliferation, migration, and invasion in non-small cell lung cancer[J]. Tumour Biol, 2015, 36(12): 9179–88. DOI:10.1007/s13277-015-3453-8 |
| [21] | Qiu M, Xu Y, Yang X, et al. CCAT2 is a lung adenocarcinoma-specific long non-coding RNA and promotes invasion of non-small cell lung cancer[J]. Tumour Biol, 2014, 35(6): 5375–80. DOI:10.1007/s13277-014-1700-z |
| [22] | Shi X, Sun M, Liu H, et al. A critical role for the long non-coding RNA GAS5 in proliferation and apoptosis in non-small-cell lung cancer[J]. Mol Carcinog, 2015, 54(Suppl 1): E1–E12. |
| [23] | Qiu H, Wang X, Guo R, et al. HOTAIR rs920778 polymorphism is associated with ovarian cancer susceptibility and poor prognosis in a Chinese population[J]. Future Oncol, 2016, 13(4): 347–55. DOI:10.2217/pme-2016-0014 |
| [24] | Zhang X, Li S, Dong C, et al. Knockdown of long noncoding RNA NR_026689 inhibits proliferation and invasion and increases apoptosis in ovarian carcinoma HO-8910PM cells[J]. Oncol Res, 2017, 25(2): 259–65. DOI:10.3727/096504016X14732503870766 |
| [25] | Zou A, Liu R, Wu X. Long non-coding RNA MALAT1 is up-regulated in ovarian cancer tissue and promotes SK-OV-3 cell proliferation and invasion[J]. Neoplasma, 2016, 63(6): 865–72. DOI:10.4149/neo_2016_605 |
| [26] | Li J, Yu H, Xi M, et al. Long noncoding RNA C17orf91 is a potential prognostic marker and functions as an oncogene in ovarian cancer[J]. J Ovarian Res, 2016, 9(1): 49. DOI:10.1186/s13048-016-0258-3 |
| [27] | Liu E, Liu Z, Zhou Y, et al. Overexpression of long non-coding RNA PVT1 in ovarian cancer cells promotes cisplatin resistance by regulating apoptotic pathways[J]. Int J Clin Exp Med, 2015, 8(11): 20565–72. |
| [28] | Schultz BM, Gallicio GA, Cesaroni M, et al. Enhancers compete with a long non-coding RNA for regulation of the Kcnq1 domain[J]. Nucleic Acids Res, 2015, 43(2): 745–59. DOI:10.1093/nar/gku1324 |
| [29] | Sunamura N, Ohira T, Kataoka M, et al. Regulation of functional KCNQ1OT1 lncRNA by beta-catenin[J]. Sci Rep, 2016, 6: 20690. DOI:10.1038/srep20690 |
| [30] | Bliek J, Maas SM, Ruijter JM, et al. Increased tumour risk for BWS patients correlates with aberrant H19 and not KCNQ1OT1 methylation: occurrence of KCNQ1OT1 hypomethylation in familial cases of BWS[J]. Hum Mol Genet, 2001, 10(5): 467–76. DOI:10.1093/hmg/10.5.467 |
 2017, Vol. 44
2017, Vol. 44
