文章信息
- 常见实体瘤的分子分型
- Molecular Classification of Common Solid Cancers
- 肿瘤防治研究, 2016, 43(11): 1001-1004
- Cancer Research on Prevention and Treatment, 2016, 43(11): 1001-1004
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2016.11.016
- 收稿日期: 2016-02-24
- 修回日期: 2016-03-30
2. 730050 兰州,兰州军区兰州总医院眼科
2. Department of Ophthalmology,Lanzhou General Hospital of Lanzhou Military Region,Lanzhou 730050,China
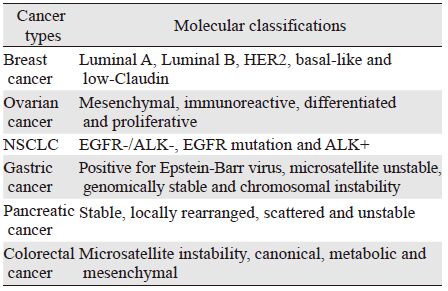
传统的肿瘤命名以器官部位为基础,亚分类则包括患者年龄、细胞类型、组织分级或分子标志物等。在过去的15年里,二代测序技术提供了肿瘤DNA、RNA、蛋白质和表观遗传等突变资料,这些资料不仅增加了我们对肿瘤异质性的理解,也拓展了肿瘤分类的新方法[1],肿瘤的分子分型也从依赖单一或少数标志物检测进入到谱学分型阶段。Hoadley等[2]分析了12种肿瘤的基因组学和蛋白质学特征,发现相同起源的细胞类型具有更多分子和遗传的相似性,提示器官分类的价值。肿瘤器官分类和分子分型的结合可能是未来肿瘤分类的方向,并将改善肿瘤治疗方法[3]。进入精准医学时代,我们更需要肿瘤的分子分型来实现精准治疗。这要求分子靶向治疗药物与个体肿瘤患者的唯一基因和分子构成精确匹配,即根据患者的分子分型指导治疗。一些常见肿瘤,如:乳腺癌、卵巢癌、肺癌、胃癌、胰腺癌和结直肠癌等的分子分型已用于指导临床治疗的选择,见表 1,而其他一些肿瘤,如:甲状腺癌、肝细胞癌、肾癌、前列腺癌、子宫内膜癌、皮肤癌和内分泌癌等的分子分型也逐渐进入临床。研究肿瘤的分子分型是实现肿瘤精准治疗的基础,本文系统介绍常见实体瘤的分子分型,为临床治疗提供指导。
乳腺癌是最早建立分子分型的肿瘤。基因表达谱的进步使人们认识到乳腺癌是一种分子异质性疾病,应用基因表达微阵列构建了乳腺癌的分子分型[4]。2009年St.Gallen会议提出乳腺癌分子分型概念,不同分子分型对不同全身治疗的有效性以及全身转移风险和生存差别得到了广泛的验证性研究[5]。2000年发表在Nature杂志上的一篇论文,开创了用基因表达谱研究乳腺癌分子分型的先河[6]。通过测定乳腺癌基因组,将乳腺癌分为:乳腺导管A型(Luminal A,ER+或PgR+,HER2-)、乳腺导管B型(Luminal B,ER+或PgR+,HER2+)、HER2阳性型(ER-/PgR-/HER2+)和基底细胞样型(Basal-like,ER-/PgR-/HER2-伴Cytokeratin5/6+或HER1+)[7],这种分型已广泛应用于乳腺癌的临床实践中。
Carlsson等[8]应用包含135个抗体的重组抗体微阵列平台,针对65个主要免疫调节蛋白,构建了一个21基因的蛋白谱,将乳腺癌患者分为高风险组和低风险组两组。研究证明,这种血清的分子特征能够较好地预测乳腺癌术后的远处转移。Dawson等[9]应用一个新的基因组为基础的整合分类法(genome-driven integrated classification)将乳腺癌分为10类,每类具有不同的预后和分子生物学特征。这种新的分类整合乳腺癌遗传和表观遗传特征,提供了研究乳腺癌分子分型的新方法。现在,乳腺癌的五类法(Luminal A、Luminal B、HER2过表达型、基底样和低Claudin)已被广泛接受并应用[10]。Anderson等[11]按照病因分类将乳腺癌分为诊断时50岁和70岁两个类型,代表了乳腺癌不同的临床特征、肿瘤特性、风险因素和分子亚型,无疑也是一种分类尝试。
2 卵巢癌Leong等[12]通过微阵列基因图谱技术将高级别浆液性卵巢癌分为四型:间叶细胞型、免疫反应型、增殖型和分化型。分化型有高度异质性,而其余三型则保持匀质性。基因组研究表明,复发高级别浆液性卵巢癌常发生基因断裂,导致RB1、NF1、RAD51B和PTEN等肿瘤抑制基因功能失活,从而出现获得性化疗抗拒;原发性化疗抗拒与难治性病例常发生CCNE1扩增。这种分子分型对临床治疗的指导作用尚待探索。
3 肺癌肺癌根据组织学分为四种类型,然而靶向肺癌分子改变的治疗明显提高了患者的生存期,提示肿瘤分型应考虑基因组信息[13]。大多数肺鳞癌都能找到其相应的驱动基因,主要的有药物治疗的靶点包括FGFR1扩增、PI3K通路改变、细胞周期检查点失衡和DDR2突变。通过分析mRNA提示即使在鳞癌的亚型中也存在异质性,包括几个亚分类:经典型(classical)、基底型(basal)、分泌型(secretory)和原始型(primitive)。对于晚期肺非鳞癌,目前按照生物标志物可将其分为三类:EGFR和ALK基因阴性、ALK融合基因阳性和EGFR基因突变阳性。按照分子分型一线治疗分别为含铂化疗、克唑替尼和厄洛替尼或阿法替尼。
未来晚期非小细胞肺癌(non-small cell lung cancer,NSCLC)将通过检测基因将患者分为:EGFR、ALK、ROS1、RET、BRAF、其他潜在基因改变和没有基因改变等亚型,并分别选择EGFR TKI、克唑替尼或二代ALK TKI、克唑替尼、Cabozantinib、BRAF抑制剂、临床试验和化疗或免疫治疗[14]。在过去的十年中,基于驱动基因标志物的靶向治疗和免疫检查点治疗使NSCLC的临床诊疗水平有了较大提高。基于驱动基因标志物的靶向治疗具有较高的缓解率,但疗效持续时间短,而免疫检查点抑制剂治疗的缓解率较低,但疗效的持续时间长。
4 胃癌2014年癌症基因组图谱研究网络的研究人员提出了胃癌分子分型,新分子分类方法将胃癌分为4种亚型:EB病毒感染型胃癌、微卫星不稳定(microsatellite instable,MSI)型胃癌、基因稳定(genomically stable,GS)型胃癌和染色体不稳定(chromosomal instability,CIN)型胃癌。其中,在EB病毒感染型胃癌中,PI3KCA呈高频突变,因此这类人群是PI3K抑制剂的靶向人群;也可能是CDK4/6抑制剂的适合人群;同时还可能是PD-L1免疫治疗的靶向人群。而GS型胃癌大多属于弥漫型胃癌,RHOA可能成为预后较差的弥漫型胃癌的潜在靶点。CIN型约占50%,胃食管交界处和贲门多发,多属肠型;因RTKs基因频扩增,可成为靶向药物的目标人群;对激酶抑制剂可能会比较敏感。MSI型则约占22%,好发于胃窦或幽门,女性多见(56%);可见DNA超甲基化,包括PIK3CA、ERBB3、ERBB2等在内的基因呈高突变,但缺乏基因扩增;治疗策略多是甲基化抑制剂、靶向基因突变的抑制剂如PI3K抑制剂[15]。Cristescu等[16]也将胃癌分为四个亚型:间质细胞型、微卫星不稳定型、TP53激活型和TP53失活型。间质细胞型包括弥漫型胃癌,常见于早期胃癌、高复发和预后差;微卫星不稳定型见于胃窦部的高突变肠型胃癌,复发率低,预后好;TP53激活型和TP53失活型有中等的复发率和预后。
5 胰腺癌Waddell等[17]通过对100例胰腺癌样本进行全基因组深度测序,将胰腺癌分为四个类型:稳定型:染色体结构变异<50,非整倍体广泛存在;局部重排型:集中于1~2个染色体基因重排;分散型:中等幅度的非随机性染色体损伤和<200染色体结构变异;不稳定型:大量的结构变异>200。然后选取与铂类药物敏感度相关的三个特征(基因组不稳定性、BRCA通路突变和BRCA信号异常)筛选出铂类药物敏感的患者群体,取得良好效果,这无疑是胰腺癌分子诊断的里程碑式研究,开启了胰腺癌诊疗的新思路。
6 结直肠癌结直肠癌也是一种异质性疾病,其分子分型主要为三类:染色体不稳定型,表现为癌基因和抑癌基因的累积突变;微卫星不稳定型,因DNA错配修复基因失调而导致基因超突变;CpG岛甲基化型,不同于超甲基化[18]。Guinney等[19]将结直肠癌分为四类:微卫星不稳定型(14%),超突变,微卫星不稳定和免疫激活;普通型(37%),上皮细胞,WNT和MYC信号通路激活;代谢型(13%),上皮细胞和代谢失调;间质型(23%),转化生长因子β激活,基质浸润和血管生成。另外,13%混合型代表了肿瘤的转化表型和肿瘤内异质型。
7 其他肿瘤除了上述常见肿瘤的分子分型外,其他肿瘤的分子分型也逐渐进入临床。甲状腺乳头状癌(papillary thyroid carcinoma,PTC)是甲状腺癌中最常见的类型。通过对496例PTC的基因组分析,发现PTC中EIF1AX、PPM1D和CHEK2等驱动基因改变和基因融合,并鉴定了BRAF突变分子亚型,开创了PTC的分子分型先河[20]。肝细胞癌(hepatocellular carcinoma,HCC)基因组分析使HCC的分子分型成为可能。Goossens将HCC分为侵袭型和非侵袭型两类,前者表现为TP53失活突变和原癌基因信号通路激活;后者以CTNNB1突变、肝特异性WNT靶标过表达为特征[21]。乳头状肾细胞癌约占肾细胞癌的15%~20%,是一类异质性明显的疾病。Linehan等[22]通过对乳头状肾细胞癌全面的分子特征分析,将其分为两型:Ⅰ型表现为MET基因改变,Ⅱ型则联系着CDKN2A沉默、SETD2突变、TFE3融合和NRF2抗氧化反应通路激活等。Ⅱ型中还有CpG岛甲基化表型,预后差。前列腺癌分为ETS融合基因阳性、SPINK1过表达和CHD1缺失等各型[23]。
未来多个候选预测分子标志物联合检测、新标志物应用和不同时间点转移部位的活检可能是实现“适合的患者应用适合的药物”这一精准治疗概念的关键步骤。子宫内膜癌传统的分类方法或基于临床和内分泌特征(Ⅰ型和Ⅱ型),或基于组织病理特征(子宫内膜样癌、浆液癌和透明细胞腺癌)。新的分类系统整合基因组和组织病理特征更有助于子宫内膜癌精准治疗的实现[24]。子宫内膜癌也重新分为四个类型:POLE强突变、微卫星不稳定超突变、低拷贝数和高拷贝数[25]。Jee等[26]通过对皮肤癌基因表达谱比较分析,将皮肤基底细胞癌分为三型:传统型、鳞状细胞癌样型和类正常基底细胞癌型。传统型表现为Wnt和Hedgehog信号通路激活;鳞状细胞癌样型则为免疫反应和氧化压力相关基因表达,PLAU/PLAUR是其关键调节因子;类正常基底细胞癌型表现为代谢尤其是脂肪酸代谢旺盛。
8 结语肿瘤的异质性是肿瘤分子分型的基础,即使同一患者的同一瘤体内也无法寻找到两个一模一样的肿瘤细胞[27]。Sottoriva等[28]提出了一个“大爆炸”模型,认为肿瘤早期快速膨胀生长,产生多个非选择的异质亚克隆。一些肿瘤显然“天生就是坏的”,早期的亚克隆已呈现恶性潜能。当然,肿瘤的异质性也是肿瘤在不同压力下克隆进化的结果,如化疗可能促进某些微小或休眠的细胞克隆占优势,多种自然选择压力使肿瘤在达尔文式选择过程中发生有利于自身生长和增殖的适应性改变。分子分型即将肿瘤的异质性根据分子特征分类。
肿瘤的分子分型改变了传统的分类方法和治疗策略,组学的发展使肿瘤分子分型日益完善。肿瘤基因组学包括基因点突变和复制的DNA改变,而肿瘤转录组学则涉及包含mRNA在内的RNA改变,两者都提供了肿瘤患者个体的全面信息,从而实现实时的精准治疗[29]。单细胞方法亦为肿瘤的分子分类提供了新的平台[30]。基于生物网络的数学模型和肿瘤细胞分子通路的高通量数据库可能会提高肿瘤分子分型和精准治疗的效果[31]。但是,以基因组为基础的肿瘤分子分型也有诸多问题:肿瘤的分子分型究竟有多少?如何评估一个新分子分型的优劣?分类系统如何受肿瘤异质性和进化的影响?如何理解肿瘤分子分型?是否存在多个分子分型?理解这些问题可能帮助我们提出更好的肿瘤分子分型并实现肿瘤精准治疗。
| [1] | Song Q, Merajver SD, Li JZ. Cancer classification in the genomic era: five contemporary problems[J]. Hum Genomics, 2015, 9 : 27. DOI:10.1186/s40246-015-0049-8 |
| [2] | Hoadley KA, Yau C, Wolf DM, et al. Multiplatform analysis of 12 cancer types reveals molecular classification within and across tissues of origin[J]. Cell, 2014, 158 (4) : 929–44. DOI:10.1016/j.cell.2014.06.049 |
| [3] | Cortés J, Calvo E, Vivancos A, et al. New approach to cancer therapy based on a molecularly defined cancer classification[J]. CA Cancer J Clin, 2014, 64 (1) : 70–4. DOI:10.3322/caac.21211 |
| [4] | Cianfrocca M, Gradishar W. New molecular classifications of breast cancer[J]. CA Cancer J Clin, 2009, 59 (5) : 303–13. DOI:10.3322/caac.20029 |
| [5] | Goldhirsch A, Wood WC, Coates AS, et al. Strategies for subtypes--dealing with the diversity of breast cancer: highlights of the St. Gallen International Expert Consensus on the Primary Therapy of Early Breast Cancer 2011[J]. Ann Oncol, 2011, 22 (8) : 1736–47. DOI:10.1093/annonc/mdr304 |
| [6] | Perou CM, S?rlie T, Eisen MB, et al. Molecular portraits of human breast tumours[J]. Nature, 2000, 406 (6797) : 747–52. DOI:10.1038/35021093 |
| [7] | S?rlie T, Perou CM, Tibshirani R, et al. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications[J]. Proc Natl Acad Sci U S A, 2001, 98 (19) : 10869–74. DOI:10.1073/pnas.191367098 |
| [8] | Carlsson A, Wingren C, Kristensson M, et al. Molecular serum portraits in patients with primary breast cancer predict the development of distant metastases[J]. Proc Natl Acad Sci U S A, 2011, 108 (34) : 14252–7. DOI:10.1073/pnas.1103125108 |
| [9] | Dawson SJ, Rueda OM, Aparicio S, et al. A new genome-driven integrated classification of breast cancer and its implications[J]. EMBO J, 2013, 32 (5) : 617–28. DOI:10.1038/emboj.2013.19 |
| [10] | Prat A, Pineda E, Adamo B, et al. Clinical implications of the intrinsic molecular subtypes of breast cancer[J]. Breast, 2015, 24 : S26–35. |
| [11] | Anderson WF, Rosenberg PS, Prat A, et al. How many etiological subtypes of breast cancer: two,three,four,or more?[J]. J Natl Cancer Inst, 2014, 106 (8) : Pii: dju 165. DOI:10.1093/jnci/dju165 |
| [12] | Leong HS, Galletta L, Etemadmoghadam D, et al. Efficient molecular subtype classification of high-grade serous ovarian cancer[J]. J Pathol, 2015, 236 (3) : 272–7. DOI:10.1002/path.4536 |
| [13] | Bunn PA Jr, Franklin W, Doebele RC. The evolution of tumor classification: a role for genomics?[J]. Cancer Cell, 2013, 24 (6) : 693–4. DOI:10.1016/j.ccr.2013.11.019 |
| [14] | Kris MG, Johnson BE, Berry LD, et al. Using multiplexed assays of oncogenic drivers in lung cancers to select targeted drugs[J]. JAMA, 2014, 311 (19) : 1998–2006. DOI:10.1001/jama.2014.3741 |
| [15] | Cancer Genome Atlas Research Network. Comprehensive molecular characterization of gastric adenocarcinoma[J]. Nature, 2014, 513 (7517) : 202–9. DOI:10.1038/nature13480 |
| [16] | Cristescu R, Lee J, Nebozhyn M, et al. Molecular analysis of gastric cancer identifies subtypes associated with distinct clinical outcomes[J]. Nat Med, 2015, 21 (5) : 449–56. DOI:10.1038/nm.3850 |
| [17] | Waddell N, Pajic M, Patch AM, et al. Whole genomes redefine the mutational landscape of pancreatic cancer[J]. Nature, 2015, 518 (7540) : 495–501. DOI:10.1038/nature14169 |
| [18] | Bogaert J, Prenen H. Molecular genetics of colorectal cancer[J]. Ann Gastroenterol, 2014, 27 (1) : 9–14. |
| [19] | Guinney J, Dienstmann R, Wang X, et al. The consensus molecular subtypes of colorectal cancer[J]. Nat Med, 2015, 21 (11) : 1350–6. DOI:10.1038/nm.3967 |
| [20] | Cancer Genome Atlas Research Network. Integrated genomic characterization of papillary thyroid carcinoma[J]. Cell, 2014, 159 (3) : 676–90. DOI:10.1016/j.cell.2014.09.050 |
| [21] | Goossens N, Sun X, Hoshida Y. Molecular classification of hepatocellular carcinoma: potential therapeutic implications[J]. Hepat Oncol, 2015, 2 (4) : 371–9. DOI:10.2217/hep.15.26 |
| [22] | Cancer Genome Atlas Research Network, Linehan WM, Spellman PT, et al. Comprehensive Molecular Characterization of Papillary Renal-Cell Carcinoma[J]. N Engl J Med, 2016, 374 (2) : 135–45. DOI:10.1056/NEJMoa1505917 |
| [23] | Attard G, Parker C, Eeles RA, et al. Prostate cancer[J]. Lancet, 2016, 387 (10013) : 70–82. DOI:10.1016/S0140-6736(14)61947-4 |
| [24] | Murali R, Soslow RA, Weigelt B. Classification of endometrial carcinoma: more than two types[J]. Lancet Oncol, 2014, 15 (7) : e268–78. DOI:10.1016/S1470-2045(13)70591-6 |
| [25] | Cancer Genome Atlas Research Network, Kandoth C, Schultz N, et al. Integrated genomic characterization of endometrial carcinoma[J]. Nature, 2013, 497 (7447) : 67–73. DOI:10.1038/nature12113 |
| [26] | Jee BA, Lim H, Kwon SM, et al. Molecular classification of basal cell carcinoma of skin by gene expression profiling[J]. Mol Carcinog, 2015, 54 (12) : 1605–12. DOI:10.1002/mc.22233 |
| [27] | Wang Y, Waters J, Leung ML, et al. Clonal evolution in breast cancer revealed by single nucleus genome sequencing[J]. Nature, 2014, 512 (7513) : 155–60. DOI:10.1038/nature13600 |
| [28] | Sottoriva A, Kang H, Ma Z, et al. A big bang model of human colorectal tumor growth[J]. Nat Genet, 2015, 47 (3) : 209–16. DOI:10.1038/ng.3214 |
| [29] | Roychowdhury S, Chinnaiyan AM. Translating cancer genomes and transcriptomes for precision oncology[J]. CA Cancer J Clin, 2016, 66 (1) : 75–88. DOI:10.3322/caac.v66.1 |
| [30] | Koh J, Allbritton NL, Sosa JA. Single-cell approaches for molecular classification of endocrine tumors[J]. Curr Opin Oncol, 2016, 28 (1) : 43–9. DOI:10.1097/CCO.0000000000000246 |
| [31] | Workman P, Al-Lazikani B, Clarke PA. Genome-based cancer therapeutics: targets,kinase drug resistance and future strategies for precision oncology[J]. Curr Opin Pharmacol, 2013, 13 (4) : 486–96. DOI:10.1016/j.coph.2013.06.004 |
 2016, Vol. 43
2016, Vol. 43