文章信息
- miR-23a靶向NID2通过Notch通路调控肺癌细胞的侵袭和迁移
- miR-23a Targeting NID2 Regulates Invasion and Migration of Lung Cancer Cells Through Notch Pathway
- 肿瘤防治研究, 2017, 44(6): 381-386
- Cancer Research on Prevention and Treatment, 2017, 44(6): 381-386
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2017.17.0035
- 收稿日期: 2017-01-11
- 修回日期: 2017-03-08
肺癌是呼吸系统发病率最高最常见的原发性肿瘤之一,危险因素众多,国内发病率约为26.7/10万,大约占所有呼吸系统肿瘤的68%[1-2]。由于早期肺癌患者症状缺乏特异性,多数患者确诊时已进入中晚期,并且伴有多处转移。虽然近些年手术、化疗、放疗、靶向治疗等综合治疗手段不断进步,但肺癌患者尤其是晚期肺癌患者的5年生存率仍然很低[3]。肺癌的术后复发率也比较高,且复发后患者的平均生存期更加短,甚至只有3~6月[4]。随着对肺癌发病相关基因和分子机制的不断认识,寻找调控肺癌细胞发生和发展的分子靶点已经成为肺癌治疗和改善预后的新研究方向。
miR-23a在多种类型的肿瘤中表达失调,例如胃癌﹑肠癌﹑乳腺癌[5-7],其通过与靶基因的3'UTR结合抑制mRNA的后续翻译。有研究发现miR-23a的潜在靶基因有PPP2R5E、IRF1、MTMS1、APAF1、RUNX1等,可以参与调控多种肿瘤生物学行为[8]。
Nidogen-2(NID2)是Nidogen家族成员,是构成基底膜的主要成分之一,对维持基底膜的稳定性有重要意义[9]。Nidogen家族成员在哺乳动物中广泛存在并且呈高度保守序列,目前已经发现的家族成员包括nidogen-1和nidogen-2,两者有46%的同源性[10]。基底膜作为细胞外基质的主要成分,是肿瘤侵袭转移过程中必须穿过的物理屏障,也是肿瘤侵袭转移过程中必须破坏的,因此Nidogen家族与肿瘤侵袭转移密切相关。
Notch信号是一个在进化过程中高度保守的信号通路,广泛存在于无脊椎动物和脊椎动物的不同物种中,通过影响下游靶基因的表达水平,从而调控生物体细胞的增殖、迁移和分化[11]。Notch信号通路的异常激活或结构性活化与多种组织恶性肿瘤的发病有关。有文献报道,NID2调控机制可能与Notch信号通路相关[12]。
本研究通过检测miR-23a在肺癌细胞中表达情况,然后检测过表达miR-23a后NID2蛋白水平的变化和肺癌细胞生物学行为的改变,推测miR-23a通过NID2对肺癌细胞的影响,最后通过检测Notch信号蛋白水平变化验证猜想是否正确。
1 资料与方法 1.1 临床标本采集及处理收集2015年3月—2016年5月在南阳市中心医院收治的40例肺癌患者肺癌组织及相对应的癌旁正常组织。患者年龄39~68岁,中位年龄52岁,平均年龄(53.23±4.95)岁。其中37例鳞癌,3例腺癌。根据肺癌TNM分期标准,Ⅰ期5例,Ⅱ期8例,Ⅲ期24例,Ⅳ期3例。男性34例,女性6例。所有患者术前均未行化疗或放疗,全部患者术后病理分期均经两名副高以上病理科医师共同阅片确定。肿瘤组织离体后迅速投入液氮中保存。患者及家属签署知情同意书。
1.2 细胞株与主要试剂人肺癌A549、H1650、DMS53、H889细胞购自武汉大学中国典型培养物保藏中心。细胞培养条件:含10%胎牛血清的RPMI 1640培养液,37℃、5%CO2条件下培养。胎牛血清、RPMI 1640培养液均购自美国Gibco公司。NID2、NICD和hes1兔单克隆抗体均购自美国Abcam(ab14513、ab27526、ab71559)。Transwell小室购自美国Millipore公司,Matrigel购自美国BD公司。miR-23a mimics、NID沉默、LV5-NID2及对照慢病毒购自上海吉凯制药技术有限公司。miR-23a、NID及Notch qPCR引物以及RNA提取试剂盒、反转录试剂盒、PCR试剂盒均购自广州复能基因有限公司。
1.3 免疫组织化学法将石蜡包埋好的组织行4 μm连续切片,60℃烤箱中烘烤3~4 h;二甲苯溶液脱蜡3次,每次15 min,梯度酒精(100%~80%)逐级水化,每级3 min;PBS液洗3次,每次3 min;将切片置入pH6.0柠檬酸盐缓冲液高压修复2 min 15 s后,置于PBS液中浸洗33次,每次3 min;3%过氧化氢甲醇溶液封闭10 min,PBS液洗3次,每次3 min;滴加非免疫山羊血清后静置15 min后滴加一抗,4℃孵育过夜;37℃下复温30 min,PBS+1‰Tween20液浸洗3次,每次3 min;室温下滴加二抗孵育15 min,PBS+1‰Tween20液浸洗3次,每次3 min;滴加二氨基联苯胺(DAB),显微镜下观察染色,及时终止反应;苏木精对比染色,流水冲洗,氨水碱化;梯度酒精(80%~100%)逐级脱水,每级3 min;二甲苯冲片3次,每次3 min;风干二甲苯,用中性树胶进行封片。
1.4 qPCR法检测不同肺癌细胞中miR-23a、NID2、NICD和hes1的表达水平按照TRIzol操作说明提取组细胞总RNA,使用紫外分光光度计检测核酸的浓度和纯度。以DEPC将各组总RNA稀释调整为同一浓度,按步骤加入试剂,反应完成后将cDNA于-20℃储存备用。根据美国Sigma公司荧光定量试剂盒(GoTaq@qPCRMaster Mix)操作说明配制反应体系,每组反应体系的体积为20 μl,并各设置3个复孔,每组样本cDNA均需行目的RNA和内参基因GAPDH荧光定量PCR扩增。根据2-ΔΔCt法计算检测基因反应条件为:37℃ 15 min,98℃ 5 min。后根据PCR试剂盒说明书进行PCR反应。获得数据以RQ=2-ΔΔCt计算mRNA表达量。
1.5 肺癌H1650细胞中NICD和hes1蛋白检测分别提取miR-23a-mimic组、LV5-NID2组和对照组(NC组)的细胞蛋白。配制10%SDS-PAGE,每孔加入25 μg蛋白样品。使用湿转法电转至PVDF膜,5%脱脂奶粉封闭2 h,1:2000 TBST稀释一抗(羊抗人NICD和hes1单克隆抗体),4℃过夜;加入辣根过氧化酶物标记兔抗羊IgG(1:5000)稀释,室温孵育2 h;ECL发光。实验重复3次。
1.6 Transwell侵袭实验检测肺癌H1650细胞侵袭能力所有试剂及器材均于冰上预冷,将Transwell小室置于24孔板内,将Transwell小室内膜均匀涂抹Matrigel胶50 μl(0.2 μg/ul),37℃孵育15 min,使胶凝固;将肺癌H1650细胞分为对照组(NC组)和miR-23a-mimic组,消化、离心、计数细胞后,按照2.5×104个/毫升用无血清培养液稀释细胞,制成细胞悬液;按照每孔200 μl,将细胞悬液加入Transwell上室,同时在Transwell下室加入10%FBS+培养液600 μl,放入37℃培养箱培养;甲醛固定,结晶紫染色15 min,然后用棉签轻轻擦拭内膜上的细胞。显微镜下计数,计数5个高倍视野(×40)下穿过滤膜的细胞数。实验重复3次。
1.7 荧光素酶活性检测将荧光素酶报告载体与miR-23a-mimic共转染H1650细胞。以转染pRL-TK作为标准内质控。转染36 h后,收获细胞。按Promega公司荧光素酶活性检测试剂盒说明书检测H1650细胞荧光素酶活性。计算相对荧光素酶活性=萤火虫荧光素酶活性值/海肾荧光素酶活性值。
1.8 划痕实验检测肺癌H1650细胞迁移能力划痕实验:将肺癌H1650细胞分为对照组(NC组)和miR-23a-mimic组分别接种于6孔板,待细胞生长融合度在90%时,用200 μl消毒枪头从上而下划线,并在显微镜下观察,测量划痕的初始距离(0 time);在24、48和72 h后,测量划痕的距离,并拍照,计算细胞的迁移率。迁移率=(迁移距离(24、48和72 h)-迁移距离(0 h))/迁移距离(0 h)×100%。实验重复3次。
1.9 统计学方法采用SPSS20.0软件进行统计分析,计量数据以(x± s)表示,两组间均数比较采用t检验,以P < 0.05为差异具有统计学意义。
2 结果 2.1 NID2在肺癌和癌旁正常组织中的表达免疫组织化学结果显示:NID2主要定位在细胞质。在肺癌组织中NID2的表达明显高于正常肺组织,两者比较差异具有统计学意义(P=0.0198),见图 1。
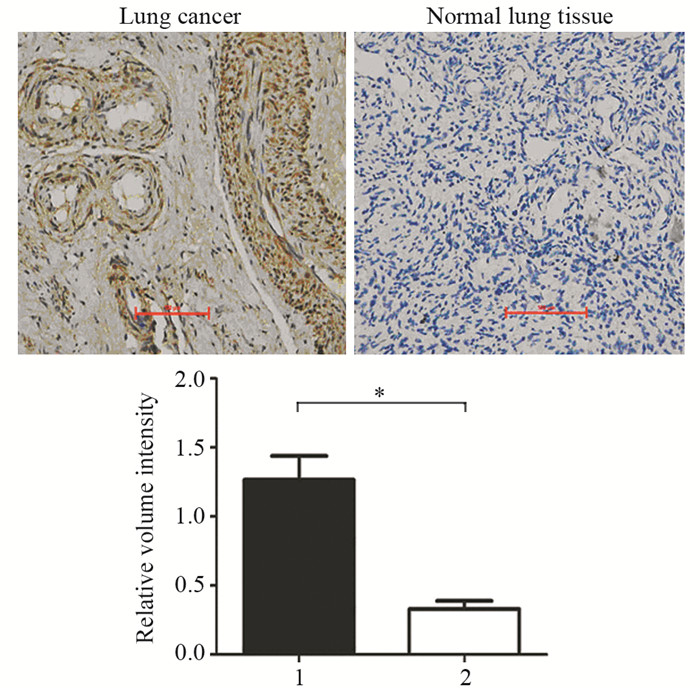
|
| 1: lung cancer; 2: normal lung tissue; *: P=0.0198 图 1 免疫组织化学检测NID2蛋白在肺癌和癌旁正常肺组织的表达(DAB ×40) Figure 1 NID2 expression in lung cancer and adjacent normal lung tissues detected using immunohistochemical staining (DAB ×40) |
Western blot实验结果显示:相比A549、H1650、DMS53、H889细胞株,NID2蛋白在H1650细胞株中表达水平最高,所以后续实验我们选取H1650作为实验细胞株,见图 2。
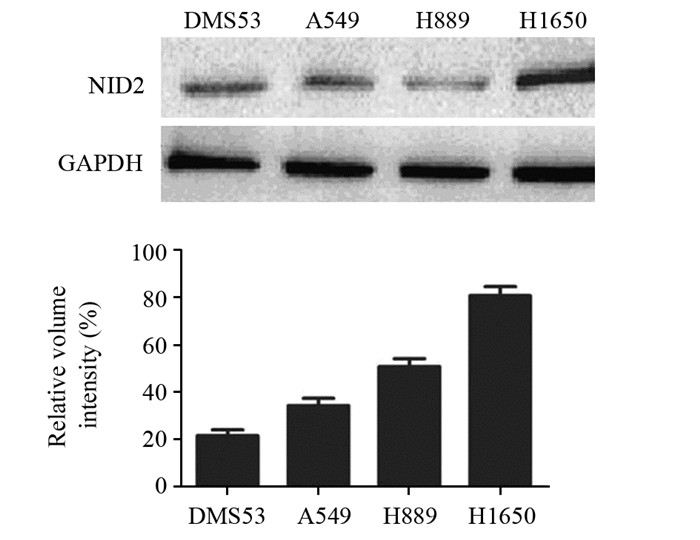
|
| The expression level of NID2 was the highest in H1650 cell line 图 2 NID2蛋白在不同肺癌细胞株中的表达 Figure 2 NID2 protein expression in different lung cell lines |
许多研究表明,NID2与miRNA在恶性肿瘤细胞中可相互作用,通过生物信息学进行预测(TargetScan),与NID2相关的miRNA中,NID2是miR-23a的直接靶点。为了进一步证实NID2是miR-23a下游的靶向基因,我们使用双荧光素酶报告系统检测H1650细胞发现,结果提示,NID2是miR-23a的直接靶点,NID2可被上游miR-23a调控其表达,见图 3。
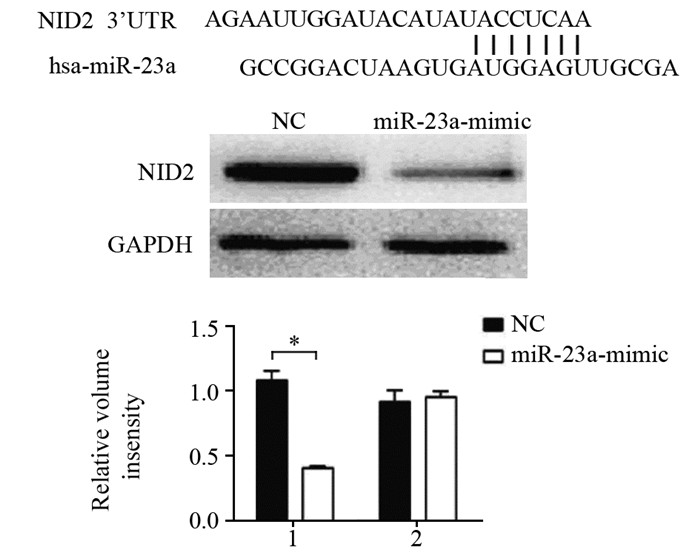
|
| 1: preporter-miR-NID2-3'UTR; 2: preporter-miR-NID2-3'UTR mutant; *: P=0.0225 图 3 双荧光素酶验证NID2是miR-23a下游的靶向基因 Figure 3 NID2 was a downstream target of miR-23a verified by dual luciferase |
Transwell实验结果显示:miR-23a-mimic组穿透过基质胶的细胞数量明显少于NC组(48.75±5.76)% vs.(226.25±7.75)%,差异有统计学意义(P=0.0086),见图 4。结果表明,过表达miR-23a的表达后可以有效抑制肺癌H1650细胞的侵袭能力。
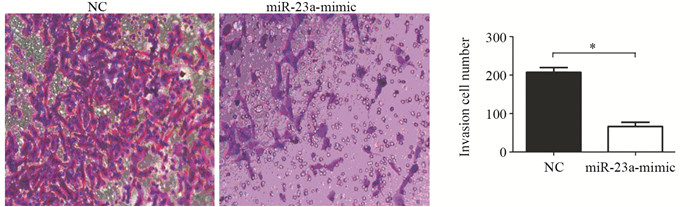
|
| *: P=0.0086 图 4 Transwell侵袭实验检测过表达miR-23a对肺癌H1650细胞侵袭能力的影响 Figure 4 Effect of miR-23a overexpression on invasion ability of H1650 cells detected by Transwell invasion assays |
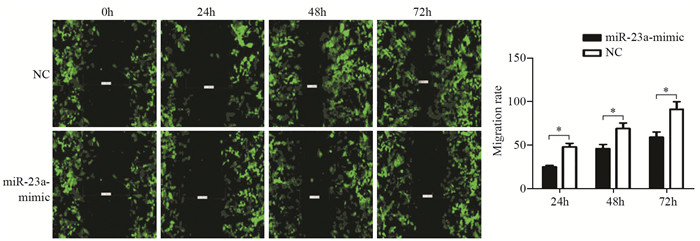
划痕实验结果显示:在显微镜下观察,两组细胞任意三个部位的划痕宽度,迁移率=[D(t=24, 48和72 h)-D(t=0h)]/D(t=0h)。与对照组(NC组)相比,miR-23a-mimic组迁移比率明显降低,在24、48和72 h结果分别是(28.67±6.32)% vs.(45.75±4.34)%, P < 0.05;(48.45±6.35)% vs.(67.64±6.85)%, P < 0.05;(56.84±2.75)% vs.(87.12±9.09)%, P < 0.05,差异有统计学意义,见图 5。结果表明,过表达miR-23a后可以有效抑制肺癌H1650细胞的迁移能力。
|
| *: all P=0.0119 图 5 划痕实验检测过表达miR-23a对肺癌细胞H1650转移能力的影响 Figure 5 Effect of miR-23a overexpression on migration ability of H1650 cells detected by wound healing assays |
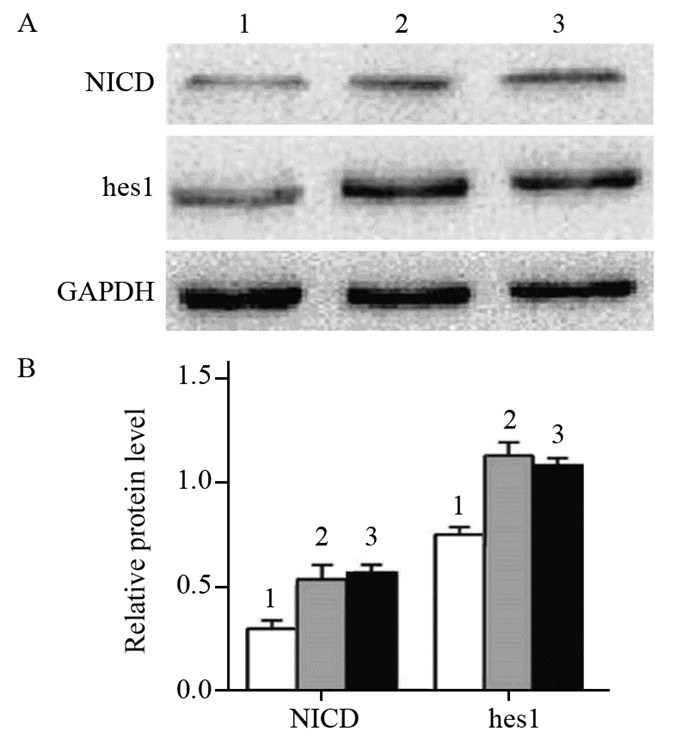
结果发现,和对照组(NC)相比,肺癌细胞株H1650转染miR-23a-mimic后,NICD和hes1的蛋白水平明显受到抑制,见图 6A,同时再加入LV5-NID2后,结果发现(miR-23a-mimic vs. miR-23a-mimic+LV5-NID2 vs. NC):NICD和hes1的表达水平在一定程度上得到恢复(NICD(0.28±0.06)vs.(0.56±0.09)vs.(0.59±0.07),P < 0.05; hes1(0.59±0.09)vs.(1.36±0.25)vs.(1.10±0.17),P < 0.05),见图 6B。表明miR-23a可以通过NID2调控Notch通路的激活情况。
|
| 1: miR-23a-mimic; 2: miR-23a-mimic+LV5-NID2; 3: NC 图 6 过表达miR-23a和NID2对Notch通路蛋白的调控作用(A)及对NICD和hes1蛋白表达的影响(B) Figure 6 Regulation of miR-23a and NID2 overexpression on Notch pathway proteins(A) and their effect on NICD and hes1 protein expression(B) |
目前关于miRNA的研究包括生物信息学预测以及生物芯片筛选,但两者都需要通过后续实验验证[13-14]。由于miRNA的作用机制非常复杂,比如调控蛋白的稳定性或者定位[15]和DNA以及RNA之间相互作用调控基因表达[16-17]、形成mRNA调控基因的表达[18]、调控mRNA前体的拼接、作为内源性竞争性RNA发挥调控作用[19]等。Qin等[20]研究发现miR-23a在卵巢颗粒肿瘤中通过调控XIAP的表达促进细胞凋亡,并且通过荧光素酶报告基因活性分析提示XIAP是miR-23a的靶基因。Liu等[21]研究发现miR-23a下调PPP2R5E的表达,促进胃癌细胞的生存,抑制胃癌细胞凋亡,PPP2R5E被认为是miR-23a新的靶基因,在调控肺癌细胞增殖与凋亡方面发挥了重要的作用。鉴于以上miR-23a的潜在靶点,让我们联想到miR-23a可能参与调控细胞凋亡的过程。后续需要探讨miR-23a在自噬与凋亡的平衡中的具体作用机制。
Wang等[22]在卵巢癌组织中发现,随着其恶性程度升高,NID2的表达也是随之降低的。推测NID2在恶性肿瘤中侵袭转移过程中可能会对基底膜造成不同程度的破坏,调控肿瘤细胞的侵袭和迁移,NID2有可能成为早期诊断部分恶性肿瘤的重要预测分子。本研究通过检测miR-23a和NID2的关系,明确miR-23a可以调控NID2的表达水平,过表达miR-23a后,NID2水平相应下调,肺癌细胞的侵袭迁移能力受到明显抑制。这与在其他肿瘤中的表达情况相一致。
Notch信号通路是一种具有高度保守型的信号通路,在肿瘤细胞的发生发展中发挥重要作用[23]。通常认为Notch信号通路与生物个体的生长发育及新生组织的分化再生行为相关[24]。Notch信号通路的下游基因很多包括:hes1、NICD等。Notch信号通路的经典激活方式是经过Notch受体与其相应的配体相结合后,通过胞内水解酶的作用,分解释放NICD小段,NICD进入细胞核作用于下游基因靶点,激活下游相关生物学效应[25]。Shih报道在胶质瘤中NID2及同型分子可以激活Notch信号通路中的活化蛋白,继而影响胶质瘤的表达和转移能力[26]。还有文献报告在胰腺囊性瘤中NID2的表达和Notch信号通路的激活相关,具体分子机制还需后续研究[27]。本研究通过Western blot检测沉默NID2后Notch信号通路中关键蛋白的表达水平,发现NICD和hes1的表达水平相应下调,所以推测miR-23a可以靶向调控NID2的表达通过Notch信号通路的激活调节肺癌细胞的生物学行为。由于本实验只验证了H1650一种肺癌细胞株中miR-23a和NID2的相互关系,对其他肺癌细胞系的预测和推测存在一定局限性,后续可以逐步完善其他肺癌细胞系的检测,对本研究结果进行佐证。
综上所述,miR-23a的过表达可以抑制肺癌的迁移和侵袭,通过前面实验的结果,发现miR-23a在肺癌细胞中表达下调,NID2在肺癌细胞中上调。我们推测,在生理状态下,这个miR-23a/NID2调节环路维持表达情况处于低表达的水平,如果在病理状态下,一旦这个环路平衡被打破,可能通过激活Notch信号通路导致肺癌的发生或进展。本实验阐明了miR-23a/NID2/Notch在肺癌中发生发展中的作用,为进一步研究miR-23a和NID2在肺癌中的作用提供了理论基础。
| [1] | Roth JA, Atkinson EN, Fossella F, et al. Long-term follow-up of patients enrolled in a randomized trial comparing perioperative chemotherapy and surgery with surgery alone in resectable stage ⅢA non-small-cell lung cancer[J].Lung cancer, 1998, 21(1): 1–6. DOI:10.1016/S0169-5002(98)00046-4 |
| [2] | Zatloukal P, Petruzelka L, Zemanova M, et al. Concurrent versus sequential chemoradiotherapy with cisplatin and vinorelbine in locally advanced non-small cell lung cancer: a randomized study[J].Lung cancer, 2004, 46(1): 87–98. DOI:10.1016/j.lungcan.2004.03.004 |
| [3] | Lortet-Tieulent J, Soerjomataram I, Ferlay J, et al. International trends in lung cancer incidence by histological subtype: adenocarcinoma stabilizing in men but still increasing in women[J].Lung cancer, 2014, 84(1): 13–22. DOI:10.1016/j.lungcan.2014.01.009 |
| [4] | Markou A, Sourvinou I, Vorkas PA, et al. Clinical evaluation of microRNA expression profiling in non small cell lung cancer[J].Lung cancer, 2013, 81(3): 388–96. DOI:10.1016/j.lungcan.2013.05.007 |
| [5] | Yang Z, Wang X, Bai R, et al. miR-23a promotes IKKα expression but suppresses ST7L expression to contribute to the malignancy of epithelial ovarian cancer cells[J].Br J Cancer, 2016, 115(6): 731–40. DOI:10.1038/bjc.2016.244 |
| [6] | Croce CM, Calin GA, Volinia S. Methods for diagnosing colon cancer using MicroRNA signatures: U.S. Patent 9, 017, 940[P]. 2015-04-28. |
| [7] | Ma S, Liu M, Xu Z, et al. A double feedback loop mediated by microRNA-23a/27a/24-2 regulates M1 versus M2 macrophage polarization and thus regulates cancer progression[J].Oncotarget, 2016, 7(12): 13502–19. |
| [8] | Li X, Liu X, Xu W, et al. c-MYC-regulated miR-23a/24-2/27a cluster promotes mammary carcinoma cell invasion and hepatic metastasis by targeting Sprouty2[J].J Biol Chem, 2013, 288(25): 18121–33. DOI:10.1074/jbc.M113.478560 |
| [9] | Scher M, Trama J. Detecting DNA methylation of BCL2, CDKN2A and NID2 genes to predict bladder cancer in humans: U.S. Patent 9, 096, 905[P]. 2015-08-04. |
| [10] | Scher MB, Elbaum MB, Mogilevkin Y, et al. Detecting DNA methylation of the BCL2, CDKN2A and NID2 genes in urine using a nested methylation specific polymerase chain reaction assay to predict bladder cancer[J].J Urol, 2012, 188(6): 2101–7. DOI:10.1016/j.juro.2012.08.015 |
| [11] | Hadland BK, Varnum-Finney B, Moon RT, et al. Notch signaling by either Notch1 or Notch2 mediates expansion of AGM-Derived long-term HSC populations in vitro[J].Blood, 2014, 124(21): 2897. |
| [12] | Xue YK, Tan J, Dou DW, et al. Effect of Kruppel-like factor 4 on Notch pathway in hepatic stellate cells[J].J Huazhong Univ Sci Technolog Med Sci, 2016, 36(6): 811–6. |
| [13] | Frampton GM, Fichtenholtz A, Otto GA, et al. Development and validation of a clinical cancer genomic profiling test based on massively parallel DNA sequencing[J].Nat Biotechnol, 2013, 31(11): 1023–31. DOI:10.1038/nbt.2696 |
| [14] | Köttgen A, Albrecht E, Teumer A, et al. Genome-wide association analyses identify 18 new loci associated with serum urate concentrations[J].Nat Genet, 2013, 45(2): 145–54. |
| [15] | Martin-Belmonte F, Perez-Moreno M. Epithelial cell polarity, stem cells and cancer[J].Nat Rev Cancer, 2012, 12(1): 23–38. |
| [16] | Jeselsohn R, Yelensky R, Buchwalter G, et al. Emergence of constitutively active estrogen receptor-α mutations in pretreated advanced estrogen receptor-positive breast cancer[J].Clin Cancer Res, 2014, 20(7): 1757–67. DOI:10.1158/1078-0432.CCR-13-2332 |
| [17] | Kostović I, Jovanov-Milošević N, Radoš M, et al. Perinatal and early postnatal reorganization of the subplate and related cellular compartments in the human cerebral wall as revealed by histological and MRI approaches1[J].Brain Struct Funct, 2014, 219(1): 231–53. DOI:10.1007/s00429-012-0496-0 |
| [18] | Pietra G, Manzini C, Rivara S, et al. Melanoma cells inhibit natural killer cell function by modulating the expression of activating receptors and cytolytic activity[J].Cancer Res, 2012, 72(6): 1407–15. DOI:10.1158/0008-5472.CAN-11-2544 |
| [19] | Marinić M, Aktas T, Ruf S, et al. An integrated holo-enhancer unit defines tissue and gene specificity of the Fgf8 regulatory landscape[J].Dev Cell, 2013, 24(5): 530–42. DOI:10.1016/j.devcel.2013.01.025 |
| [20] | Qin LX, Levine DA. Study design and data analysis considerations for the discovery of prognostic molecular biomarkers: a case study of progression free survival in advanced serous ovarian cancer[J].BMC Med Genomics, 2016, 9(1): 27. DOI:10.1186/s12920-016-0187-4 |
| [21] | Liu X, Liu Q, Fan Y, et al. Downregulation of PPP2R5E expression by miR-23a suppresses apoptosis to facilitate the growth of gastric cancer cells[J].FEBS Lett, 2014, 588(17): 3160–9. DOI:10.1016/j.febslet.2014.05.068 |
| [22] | Wang ZQ, Bachvarova M, Morin C, et al. Role of the polypeptide N-acetylgalactosaminyltransferase 3 in ovarian cancer progression: possible implications in abnormal mucin O-glycosylation[J].Oncotarget, 2014, 5(2): 544–60. DOI:10.18632/oncotarget |
| [23] | Guruharsha KG, Kankel MW, Artavanis-Tsakonas S. The Notch signalling system: recent insights into the complexity of a conserved pathway[J].Nat Rev Genet, 2012, 13(9): 654–66. DOI:10.1038/nrg3272 |
| [24] | Hori K, Sen A, Artavanis-Tsakonas S. Notch signaling at a glance[J].J Cell Sci, 2013, 126(10): 2135–40. DOI:10.1242/jcs.127308 |
| [25] | Bolós V, Mira E, Martínez-Poveda B, et al. Notch activation stimulates migration of breast cancer cells and promotes tumor growth[J].Breast Cancer Res, 2013, 15(4): R54. DOI:10.1186/bcr3447 |
| [26] | Shih AH, Holland EC. Notch signaling enhances nestin expression in gliomas[J].Neoplasia, 2006, 8(12): 1072–82. DOI:10.1593/neo.06526 |
| [27] | Fukushima N, Fukayama M. Mucinous cystic neoplasms of the pancreas: pathology and molecular genetics[J].J Hepatobiliary Pancreatic Surg, 2007, 14(3): 238–42. DOI:10.1007/s00534-006-1168-3 |
 2017, Vol. 44
2017, Vol. 44


