| 植物络合素合酶及其基因研究进展 |
2. 剑桥大学植物系,英国 剑桥 CB23EA;
3. 邵阳市烟草专卖局,湖南 邵阳 422000
2. Department of Plant Sciences, University of Cambridge, Cambridge CB23EA, United Kingdom;
3. Shaoyang Tobacco Monopoly Bureau, Shaoyang 422000, Hunan, China
植物在生活期从环境中吸收和利用各种必需金属元素的同时,会不可避免地摄入某些有害重金属元素。经过长期的自然选择与进化,植物拥有了一套复杂而精细的应对有害重金属的策略,其中,通过合成配体小分子化合物与之络合并固定到类似于液泡等细胞特定区域的方式,在植物保持体内金属离子平衡的过程中起着关键作用[1]。在多种植物及真菌中发现的植物络合素(phytochelatin, PC)是一类由酶促反应合成的、富含半胱氨酸(cysteine, Cys)的多肽,被认为是植物解除重金属毒害的最重要的小分子化合物之一。植物络合素具有(γ-Glu-Cys)n-X (其中n为2~11,X可以缺失或以Gly、β-Ala、Ser、Clu和Cln这5种形式存在)的一般结构通式,能以胱氨酸(cystine)的巯基与重金属发生络合作用而实现解毒[1-3]。植物络合素在植物或者酵母体内以谷胱甘肽(glutathione, GSH)为前体,由植物络合素合酶(phytochelatin synthase, PCS, EC 2.3.2.15)催化合成。自GRILL等[4]于1989年首次从膀胱麦瓶草中鉴定到植物络合素合酶的活性以来,该酶的同源物和基因陆续在数十种物种中被鉴定发掘,极大地丰富了人类对PCS功能的认知。结合前人的研究成果,本文从PCS的双功能酶特性、基因表达和酶学特征、物种分布和亲缘关系、催化活性中心及激活模式5个方面作简要评述。
1 PCS双功能酶特性一直以来,人们普遍认为PCS只具有以谷胱甘肽为底物催化合成植物络合素的作用[5]。PCS催化2个GSH分子间的N-基末端和C-基末端生成由α-酰胺键连接的(γ-Glu-Cys)2-Gly和另一个Gly分子,在后续的过程中催化(γ-Glu-Cys)n-1-Gly分子连接GSH中的γ-Glu-Cys部分形成PCn(n=2~11)寡聚体,同时生成1个Gly分子;在这个过程中,PCS行使γ-谷氨酰半胱氨酸二肽转移酶(transpeptidase)的功能(图 1) [2, 5-6]。VATAMANIUK等[3]于1999年将从拟南芥中分离的PCS1酶基因转移到酿酒酵母中,其酶蛋白作为γ-谷氨酰半胱氨酸二肽转移酶催化合成PC,然后,植物络合素合成酶的γ-谷氨酰半胱氨酸二肽转移酶作用被大量报道[3, 7-11]。几乎在所有植物中均发现了植物络合素合成酶基因的存在,即使在偏远无污染地区的植物中也一样存在该酶蛋白的表达。科学家们对于植物在无需解毒的情况下也“愿意浪费”如此多的能量在该酶参与的通路中产生疑虑,促使了植物络合素合成酶肽酶作用的发现。在此酶促反应中,植物络合素合酶起着植物特有肽酶的作用,从谷胱甘肽配合物(GS-conjugates)中剪切掉羧基端的甘氨酸残基,从而得到相应的γ-谷氨酰半胱氨酸结合物(γ-ECconjugate)。BLUM等[12]在2007年用异源生物的双满(xenobiotic bimane,巯基结合荧光剂)追踪谷胱甘肽配合物在生物体内的转化,发现拟南芥PCS1基因缺失系中γ-EC-bimane缺失,该突变体恢复系中相应的γ-EC-bimane得到恢复,很好地支持了该酶具有肽酶副功能的猜想[5]。
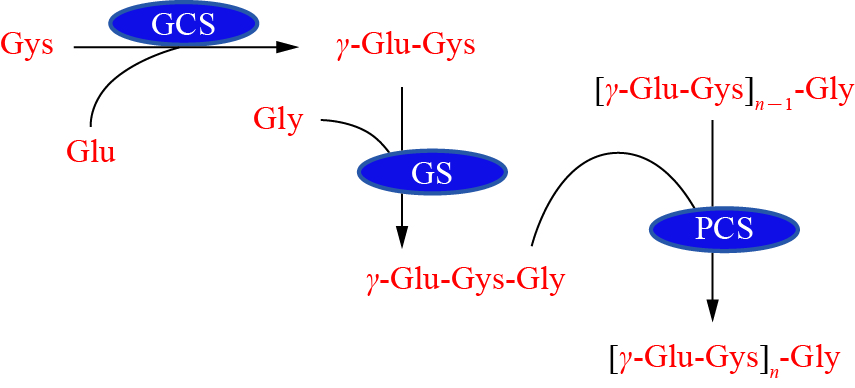 |
| GCS、GS和PCS分别表示γ-谷胱酰半胱氨酸合成酶、谷胱甘肽合成酶和植物络合素合酶;n=2~11。 GCS, GS and PCS represent γ-glutamylcysteine synthetase, glutathione synthetase and phytochelatin synthase, respectively; n = 2-11. 图1 植物中植物络合素的生物合成[2, 5-6] Fig. 1 Biosynthesis of phytochelatin in plants[2, 5-6] |
早期的研究表明,PCS基因的表达是组成型的,在PC合成过程中不存在转录水平的调控[1]。AtPCS1表达产物的实时荧光定量聚合酶链式反应和RNA印迹法(Northern blotting)结果均表明,Cd2+ (镉离子)存在与否对其转录水平没有明显影响,且在不同组织部位(叶或根)中的表达也没有明显差异[3],相同的结果也在番茄中被发现[13]。然而,后期的一些研究表明,PCS基因的表达也可能受Cd2+的轻微差异化调控。LEE等[14]将AtPCS1启动子与GUS基因相连后,检测到5天龄拟南芥经50 mmol/L Cd2+处理5 d后的GUS活性比对照提高了1倍,后期差异逐渐消失,表明AtPCS1的表达在植物发育早期受Cd2+正调控;冯保民等[1]也初步认为在大蒜中PCS基因表达可能受Cd2+和As3+(铯离子)的影响而上调,但更深入的研究未见报道。最初翻译合成的PCS没有催化活性,其催化活性受诱导PC合成的金属离子激活,但不同金属离子激活效率差异很大[4]。Cd2+被认为是最强的激活剂,其对PCS的激活效率是Pb2+(铅离子)的50倍,其次是Ag+(银离子)、Bi3+(铋离子)、Pb2+、Zn2+(锌离子)、Cu2+(铜离子)、Hg+(汞离子)、AuCl4- (四氯合金络阴离子)等,而Na+(钠离子)、K+(钾离子)、Ca2+ (钙离子)、Mg2+(镁离子)等则对PCS没有激活作用[1, 4]。PCS的激活特性起着调节植物体内PC水平的作用,该催化合成是一个自身反馈环(selffeedback loop):一旦植物体内重金属离子含量超过阈值浓度,PCS将被激活,从而产生更多的PC与重金属进行络合,降低细胞内重金属离子水平;当重金属离子浓度降低后便失去了激活PCS的能力,进而减少了PC的合成。有报道表明,当Cd2+浓度以低于2:1的摩尔比结合PC中的Cys残基时,PCS的催化活性趋于停止[1, 5]。
3 PCS物种分布及亲缘关系最早普遍认为植物络合素合酶只存在于植物中[4],之后在酵母、小麦和拟南芥中的植物络合素合酶基因几乎同时被克隆分离出来,颠覆了早期人们对PCS的认知[5];后来从藻青菌(Nostoc sp. PCC 7120) [15]、锡兰钩口线虫(Caenorhabditis elegans) [16]和小眼虫(Euglena gracilis) [17]中也相继分离鉴定出PCS,可见PCS可能是广泛存在于自然界中的,不过到目前为止还未有从高等动物中分离出PCS的相关报道。自膀胱麦瓶草PCS被报道以来,已有近100种PCS或相关基因被预测或鉴定出来,其中具有代表性和研究较为深入的物种有拟南芥[3, 7, 18]、小麦[11, 19-20]、大豆[21]、土豆[22]、烟草[10, 23]和水稻[24-26]等(表 1)。这些PCS具有同源性N-基端高于C-基端的特点,N、C两端分别具有1个保守结构域,其中N-端保守结构域为该酶的功能中心(pfam05023),C-端结构域含有1个功能未知的保守结构域,被认为可能具有增强酶活性的功能(pfam09328),C-端保守结构域的保守性低于N-端保守结构域[27]。大部分物种的PCS均由长度为414~505 aa (氨基酸)组成,其中植物均同时含有PCS必备的N-基端保守结构域(pfam05023)和C-基端保守结构域(pfam09328)。在NCBI基因库中发现一个长度为226 aa的以OsPCS2b命名的短片段,经比对分析,认为可能是由OsPCS2a基因片段丢失造成的,需要进一步核实。粟酒裂殖酵母(Schizosaccharomyces pombe) [28]、锡兰钩口线虫(C. elegans) [16]和小眼虫(E. gracilis) [17]具有C-端氨基酸残基链,但没发现PCS共有的C-端保守结构域(pfam09328);藻青菌(Nostoc sp. PCC 7120)只单独含有一个长度为242 aa的N-端催化中心[15]。由图 2可知,植物间PCS具有较高的相似性,同源性较高。拟南芥中2个旁系同源蛋白AtPCS1和AtPCS2的相似性达74%,水稻中2个旁系同源蛋白OsPCS和OsPCS2a的相似性达69.5%,大豆、田菁属、拟南芥和芥菜直系同源蛋白GmhPCS1、SrPCS、BjPCS1和AtPCS1/AtPCS2的相似性为65%~92%,烟草和土豆直系同源PCS蛋白的相似性高达91%,莲藕、金鱼藻、水稻和小麦直系同源蛋白间的相似性为52%~78%;而酵母、线虫、小眼虫和藻青菌直系同源PCS间的相似性均较低,为26%~30%,且它们与植物类PCS的相似性也相对较低,这可能是这些生物体抗重金属能力低于植物的原因之一。可见,PCS在植物对周围环境的适应中可能有着更加复杂和重要的意义。
| 表1 PCS在物种中的鉴定和克隆 Table 1 Identification and cloning of PCS in speciess |
 |
| 点击放大 |
 |
| 左边亲缘关系树由ClustalX 2.0和MEGA 6.06软件通过多序列比对后用邻位连接法构建,其中红色数字表示频率,黑色数字表示分支长度,分类名参照表 1;右边保守位点借助NCBI中的“Web CD-Search Tool”获得[38],其中绿色条表示N-端保守酶功能结构区域(pfam05023),蓝色和黑色条表示C-端保守功能未知结构域(pfam09328),背景灰色条表示相关PCS蛋白链,红色矩形虚线框表示非植物类生物PCS缺少C-端保守结构域。 Polygenetic tree on the left was generated by softwares ClustalX 2.0 and MEGA 6.06 via multiple sequence alignment and neighbor jointing method, in which red numbers show statistic frequency, black numbers show branch length, and taxon names refer to the Table 1. Conserved domain sites on the right were obtained by Web CD-Search Tool in NCBI[38], in which green bars show pfam05023 located on N-terminal of conservative enzyme' s functional structure area, and blue and black bars show pfam09328 located on C-terminal of conservative enzyme' s functional unknown area; grey bars show PCS protein chains, and red dash rectangle shows organisms lacking PCS C-terminal conserved domain. 图2 代表性物种中PCS亲缘关系树和特征保守结构域 Fig. 2 Polygenetic tree and conserved domains of PCS in representative species |
研究的深入为N-基端是PCS的催化中心提供了有力证据:1) N-基端保守性明显高于C-基端;2)藻青菌中只含有N-基端片段,然而仍然具有PCS酶活性(尽管活性较弱);3)拟南芥突变体cad1-5等位基因编码的蛋白终止于280氨基酸残基处,仅为野生型蛋白长度的3/5,其N-基端未发生突变,仍然表现出一定的Cd2+抗性,可见突变产物仍旧保留了部分催化活性(合成PCS的能力为野生型的33%);4)拟南芥中N-基端发生突变的突变体cad1-1、cad1-3和cad1-4对Cd2+的敏感性明显比cad1-5强[4, 7]。藻青菌PCS为只含有N-基端催化中心的、长度为242 aa的短结构,为解析PCS蛋白空间结构域提供了理想材料。REA等[39]于2004年提出PCS酶活性中心可能类似于木瓜蛋白酶超家族的猜想,这个猜测翌年被VIVARES等[40]通过对藻青菌PCC 7120的PCS蛋白进行空间结构解析而得到证明。藻青菌PCC 7120的PCS是由2个新月形的单体组成的二聚体,空间大小均大概为50×30×30 Å,PCS二聚体一共含有8个α螺旋和6个β折叠条(图 3A);PCS的酶活性位点与木瓜蛋白酶的酶活性位点表现出高度相似性,这些酶激活区功能相似的氨基酸残基是二者具有相似作用机制的重要基础[39],其中典型氨基酸残基在NsPCS和木瓜蛋白酶的对应位点分别有Gln64和Gln19,Cys70和Cys25,Arg173和Ala136,His183和His159,Asp201和Asn175,Ser203和Trp177 (图 3B) [41]。在酰基化(转肽)过程中,NsPCS中参与γ-EC形成氢键的氨基酸残基有Cys70、Met123、Gly182、Asp225和Arg232 (图 3C和D),其中酶催化位点中最重要的3个氨基酸残基为Cys70、His183和Asp201,他们在空间结构上形成结合底物的“小洞”,这2种类型的氨基酸残基在真核生物(表 1)和原核生物(酵母和藻青菌等)中都严格保守(图 4),可见这些位点在PC形成过程中起了关键作用。
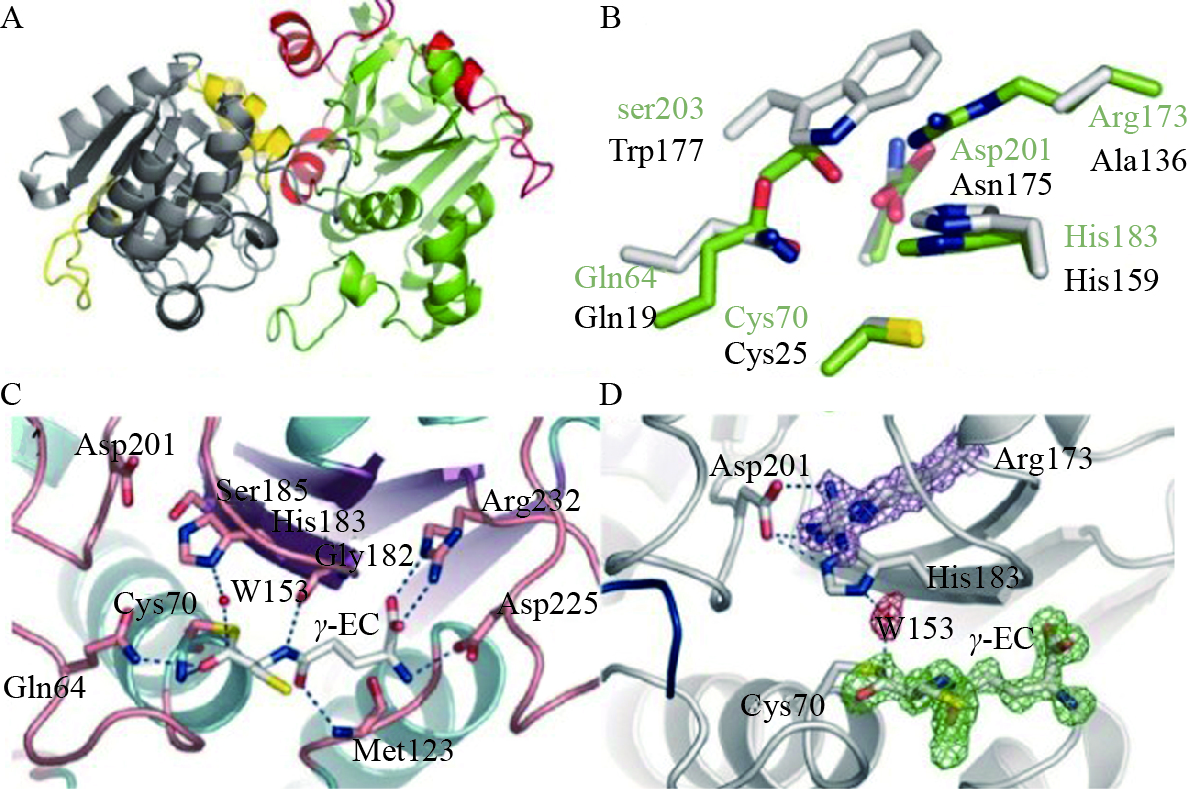 |
| A:自然条件下NsPCS的二聚体全局示意结构,2个单体的条带分别用灰色和绿色表示,其中类似于木瓜蛋白酶折叠出额外的次级结构,左边单体用黄色表示,右边单体用红色表示;B:NsPCS和木瓜蛋白酶活性位点的比较,分别用绿色和白色表示;C:围绕γ-EC主要结合位点中的氨基酸残基,蛋白和γ-EC间的氢键用蓝色虚线表示;D:围绕在γ-EC周围的电子云密度图。以上图片参考VIVARES等[40]进行修改。 A: Overall structure of NsPCS dimer under natural condition. Two monomers of the ribbon are colored in gray and green; furthermore, a comparison with the papain fold shows additional secondary structures drawn here in yellow or red depending on the monomer to which they belong. B: Comparison of the active-sites in the NsPCS (green) and papain (white). C: Selected amino acid residues around the γ-EC main binding site. Hydrogen bonds between the protein and γ-EC moiety are depicted by blue dotted lines. D: Electron-density map around the γ-EC moiety. Above figures were modified from VIVARES et al.[40]. 图3 PCS活性N-端催化中心蛋白的空间结构和转肽作用位点 Fig. 3 Spatial structures and enzyme active-sites from N-terminal catalytic center of PCS |
 |
| 红色条、蓝色箭头和橘黄色线条分别表示α螺旋、β折叠和β转角;红星表示酶催化“小洞”相关的氨基酸残基,蓝星和橘黄星分别表示与γ-EC形成氢键相关氨基酸残基的主链和支链。比对序列上面的灰色条表示比对序列间的保守性程度,其中黑星表示此处完全保守。TaPCS1、AtPCS1、NsPCS和SpPCS分别表示来自小麦、拟南芥、藻青菌和粟酒裂殖酵母的植物络合素合酶。 Red bar, blue arrow and orange line represent α-helix, β-strand and β-turn, respectively. Red star represents the position of catalytic triad; blue and orange stars represent the residues whose main or side chain is related to hydrogen-bonded to γ-EC, respectively. Grey bar on the head of sequence alignment represents conservative degree of amino acid, in which black star shows totally conserved position. TaPCS1, AtPCS1, NsPCS and SpPCS represent PCS from Triticum aestivum, Arabidopsis thaliana, Nostoc sp. PCC 7120 and Schizosaccharomyces pombe. 图4 物种中代表性PCS的N-基端点序列与功能位点对比 Fig. 4 N-terminal sequence alignment and structural site interpretation of selected and representative PCS in organisms |
半胱氨酸(cysteine, Cys)是20种氨基酸中唯一含有巯基的氨基酸,可以与重金属通过巯基实现不同形式的结合,许多金属结合原件具有富含Cys残基的特征,在有金属参与的生命过程中扮演着重要角色。富含特征Cys是PCS的重要特征之一,Cys被认为与PCS的激活有关,其在不同物种中和N、C-基端的分布呈现明显的差异化。在植物PCS中,除拟南芥旁系同源AtPCS2只含有13个Cys外,其他PCS均包含18~22个Cys,这可能是AtPCS2酶催化效率低于AtPCS1的原因;酵母和线虫PCS的Cys含量分别为18和22个,小眼虫为13个,而PCS酶催化活性低的藻青菌只含2个。PCS中Cys分布的另一个特征就是成对在N和C-基端出现,该特征在C-基端表现更为明显。随着对N-基端催化结构的解析和对越来越多物种PCS基因克隆研究的深入,PCS激活机制逐步形成了两大假说。一种认为重金属直接与N-基端酶活性中心的Cys结合,激活PCS活性,此外,C-基端的Cys也能与重金属结合,通过空间构想的改变,将重金属送到离活性中心更近的位置。这个假说很好地解释了为什么物种中N-基端保守性都非常高,而C-基端只要求含有数个Cys残基。另一种假说认为重金属离子并不直接接触PCS,而是通过一种双底物反应模式合成PC,与此同时,金属离子也可能直接结合在PCS的Cys残基上以促进反应。该假说中第一和第二底物分别是GSH和GSH巯基结合物。第一底物分子上的Cly残基脱落分离,产生中间产物γ-谷氨酰半胱氨酰基PCS结合体(γ-EC acyl-PCS),然后中间产物的γ-EC基团转移到第二个底物分子上产生双γ-EC基团,最后被从细胞质运送到液泡中。VATAMANIUK等[3, 35]通过构建AtPCS1-FLAG (拟南芥PCS1的C-基末端融合了一段DYKDDDDK的标记肽)发现,体外反应体系中金属离子与GSH或PCS的结合比与AtPCS1-FLAG的结合稳定得多,而且在完全不含金属离子的条件下,AtPCS-FLAG仍然能够以S-烷基-GSH为底物催化合成S-烷基-PC2,间接地证明了第二种假说的合理性。
6 展望从目前的研究来看,PCS作为植物络合素合酶的功能已被普遍证实,而基于该功能的基因表达模式、酶的活化模式及在不同物种中的差异性等在学界还存在严重分歧。PCS的肽酶特性的发现丰富了我们对PCS的认识与理解,但至今仍缺乏直接的关于该肽酶引起生物体内(植物)生理活动与表型变化的直接证据。而最新的研究表明,拟南芥AtPCS1突变体在面对生物胁迫时的非宿主抗性免疫系统遭受破坏,这可能是由于PCS作为肽酶参与到与之相关的吲哚、硫苷代谢途径中,且参与相关微生物维持养分平衡[5]。许多文献表明,PCS在植物中呈组成型表达,即无论有无外刺激物,植物中的转录量始终维持在较高水平,从而认定其在植物对重金属抗性作用中的差异主要由后期蛋白活性的调控决定,这也是目前的主流观点。尽管PCS的双功能酶特性从侧面支持了PCS在植物中持续高表达的合理性,但PCS家族基因的表达在某些物种中也受到不同重金属离子的调控,反映出该酶基因可能不是简单的组成型表达,而是具有“组成诱导型”表达的特点,即在温和条件下这些基因本生具有相对较高的表达量,而在外界刺激下,它们的表达又能受到进一步调控,这与传统意义上的平时低表达或不表达、刺激后大量表达的“诱导型表达”存在明显差别。植物络合素合酶在未经活化时不具有生物酶活性,其中关于重金属离子直接和不直接接触PCS活化的假设都有各自的证据,且并不能相互否认,据此推测PCS的活化极有可能同时存在以上2种模式。
植物体内植物络合素含量被认为和重金属污染程度密切相关。美国东部森林蓑萎树木体内PC含量均高于正常树木,水藓(Fontinalis antipyretica)中PC含量水平表现为与Cd2+水平呈依赖性剂量表达的动力学关系,长心卡帕藻(Kappaphycus alvarezii)中的PC含量随着暴露在Cd2+中的时间延长而增加,这些都为PC可作为环境重金属污染的生物指示剂提供了强力证据。植物体内PC含量除与金属差异诱导相关外,还呈现出由PCS引起的物种差异性,因而深入研究PCS催化机制和活性特征对于PC作为重金属污染的生物指示剂具有重要意义[4]。据GRILL等[42]在1985的报道,蛇根木(Rauvolfia serpentina)细胞悬浮培养液在200 μmol/L CdSO4(硫酸镉)胁迫下,90%以上吸收的重金属被植物络合素所吸附,可见植物络合素在植物主动抗重金属胁迫过程中占主导地位。因此,通过生物工程学手段,在粮食作物中抑制表达或者敲除PCS基因,创造重金属敏感作物,可降低可食用部分重金属含量。对于PCS基因抑制表达后对重金属表现为超级敏感的工程植物,可以开发为重金属污染土壤指示物;相应地,通过PCS过表达工程植物的创制,筛选获得强重金属吸附能力植物,是对重金属污染土壤进行植物修复的强有力措施。最后,作为富含Cys氨基残基的金属相关功能酶,PCS与金属的作用具有典型性,对我们研究锌指类蛋白(Cys与重金属Zn作用)等诸多金属结合原件的调控作用具有借鉴意义。
| [1] |
冯保民, 麻密. 植物络合素及其合酶在重金属抗性中的功能研究进展. 应用与环境生物学报, 2003(6): 657-661. FENG B M, MA M. Research advance of phytochelatins and phytochelatin synthase on heavy metal tolerance. Chinese Journal of Applied and Environmental Biology, 2003(6): 657-661. (in Chinese with English abstract) DOI:10.3321/j.issn:1006-687X.2003.06.023 |
| [2] |
BRUNETTI P, ZANELLA L, PROIA A, et al. Cadmium tolerance and phytochelatin content of Arabidopsis seedlings over-expressing the phytochelatin synthase gene AtPCS1. Journal of Experimental Botany, 2011, 62(15): 5509-5519. DOI:10.1093/jxb/err228 |
| [3] |
VATAMANIUK O K, MARI S, LU Y P, et al. AtPCS1, a phytochelatin synthase from Arabidopsis: Isolation and in vitro reconstitution. Proceedings of the National Academy of Sciences of the USA, 1999, 96(12): 7110-7115. DOI:10.1073/pnas.96.12.7110 |
| [4] |
GRILL E, LÖFFLER S, WINNACKER E L, et al. Phytochelatins, the heavy-metal-binding peptides of plants, are synthesized from glutathione by a specific γ-glutamylcysteine dipeptidyl transpeptidase (phytochelatin synthase). Proceedings of the National Academy of Sciences of the USA, 1989, 86(18): 6838-6842. DOI:10.1073/pnas.86.18.6838 |
| [5] |
高华, 张玉秀, 柴团耀. 植物络合素和植物络合素合酶的研究. 西北植物学报, 2001, 21(4): 779-790. GAO H, ZHANG Y X, CHAI T Y. Research advances in phytochelatins and phytochelatin synthase. Acta Botanica Boreali-Occidentalia Sinica, 2001, 21(4): 779-790. (in Chinese with English abstract) DOI:10.3321/j.issn:1000-4025.2001.04.031 |
| [6] |
CLEMENS S, PERŠOH D. Multi-tasking phytochelatin synthases. Plant Science, 2009, 177(4): 266-271. DOI:10.1016/j.plantsci.2009.06.008 |
| [7] |
KÜHLENZ T, SCHMIDT H, URAGUCHI S, et al. Arabidopsis thaliana phytochelatin synthase 2 is constitutively active in vivo and can rescue the growth defect of the PCS1-deficient cad1-3 mutant on Cd- contaminated soil. Journal of Experimental Botany, 2014, 65(15): 4241-4253. DOI:10.1093/jxb/eru195 |
| [8] |
LI A M, YU B Y, CHEN F H, et al. Characterization of the Sesbania rostrata phytochelatin synthase gene: Alternative splicing and function of four isoforms. International Journal of Molecular Sciences, 2009, 10(8): 3269-3282. DOI:10.3390/ijms10083269 |
| [9] |
LIU Z L, GU C S, CHEN F D, et al. Heterologous expression of a Nelumbo nucifera phytochelatin synthase gene enhances cadmium tolerance in Arabidopsis thaliana. Applied Biochemistry and Biotechnology, 2012, 166(3): 722-734. DOI:10.1007/s12010-011-9461-2 |
| [10] |
LEE B D, HWANG S. Tobacco phytochelatin synthase (NtPCS1) plays important roles in cadmium and arsenic tolerance and in early plant development in tobacco. Plant Biotechnology Reports, 2015, 9(3): 107-114. DOI:10.1007/s11816-015-0348-5 |
| [11] |
WANG F J, WANG Z B, ZHU C. Heteroexpression of the wheat phytochelatin synthase gene (TaPCS1) in rice enhances cadmium sensitivity. Acta Biochimica et Biophysica Sinica, 2012, 44(10): 886-893. DOI:10.1093/abbs/gms073 |
| [12] |
BLUM R, BECK A, KORTE A, et al. Function of phytochelatin synthase in catabolism of glutathioneconjugates. The Plant Journal, 2007, 49(4): 740-749. DOI:10.1111/j.1365-313X.2006.02993.x |
| [13] |
CHEN J J, ZHOU J M, GOLDSBROUGH P B. Characterization of phytochelatin synthase from tomato. Physiologia Plantarum, 1997, 101(1): 165-172. DOI:10.1111/ppl.1997.101.issue-1 |
| [14] |
LEE S, KORBAN S. Transcriptional regulation of Arabidopsis thaliana phytochelatin synthase (AtPCS1) by cadmium during early stages of plant development. Planta, 2002, 215(4): 689-693. DOI:10.1007/s00425-002-0821-6 |
| [15] |
TSUJI N, NISHIKORI S, IWABE O, et al. Characterization of phytochelatin synthase-like protein encoded by alr0975 from a prokaryote, Nostoc sp. PCC 7120. Biochemical and Biophysical Research Communications, 2004, 315(3): 751-755. DOI:10.1016/j.bbrc.2004.01.122 |
| [16] |
RIGOUIN C, VERMEIRE J J, NYLIN E, et al. Characterization of the phytochelatin synthase from the human parasitic nematode Ancylostoma ceylanicum. Molecular and Biochemical Parasitology, 2013, 191(1): 1-6. DOI:10.1016/j.molbiopara.2013.07.003 |
| [17] |
GARCIA-GARCIA J D, GIRARD L, HERNÁNDEZ G, et al. Zn-bis-glutathionate is the best co-substrate of the monomeric phytochelatin synthase from the photosynthetic heavy metalhyperaccumulator Euglena gracilis. Metallomics, 2014, 6(3): 604-616. DOI:10.1039/c3mt00313b |
| [18] |
LEE S, KANG B S. Expression of Arabidopsis phytochelatin synthase 2 is too low to complement an AtPCS1-defective cad1-3 mutant. Molecules and Cells, 2005, 19(1): 81-87. |
| [19] |
CLEMENS S, KIM E J, NEUMANN D, et al. Tolerance to toxic metals by a gene family of phytochelatin synthases from plants and yeast. The EMBO Journal, 1999, 18(12): 3325-3333. DOI:10.1093/emboj/18.12.3325 |
| [20] |
COUSELO J L, NAVARRO-AVIÑÓ J, BALLESTER A. Expression of the phytochelatin synthase TaPCS1 in transgenic aspen, insight into the problems and qualities in phytoremediation of Pb. International Journal of Phytoremediation, 2010, 12(4): 358-370. DOI:10.1080/15226510902968134 |
| [21] |
OVEN M, PAGE J E, ZENK M H, et al. Molecular characterization of the homo-phytochelatin synthase of soybean Glycine max: Relation to phytochelatin synthase. Journal of Biological Chemistry, 2002, 277(7): 4747-4754. DOI:10.1074/jbc.M108254200 |
| [22] |
STROIŃSKI A, ZIELEZIŃSKA M. Cadmium and oxidative stress influence on phytochelatin synthase activity in potato tuber. Acta Physiologiae Plantarum, 2001, 23(2): 157-160. DOI:10.1007/s11738-001-0003-3 |
| [23] |
KIM Y J, CHANG K S, LEE M R, et al. Expression of tobacco cDNA encoding phytochelatin synthase promotes tolerance to and accumulation of Cd and as in Saccharomyces cerevisiae. Journal of Plant Biology, 2005, 48(4): 440-447. DOI:10.1007/BF03030586 |
| [24] |
LI J C, GUO J B, XU W Z, et al. RNA interferencemediated silencing of phytochelatin synthase gene reduce cadmium accumulation in rice seeds. Journal of Integrative Plant Biology, 2007, 49(7): 1032-1037. DOI:10.1111/jipb.2007.49.issue-7 |
| [25] |
KNYAZEV A V, KULUEV B R, CHEMERIS A V, et al. Effect of cadmium on promoter activity of rice phytochelatin synthase gene in transgenic tobacco plants. Russian Journal of Plant Physiology, 2013, 60(5): 701-705. DOI:10.1134/S1021443713030126 |
| [26] |
DAS N, BHATTACHARYA S, BHATTACHARYYA S, et al. Identification of alternatively spliced transcripts of rice phytochelatin synthase 2 gene OsPCS2 involved in mitigation of cadmium and arsenic stresses. Plant Molecular Biology, 2017, 94(1/2): 167-183. |
| [27] |
VESTERGAARD M, MATSUMOTO S, NISHIKORI S, et al. Chelation of cadmium ions by phytochelatin synthase: Role of the cystein-rich C-terminal. Analytical Sciences, 2008, 24(2): 277-281. DOI:10.2116/analsci.24.277 |
| [28] |
HA S B, SMITH A P, HOWDEN R, et al. Phytochelatin synthase genes from Arabidopsis and the yeast Schizosaccharomyces pombe. The Plant Cell, 1999, 11(6): 1153-1164. DOI:10.1105/tpc.11.6.1153 |
| [29] |
HEISS S, WACHTER A, BOGS J, et al. Phytochelatin synthase (PCS) protein is induced in Brassica juncea leaves after prolonged Cd exposure. Journal of Experimental Botany, 2003, 54(389): 1833-1839. DOI:10.1093/jxb/erg205 |
| [30] |
GASIC K, KORBAN S S. Expression of Arabidopsis phytochelatin synthase in Indian mustard (Brassica juncea) plants enhances tolerance for Cd and Zn. Planta, 2007, 225(5): 1277-1285. DOI:10.1007/s00425-006-0421-y |
| [31] |
GOTO F, ARASHIMA Y, SHIMADA H, et al. The expression analysis of phytochelatin synthase gene in Athyrium yokoscense//FUKADA H. Plant and Cell Physiology. Oxford, England: Oxford University Press, 2004: S168.
|
| [32] |
SHUKLA D, KESARI R, MISHRA S, et al. Expression of phytochelatin synthase from aquatic macrophyte Ceratophyllum demersum L. enhances cadmium and arsenic accumulation in tobacco. Plant Cell Reports, 2012, 31(9): 1687-1699. DOI:10.1007/s00299-012-1283-3 |
| [33] |
SHRI M, DAVE R, DIWEDI S, et al. Heterologous expression of Ceratophyllum demersum phytochelatin synthase, CdPCS1, in rice leads to lower arsenic accumulation in grain. Scientific Reports, 2014, 4: 5784. |
| [34] |
SHUKLA D, KESARI R, TIWARI M, et al. Expression of Ceratophyllum demersum phytochelatin synthase, CdPCS1, in Escherichia coli and Arabidopsis enhances heavy metal- (loid)s accumulation. Protoplasma, 2013, 250(6): 1263-1272. DOI:10.1007/s00709-013-0508-9 |
| [35] |
VATAMANIUK O K, BUCHER E A, WARD J T, et al. A new pathway for heavy metal detoxification in animals: Phytochelatin synthase is required for cadmium tolerance in Caenorhabditis elegans. Journal of Biological Chemistry, 2001, 276(24): 20817-20820. DOI:10.1074/jbc.C100152200 |
| [36] |
CLEMENS S, SCHROEDER J I, DEGENKOLB T. Caenorhabditis elegans expresses a functional phytochelatin synthase. The FEBS Journal, 2001, 268(13): 3640-3643. |
| [37] |
ESSIG Y J, WEBB S M, STURZENBAUM S R. Deletion of phytochelatin synthase modulates the metal accumulation pattern of cadmium exposed C. elegans. International Journal of Molecular Sciences, 2016, 17(2): 257. DOI:10.3390/ijms17020257 |
| [38] |
MARCHLER-BAUER A, LU S, ANDERSON J B, et al. CDD: A Conserved Domain Database for the functional annotation of proteins. Nucleic Acids Research, 2010, 39(Suppl. 1): D225-D229. |
| [39] |
REA P A, VATAMANIUK O K, RIGDEN D J. Weeds, worms, and more. Papain' s long-lost cousin, phytochelatin synthase. Plant Physiology, 2004, 136(1): 2463-2474. DOI:10.1104/pp.104.048579 |
| [40] |
VIVARES D, ARNOUX P, PIGNOL D. A papain-like enzyme at work: Native and acyl-enzyme intermediate structures in phytochelatin synthesis. Proceedings of the National Academy of Sciences of the USA, 2005, 102(52): 18848-18853. DOI:10.1073/pnas.0505833102 |
| [41] |
YAMAMOTO D, MATSUMOTO K, OHISHI H, et al. Refined X-ray structure of papain. E-64-c complex at 2.1-A resolution. Journal of Biological Chemistry, 1991, 266(22): 14771-14777. |
| [42] |
GRILL E, WINNACKER E, ZENK M H. Phytochelatins: The principal heavy-metal complexing peptides of higher plants. Science, 1985, 230(4726): 674-676. DOI:10.1126/science.230.4726.674 |
 2018, Vol. 44
2018, Vol. 44


