| 基于人工智能的肺磨玻璃密度结节三维测量与病理切片测量的相关性研究 |
2. 上海市嘉定区中医医院放射科,上海 201800;
3. 上海中医药大学附属曙光医院病理科,上海 201203
2. Department of Radiology, Shanghai Jiading District Hospital of TCM, Shanghai 201800, China;
3. Department of Pathology, Shuguang Hospital Affiliated to Shanghai University of Traditional Chinese Medicine, Shanghai 201203, China
肺癌是死亡率最高(18%)的癌症[1]。肺腺癌是非小细胞肺癌中最常见的组织学亚型[2],早期CT常表现为磨玻璃密度结节,而磨玻璃密度结节的大小是鉴别病灶良恶性的一个重要指标[3-4]。临床工作中结节的直径可在PACS上使用电子卡尺手动测量,而结节体积的测量则相对复杂。随着人工智能的迅速发展,尤其是深度学习和卷积神经网络技术的应用,使得对肺结节的检出及分割能力明显提升[5-6]。但关于人工智能肺结节测量与病理切片测量之间的相关性分析尚未见报道。本研究拟对经手术病理证实的T1a期肺腺癌的人工智能肺部影像分析系统(artificial intelligence lung image analysis system,AILIAS)测量、半自动分割技术(subsolid lung lesion segmentation,SLLS)测量与病理数字切片测量的数据进行对照分析,以评估人工智能对肺磨玻璃结节测量的可靠性。
1 资料与方法 1.1 一般资料回顾性分析2017年1月至2021年12月在上海中医药大学附属曙光医院行CT检查发现肺结节并行手术治疗的T1a期肺腺癌患者,包含原位癌、微浸润腺癌及浸润性腺癌。纳入标准:①均于术前2周内行胸部CT扫描。②术前未行穿刺或抗肿瘤治疗。排除标准:①病灶内出现粗大血管影。②ROI层面CT图像为呼气相或出现呼吸运动伪影。
最终纳入91例,其中男24例,女67例;年龄17~80岁,平均(45.6±15.1)岁;原位癌28例(30.8%)、微浸润腺癌51例(56.0%)、浸润性腺癌12例(13.2%)。本研究经医院伦理委员会批准(2021-979-54-01),免除知情同意。
1.2 仪器与方法采用Siemens Somatom Force 384层双源CT、Philips 256层CT(Brilliance iCT)、GE 64层CT(LightSpeed VCT 64)及联影64层CT(uCT760)。患者取仰卧位,头先入,行全肺容积扫描。扫描参数:120 kV,自动管电流,层厚、层距均为1 mm,矩阵512×512;标准算法重建。
1.3 图像分析将DICOM格式的CT图像导入Siemens工作站,使用多模态肿瘤学分析模块对结节行定量分析。由1位具有15年胸部影像诊断经验的副主任医师在肺窗(-450 HU,1 500 HU)对磨玻璃密度结节行半自动分割,手动调整计算机勾画不满意的层面。分割时尽可能地去除病灶边缘的小血管。勾画完成后,采集病灶体积、平均直径(最大径与其最大垂直径的平均值)(图 1a)。间隔1个月行第2次测量,取2次结果的平均值。
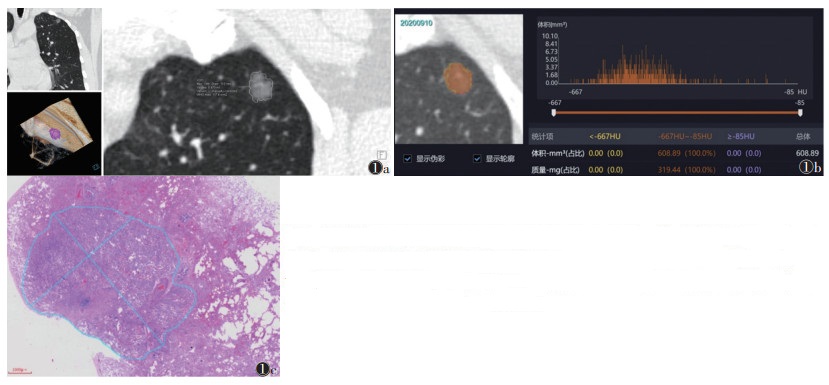 |
| 注:患者,男,53岁,微浸润腺癌。图 1a为半自动分割技术测量结节平均直径9 mm,体积479 mm3;图 1b为人工智能分析系统测量结节平均直径11 mm,体积609 mm3;图 1c为病理数字切片上测量的结节平均直径5 mm,面积166.6 mm2 图 1 磨玻璃密度结节图像分割及病理切片测量示意图 |
将DICOM格式的CT图像导入联影肺结节人工智能系统(型号:uAI-ChestCare),采集、记录病灶体积、平均直径(图 1b)。
对所有结节的最大层面切片使用麦克奥迪EASY SCAN数字切片扫描与应用系统扫描成电子影像,手动勾勒结节边缘,由软件自动测量勾勒区域平均直径及面积(图 1c)。
1.4 统计学分析采用SPSS 18.0软件行统计分析。对结节平均直径、面积及体积行正态性检验,正态分布数据以x±s表示,非正态分布数据以M(QL,QU)表示。AILIAS、SLLS及病理数字切片平均直径测量值两两比较、AILIAS与SLLS体积测量值间两两比较采用Wilcoxon符号秩检验。AILIAS及SLLS体积测量值与病理数字切片面积测量值间的相关性分析采用Spearman秩相关。以P<0.05为差异有统计学意义。
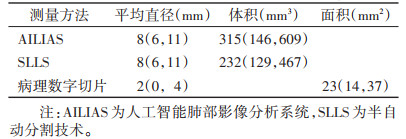
2 结果AILIAS与SLLS所测平均直径均大于病理数字切片,差异均有统计学意义(Z=-7.310,-8.577;均P<0.001)。AILIAS与SLLS所测直径差异无统计学意义(Z=-0.744,P=0.457)。
SLLS所测体积小于AILIAS,差异有统计学意义(Z=-6.218,P<0.001)(表 1)。
| 表 1 3种方法测量值及非参数比较[M(QL,QU)] |
 |
AILIAS、SLLS体积测量值与病理数字切片面积测量值均呈正相关(rs=0.729,0.727;均P<0.001)(图 2,3)。
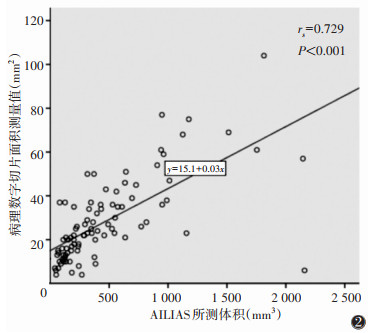 |
| 注:AILIAS为人工智能肺部影像分析系统 图 2 AILIAS所测体积与病理数字切片面积测量值散点图 |
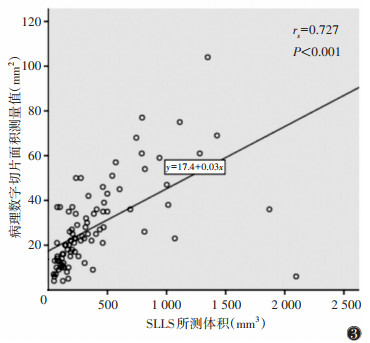 |
| 注:SLLS为半自动分割技术 图 3 SLLS所测体积与病理数字切片面积测量值散点图 |
3 讨论
本研究回顾性分析91例以磨玻璃密度结节为表现的T1a期(病理切片测量直径≤10 mm的结节)肺腺癌,分别以AILIAS及SLLS测量CT图像上结节的体积、平均直径,并在病理数字切片电子图像上测量病灶的平均直径和面积。AILIAS及SLLS测量的体积与病理数字切片测得的面积均呈正相关。AILIAS及SLLS测量的平均直径差异无统计学意义(P>0.05)。
磨玻璃结节的大小是判断肿瘤良恶性的重要指标之一[2-3],也是随访时判断病变进展的重要依据,因此精确测量至关重要[7]。结节大小的测量包括一维测量(直径测量)、二维测量(平均直径测量)和体积测量。
美国国家肺癌筛查试验、Fleischner协会及肺部影像报告和数据系统(Lung-RADS)等均以结节的平均直径作为随访频率调整和临床介入的依据[8-10]。在临床工作中可通过PACS的卡尺手动测量肺结节平均直径,耗时且不同观察者之间可能存在差异。而AILIAS可自动测量结节的最大径、平均直径,较手动测量更快捷、且一致性较好[11]。本研究发现,AILIAS测量肺结节的直径与SLLS差异无统计学意义,与吴林玉等[12]研究结果一致。AILIAS、SLLS测量的平均直径均大于在病理数字切片中所测的平均直径,与文献[13]报道一致。可能是由于切片中的病灶为离体标本,病灶内肺泡腔里的气体排出,导致病灶塌陷,故其平均直径小于活体组织;另外,病理数字切片标本制作时采用的固定方法也可导致病灶缩小。
相对于二维测量,结节体积的测量对恶性结节变化更敏感[14]。精确的结节分割是结节体积测量的必要因素,也是结节管理和倍增时间计算的重要因素[15-16]。传统的体积测量方法包括手动测量和半自动测量。手动测量须勾画每一个层面结节的轮廓后经软件计算出结节的体积;半自动测量则通过勾画一个层面中结节轮廓、直径或一个点,软件自动识别其他层面结节轮廓并计算体积,同时后处理医师可手动修改调整各层面结节的轮廓使分割更精确。近年来,基于深度学习的人工智能全自动分割技术可在无需手动干预的情况下自动分割、测量结节的体积,且观察者间和观察者内一致性较高[17-20]。目前,结节的体积测量已是欧洲肺癌筛查声明中的推荐方法[21],Fleischner协会及Lung-RADS在倡导平均直径测量法的同时也提供了体积阈值。但关于体积测量与病理切片测量的相关性报道较少。本研究在数字病理切片中手动勾画结节的轮廓,通过软件自动计算结节面积,并与AILIAS、SLLS测得的结节体积行相关性分析,发现ALIAS、SLLS所测结节体积与病理切片面积测量值均具有良好的相关性,且AILIAS的相关系数略高于SLLS。这表明人工智能技术可方便快捷地获得结节的体积值,大大缩减时间成本,且结果与病理切片有良好的相关性,甚至略优于半自动测量。另外,本研究发现AILIAS体积测量值显著大于SLLS测量值,推测可能由以下因素导致:磨玻璃结节边缘体素点与背景间密度差异较小,导致靠基于视觉或密度阈值分割时切缘小于人工智能的判别;人工智能在分割血管旁结节时,往往将血管判定为结节的一部分,而半自动分割时可手动调整结节的边缘。因此,在肺磨玻璃结节的随访过程中,为使病灶体积多次测量值更具可比性,需采用同一种测量方法。
本研究存在的不足:例数相对较少,所测定量数据均为非正态分布数据,可能导致检验效能降低;由于本研究中为非连续病理切片,故无法对结节病理切片体积与人工智能体积测量行相关分析,且未对病灶内实性成分体积与病理浸润区行对照分析。后续将增大样本量验证结果。
综上所述,对于肺磨玻璃结节的测量,人工智能测量与传统半自动测量一致性较好,且人工智能测量与病理切片测量具有良好的相关性,略高于传统的半自动测量。基于人工智能的二维和三维测量可为磨玻璃结节临床管理提供可靠的影像学依据。
| [1] |
SUNG H, FERLAY J, SIEGEL R L, et al. Global Cancer Statistics 2020:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
CHENG T Y, CRAMB S M, BAADE P D, et al. The international epidemiology of lung cancer: latest trends, disparities, and tumor characteristics[J]. J Thorac Oncol, 2016, 11(10): 1653-1671. DOI:10.1016/j.jtho.2016.05.021 |
| [3] |
MCWILLIAMS A, TAMMEMAGI M C, MAYO J R, et al. Probability of cancer in pulmonary nodules detected on first screening CT[J]. N Engl J Med, 2013, 369(10): 910-919. DOI:10.1056/NEJMoa1214726 |
| [4] |
HE W, GUO G, DU X, et al. CT imaging indications correlate with the degree of lung adenocarcinoma infiltration[J]. Front Oncol, 2023, 13: 1108758. DOI:10.3389/fonc.2023.1108758 |
| [5] |
CHEN L, GU D, CHEN Y, et al. An artificial-intelligence lung imaging analysis system (AILIAS) for population-based nodule computing in CT scans[J]. Comput Med Imaging Graph, 2021, 89: 101899. DOI:10.1016/j.compmedimag.2021.101899 |
| [6] |
HUANG W, XUE Y, WU Y. A CAD system for pulmonary nodule prediction based on deep three-dimensional convolutional neural networks and ensemble learning[J]. PLoS One, 2019, 14(7): e0219369. DOI:10.1371/journal.pone.0219369 |
| [7] |
NAIR A, DYER D S, HEUVELMANS M A, et al. Contextualizing the role of volumetric analysis in pulmonary nodule assessment: AJR expert panel narrative review[J]. AJR Am J Roentgenol, 2023, 220(3): 314-329. DOI:10.2214/AJR.22.27830 |
| [8] |
AMERICAN COLLEGE OF RADIOLOGY. Lung CT screening reporting and data system (Lung-RADS)[EB/OL]. https://www.acr.org/Quality-Safety/Resources/LungRADS.
|
| [9] |
MACMAHON H, NAIDICH D P, GOO J M, et al. Guidelines for management of incidental pulmonary nodules detected on CT images: from the Fleischner society 2017[J]. Radiology, 2017, 284(1): 228-243. DOI:10.1148/radiol.2017161659 |
| [10] |
NATIONAL LUNG SCREENING TRIAL RESEARCH TEAM. Reduced lung-cancer mortality with low-dose computed tomographic screening[J]. N Engl J Med, 2011, 365(5): 395-409. DOI:10.1056/NEJMoa1102873 |
| [11] |
CHEN L, GU D, CHEN Y, et al. An artificial-intelligence lung imaging analysis system (AILIAS) for population-based nodule computing in CT scans[J]. Comput Med Imaging Graph, 2021, 89: 101899. DOI:10.1016/j.compmedimag.2021.101899 |
| [12] |
吴林玉, 叶剑锋, 郑思思, 等. 应用人工智能对肺结节直径测量: 观察者内、观察者间差异[J]. 医学影像学杂志, 2020, 30(1): 47-51. |
| [13] |
GARZELLI L, GOO J M, AHN S Y, et al. Improving the prediction of lung adenocarcinoma invasive component on CT: value of a vessel removal algorithm during software segmentation of subsolid nodules[J]. Eur J Radiol, 2018, 100: 58-65. DOI:10.1016/j.ejrad.2018.01.016 |
| [14] |
KO J P, BERMAN E J, KAUR M, et al. Pulmonary Nodules: growth rate assessment in patients by using serial CT and three-dimensional volumetry[J]. Radiology, 2012, 262(2): 662-671. DOI:10.1148/radiol.11100878 |
| [15] |
CHARTRAND G, CHENG P M, VORONTSOV E, et al. Deep learning: a primer for radiologists[J]. Radiographics, 2017, 37(7): 2113-2131. DOI:10.1148/rg.2017170077 |
| [16] |
CALLISTER M E, BALDWIN D R, AKRAM A R, et al. British Thoracic Society guidelines for the investigation and management of pulmonary nodules[J]. Thorax, 2015, 70(Suppl 2): ⅱ1-ⅱ54. DOI:10.1136/thoraxjnl-2015-207168 |
| [17] |
LANCASTER H L, ZHENG S, ALESHINA O O, et al. Outstanding negative prediction performance of solid pulmonary nodule volume AI for ultra-LDCT baseline lung cancer screening risk stratification[J]. Lung Cancer, 2022, 165: 133-140. DOI:10.1016/j.lungcan.2022.01.002 |
| [18] |
RITCHIE A J, SANGHERA C, JACOBS C, et al. Computer vision tool and technician as first reader of lung cancer screening CT scans[J]. J Thorac Oncol, 2016, 11(5): 709-717. DOI:10.1016/j.jtho.2016.01.021 |
| [19] |
MARTINS JARNALO C O, LINSEN P V M, BLAZIS S P, et al. Clinical evaluation of a deep-learning-based computer-aided detection system for the detection of pulmonary nodules in a large teaching hospital[J]. Clin Radiol, 2021, 76(11): 838-845. DOI:10.1016/j.crad.2021.07.012 |
| [20] |
JACOBS C, SCHREUDER A, VAN RIEL S J, et al. Assisted versus manual interpretation of low-dose CT scans for lung cancer screening: impact on lung-rads agreement[J]. Radiol Imaging Cancer, 2021, 3: e200160. DOI:10.1148/rycan.2021200160 |
| [21] |
OUDKERK M, DEVARAJ A, VLIEGENTHART R, et al. European position statement on lung cancer screening[J]. Lancet Oncol, 2017, 18(12): e754-e766. DOI:10.1016/S1470-2045(17)30861-6 |
 2024, Vol. 22
2024, Vol. 22


