

2. 沈阳医学院生物化学与分子生物学教研室,辽宁 沈阳 110034;
3. 沈阳医学院病原生物学教研室,辽宁 沈阳 110034
2. Dept of Biochemistry and Molecular Biology, Shenyang Medical College, Shenyang 110034, China;
3. Dept of Pathogen Biology, Shenyang Medical College, Shenyang 110034, China
癌症被认为是世界上每个国家的主要死亡原因和延长预期寿命的重要障碍,据我国肿瘤登记中心的数据资料显示,乳腺癌是最常见的恶性肿瘤之一,其发病率在近十几年呈现上升趋势,居于城乡女性第一位[1]。辛二酰苯胺异羟肟酸(suberoylanilide hydroxamic acid,SAHA)是一种二代组蛋白去乙酰化酶抑制剂,通过对细胞的核小体中的组蛋白乙酰化水平的调节,从而改变细胞染色质的结构,起到调节细胞转录因子的表达、细胞凋亡及细胞的诱导分化等相关的生理生化效应[2]。由334个氨基酸残基组成的人组织蛋白酶V (cathepsin V,CTSV),分子量约为37 ku,是一种溶酶体蛋白水解酶,定位于9q22.2染色体[3]。但是,CTSV在自噬中的作用还未见深入了解。本文将SAHA作用于乳腺癌MCF-7细胞,研究CTSV在SAHA诱导乳腺癌MCF-7细胞自噬中的作用和机制,为进一步明确SAHA-CTSV轴通过诱导过度自噬抑制乳腺癌MCF-7细胞生长的分子作用机制,提供相应的理论基础和实验数据。
1 材料与方法 1.1 材料人乳腺癌细胞株MCF-7(ATCC细胞库);RPMI 1640培养基(A4192301)、胎牛血清(A3160902)、青霉素及链霉素(KGY0023)(Thermo Scientific);SAHA(1604-1)(Sigma-Aldrich);Muse Count & Viability试剂盒(13-0618)(Millipore);RNA提取试剂盒(1182866500)(Roche);Power SYBR Green PCR Master mix(4367659)(Life Technologies);抗体(Abcam Inc);ECL化学发光试剂盒(B2162617)(Roche);CTSV小干涉RNA(small interfering RNA,siRNA) (SC-44526-SH)(Santa Cruz)。
1.2 方法 1.2.1 细胞培养人乳腺癌MCF-7细胞使用含10 %胎牛血清的RPMI 1640培养基(添加100 U·mL-1青霉素和100 mg·L-1链霉素),在含有5 % CO2的培养箱中37 ℃培养。实验时当细胞长至培养瓶底70%时,收集细胞调整浓度并接种到96孔板(1×107 L-1)或6孔板(5×108 L-1)。siRNA干扰实验需将已贴壁细胞更换为无血清培养基进行同步化处理后再进行实验。
1.2.2 Muse自动细胞分析仪检测细胞活力配制浓度为0、0.5、1、2、5、10、20、50 μmol·L-1的SAHA,加入到细胞已贴壁的培养板中培养24 h,胰酶消化收集细胞,每组约2×105个,每组样品加入Muse自动细胞分析仪检测试剂Count & Viability reagent 450 μL,混匀室温静止,然后检测细胞活力。
1.2.3 CTSV siRNA转染MCF-7细胞根据Lipofectamine 3000转染试剂使用说明,先将无血清培养基与适量lipofectamine 3000转染试剂和CTSV siRNA分别混匀并室温静止5 min,然后再将两种混合液移到1.5 mL离心管中混匀后静止5 min。将转染复合物加至培养板中培养24 h后继续后面操作。
1.2.4 RNA提取和实时荧光定量PCR检测mRNA水平将SAHA与MCF-7细胞共同孵育24 h,按照RNA提取试剂盒说明进行,合成第一链cDNA后应用SYBR Green方法进行实时荧光定量PCR,以GAPDH为内参进行作用,观察mRNA变化。每个样品设3个复孔,数据分析采用2-ΔΔCT方法。Beclin 1引物序列:Forward为5′-AGGTTGAGAAAGGCG AGACA-3′,Reverse为5′-GCTTTTGTCCACTGCTCCT C-3′;ATG12引物序列:Forward为5′-AGGTCTGT AGTCGCGGAGAA-3′,Reverse为5′-GTTCCCGGCT AGTCATTCAA-3′;ATG10引物序列:Forward为5′-GCATTGTAGGGCCAGTTGTT-3′,Reverse为5′-GCT GGCCAGGTAAACTCTTG-3′;ATG9A引物序列:Forward为5′-GTCAGCTGCGTGGACTATGA-3′,Reverse为5′-GACTTGAGCAGGCAAAAAGG-3′;ATG7引物序列:Forward为5′-GAACATGGTGCTGGTTTCCT-3′,Reverse为5′-CATCCAGGGTACTGGGCTAA-3′;ATG5引物序列:Forward为5′-GCAAGCCAGACAGGAAA AAG-3′,Reverse为5′-GACCTTCAGTGGTCCGGTA A-3′;ATG4B引物序列:Forward为5′-GAGCTGCC AAGTCCAGATTC-3′,Reverse为5′-CCAGGATTTTCA AAGGGACA-3′;ATG4C引物序列:Forward为5′-CCTTGGCTCTTTTTGGCTTA-3′,Reverse为5′-AGG ATGCCTTGCTTCTTCAA-3′;ATG3引物序列:Forward为5′-TTTGGCTATGATGAGCAACG-3′,Reverse为5′-GTGGCAGATGAGGGTGATTT-3′;ULK1引物序列:Forward为5′-GTAAACTGGGGTCGCATTGT-3′,Reverse为5′-TGGATCCAAGGCTCTAGGTG-3′;GAPDH引物序列:Forward为5′-ACAGTCAGCCGCATCTTCTT-3′,Reverse为5′-ACGACCAAATCCGTTGACTC-3′。
1.2.5 Western blot检测蛋白表达水平SAHA作用细胞24h后,收集细胞提取蛋白测定蛋白量;将蛋白含量调整一致后,SDS-聚丙酰胺凝胶电泳,将蛋白转移至PVDF膜,4℃封闭过夜,然后经过一抗及二抗孵育,ECL化学发光,凝胶成像系统成像。
1.2.6 细胞免疫荧光法检测自噬水平SAHA作用细胞24 h后,细胞清洗后用4 %甲醛室温固定30 min,5 % BSA室温封闭1 h,加入LC3抗体试剂4 ℃过夜孵育,细胞清洗后激光共聚焦显微镜扫描成像。
1.2.7 自噬通量阻滞试验将0-40 nmol·L-1不同剂量的Bafilomycin A1与SAHA共同作用MCF-7细胞24 h。一部分细胞收集提取蛋白,利用Western blot检测细胞自噬相关蛋白表达量从而确定最佳自噬抑制剂量。另一部分细胞在Bafilomycin A1存在下做CTSV-siRNA转染实验,实验结束后收集细胞,然后检测p62蛋白水平并进行细胞活力分析。
1.3 统计学方法所有数据以x±s表示,采用SPSS 25.0软件对实验结果进行统计分析,通过Student’s t检验和方差分析进行统计学处理。
2 结果 2.1 SAHA对MCF-7细胞增殖的影响我们将SAHA(0-50 μmol·L-1)与MCF-7细胞作用24 h,通过Muse细胞分析仪测定细胞活力,以期找到SAHA对MCF-7细胞的最适浓度。如Tab 1数据结果显示,不同浓度的SAHA作用细胞后,细胞的活力随着浓度增加而逐渐降低,其中在5 μmol·L-1浓度的SAHA组,MCF-7细胞活率为70.8%,与对照组细胞活率88.5 %相比,细胞活率显著降低,见Tab 1。结果表明,SAHA能显著抑制MCF-7细胞的增殖。
| Sample | Cell viability | |
| Live/% | Dead /% | |
| Basal | 88.5±2.8 | 11.5±1.7 |
| SAHA 0.5 μmol·L-1 | 87.1±2.9 | 12.9±2.1 |
| SAHA 1 μmol·L-1 | 85.0±1.5* | 15.0±1.9* |
| SAHA 2 μmol·L-1 | 83.4±2.3* | 16.6±1.9* |
| SAHA 5 μmol·L-1 | 70.8±2.4* | 29.2±2.3* |
| SAHA 10 μmol·L-1 | 71.2±2.6* | 28.8±2.1* |
| SAHA 20 μmol·L-1 | 72.0±2.3* | 28.0±2.5* |
| SAHA 50 μmol·L-1 | 63.7±2.1* | 36.3±2.4* |
| *P<0.05 vs Basal | ||
为明确SAHA诱导乳腺癌细胞增殖中是否伴随CTSV表达量的改变,我们测定了不同剂量SAHA诱导的MCF-7细胞CTSV的mRNA和蛋白水平。结果表明,SAHA处理后CTSV的mRNA和蛋白水平显著升高。SAHA 5 μmol·L-1作用24 h后,检测CTSV的mRNA水平,与对照组比较达到其93.7倍,同时CTSV的蛋白表达水平也显著上升。实验结果表明SAHA能促使MCF-7细胞中CTSV的基因和蛋白的表达增高,见Fig 1。此外,综合SAHA的毒副作用,我们最终确定了SAHA对MCF-7细胞作用的最适浓度为5 μmol·L-1。

|
| 图 1 Assays for CTSV expressions induced by different doses of SAHA for 24 h treatment (x±s, n=3) A: Western blot assay for MCF-7 cells; B: Real-time PCR assay for MCF-7 cells. *P<0.05 vs Basal. |
为验证CTSV-siRNA是否成功转染MCF-7细胞,我们用Real-time PCR和Western blot检测敲减效果。实验结果显示,与对照组比较,SAHA组CTSV mRNA转录水平和蛋白表达水平最高,CTSV-siRNA组的CTSV mRNA转录水平和蛋白表达水平显著下降,而SAHA+CTSV-siRNA组的CTSV mRNA转录水平和蛋白表达水平高于对照组,但明显低于SAHA组,见Fig 2。这些结果表明,SAHA对CTSV具有上调作用,CTSV-siRNA可有效抑制CTSV mRNA转录和蛋白表达水平。

|
| Fig 2 Effect of SAHA on MCF-7 breast cancer cells accompanied by increase of CTSV function (x±s, n=3) A: Western blot assay for MCF-7 cells; B: Real-time PCR assay for MCF-7 cells. *P<0.05 vs Basal; #P<0.05 vs SAHA. (CVi: CTSV siRNA). |
为了进一步探讨SAHA-CTSV诱导乳腺癌细胞自噬的作用,利用激光共聚焦显微镜成像观察SAHA-CTSV对细胞自噬标志分子LC3蛋白表达的影响。如Fig 3所示,SAHA作用细胞后,细胞自噬强度明显增强;而CTSV-siRNA转染后,LC3蛋白集中分布在细胞膜周围,并呈散簇状,荧光亮度也有所减弱。此外,观察SAHA诱导CTSV siRNA干扰后的细胞自噬的影响发现,LC3蛋白荧光也分布在细胞膜周围,但分布范围小于未经SAHA处理细胞组,不过LC3蛋白荧光仍为块状分布,无明显散簇状,而且荧光亮度也强于未经SAHA处理细胞组。这些结果表明SAHA-CTSV能够诱发MCF-7细胞发生高强度的自噬,升高LC3蛋白的表达水平。

|
| 图 3 Fluorescence microscopy for SAHA-CTSV induced autophagy(CVi: CTSV siRNA). |
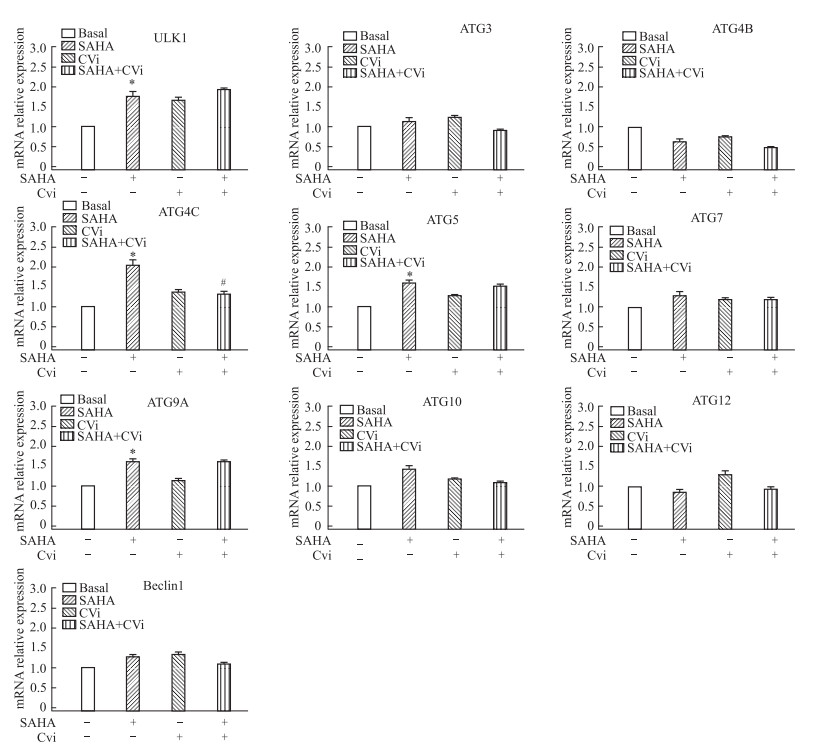
为更深入研究SAHA-CTSV诱导乳腺癌发生自噬的机制,特别是自噬相关分子ATGs和LC3在自噬过程中的变化。我们通过Real-time PCR和Western blot进行检测,结果显示,在MCF7细胞中,SAHA增强了ULK1、ATG4C、ATG5和ATG9A的功能,转染的CTSV siRNA只抑制了ATG4C的表达,对其他ATGs活性没有影响,见Fig 4。对相关蛋白表达水平分析,结果显示当SAHA作用于MCF-7细胞时,细胞中ATG4C和Beclin1的表达量上升,而同时LC3B蛋白的表达量也随之增加,但mTOR蛋白的表达量却降低。CTSV表达被抑制后,SAHA诱导强自噬效应也随之被降低,见Fig 5。结果表明SAHA-CTSV在MCF-7细胞自噬过程中起到重要作用。
|
| Fig 4 The mRNA expressions of autophagy related molecules induced by SAHA-CTSV in MCF-7 breast cancer cells (x±s, n=3) *P<0.05 vs Basal; #P<0.05 vs SAHA (CVi: CTSV siRNA). |

|
| Fig 5 Effect of SAHA-CTSV on protein expressions of autophagy related molecules Western blot assay for MCF-7 cells (x±s, n=3) *P<0.05 vs Basal; #P<0.05 vs SAHA (CVi: CTSV siRNA). |
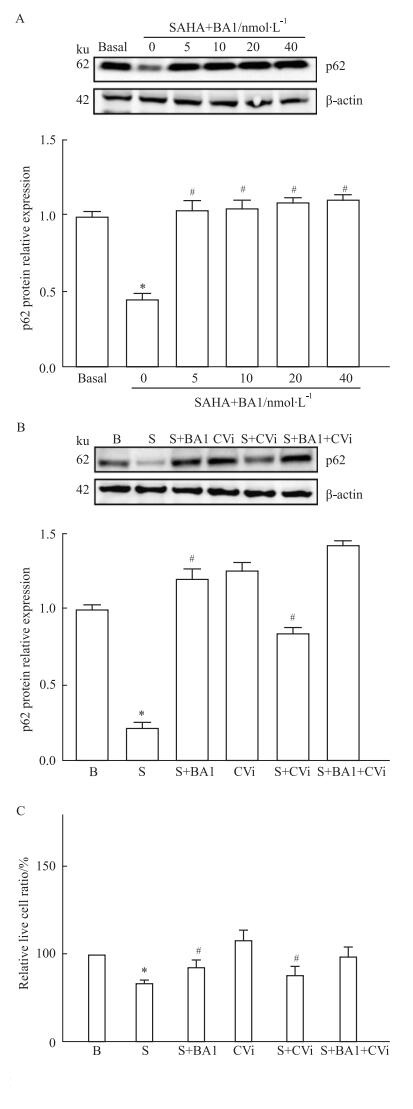
我们通过Bafilomycin A1阻断自噬的传递来验证SAHA-CTSV对MCF-7细胞自噬的促进作用。首先,利用细胞自噬抑制剂Bafilomycin A1抑制SAHA对MCF-7细胞的自噬诱导作用,使用浓度分别为0、5、10、20、40 nmol·L-1的Bafilomycin A1与SAHA孵育的MCF-7细胞作用24 h。Western blot检测结果显示,与SAHA组相比,从5 nmol·L-1 Bafilomycin A1开始,随着抑制剂浓度的增加,p62蛋白的表达水平也随之有显著增加。其次,将5 nmol·L-1 Bafilomycin A1和SAHA共同加入CTSV-siRNA的MCF-7细胞24 h,结果分析显示在CTSV表达被抑制后,SAHA诱导细胞自噬并导致细胞死亡的作用被明显降低,无法达到显著的抗肿瘤作用。随后的细胞活力分析结果显示,与SAHA组相比,Bafilomycin A1和SAHA共孵育组中活细胞数量增加。此外,CTSV mRNA转录被抑制后,SAHA诱导细胞自噬的强度也随之减弱,见Fig 6。结果表明,CTSV在SAHA诱导的MCF-7细胞自噬过程中起着重要的作用。
|
| Fig 6 The anti-tumor effect of SAHA-CTSV in environment that autophagic flux was block by Bafilomycin A1 (x±s, n=3) *P<0.05 vs Basal; #P<0.05 vs SAHA. (B: Basal, S: SAHA, CVi: CTSV siRNA, BA1: Bafilomycin A1). |
癌症的发病率出现逐年增多的趋势,全球每年新发病例将达到1406.8万,其中乳腺癌是全世界女性癌症发病之首[4-6]。作为二代组蛋白去乙酰化酶抑制剂,SAHA在许多生物学功能上起着极其重要的作用,已知的研究表明SAHA是通过抑制组蛋白去乙酰化酶活性,从而改变染色质的结构,在基因水平上调节细胞活动[7-9]。我们将SAHA与乳腺癌MCF-7细胞共同孵育,利用Muse自动细胞分析仪发现,SAHA可以降低MCF-7细胞活力,抑制MCF-7细胞增殖。CTSV又称为组织蛋白酶L2,属于半胱胺酸蛋白酶体系,是11种人半胱氨酸组织蛋白酶中的一员[10],我们通过实时荧光定量PCR和Western blot证实,在SAHA抑制MCF-7细胞增殖时,CTSV的表达量显著增加。
自噬是一种细胞内降解系统,通过降解蛋白质、细胞器和微生物等多种靶点,促进细胞内稳态[11-13]。自噬具有双重性,一方面,自噬是一种保护机制,保护细胞受到药物等外界一些刺激因素引起的死亡,进而产生耐药。另一方面,保护也不是绝对的,当超过一定限度时,自噬可以诱导或参与细胞死亡,这就是自噬性细胞死亡,这也是肿瘤化疗时的一种重要死亡方式。为了验证自噬是否与SAHA诱导的细胞死亡有关,我们检测了自噬标志物LC3,结果表明,SAHA能显著促进MCF-7细胞中LC3的表达。利用siRNA干扰技术抑制CTSV转录后,在SAHA作用细胞时LC3蛋白的表达量也呈下降趋势,与此同时细胞的死亡率也随之下降。这些结果说明了在乳腺癌细胞因自噬而死亡的过程中SAHA-CTSV轴起着关键作用。
为进一步发现SAHA-CTSV轴诱导自噬发生发展的作用机制,本研究采用实时定量PCR和Western blot检测自噬过程中的主要分子转录及表达情况。数据显示当SAHA作用于MCF-7细胞时,细胞中ATG4C和Beclin1的表达量上升,而同时LC3B蛋白的表达量也随之增加,但mTOR蛋白的表达量却随之降低。此外,我们用Bafilomycin A1阻断自噬通量后,发现被SAHA抑制的细胞活力和p62蛋白的表达显著增加。以上结果提示,SAHA-CTSV轴通过诱导乳腺癌MCF-7细胞的过度自噬发挥抗肿瘤作用。
总之,我们的实验结果说明CTSV参与并介导了SAHA诱发MCF-7细胞自噬并死亡的过程。在SAHA诱导细胞自噬过程中,首先通过CTSV激活ATG4C和Beclin1调高其蛋白表达量,然后激活并诱导下游自噬关键分子LC3的合成,并伴有mTOR相关信号通路的转导,最终引起MCF-7细胞过度自噬从而引发细胞死亡。综上所述,本研究深入地阐明了SAHA-CTSV轴作用于乳腺癌MCF-7细胞调控增殖、诱导自噬性细胞死亡的分子机制,同时为解决SAHA的耐药问题提供理论依据,拓宽乳腺癌研究方向,为更好地理解SAHA-CTSV轴在乳腺癌发生、发展中的规律提供了实验依据。
( 致谢: 衷心感谢沈阳医学院辽宁省环境污染与微生态重点实验室为本实验提供的实验设备,感谢实验室全体工作人员对本实验的大力协助。)
| [1] |
陈万青, 郑荣寿. 中国女性乳腺癌发病死亡和生存状况[J]. 中国肿瘤临床, 2015, 42(13): 668-74. Chen W Q, Zheng R S. Incidence, mortality and survival analysis of breast cancer in China[J]. Chin J Clin Oncol, 2015, 42(13): 668-74. |
| [2] |
Goldstein L J, Zhao F, Wang M, et al. A Phase Ⅰ/Ⅱ study of suberoylanilide hydroxamic acid(SAHA)in combination with trastuzumab(Herceptin)in patients with advanced metastatic and/or local chest wall recurrent HER2-amplified breast cancer: A trial of the ECOG-ACRIN Cancer Research Group(E1104)[J]. Breast Cancer Res Treat, 2017, 165(2): 375-82. doi:10.1007/s10549-017-4310-9 |
| [3] |
Bromme D, Li Z, Barnes M, et al. Human cathepsin V functional expression, tissue distribution, electrostatic surface potential, enzymatic characterization, and chromosomal localization[J]. Biochemistry, 1999, 38(8): 2377-85. doi:10.1021/bi982175f |
| [4] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. doi:10.3322/caac.21492 |
| [5] |
Torre L A, Bray F, Siegel R L, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87-108. doi:10.3322/caac.21262 |
| [6] |
DeSantis C, Ma J, Bryan L, et al. Breast cancer statistics, 2013[J]. CA Cancer J Clin, 2014, 64(1): 52-62. doi:10.3322/caac.21203 |
| [7] |
Han H, Zhou H, Li J, et al. TRAIL DR5-CTSB crosstalk participates in breast cancer autophagy initiated by SAHA[J]. Cell Death Discov, 2017, 3: 17052. doi:10.1038/cddiscovery.2017.52 |
| [8] |
Han H, Li J, Feng X, et al. Autophagy-related genes are induced by histone deacetylase inhibitor suberoylanilide hydroxamic acid via the activation of cathepsin B in human breast cancer cells[J]. Oncotarget, 2017, 8: 53352-65. doi:10.18632/oncotarget.18410 |
| [9] |
李静, 周伟强. SAHA、TRAIL与乳腺癌细胞凋亡[J]. 沈阳医学院学报, 2016, 18(3): 183-6. Li J, Zhou W Q. SAHA, TRAIL and apoptosis in breast cancer[J]. J Shenyang Med Coll, 2016, 18(3): 183-6. |
| [10] |
周慧, 翰翰, 周伟强. SAHA靶定组织蛋白酶V诱导乳腺癌MDA-MB-231细胞自噬[J]. 中国药理学通报, 2020, 36(11): 1563-8. Zhou H, Han H, Zhou W Q. SAHA targeted CTSV for autophagy induction in MDA-MB-231 breast cancer cells[J]. Chin Pharmacol Bull, 2020, 36(11): 1563-8. doi:10.3969/j.issn.1001-1978.2020.11.016 |
| [11] |
Goodwin J M, Dowdle W E, DeJesus R, et al. Autophagy-independent lysosomal targeting regulated by ULK1/2-FIP200 and ATG9[J]. Cell Rep, 2017, 20: 2341-56. doi:10.1016/j.celrep.2017.08.034 |
| [12] |
Nikoletopoulou V, Markaki M, Palikaras K, Tavernarakis N. Crosstalk between apoptosis, necrosis and autophagy[J]. Biochim Biophys Acta, 2013, 1833: 3448-59. doi:10.1016/j.bbamcr.2013.06.001 |
| [13] |
于存治, 戚新明, 任进. 靶向乳腺癌耐药蛋白逆转肿瘤耐药: 研究进展与药物开发[J]. 中国药理学通报, 2014, 30(5): 615-8. Yu C Z, Qi X M, Ren J. BCRP-targeted reverse of multidrug resistance: Research progress and drug development[J]. Chin Pharmacol Bull, 2014, 30(5): 615-8. doi:10.3969/j.issn.1001-1978.2014.05.006 |

