

流行病学研究表明,高尿酸血症已成为危害工业化国家健康的主要问题,其患病率在全世界范围内逐年升高[1]。近年来,有研究通过纵向观察和介入研究提出,高尿酸血症与慢性肾病之间的关系,并提示血清尿酸水平与肾功能损伤的发生和发展具有密切相关性[2]。组织病理学研究显示,79%~99%的高尿酸血症患者都伴有肾脏损伤[3]。因此,防治高尿酸血症慢性肾病具有十分重要的意义。
高尿酸血症诱导慢性肾病的发病机制十分复杂,且有多种途径,如激活白细胞介素(interleukin,IL)-6,信号传导与转录激活因子3和活性氧簇[4]。在肾脏中,尿酸能在人肾近端管状上皮细胞中诱导单核细胞趋化蛋白1的产生[5]和激活丝裂原活化蛋白激酶信号通路[6]。尿酸还可激活NOD样受体蛋白3,并刺激分泌IL-1β和IL-18。而IL-1β和IL-18的激活也是导致肾功能损伤的原因之一[7]。尽管很多致病因素已经被发现,但高尿酸血症诱发慢性肾病的机制仍然需要深入研究。
代谢组学是继基因组学、蛋白质组学和转录组学之后的一门新兴学科,主要用于发现生物体整体代谢变化,揭示机体生命活动的代谢本质,通常被用作医学领域的研究,包括疾病诊断、生物标志物筛查,以及化学物质安全性评估[8]。
本文利用1H-NMR技术研究慢性高尿酸血症肾病模型大鼠尿液和肾组织中的代谢特征,鉴定其差异代谢物和代谢通路。旨在为高尿酸血症所致慢性肾病的临床诊断和治疗提供潜在生物标志物,并为进一步机制研究奠定坚实理论基础。
1 材料与方法 1.1 材料 1.1.1 实验动物SPF级Wistar大鼠16只,♂,体质量(200±20)g,购于北京维通利华实验动物技术有限公司,许可证号:SCXK(京) 2016-0006。饲养温度(22 ±2) ℃,湿度(55±5)%,噪音 < 85 dB,提供12 h/12 h白天和黑夜循环照明,自由饮食饮水。将大鼠分为两组,对照组饲喂正常基础饲料,高尿酸血症组饲喂2%尿酸和2%氧嗪酸钾的高尿酸复合饲料,连续饲喂12周,本实验所有动物操作均严格按照西南民族大学动物保护和使用规定。
1.1.2 主要试剂尿酸(uric acid)、氧嗪酸钾(oxonic acid)(南京康满林化工实业有限公司);血清尿酸(serum uric acid,SUA)、血清葡萄糖(serum glucose,GLU)、血清尿素氮(blood urea nitrogen,BUN)、血清肌酐(serum creatinine,CREA)血生化试剂盒(四川迈克生物科技股份有限公司);氘水(D2O)(美国Sigma公司)。
1.1.3 仪器DP80显微镜(日本Olympus公司);石蜡组织包埋机(德国Leica公司);石蜡组织冷冻机(德国Leica公司);病理切片机(德国Leica公司);PHY-Ⅲ病理组织漂烘处理仪(常州中威电子仪器厂);高速低温离心机(德国Heraeus sepatech公司);7020全自动血生化仪(日本Hitachi公司);Bruker 600 MHz AVANCE Ⅱ NMR (德国Bruker公司)。
1.2 血清生化指标水平检测10%水合氯醛腹腔注射麻醉慢性高尿酸血症肾病模型大鼠,抽取腹主动脉血,经凝固(4 ℃, 60 min)后离心(3 000 r·min-1, 15 min, 4 ℃)得到血清。SUA、GLU、BUN和CREA测定严格按照血生化试剂盒说明书操作。
1.3 肾脏组织病理学观察取大鼠肾脏,固定于4%多聚甲醛中,梯度乙醇脱水,石蜡包埋。将蜡块切成4 μm厚切片,经HE染色,然后用DP80显微镜进行肾脏组织病理学观察。
1.4 代谢组学检测及分析 1.4.1 尿液采集和预处理将大鼠尿液样本收集到含有1%叠氮化钠容器中低温放置过夜。离心(3 500 r·min-1, 15 min, 4 ℃),除去沉淀,收集上清液。为了尽量减少尿液pH差异所引起的化学位移变化,将400 μL尿液与200 μL磷酸盐缓冲液(0.2 mol·L-1 Na2HPO4-0.2 mol·L-1 NaH2PO4, pH 7.4)混合,将混悬液静置20 min后离心(12 000 r·min-1, 10 min, 4 ℃)。然后将500 μL上清液与50 μL D2O(含有0.05% W/V TSP-d4)加入5 mm NMR管中,振荡混匀,待测。
1.4.2 肾脏提取和预处理大鼠在取样前应禁食过夜,且所有的样本均保存在-80 ℃,待分析时取用。将肾组织样品称重,冰水浴中用50%氯仿-水溶液匀浆(5 mL·g-1肾组织),离心(12 000 r·min-1, 10 min, 4 ℃)。将混悬液静置冰上30 min。取上清,40 ℃氮气吹干。加入580 μL含有磷酸缓冲液(0.2 mol·L-1Na2HPO4-0.2 mol·L-1NaH2PO4, pH 7.4)和D2O(含有0.05% W/V TSP-d4)中,离心(12 000 r·min-1, 10 min, 4 ℃),取550 μL上清液转移到5 mm NMR管中,振荡混匀,待测[8]。
1.4.3 1H-NMR测定所有尿液和肾组织样品均在600.13 MHz的核磁共振氢谱下检测,实验温度为26.85 ℃,宽度为12 335.5 Hz,采集时间为2.66 s,弛豫延迟时间为7.66 s,扫采样点数64 K;采集次数128次。
1.4.4 1H-NMR图谱处理及多变量数据分析所有1H-NMR光谱用MestReNova-6.1-6384软件(西班牙Mestrelab Research公司)对相位和畸变基线进行校正。经过相位的调整和基线的校正后,尿液的谱图以加入的TSP-d4(δ 0.00)为标准,对谱图进行定标,而肾组织中乳酸峰相对稳定,故用乳酸(δ 1.336)为标准对谱图进行化学位移校正,将光谱分割为4500段,范围从δ 0.50~9.50,每个区域宽度相等(δ 0.002),水峰δ 4.70~5.02区域切除。在样品识别分析之前对集成数据进行归一化,以消除由组织重量不同而导致的样品之间的稀释差异或体积质量差异。
将归一化后生成的数据导入SIMCA-P 13.0(Umetrics,Ume,Sweden)中进行多元统计分析,首先采用PCA的无监督成分分析方法分析核磁共振光谱数据,以分离样本,随后采用PLS-DA和OPLS-DA的监督成分分析方法更好地进行样本的分离。为了防止过度拟合,通过置换分析(200次)和交叉验证法,对OPLS-DA模型进行验证。同时进行模式质量评价,用R2X、R2Y、Q2等指标进行质量评价,其中R2X表示X的变异百分比,R2Y表示Y的变异百分比,Q2表示累计预测结果的真实性。将OPLS-DA模型中所有峰值的变量和变量重要性投影(variable importance in the projection,VIP)的值作为峰值选择的系数,并认为具有VIP>1的变量与群体分化有关[9]。此外,采用Tukey’s test (P < 0.05)进行分析,仅对具有统计学意义P < 0.05的变量的数据进行鉴别,并根据P < 0.05和VIP>1筛选出差异代谢物。
1.4.5 差异代谢物识别采用双参数法归属差异代谢物,参照前期相关的文献和代谢组学的数据库网站(http://www.hmdb.ca/),只有同时满足相应的化学位移和峰多态性的化合物,才被认为是真实发生变化的代谢物。再结合多变量数据和统计学分析结果,以VIP>1和P < 0.05为筛选条件,最终确定尿液和肾脏中的差异代谢物。
1.5 统计学方法所有实验结果均采用SPSS 19.0软件进行统计学分析,两组之间的比较采用独立样本t检验,并用x±s表示。
2 结果 2.1 慢性高尿酸血症肾病模型大鼠肾脏组织病理学及血清生化水平检测肾脏组织病理学检测显示,慢性高尿酸血症大鼠的肾脏已产生病理性萎缩,肾脏表面呈现不规则凹陷,髓质出现淡黄色斑点和放射状纹路,而皮质边缘变模糊(Fig 1A-D),并观察到肾髓质、乳头和锥体中的尿酸晶体沉积以及肾间质的炎症,且有肾小球硬化、间质纤维化、肾小动脉硬化(Fig 2A-D)。以上结果表明,高尿酸血症可诱发大鼠慢性肾病。
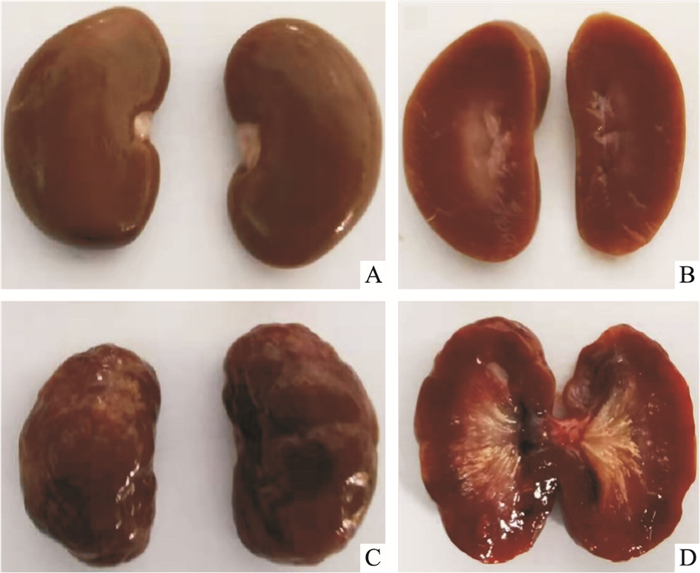
|
| Fig 1 Kidney typical photos of control and hyperuricaemia rats A: Typical kidney photo of control rats; B: Kidney longitudinal section photo of control rats; C: Typical kidney photo of hyperuricaemia rats; D: Kidney longitudinal section photo of hyperuricaemia rats. |
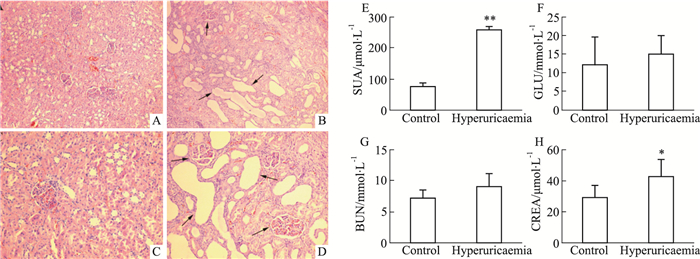
|
| Fig 2 Renal histopathology and serum biochemical results of hyperuricaemia and control rats(x±s, n=8) A: Kidney microscopic image of control rats (×200); B: Kidney microscopic image of hyperuricaemia rats (×200); C: Kidney microscopic image of control rats (×400); D: Kidney microscopic image of hyperuricaemia rats (×400); E: SUA; F: GLU; G: BUN; H: CREA. *P < 0.05, **P < 0.01 vs control. |
采用血生化方法测定慢性高尿酸血症模型大鼠相关指标水平。正常大鼠SUA为75.8 μmol·L-1,而模型大鼠为258.3 μmol·L-1,相比增加3倍,表明高尿酸血症大鼠模型构建成功(Fig 2E)。对照组大鼠BUN和CREA水平分别为7.2 mmol·L-1和29.2 μmol·L-1,而模型大鼠分别为9.0 mmol·L-1和42.3 μmol·L-1,表明高尿酸血症大鼠出现肾功能损伤(Fig 2G, 2H)。高尿酸血症大鼠血清GLU也增加20%(Fig 2F)。
2.2 尿液和肾组织代谢轮廓分析慢性高尿酸血症肾病大鼠尿液和肾组织的核磁图谱见Fig 3。根据化学位移、峰形、偶合常数,并结合Metabolome数据库对图谱进行确认,在大鼠尿液中发现51个差异代谢物,肾组织中发现48个差异代谢物,包含大量参与多种生化过程的代谢物,如葡萄糖、氨基酸、NAD+和腺嘌呤等。
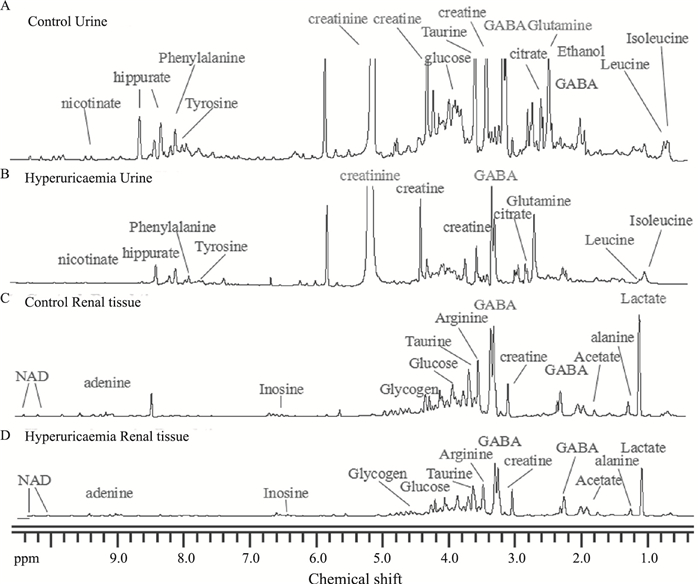
|
| Fig 3 600 MHz representative 1H-NMR spectra of urine and renal tissue samples from hyperuricaemia and control rats A: Urine samples of control rats; B: Urine samples of hyperuricaemia rats; C: Renal tissue samples of control rats; D: Renal tissue samples of hyperuricaemia rats. |
采用PCA散点图对所有样本进行分析,以显示数据的原始分类状态,可以在尿液(R2=0.872, Q2=0.702)和肾脏(R2=0.804, Q2=0.686)的PCA散点图中均观察到明显的分离(Fig 4A, 4D),表明模型组大鼠尿液和肾脏中发生明显的代谢紊乱。为优化两组分离,本研究采用7倍交叉验证法对OPLS-DA模型的可靠性进行验证,图 4B (R2X=0.758, R2Y=0.991, Q2=0.917)和图 4E (R2X=0.776, R2Y=0.923, Q2=0.765)分别为尿液和肾脏的OPLS-DA散点图,同样观察到明显的分离,表明慢性高尿酸血症肾病大鼠机体生理及物质代谢发生明显改变。
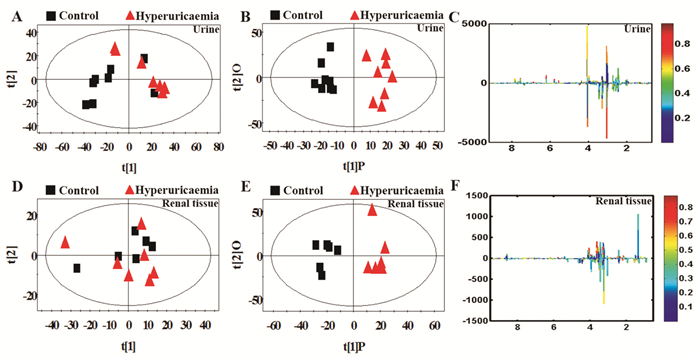
|
| Fig 4 Metabolite profiles of urine and renal tissue samples between hyperuricaemia and control rats A: PCA scores plot, B: OPLS-DA scores plots, C: Color map of the urine samples between hyperuricaemia and control rats; D: PCA scores plot, E: OPLS-DA scores plots, F: Color map of the renal tissue samples between hyperuricaemia and control rats. |
为进一步确定正常组和模型组的主要差异代谢物,从OPLS-DA分析中得到评分和相关系数载荷图,根据UV模型的可变权重对载荷进行着色,如图(Fig 4C, 4F)显示出两组差异代谢物,根据两组之间的化学位移(VIP>1和P < 0.05),鉴定出多种差异代谢物(Tab 1, 2)。这些代谢产物均参与糖酵解、三羧酸循环(tricarboxylic acid cycle,TCA)、尿素循环、氨基酸和蛋白质代谢、能量代谢以及抗氧化活性等关键代谢途径。
| Metabolite | Chemical shift (ppm) | Mutiplicity | VIPa | P-valueb | FCc |
| α-Hydroxy-N-valerate | 1.68 | m | 2.13 | 0.001 8 | -1.20 |
| β-Hydroxybutyrate | 1.20 | d | 1.84 | 0.009 3 | -1.03 |
| 4.16 | m | 2.17 | 0.003 8 | -1.01 | |
| 3-Hydroxybutyrate | 1.20 | d | 2.17 | 0.003 8 | -1.01 |
| 4.13 | m | 1.84 | 0.009 3 | -1.03 | |
| 3-Hydroxyisovalerate | 1.271 | s | 1.06 | 0.046 5 | 1.04 |
| 2-Hydroxyisovalenc acid | 4.13 | d | 1.44 | 0.008 8 | -1.06 |
| 2-Hydroxybutyric acid | 2.30 | m | 1.48 | 0.028 0 | 1.07 |
| 2-Hydroxy-glutarate | 7.18 | d | 2.02 | 0.005 9 | 1.09 |
| 4-Hydroxybutyric acid | 1.83 | m | 1.42 | 0.041 0 | 1.24 |
| 2.44 | t | 2.02 | 0.005 9 | 1.09 | |
| 4-Hydroxyphenylactate | 1.541 | m | 1.85 | 0.008 1 | 1.59 |
| Caprylate | 1.541 | m | 1.20 | 0.021 3 | 1.13 |
| 2-Aminoadipic acid | 1.64 | m | 1.48 | 0.028 0 | 1.07 |
| 1.89 | m | 1.40 | 0.039 8 | 1.06 | |
| GABA (γ-aminobutyric acid) | 2.10 | m | 1.56 | 0.003 9 | -1.11 |
| N-Acetylglutamate | 1.84 | m | 2.02 | 0.005 9 | 1.09 |
| 2.04 | s | 1.62 | 0.034 2 | 1.04 | |
| Glutamate | 2.35 | m | 1.45 | 0.031 4 | 1.09 |
| 3.77 | m | 1.47 | 0.026 1 | 1.06 | |
| Glutamine | 2.14 | m | 1.47 | 0.026 1 | 1.06 |
| 3.77 | m | 1.42 | 0.041 0 | 1.21 | |
| Malate | 2.41 | m | 1.56 | 0.043 2 | -1.17 |
| Malonate | 3.133 | s | 1.41 | 0.046 0 | -1.05 |
| Aspartate | 2.82 | dd | 2.00 | 0.006 6 | 1.10 |
| Guanidinosuccinic acid | 2.83 | dd | 2.09 | 0.001 1 | 1.32 |
| 4.24 | dd | 2.00 | 0.006 6 | 1.10 | |
| Taurine | 3.27 | t | 1.68 | 0.014 8 | -1.36 |
| Arginine | 3.25 | t | 1.68 | 0.014 8 | -1.36 |
| β-Glucose | 4.65 | d | 1.92 | 0.011 5 | -1.21 |
| Myo-Inositol | 3.55 | dd | 1.68 | 0.014 8 | -1.36 |
| o-HPA | 3.66 | s | 1.31 | 0.026 2 | -1.32 |
| p-HPA | 3.90 | s | 1.44 | 0.003 5 | -1.16 |
| 7.28 | m | 1.60 | 0.013 4 | -1.02 | |
| 7.71 | d | 1.32 | 0.007 1 | -1.05 | |
| Mannitol | 3.77 | m | 1.26 | 0.023 9 | -1.01 |
| N-Methylnicotinamide | 8.21 | m | 2.02 | 0.001 7 | -1.95 |
| 8.90 | d | 1.65 | 0.013 3 | -1.86 | |
| 8.97 | d | 1.89 | 0.007 0 | -1.79 | |
| N-Acetyltyrosine | 7.16 | d | 1.85 | 0.008 1 | 1.59 |
| Hippurate | 3.97 | d | 1.60 | 0.003 3 | 1.45 |
| 7.55 | t | 1.49 | 0.048 4 | 1.35 | |
| 7.64 | t | 2.41 | 0.000 0 | 1.24 | |
| Glycine | 3.57 | s | 1.71 | 0.027 4 | 1.34 |
| Glycolate | 3.93 | s | 2.23 | 0.000 8 | 1.20 |
| PAG(Phenylacetylglycine) | 7.33 | d | 1.31 | 0.026 2 | -1.32 |
| Phenylalanine | 7.33 | d | 1.31 | 0.026 2 | -1.32 |
| Creatine | 3.04 | s | 1.62 | 0.017 7 | 1.30 |
| 3.94 | s | 2.23 | 0.000 8 | 1.20 | |
| 2.20 | 0.000 3 | 1.01 | |||
| Creatinine | 3.04 | s | 1.90 | 0.005 3 | 1.10 |
| 4.05 | s | 2.20 | 0.000 3 | 1.01 | |
| 2-Oxoisovaleric acid | 1.13 | d | 1.59 | 0.034 0 | -1.27 |
| Isobutyrate | 1.12 | d | 1.59 | 0.034 0 | -1.26 |
| Isoleucine | 0.95 | t | 2.13 | 0.001 8 | -1.20 |
| 3-Methyladenine | 3.97 | d | 2.41 | 0.000 0 | 1.24 |
| 7-Methyladenine | 8.20 | s | 1.90 | 0.005 3 | 1.10 |
| Methylmalonate | 1.25 | d | 1.84 | 0.009 3 | -1.03 |
| Leucine | 0.96 | t | 2.13 | 0.001 8 | -1.20 |
| Phosphocreatine | 3.04 | s | 2.23 | 0.000 8 | 1.20 |
| 3.93 | s | 2.20 | 0.0003 | 1.01 | |
| Triacylglycerol | 4.28 | m | 2.23 | 0.000 9 | 1.10 |
| Ethanol | 1.19 | t | 1.91 | 0.017 6 | -1.15 |
| Trytophan | 7.29 | m | 1.44 | 0.003 5 | -1.16 |
| Nicotinate | 8.84 | d | 2.29 | 0.000 4 | -1.15 |
| Butyrate | 1.563/1.613 | m | 1.20 | 0.021 3 | 1.13 |
| Citrulline | 1.57 | m | 1.20 | 0.021 3 | 1.13 |
| Proprionate | 1.04 | t | 1.64 | 0.010 6 | 1.11 |
| Fumarate | 6.53 | s | 1.55 | 0.025 8 | -1.11 |
| aVariable importance in the projection (VIP>1) was obtained from OPLS-DA analysis; bp-value determined from Tukey’s-test; cFC: Fold change between hyperuricaemia and control rats. Positivesign indicates a higher level in hyperuricaemia rats and a negative value indicates a lower level. s, singlet; d, doublet; t, triplet; q, quartet; dd, doublet of doublets; m, multiplet. | |||||
| Metabolite | Chemical shift (ppm) | Mutiplicity | VIPa | P-valueb | FCc |
| α-Hydroxy-N-valerate | 1.68 | m | 2.12 | 0.008 5 | -1.03 |
| 4.03 | m | 1.95 | 0.019 1 | -1.86 | |
| 1.20 | d | 1.73 | 0.043 6 | -1.18 | |
| Cholate | 1.20 | d | 1.77 | 0.038 9 | 1.08 |
| β-Hydroxybutyrate | 1.73 | m | 1.85 | 0.029 1 | 1.38 |
| 3-Hydroxybutyrate | 1.85 | m | 1.85 | 0.029 1 | 1.38 |
| 2-Hydroxybutyric acid | 2.30 | m | 1.80 | 0.034 1 | -1.04 |
| 2-Hydroxy-glutarate | 3.62 | t | 2.15 | 0.007 5 | 1.04 |
| 7.18 | d | 2.12 | 0.008 7 | 1.19 | |
| 4-Hydroxybutyric acid | 1.48 | d | 2.51 | 0.000 6 | 1.01 |
| 4-Hydroxyphenylactate | 1.541 | m | 1.81 | 0.033 6 | 1.03 |
| Alanine | 1.89 | m | 1.69 | 0.049 7 | -1.19 |
| Caprylate | 1.91 | m | 2.42 | 0.001 3 | -1.62 |
| 2-Aminoadipic acid | 3.01 | t | 2.15 | 0.007 5 | 1.04 |
| GABA (γ-aminobutyric acid) | 2.10 | m | 1.97 | 0.017 6 | -1.28 |
| N-Acetylglutamate | 2.35 | m | 2.06 | 0.011 8 | -1.09 |
| Glutamate | 2.14 | m | 2.53 | 0.000 5 | 1.03 |
| Glutamine | 3.77 | m | 2.53 | 0.000 5 | 1.03 |
| 2.56 | m | 2.36 | 0.001 9 | 1.16 | |
| Glutathione | 2.82 | br | 2.27 | 0.003 7 | 2.17 |
| D-Ribose | 2.23 | s | 2.21 | 0.005 3 | 1.30 |
| Acetone | 2.23 | s | 2.21 | 0.005 3 | 1.30 |
| Malate | 2.41 | m | 2.22 | 0.004 8 | -1.14 |
| Aspartate | 2.82 | dd | 1.72 | 0.045 9 | 1.18 |
| Guanidinosuccinic acid | 2.83 | dd | 1.72 | 0.045 9 | 1.18 |
| 6-Methyladenine | 2.95 | br | 2.27 | 0.003 7 | 2.17 |
| α-Ketoglutarate | 3.01 | t | 1.97 | 0.017 6 | -1.28 |
| Albumin lysyl | 3.01 | t | 1.97 | 0.017 6 | -1.28 |
| Taurine | 3.27 | t | 2.10 | 0.009 7 | -1.09 |
| Arginine | 3.25 | t | 2.10 | 0.009 7 | -1.09 |
| α-Glucose | 3.54 | dd | 2.13 | 0.008 0 | -1.01 |
| 3.72 | t | 1.89 | 0.024 8 | -1.48 | |
| 3.74 | m | 1.76 | 0.039 8 | -1.20 | |
| 3.84 | m | 2.25 | 0.004 1 | -1.31 | |
| β-Glucose | 4.65 | d | 1.83 | 0.031 3 | -1.60 |
| Myo-Inositol | 3.55 | dd | 2.13 | 0.008 0 | -1.01 |
| o-HPA | 3.66 | s | 2.41 | 0.001 4 | -1.40 |
| 7.33 | d | 1.78 | 0.037 6 | -1.02 | |
| 7.36 | t | 1.89 | 0.024 4 | -1.04 | |
| 7.42 | dd | 2.01 | 0.014 4 | -1.03 | |
| p-HPA | 7.28 | m | 1.99 | 0.016 3 | 1.08 |
| Mannitol | 3.69 | m | 2.22 | 0.004 9 | -1.31 |
| 3.77 | m | 1.89 | 0.024 8 | -1.25 | |
| 3.81 | m | 2.25 | 0.004 1 | -1.31 | |
| Lysine | 3.77 | m | 1.89 | 0.024 8 | -1.25 |
| Glycogen | 3.97 | m | 2.26 | 0.003 9 | -1.01 |
| Serine | 3.98 | m | 2.26 | 0.003 9 | -1.01 |
| Lactate | 4.11 | q | 1.79 | 0.035 6 | -1.15 |
| Glyceryl | 4.30 | m | 2.35 | 0.002 2 | -1.24 |
| CH2OCOR | 4.30 | m | 2.35 | 0.002 2 | -1.24 |
| Urea | 5.78 | br | 2.45 | 0.001 0 | 13.61 |
| Inosine | 6.11 | d | 2.09 | 0.010 2 | -1.19 |
| 8.24 | s | 1.79 | 0.035 4 | -1.24 | |
| Tyrosine | 7.20 | d | 1.94 | 0.019 9 | 1.26 |
| PAG(Phenylacetylglycine) | 7.33 | d | 1.78 | 0.037 6 | -1.02 |
| 7.36 | t | 1.89 | 0.024 4 | -1.04 | |
| 7.42 | dd | 2.01 | 0.014 4 | -1.03 | |
| Phenylalanine | 7.33 | d | 1.78 | 0.037 6 | -1.02 |
| 7.38 | t | 1.89 | 0.024 4 | -1.04 | |
| 7.42 | dd | 2.01 | 0.014 4 | -1.03 | |
| Adenine | 8.12 | m | 1.96 | 0.018 5 | 1.43 |
| N-Methylnicotinamide | 8.21 | m | 1.72 | 0.044 8 | -1.39 |
| 7-Methyladenine | 8.20 | s | 1.79 | 0.035 4 | -1.24 |
| Nicotinate | 8.84 | d | 1.82 | 0.031 4 | -1.52 |
| NAD+ | 8.83 | d | 1.82 | 0.031 4 | -1.52 |
| 9.35 | s | 1.85 | 0.028 9 | -1.47 | |
| aVariable importance in the projection (VIP>1) was obtained from OPLS-DA analysis; bp-value determined from Tukey’s-test; cFC: fold change between hyperuricaemia and control rats. Positivesign indicates a higher level in hyperuricaemia rats and a negative value indicates a lower level. s, singlet; d, doublet; t, triplet; q, quartet; dd, doublet of doublets; m, multiplet. | |||||
综合分析以上结果显示,在慢性高尿酸血症大鼠尿液和肾脏中分别发现51和48个潜在差异代谢物,在尿液中,马尿酸、谷氨酸、天冬氨酸、甘氨酸、谷氨酰胺以及甲基腺嘌呤等代谢物明显增加;而葡萄糖、γ-氨基丁酸(GABA)、牛磺酸、精氨酸、苹果酸、富马酸、烟酸盐、甘露醇和肌醇则明显减少。在肾脏中,而尿素、天冬氨酸和酪氨酸明显增加;而葡萄糖、GABA、牛磺酸、精氨酸、甘露醇、NAD+、烟酸、苹果酸以及α-酮戊二酸等代谢物则明显减少。综合两组数据发现,葡萄糖、牛磺酸、精氨酸、甲基腺嘌呤和烟酸在尿液和肾脏中都发生明显变化,且主要涉及糖酵解、TCA循环和氨基酸代谢途径。这些代谢物可能是慢性高尿酸血症肾病大鼠的潜在生物标志物(Fig 5)。

|
| Fig 5 Fold change of metabolites of urine and renal tissue samples from hyperuricaemia and control rats |
基于1H-NMR的代谢组学技术对慢性高尿酸血症大鼠的尿液和肾脏进行检测,分析其代谢物和代谢通路动态变化情况。造模结束后,模型组和对照组的代谢物得到良好分离。可见,代谢组学研究可以从慢性高尿酸血症肾病复杂的内源性代谢物中发现其变化规律,也能够促进此方法的临床应用(Fig 6)。
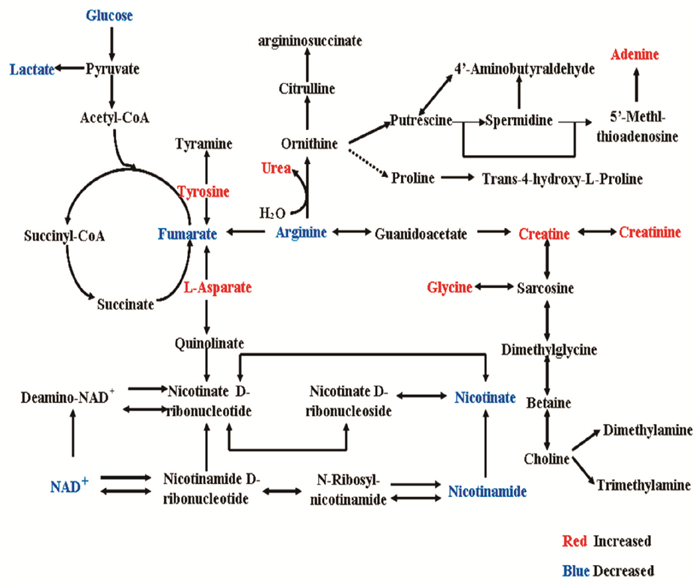
|
| Fig 6 Metabolic pathways of most relevant metabolites between hyperuricaemia and control rats |
牛磺酸是一种游离氨基酸,能抵消组织中的氧化活性。据报道,为减轻肾损伤患者的氧化活性,会用升高患者体内牛磺酸的方法进行治疗[10]。在慢性高尿酸血症大鼠的尿液和肾脏中都检测出牛磺酸明显降低,表明牛磺酸减轻氧化活性作用的潜在机制。因此,牛磺酸可作为高尿酸血症大鼠氧化活性的生物标志物,通过氧化应激过程,导致肾功能受损。
3.2 能量代谢TCA循环是机体ATP产生的主要来源。慢性高尿酸血症肾病大鼠的尿液和肾脏中TCA循环的多个中间代谢物,包括苹果酸、富马酸、α-酮戊二酸和枸橼酸等明显降低。此外,通过糖酵解降低葡萄糖的利用率可能会降低丙酮酸转化为乙酰辅酶A的可能性,可见大鼠的TCA循环总体上受到抑制。
在能量代谢中,谷氨酰胺可以转化为谷氨酸,谷氨酸是主要的细胞能量底物,可进入TCA循环[11]。而在高尿酸血症大鼠中,谷氨酰胺的转化和谷氨酸进入TCA循环可能有助于维持TCA循环的稳态。此外,谷氨酰胺水解也有助于氧化磷酸化过程中ATP和NAD+的产生。而NAD+作为能量代谢的关键组成部分[12],在高尿酸血症大鼠肾脏中明显降低,可能与肾脏ATP水平降低密切相关。NAD+是线粒体呼吸、氧化磷酸化、DNA修复和核信号传递等各项生理过程所需的重要底物。NAD+的减少与老年人和啮齿类动物氧化损伤的增加以及沉默信息调节因子2相关酶1(silent information regulator 2 homolog1,SIRT1)的活性降低有关[13]。SIRT1是NAD+依赖的组蛋白去乙酰化酶,与代谢功能障碍和能量代谢有关。此外,SIRT1还参与肾损伤的调节,包括与衰老相关的肾损伤、肾组织缺氧、肾间质纤维化和肾小管细胞凋亡[14]。虽然机制尚不清楚,但研究表明,NAD+的降低及其相关的SIRT1功能障碍可能与慢性高尿酸血症肾病的发病机制有关。因此,SIRT1的活化可能成为防治肾损伤发生和发展的潜在靶点。
3.3 氨基酸代谢精氨酸是一种半必需氨基酸,内源性精氨酸的合成集中在肝脏和肾皮质上,肾脏中产生的大多数精氨酸被释放到血液中,并分布在全身各处。在肾损伤大鼠中,精氨酸进入内皮细胞的运输受到抑制,而促进精氨酸摄取的是阳离子氨基酸转运蛋白1(cationic amino acid transporter 1,CAT1)。CAT1可转运多种阳离子氨基酸,这可能竞争性抑制精氨酸的摄取。Schwartz等[15]的研究表明,精氨酸在肾损伤大鼠的主动脉和肾小球中转运降低,并伴随着CAT1水平降低。虽然其潜在机制尚未确定,但实验研究表明,精氨酸的降低可能与慢性高尿酸血症肾病的发病机制相关。
此外,大鼠犬尿氨酸途径中的代谢物有所升高,而此途径是由吲哚胺2, 3-双加氧酶(indoleamine 2, 3-dioxygenase,IDO)所介导,IDO水平的升高与肾损伤中炎症和免疫调节能力不足有关。有研究表明,血清中高水平IDO与肾病严重程度和肾损伤有关[16]。因此,IDO可能是防治慢性高尿酸血症肾病很有前途的靶点,而未来研究可集中在IDO的调控机制上。
| [1] |
So A, Thorens B. Uric acid transport and disease[J]. J Clin Invest, 2010, 120(6): 1791-9. doi:10.1172/JCI42344 |
| [2] |
Kang D H, Nakagawa T, Feng L, et al. A role for uric acid in the progression of renal disease[J]. J Am Soc Nephrol, 2002, 13(12): 2888-97. doi:10.1097/01.ASN.0000034910.58454.FD |
| [3] |
Gonick H C, Rubini M E, Gleason I O, et al. The renal lesion in gout[J]. Ann Intern Med, 1965, 62: 667-74. doi:10.7326/0003-4819-62-4-667 |
| [4] |
Hu Q H, Zhang X, Pan Y, et al. Allopurinol, quercetin and rutin ameliorate renal NLRP3 inflammasome activation and lipid accumulation in fructose-fed rats[J]. Biochem Pharmacol, 2012, 84(1): 113-25. |
| [5] |
Cirillo P, Gersch MS, Mu W, et al. Ketohexokinase-dependent metabolism of fructose induces proinflammatory mediators in proximal tubular cells[J]. J Am Soc Nephrol, 2009, 20(3): 545-53. |
| [6] |
Quan H, Peng X, Liu S, et al. Differentially expressed protein profile of renal tubule cell stimulated by elevated uric acid using SILAC coupled to LC-MS[J]. Cell Physiol Biochem, 2011, 27(1): 91-8. |
| [7] |
Gasse P, Riteau N, Charron S, et al. Uric acid is a danger signal activating NALP3 inflammasome in lung injury inflammation and fibrosis[J]. Am J Respir Crit Care Med, 2009, 179(10): 903-13. doi:10.1164/rccm.200808-1274OC |
| [8] |
Beckonert O, Keun H C, Ebbels T M, et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts[J]. Nat Protoc, 2007, 2(11): 2692-703. doi:10.1038/nprot.2007.376 |
| [9] |
罗良, 陈嘉辉, 王园园, 等. 脾气虚证模型大鼠尿液的~1H-NMR代谢组学研究[J]. 中国药理学通报, 2017, 33(10): 1363-70. Luo L, Chen J, Wang Y, et al. 1H-NMR-based metabonomics study on urine of rat with Spleen-Qi deficiency pattern[J]. Chin Pharmacol Bull, 2017, 33(10): 1363-70. |
| [10] |
Han X, Chesney R W. The role of taurine in renal disorders[J]. Amino Acids, 2012, 43(6): 2249-63. doi:10.1007/s00726-012-1314-y |
| [11] |
龚梦鹃, 巫圣乾, 岳贺, 等. 基于~1H-NMR护肝片抗大鼠急性肝损伤的代谢组学研究[J]. 中国药理学通报, 2017, 33(12): 1766-70. Gong M J, Wu S Q, Yue H, et al. Metabonomic analysis of Hugan Tablets on CCl4-induced acute hepatic injury in rats based on 1H-NMR[J]. Chin Pharmacol Bull, 2017, 33(12): 1766-70. |
| [12] |
Xu W, Wu J, An Y, et al. Streptozotocin-induced dynamic metabonomic changes in rat biofluids[J]. J Proteome Res, 2012, 11(6): 3423-35. doi:10.1021/pr300280t |
| [13] |
Braidy N, Guillemin G J, Mansour H, et al. Age related changes in NAD+ metabolism oxidative stress and Sirt1 activity in wistar rats[J]. PLoS One, 2011, 6(4): e19194. doi:10.1371/journal.pone.0019194 |
| [14] |
Kitada M, Kume S, Takeda-Watanabe A, et al. Sirtuins and renal diseases: Relationship with aging and diabetic nephropathy[J]. Clin Sci, 2013, 124(3): 153-64. doi:10.1042/CS20120190 |
| [15] |
Schwartz I F, Ayalon R, Chernichovski T, et al. Arginine uptake is attenuated through modulation of cationic amino-acid transporter-1, in uremic rats[J]. Kidney Int, 2006, 69(2): 298-303. doi:10.1038/sj.ki.5000067 |
| [16] |
Outinen T K, Mäkelä S M, Ala-Houhala I O, et al. High activity of indoleamine 2, 3-dioxygenase is associated with renal insufficiency in puumala hantavirus induced nephropathia epidemica[J]. J Med Virol, 2011, 83(4): 731-7. doi:10.1002/jmv.22018 |

