2. Dept of Rehabilitation, Affiliated Hospital of Qingdao University, Qingdao Shandong 266003, China;
3. College of Pharmacy, Heilongjiang University of Chinese Medicine, Harbin 150040, China
随着生活节奏的加快、生活方式的改变以及社会压力的增加,睡眠剥夺(sleep deprivation, SD)或者睡眠不足已经成为一种普遍的社会现象。据世界卫生组织调查结果显示,全世界约27%的人有睡眠问题。SD给人们带来的负面影响也日益受到关注。研究表明[1],短期SD会导致疲劳、反应迟钝;而长期、连续的SD会引起机体多种生理功能改变,如内分泌、免疫系统的失调、加重炎症反应,引发糖尿病[2]等。睡眠与学习记忆关系尤为密切[3],良好的睡眠有助于学习内容的记忆和巩固。睡眠缺失导致的记忆障碍也与多种神经精神系统疾病密切相关, 如阿尔茨海默病[4]、帕金森病[5]及抑郁症[6]等。SD对学习记忆产生的负性作用,涉及到多种基因的转录和蛋白表达的异常[7],是多种基因和细胞信号传导通路相互作用的结果。目前研究表明,异相睡眠(paradoxical sleep, PS)在记忆形成中具有重要的作用,特别是杏仁核相关的情感记忆[8]。但异相睡眠剥夺(paradoxical sleep deprivation,PSD)导致记忆障碍的确切机制,目前还不是很清楚。为了更好地预防和治疗睡眠剥夺相关疾病,阐明PSD造成记忆障碍的分子机制,课题组利用改良多平台水环境法(modified multiple platform method, MMPM)制备PSD动物模型,再利用六角迷宫评价PSD对大鼠学习记忆行为上的影响;在此基础上,通过基因芯片技术,研究其对海马全基因组表达谱的影响,筛选出差异表达基因,对部分差异表达基因进行实时荧光定量PCR验证;对筛选的差异基因进行GO和KEGG富集分析,以判定差异基因主要影响的生物学功能及信号转导通路。研究结果为初步阐明PSD影响学习记忆可能的分子机制、临床治疗睡眠障碍相关疾病奠定了理论和实验基础,并为相关新药研发提供了治疗靶点。
1 材料 1.1 药品与试剂HiScript Ⅱ Q RT SuperMix for qPCR(Vazyme,R223-01);QuantiFast® SYBR® Green PCR试剂盒(Qiagen,204054);Agilent SurePrint G3 Rat GE(8*60K,Design ID:028279) 芯片,由上海欧易生物医学科技有限公司提供并检测。
1.2 实验动物与分组清洁级SD大鼠16只,♂,体质量(200±20) g,由黑龙江中医药大学GLP实验室提供,许可证号:黑动字第P00700619。将大鼠随机分为异相睡眠剥夺组(PSD)和大平台对照组(TC),每组8只。实验期间自由饮食饮水,室温(24±2)℃,湿度(55±15)%,12 h :12 h明暗交替。
1.3 仪器NanoDrop 2000分光光度计(Thermo Scientific,USA);荧光定量PCR仪(Roche,Swiss,LC480Ⅱ型);PCR仪(ABI,9700型);Agilent扫描仪(Agilent Technologies,G2505C);睡眠剥夺箱(自制,箱体为90 cm×70 cm×50 cm),箱内有12个直径6.3 cm的小平台(大平台直径为12 cm,大鼠能够正常睡眠)。箱内注水,水面低于平台1.0 cm,水温(20±1)℃,每天更换。
2 方法 2.1 睡眠剥夺模型的建立及行为学训练、测试每天将大鼠放入睡眠剥夺箱适应10 min,1周后放入睡眠剥夺箱剥夺异相睡眠[9],连续剥夺4 d,每天更换箱体中的水。PSD 1 d后进行六角迷宫训练(每天上午进行,结束后立即放回睡眠剥夺箱,连续训练3 d),4 d后进行测试。实验流程见Fig 1。
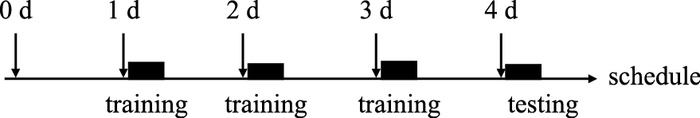
|
| Fig 1 The experimental process |
训练与测试方法d 1,将大鼠放入六角迷宫,不给予时间限制,只要找到6个出口当中的1个并逃出即可。d 2,条件刺激(CS)和非条件刺激(US)相结合:大鼠进入迷宫3 min后,给予持续3s的铃声(CS),随后给予50V、持续1s的电击(US);此后每间隔2 s将CS+US配对循环1次,直至大鼠找到其中1个出口并逃出。d 3,重复d 2的训练方法,并依次将大鼠找到的出口关闭,直至完成寻找到全部6个出口的任务[15]。d 4,测试:方法与d 3的训练方法相同,记录大鼠寻找到6个出口所用的总时间(即寻找时间,min)(time)和犯错误次数(fail times),计算认知率(cognitive rate)。认知率/%=正确出逃次数/寻找总次数×100%。寻找总次数=正确次数+错误次数。正确次数为6,错误次数为大鼠每重复寻找1个已经关闭的出口记为1次错误。如果大鼠1次错误也没出现,则认知率为100%。
2.2 样本处理及芯片杂交、扫描行为学测试后,立即断头处死大鼠,冰浴上剥取海马,液氮中保存。用mirVanaTM RNA Isolation Kit AM1561提取总RNA,NanoDrop ND-2000超微量分光光度计定量,并经Agilent 2100生物分析仪检测RNA完整性。RNA质检合格后,将总RNA反转录成双链cDNA,再进一步合成用Cyanine-3-CTP(Cy3) 标记的cRNA。标记好的cRNA和芯片杂交,洗脱后利用Agilent G2505C扫描仪扫描得到原始图像。
2.3 实时荧光定量PCR验证选取PSD组与TC组的差异表达基因S100a9、Ret、Mrvi1和Oxt为目的基因,ACTB作为管家基因。引物采用Roche LCPDS2软件设计并由上海捷瑞生物工程有限公司合成,S100a9的引物序列:正义5′-ATGATGCTGATGGGAAAGTT-3′,反义5′-GGGCAGATGTTACATGGC-3′;Casr的引物序列:正义5′-AGCCTGCATCTTCTTCAAC-3′,反义5′-TTTGAAAGCATGTGCTGCG-3′;Hbb的引物序列:正义5′-CTGAGAAGGCTGCTGTTAATG-3′,反义5′-TCTGGGTCCAAGGGTAGACAA-3′;Alox15的引物序列:正义5′-ATCGAGACTTGCTTCTATGCTA-3′,反义5′-ACAGCTTTGTCGGTCTTGTA-3′。以与芯片杂交相同的总RNA为模板,逆转录成cDNA,按照定量PCR试剂盒操作程序操作。反应体系:2×QuantiFast® SYBR® Green PCR Master Mix,5 μL;10 μmol·L-1 Forward primer,0.2 μL;10 μmol·L-1 Reverse primer,0.2 μL;cDNA,1 μL;Nuclease-free H2O,3.6 μL。PCR程序:95℃ 5 min;95℃ 10 s,60℃ 30 s,40个循环。循环结束后利用熔解曲线检测产物特异性:从60℃缓慢升温至97℃,每升高1 ℃采集5次荧光信号。
2.4 统计学分析行为学实验结果以x±s表示,利用SPSS13.0统计软件进行组间t检验,比较PSD组和TC组大鼠在寻找时间、错误次数及认知率3个指标上的差异。
实时荧光定量PCR验证结果采用Feature Extraction软件(version10.7.1.1, Agilent Technologies)处理原始图像提取原始数据。接着利用Genespring软件(version 12.5;Agilent Technologies)进行quantile标准化和后续处理。标准化后的数据进行过滤,在用于比较的每组样本中至少有一组100%标记为Detected的探针留下进行后续分析。利用T检验的P值和倍数变化值进行差异基因筛选,筛选的标准为上调或者下调倍数变化值≥2.0且P值≤0.05。接着,对差异基因进行GO和KEGG富集分析,以判定差异基因主要影响的生物学功能或者通路。最后,对差异基因进行非监督层次聚类,利用热图的形式展示差异基因在不同样本间的表达模式。
3 结果 3.1 PSD对大鼠学习记忆能力的影响经组间t检验,PSD组和TC组大鼠在寻找时间、错误次数及认知率3个指标上,差异存在显著性:PSD组大鼠寻找到全部6个出口所用的总时间明显延长(P<0.05);犯错误次数增多(P<0.01);总体认知率下降(P<0.01)。见Fig 2。
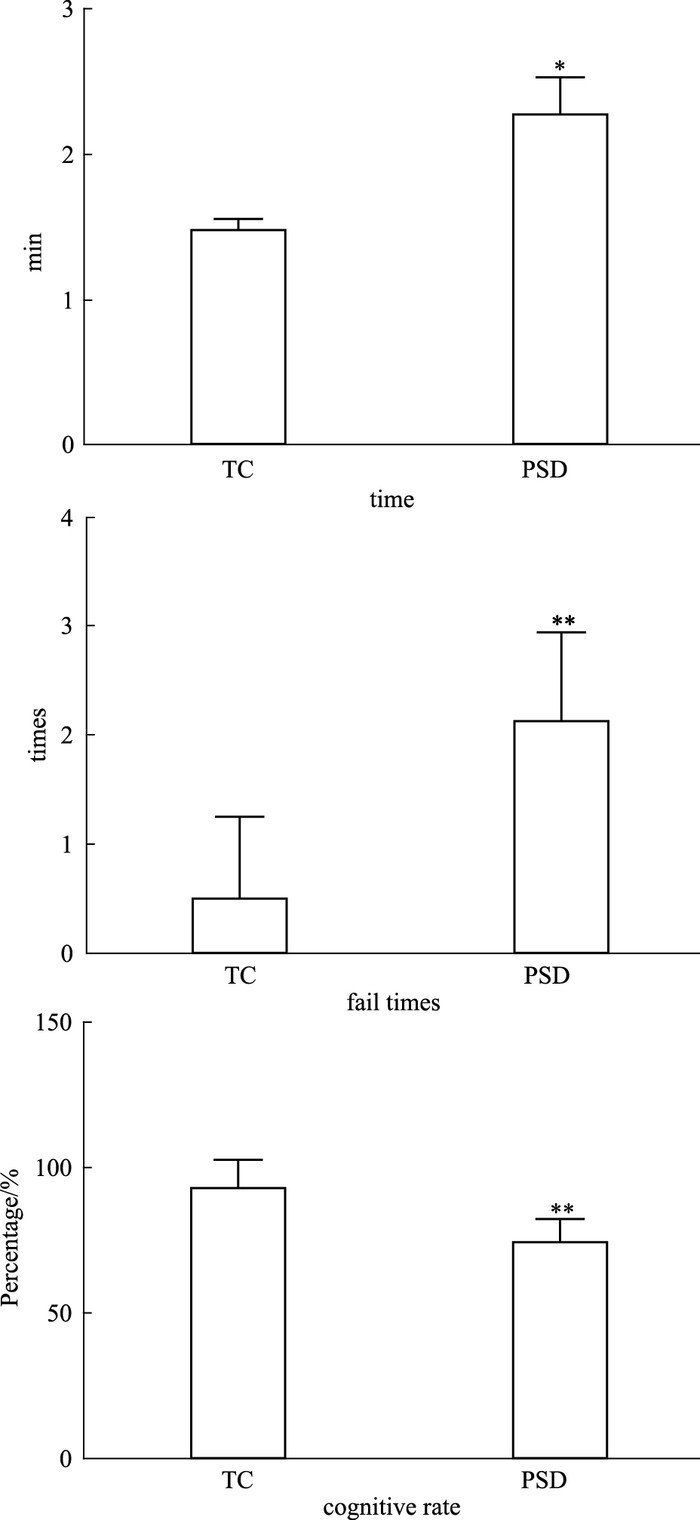
|
| Fig 2 Effects of PSD on behavior of rats in Hexagonal Maze *P < 0.05, **P < 0.01 vs TC |
分光光度仪检测显示,提取组织RNA的A260/280均在1.8~2.2之间,纯度较好;Agilent Bioanalyzer 2100(Agilent Technologies)检测RNA完整性,RIN≥8且28S/18S≥1,说明RNA的质量好,能满足实验要求。见Tab 1。
| Sample | Concentration /g·L-1 | A260/280 | A260/230 | Volume/μL | Total mass/μg | Agilent 2100 | |
| 28S/18S | RIN | ||||||
| TC 1 | 0.0901 | 2.09 | 1.74 | 95 | 9 | 1.9 | 9.0 |
| TC 2 | 0.0993 | 2.05 | 1.61 | 95 | 9 | 2.0 | 8.7 |
| TC 3 | 0.1526 | 2.10 | 2.04 | 95 | 14 | 1.1 | 8.3 |
| PSD 1 | 0.0553 | 2.00 | 1.51 | 95 | 5 | 1.0 | 8.0 |
| PSD 2 | 0.1165 | 2.08 | 1.67 | 95 | 11 | 1.2 | 8.2 |
| PSD 3 | 0.1368 | 2.00 | 1.51 | 95 | 13 | 1.1 | 8.5 |
PSD组和TC组基因芯片Cy3荧光扫描图像见Fig 3。

|
| Fig 3 Chip scan gram of TC group and PSD group A:TC group; B:PSD group |
基因表达谱芯片分析显示,PSD组与TC对照组相比,差异表达基因共100个,其中上调基因25个,下调基因75个。按照GO分类标准进行基因功能分类注释,上调基因功能主要与细胞生长、分化负性调节、炎症反应、氧化应激、酶活性、转录调节等有关;下调基因功能主要与免疫应答、神经系统发育、突触传递、代谢过程、信号转导、递质分泌、细胞凋亡、氧化应激应答、转录过程等有关。部分差异表达基因的结果见Tab 2、3;利用KEGG数据库对差异基因进行Pathway分析,部分差异基因参与调控的Pathway结果见Tab 4。
| Term | Biological process | Gene number | Percentage/% |
| GO:0051384 | response to glucocorticoid | 7 | 10.14 |
| GO:0002544 | chronic inflammatory response | 3 | 4.34 |
| GO:0042060 | wound healing | 5 | 7.24 |
| GO:0042346 | positive regulation of NF-kappaB import into nucleus | 2 | 2.89 |
| GO:0042493 | response to drug | 7 | 10.14 |
| GO:0030335 | positive regulation of cell migration | 4 | 5.80 |
| GO:0051965 | positive regulation of synapse assembly | 2 | 2.89 |
| GO:2001244 | positive regulation of intrinsic apoptotic signaling pathway | 2 | 2.89 |
| GO:0042755 | eating behavior | 2 | 2.89 |
| GO:0009636 | response to toxic substance | 3 | 4.34 |
| GO:0006954 | inflammatory response | 4 | 5.80 |
| GO:0051387 | negative regulation of neurotrophin TRK receptor signaling pathway | 1 | 1.45 |
| GO:0008285 | negative regulation of cell proliferation | 4 | 5.80 |
| Term | Molecular function | Gene number | Percentage/% |
| GO:0005509 | calcium ion binding | 7 | 11.29 |
| GO:0008179 | adenylate cyclase binding | 1 | 1.61 |
| GO:0004683 | calmodulin-dependent protein kinase activity | 1 | 1.61 |
| GO:0005516 | calmodulin binding | 2 | 3.23 |
| GO:0005215 | transporter activity | 2 | 3.23 |
| GO:0046982 | protein heterodimerization activity | 4 | 6.45 |
| GO:0051721 | protein phosphatase 2A binding | 1 | 1.61 |
| GO:0004865 | protein serine/threonine phosphatase inhibitor activity | 1 | 1.61 |
| GO:0019904 | protein domain specific binding | 3 | 4.84 |
| GO:0004872 | receptor activity | 2 | 3.23 |
| GO:0005215 | transporter activity | 2 | 3.23 |
| GO:0019899 | enzyme binding | 3 | 4.84 |
| Biological pathway | Gene number | Genes |
| VEGF signaling pathway | 2 | Ptgs2;Sphk1 |
| Cell adhesion molecules(CAMs) | 3 | LOC288521;Spn; Sdc1 |
| cGMP-PKG signaling pathway | 2 | Adrb3;Mrvi1 |
| Calcium signaling pathway | 2 | Adrb3;Sphk1 |
| PI3K-Akt signaling pathway | 3 | Fn1;Chad; Angpt2 |
| Long-term depression | 1 | Ppp1r17 |
| TGF-beta signaling pathway | 1 | Inhba |
| NF-kappa B signaling pathway | 1 | Ptgs2 |
| Neuroactive ligand-receptor interaction | 2 | Adrb3;Sstr4 |
| Metabolic pathways | 4 | Adh1;Ass1;Ptgs2;Sphk1 |
| Pathways in cancer | 1 | Fn1;Ret; Ptgs2 |
| Serotonergic synapse | 1 | Ptgs2 |
| negative regulation of cell proliferation | 4 | Ptgs2;Sstr4;Inhba; Spry2 |
S100a9、Ret、Mrvi1和Oxt 4个基因的2-ΔΔCt值分别为5.1868、0.4458、0.1405、0.0042,均与芯片表达趋势一致,说明芯片数据可靠并具有可重复性。见Fig 4。
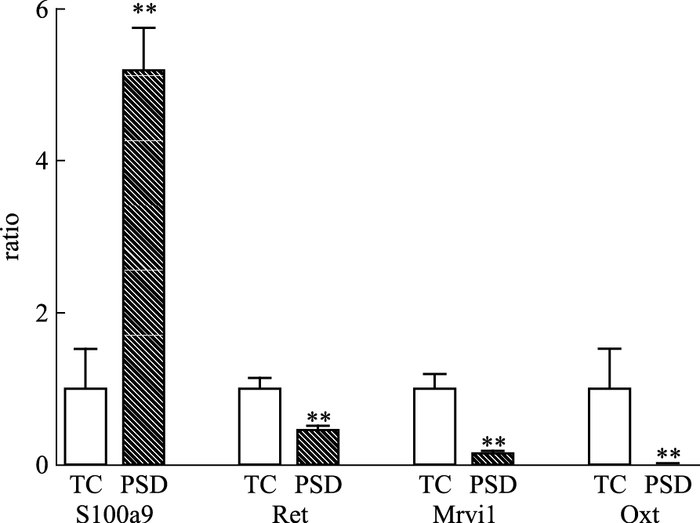
|
| Fig 4 Part of diferentially expressed genes in hippocampi in rats by real-time quantitative RT-PCR **P < 0.01 vs TC |
利用MEV 4.6软件对差异基因进行非监督层次聚类分析,结果见Fig 5。横轴方向代表样本名称:A代表TC组样本,B代表PSD组样本;纵轴方向代表 100个差异基因的探针名称:其中绿色部分代表下调基因,红色代表上调基因。
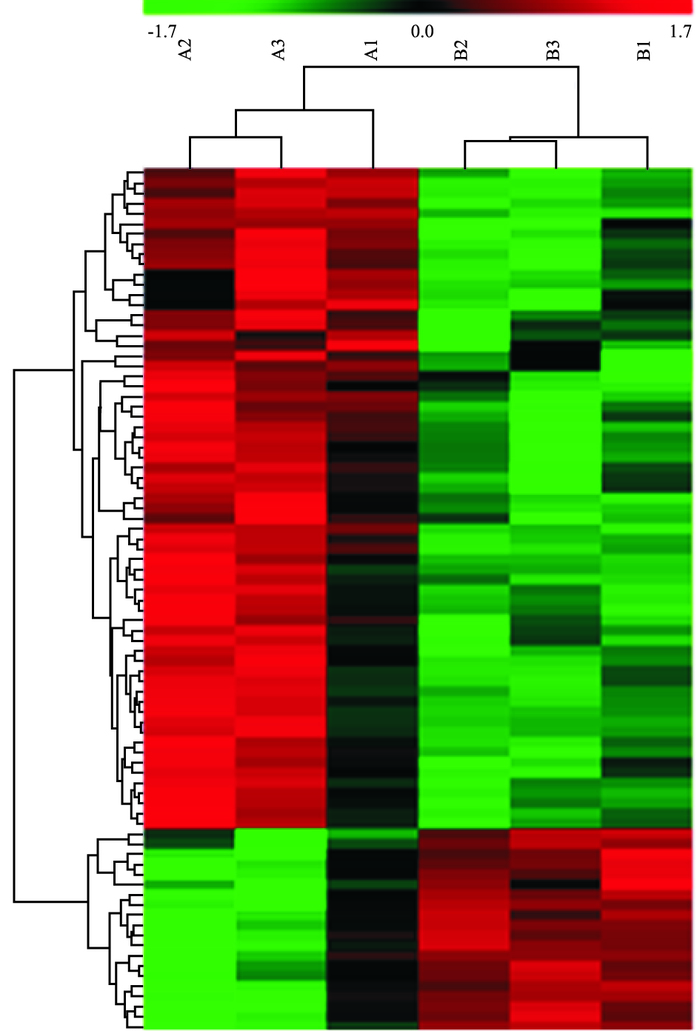
|
| Fig 5 Hierarchical clustering of differentially expressed genes |
目前研究认为[7],睡眠剥夺会影响学习记忆过程,但具体机制尚不十分清楚,认为与多种基因、蛋白表达的异常有关。以往关于睡眠剥夺对学习记忆影响的研究,多集中在全部睡眠剥夺(TSD)或慢波睡眠(SWS)剥夺阶段,对快波睡眠(REMS,也称异相睡眠)研究较少。研究表明[8],异相睡眠(PS)与学习记忆关系密切,特别是杏仁核相关的情绪记忆。为考察PS与学习记忆的关系,本研究利用改良多平台水环境法(MMPM)特异性地剥夺PS[9]。与其他睡眠剥夺方法(如药物剥夺法、轻柔刺激法等)相比,具有特异性高(对慢波睡眠影响小)、方法简单易行、无需复杂昂贵的设备等优势;通过设立大平台对照组(TC),排除了水环境应激对大鼠行为的干扰,因此在异相睡眠剥夺方面具有较为广泛的应用[10]。
研究中采用的六角迷宫行为学范式,融合了空间学习记忆(寻找6个不同方位的出口)和联想式学习记忆(CS和US结合的经典条件反射记忆)的行为学特征,因此,能够更为可靠的评价PSD造成的大鼠学习记忆和认知能力的变化[11-12]。实验结果表明,PSD能够造成大鼠六角迷宫学习记忆的障碍,与TC组比较,寻找到6个出口所需时间长、寻找过程中犯错误次数多、总体认知能力下降(Fig 2)。
关于PSD对基因表达影响的研究,目前多集中在大脑的皮质部,对海马这一记忆形成与存贮的关键脑区研究不多;由于实验目的不同,以单一基因的表达研究较多,缺乏全面、系统的研究和评价[13];现有的研究也由于睡眠剥夺的方法、时间长短及动物种属不同,结果也存在差异[14]。因此,本课题从全基因组表达的角度,全面系统的考察了PSD对学习记忆的影响及可能的分子机制。
基因芯片实验结果表明,PSD组和TC组大鼠海马差异表达基因共100个:上调基因25个,主要与细胞生长、分化负性调节、氧化应激、突触传递、转录调节等功能有关;下调基因75个,主要与免疫应答、神经系统发育、代谢过程、信号转导、递质分泌、细胞凋亡、生长调节、海马发育等功能有关。说明PSD对学习记忆的影响可能与多种基因表达异常相关,与以往研究结果是一致的[14]。本研究发现,PSD导致大鼠海马Ass1基因表达异常,而Ass1基因是细胞对cAMP反应相关的基因,说明PSD造成的大鼠记忆障碍可能与cAMP-PKA-CREB信号通路密切相关,这与Vecsey等[15]的报道一致。但学习记忆相关的信号转导通路有多条,如PLC /PKC、CaMK Ⅱ-ERK和PLC-PKC等,PSD对机体的影响比较广泛,受PSD影响的信号通路可能不仅限于cAMP-PKA-CREB这一条。我们的研究结果也发现,PSD也会影响VEGF、cGMP-PKG、Calcium及Long-term depression等多条与记忆相关的信号转导通路。因此,PSD导致的记忆障碍可能与以上多种信号通路的变化有关,具体机制有待深入研究。
总之,本研究结果表明,PSD可以导致大鼠六角迷宫学习成绩下降,涉及到多基因参与、多信号通路调节的变化。本研究在海马全基因组表达水平上,初步阐释了PSD影响大鼠学习记忆可能的分子机制,为今后深入探讨PSD影响学习记忆的神经生物学机制及相关药物研发提供了实验依据和研究靶点。
(致谢: 本文实验在黑龙江中医药大学药学院“国家中医药管理局中药行为药理三级实验室”完成,特此致谢。 )
| [1] | Namni G, Mathias B, Hengyi R, et al. Circadian rhythms, sleep deprivation, and human performance[J]. Prog Mol Biol Transl Sci, 2013, 119: 155-90. doi:10.1016/B978-0-12-396971-2.00007-5 |
| [2] | Schwarz P. Sleep deprivation can trigger diabetes[J]. MMW Fortschr Med, 2014, 156(11): 35. doi:10.1007/s15006-014-3159-7 |
| [3] | Jan B, Ines W. System consolidation of memory during sleep[J]. Psychol Res, 2012, 76(2): 192-203. doi:10.1007/s00426-011-0335-6 |
| [4] | 董静文, 史源, 张建军. 睡眠障碍与阿尔采末病相关性研究进展[J]. 中国药理学通报, 2014, 30(9): 1189-93. Dong J W, Shi Y, Zang J J. Research progress on the relationship between sleep disorders and Alzheimer′s disease[J]. Chin Pharmacol Bull, 2014, 30(9): 1189-93. |
| [5] | Willison L D, Kudo T, Loh D H, et al. Circadian dysfunction may be a key component of the non-motor symptoms of Parkinson′s disease: insights from a transgenic mouse model[J]. Exp Neurol, 2013, 243(5): 57-66. |
| [6] | Robert E R, Hao T D. The prospective association between sleep deprivation and depression among adolescents[J]. Sleep, 2014, 37(2): 239-44. |
| [7] | Vecsey C G, Peixoto L, Choi H, et al. Genomic analysis of sleep deprivation reveals translational regulation in the hippocampus[J]. Physiol Genomics, 2012, 44(20): 981-91. doi:10.1152/physiolgenomics.00084.2012 |
| [8] | Genzel L, Spoormaker V I, Konrad B N, et al. The role of rapid eye movement sleep for amygdala-related memory processing L[J]. Neurobiol Learn Mem, 2015, 122: 110-21. doi:10.1016/j.nlm.2015.01.008 |
| [9] | Mendelson W B, Guthrie R D, Frederick G, et al. The flower pot technique of rapid eye movement(REM) sleep deprivation[J]. Pharmacol Biochem Behav, 1974, 2(4): 553-6. doi:10.1016/0091-3057(74)90018-5 |
| [10] | Machado R B, Hipólide D C, Benedito-Silva A A, et al. Sleep deprivation induced by the modified multiple platform technique: quantification of sleeploss and recovery[J]. Brain Res, 2004, 1004(1-2): 45-51. doi:10.1016/j.brainres.2004.01.019 |
| [11] | 郭冷秋, 郭壮丽, 叶晓楠, 等. 刺五加总苷对REM睡眠剥夺所致大鼠记忆巩固障碍及海马群峰电位的影响[J]. 中国新药杂志, 2014, 23(19): 2304-8. Guo L Q, Guo Z L, Ye X N, et al. Effect of total glycosides of acanthopanax on memory consolidation and population spikes in the hippocampus of REM sleep deprived rats[J]. Chin J New Drugs, 2014, 23(19): 2304-8. |
| [12] | 郭冷秋, 郭壮丽, 康诗瑶, 等. 刺五加总苷对REM睡眠剥夺所致大鼠记忆获得障碍的改善作用及其LTP机制[J]. 中国实验方剂学杂志, 2014, 20(13): 125-9. Guo L Q, Guo Z L, Kang S Y. Effect of total glucosides of acanthopanax senticosus on memory acquisition deficit rats induced by REM sleep deprivation and it′s LTP mechanism[J]. Chin J Exp Tradit Med Formul, 2014, 20(13): 125-9. |
| [13] | Elliott A S, Huber D J, O′callaghan P J, et al. A review of sleep deprivation studies evaluating the brain transcriptome[J]. Springerplus, 2014, 3: 728-40. doi:10.1186/2193-1801-3-728 |
| [14] | Vecsey C G, Peixoto L, Choi J H, et al. Genomic analysis of sleep deprivation reveals translational regulation in the hippocampus[J]. Physiol Genomics, 2012, 44(20): 981-91. doi:10.1152/physiolgenomics.00084.2012 |
| [15] | Vecsey C G, Baillie G S, Jaganath D, et al. Sleep deprivation impairs cAMP signalling in the hippocampus[J]. Nature, 2009, 461(7267): 1122-5. doi:10.1038/nature08488 |


