


 ,
WU Yi-nuo,
YU Xiu,
GUO Ne,
ZHANG Ren-wei,
LIU Cui,
ZHENG Xue-ping,
DAI Qi,
LIU Pei-qing,
LI Min
,
WU Yi-nuo,
YU Xiu,
GUO Ne,
ZHANG Ren-wei,
LIU Cui,
ZHENG Xue-ping,
DAI Qi,
LIU Pei-qing,
LI Min

细胞自噬是真核细胞所特有的对细胞内受损的细胞器及长寿命蛋白通过溶酶体途径进行降解的细胞生物学过程[1]。自噬对于维持细胞稳态、调节细胞物质能量代谢具有重要意义[2]。细胞自噬是一个动态变化的过程,可大致分为以下几个阶段:诱导与成核、延伸、自噬体的成熟、自噬体与溶酶体的融合及其内容物的降解[3]。在这一过程中,有多种自噬相关基因 (autophagy-related genes, ATG) 的参与,其中两条泛素样通路ATG12-ATG5-ATG16和ATG8-PE (phosphatidylethanolamine) 在自噬体的延伸和成熟过程中起着重要的作用,而ATG12-ATG5-ATG16复合物作为E3样酶有助于ATG8与PE的结合[4]。ATG8必须经过一个蛋白水解过程暴露C末端的甘氨酸才能锚定在自噬泡膜上。ATG4作为C54家族的一种半胱氨酸蛋白酶,在ATG8连接系统中起了非常关键的作用。ATG4可以剪切ATG8的C端精氨酸,使其暴露出C端的甘氨酸残基,以便与PE共价连接形成ATG8-PE锚定在自噬泡膜上。接下来ATG4还能去脂化ATG8-PE,以便使自噬体与溶酶体融合[5]。因此,通过调控ATG4介导的ATG8-PE的去脂化过程能调控整个自噬的过程。
已有文献报道,ATG4B在肿瘤的发生发展过程中起了重要作用。ATG4B被认为是一个致癌基因,能促进结肠癌细胞的生长且独立于其自噬调节作用[6]。除此之外,在慢性髓细胞性白血病及骨肉瘤细胞中,ATG4B也被看作致癌基因[7-8]。另外使用ATG4B的小分子抑制剂NSC185058能抑制骨肉瘤细胞的生长[8]。不仅如此,ATG4B在博来霉素诱导的心肌纤维化过程中起了重要作用。由此我们可以看出,ATG4B可能是疾病治疗中的潜在靶点,抑制ATG4B可以作为疾病治疗的手段。但目前关于ATG4B小分子抑制剂的研究还少见报道,抑制剂效价偏低,缺少系统的虚拟筛选方法。本实验将建立了一种计算机虚拟筛选ATG4B抑制剂的方法,结合体外活性验证,筛选出特异性ATG4B小分子抑制剂。这些研究为ATG4B抑制剂的虚拟筛选奠定基础,也为进一步研究抑制剂在抗肿瘤等疾病治疗中的作用提供了研究工具。
1 材料与方法 1.1 材料虚拟筛选的软件Discovery Studio Client 2.5(Accelrys, San Diego, CA);分子三维结构显示软件PyMoL (DeLano Scientific, Palo Alto, CA);ATG4B的晶体结构来自PDB数据库 (Protein DataBank);化合物数据库来自SPECS (http://www.specs.net);筛选到的30个小分子化合物购自美国SPECS数据库;NEM、考马斯亮蓝R250购自上海生工生物工程有限公司;IPTG (异丙基硫代半乳糖苷) 购自美国Merck;星形孢菌素购自美国LC Laboratories;caspase-3底物肽Ac-DEVE-AFC购自中肽生化有限公司;Z-VAD-FMK购自美国Selleck公司;细胞培养所用DMEM培养基和类胎牛血清购自美国Gibco公司;表达ATG4B及其酶切底物蛋白FRET-GATE-16的质粒为本实验室前期构建并保存[9];HELA细胞由本实验室保存[10]。
1.2 数据库准备考虑到化合物的结构多样性及来源可获得性,本研究选择含有200 000个小分子的SPECS数据库作为原始数据库。首先利用MOE.2010.软件,对数据库中的小分子进行初筛。在MOE.2010.中,每个化合物将被生成250个构象,经过Lipinski类药五原则方法筛选,以下小分子将被筛除:分子质量>600,LogP<-4,LogP>8,氢键供体和受体之和>12,柔性键>7,单键链长>6,手性中心>4,自由的手性中心>3和过渡金属>8等。通过该方法,每一个化合物都能得出一组物理和生物性质的相关数据,以对数据库中的小分子进行类药性评价。筛选得到的小分子组成数据库A,所有数据库A中的小分子都将用于之后的分子对接筛选。
1.3 蛋白活性口袋的准备我们选择PDB代码为2CY7的晶体作为分子对接的受体结构。在受体蛋白结构的准备过程中,删除受体结构中的水分子,添加氢原子同时将电离残基在中性pH条件下质子化,将电场设置为CHARMm。准备好受体结构后,开始进行分子对接。
1.4 分子对接筛选我们使用Accelrys Discovery Studio Client 2.5软件里的LigandFit板块来进行分子对接。准备工作就绪后,将数据库A中的所有分子对接到蛋白口袋中,每个小分子设置50种对接构象,通过Consence Score对化合物进行打分,产生排名前500的化合物,然后考虑较好的对接打分值以及符合的结合模式 (主要与蛋白口袋中关键氨基酸残基CYS74、ASP278及HIS280的作用模式)。从而筛选出30个化合物进行购买并用于接下来的生物活性测试。
1.5 重组蛋白的表达纯化将重组质粒FRET-GATE-16和ATG4B分别转化至大肠杆菌BL21(DE3) CodonPlus和BL21(DE3) PLYSs中。LB平板挑取单克隆接种到LB液体培养基中,37℃、220 r·min-1过夜培养,1 :100进行扩增培养,当OD600达到0.6~0.8时加入0.5 mmol·L-1的IPTG进行诱导,16℃,16 h培养后收菌。离心收集菌体,加入湿菌重量5~10倍的结合缓冲液 (含5 mmol·L-1的咪唑) 稀释菌体后超声破碎菌体。离心收集上清,使用镍NTA填料进行纯化,加入菌液上清使目的蛋白挂柱,之后分别用20 mmol·L-1和50 mmol·L-1的咪唑缓冲液进行梯度洗脱,最后用200 mmol·L-1的咪唑洗脱并收集洗脱液。将收集到的洗脱液过脱盐柱后浓缩并于-80℃冰箱保存。
1.6 FRET方法检测ATG4B活性384孔黑板中加入终浓度为100 μmol·L-1的指定化合物与0.75 mg·L-1的ATG4B在Tris缓冲液中37℃共孵育30 min,之后加入50 mg·L-1的FRET-GATE-16,反应总体系为50 μL,反应时间为30 min。该体系的中含有0.1% DMSO终浓度。527/477 nm的RFUs比值在反应30 min时测定。ATG4B相对酶切活性的计算公式为:抑制率/%=(RFUmax-RFUX)/(RFUmax-RFUmin)×100%,其中RFUmax指没发生酶切反应时的527/477 nm的比值,RFUmin指酶切反应进行到最彻底的527/477 nm的比值,RFUX指在特定化合物处理条件下的527/477 nm的比值。
1.7 SDS-PAGE方法检测ATG4B活性将3 ng的ATG4B单独或与指定浓度的化合物在缓冲液中37℃共孵育30 min,之后加入4 μg的底物蛋白FRET-GATE-16,反应总体系为20 μL,反应时间为30 min。用5X上样缓冲液终止反应,将蛋白变性,采用SDS-PAGE进行电泳,电泳结束后用考马斯亮蓝染色方法对条带进行着色,之后脱色进行分析。
1.8 caspase-3活性测定HELA细胞于含有10%胎牛血清的DMED培养基中,于37 ℃,5% CO2的孵箱中培养。用星形孢菌素 (1 μmol·L-1, 5 h) 来诱导HELA细胞凋亡,对照组细胞不做任何处理,在同样的条件下培养。非变性提取细胞总蛋白,BCA试剂盒检测细胞蛋白含量,上样量为10 μg。384孔黑板中加入终浓度为100 μmol·L-1的指定化合物与10 μg的细胞裂解液在Tris缓冲液中37℃共孵育30 min,之后加入终浓度为25 μmol·L-1的荧光底物Ac-DEVE-AFC,反应总体系为50 μL,立即检测荧光值,测定时间为60 min,反应温度为37℃。分别于激发光400 nm和发射光505 nm处检测荧光AFC的动力学曲线。
2 结果 2.1 计算机虚拟筛选ATG4B抑制剂ATG4B的晶体结构已经被解析出来 (PDB ID:2CY7)。因ATG4B晶体结构中不含有与受体蛋白共结晶的小分子配体,我们使用Accelrys Discovery Studio Client 2.5软件里的Find sites模块对受体中可能存在的口袋位置进行寻找并找到10个可能的口袋位置。因site 5口袋在ATG4B的N末端尾巴部分 (1~20位氨基酸残基) 与调节环 (259~262位氨基酸残基) 之间的凹槽处,且site 5口袋包含ASP278和HIS280这两个非常重要的氨基酸残基,因此我们选择site 5为本次对接工作的口袋位置。Site 5口袋的位置如Fig 1A~B所示。Site 5口袋包含的氨基酸残基如下:THR10、LEU11、ALA14、ASN261、SER262、HIS264、TYR276、ASP278、HIS280和CYS306。将数据库A (含有149, 214个小分子) 与site 5进行对接,通过Consence Score给出打分,并对排名前500的化合物通过骨架进行分类,结合化合物与受体的结合模式,从中挑选并购买了30个有代表性的化合物进行生物活性测试。
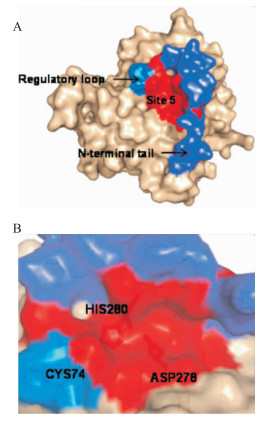
|
| Fig 1 The structure and active pocket site 5 of ATG4B A:Surface presentation of the active pocket site 5 of ATG4B. The regulatory loop was colored marine, the N-terminal tail was colored slate, and the site 5 was colored red; B:Surface model of site 5. Atom coloring was the same as in (A) |
为保证该体系的可靠性和稳定性,用考马斯亮蓝染色的方法对纯化出来的两种蛋白的纯度及活性进行了验证。FRET-GATE-16(4 μg) 与合适量的ATG4B (3 ng) 在37℃共孵育0 min或30 min。如Fig 2A所示,全长的FRET-GATE-16(0 min) 的纯度 (>90%) 可以用于接下来的实验,而30 min时全长的FRET-GATE-16(0 min) 几乎可以完全被3 ng的ATG4B酶切为CFP-GATE-16和CFP两部分,说明ATG4B的活性良好。我们选择半胱氨酸蛋白酶的通用型抑制剂N-乙基马来酰亚胺 (NEM) 作为本次筛选的阳性对照,利用该检测体系测得NEM的IC50值为134.2 μmol·L-1(Fig 2B)。接着对购买的30个小分子化合物用FRET方法测定100 μmol·L-1的浓度下对ATG4B的抑制活性。如Fig 2C所示,化合物AG-690/10400046表现出最高的抑制活性,该化合物的结构如Fig 2D所示。
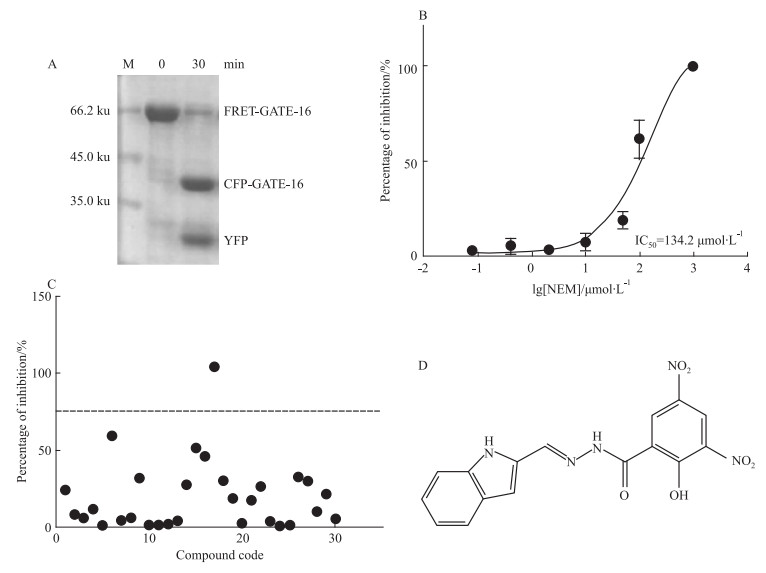
|
| Fig 2 Screening of ATG4B inhibitors by FRET assay A:Verification of the cleavage of FRET substrates by ATG4B using SDS-PAGE. B:NEM was used as a positive control with an IC50 of 134.2 μmol·L-1 for ATG4B using the FRET-based assay. C:A total of 30 compounds were further tested for their inhibitory effects on ATG4B activity using the FRET-based assay. D:The structure of AG-690/10400046 |
为进一步验证AG-690/10400046对ATG4B的体外抑制活性,采用考马斯亮蓝染色的方法检测该化合物对ATG4B的酶切抑制效果。如Fig 3A所示,AG-690/10400046能剂量依赖性地抑制ATG4B的活性,且在100 μmol·L-1的条件下几乎可以完全抑制住ATG4B的活性。FRET方法测得该化合物的IC50值为36.8 μmol·L-1(Fig 3B)。此外,从AG-690/10400046与受体的对接结果来看,该化合物与受体蛋白2CY7的口袋位置具有良好的结合模式,且与受体蛋白的PHE16及GLU312位点形成了氢键作用 (Fig 3C~D),表明该化合物与ATG4B之间有较好的相互作用。以上结果表明该化合物具有较强的ATG4B抑制能力。
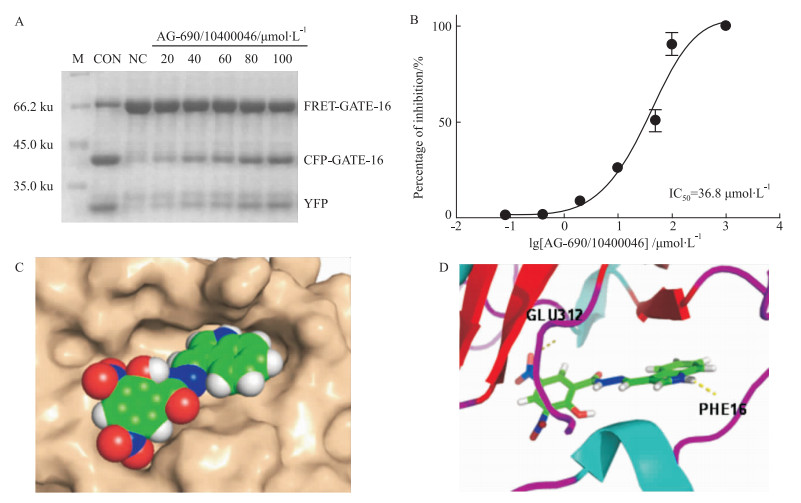
|
| Fig 3 Inhibitory effects of AG-690/10400046 on ATG4B activity A: Determination of the different concentrations of AG-690/10400046 on ATG4B activity using the SDS-PAGE assay; B: AG-690/10400046 showed an IC50 of 36.8 μmol·L-1 for ATG4B by using the FRET-based assay; C: The binding mode of AG-690/10400046 with ATG4B; D: AG-690/10400046 can form hydrophobic interactions with residues PHE16 and GLU312 of ATG4B |
为了判断AG-690/10400046是否广泛的抑制其他半胱氨酸蛋白酶,我们采用半胱氨酸蛋白酶家族的另一成员caspase-3来验证。如Fig 4A所示,星形孢菌素能有效的诱导HELA细胞产生凋亡。caspase-3的特异性抑制剂Z-VAD-FMK在50 μmol·L-1的浓度下能有效抑制caspase-3的酶切活性。而AG-690/10400046在100 μmol·L-1的浓度下对caspase-3的酶切活性没有影响,说明该化合物不是普遍的半胱氨酸蛋白酶抑制剂。
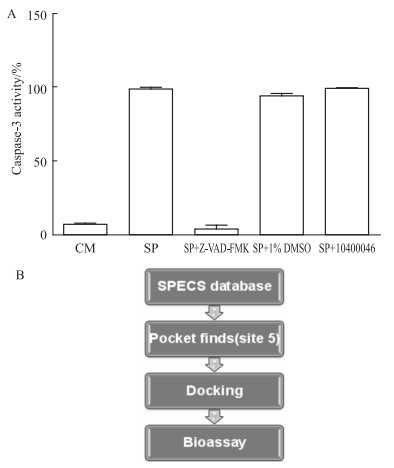
|
| Fig 4 Selectivity of AG-690/10400046and diagram of virtual screening (A) Selectivity test of AG-690/10400046 on another cysteine protease caspase-3.(SP:staurosporine).(B) The diagram of virtual screening of ATG4B inhibitors |
ATG4家族成员在脂化与去脂化ATG8家族成员以形成自噬体的过程中扮演着非常重要的作用。ATG4在酵母中仅有一个成员,其功能的缺失将阻断整个自噬的进程[11]。而在哺乳动物细胞中ATG4有4个家族成员:ATG4A、ATG4B、ATG4C和ATG4D。ATG4B作为ATG4家族中研究最为广泛的成员,对ATG8家族成员 (LC3家族及GABARAP家族) 均具有酶切活性[12]。
ATG4B的晶体结构 (PDB ID:2CY7) 已经被解析出来,其酶切活性位点为:CYS74、ASP278及HIS280[11],这些位点的突变将导致ATG4B酶切活性的丧失[13]。正常情况下,ATG4B的关键氨基酸位点CYS74被一个调节环 (259~262位氨基酸残基) 所遮盖。一旦ATG4B与LC3形成复合物,ATG4B的调节环部分被LC3的尾部残基PHE119提升起来,导致ATG4B蛋白结构发生一定的变化,使得LC3的C末端尾巴可以伸入到ATG4B的酶切位点[14]。另外ATG4B一旦与LC3结合,其N端尾巴 (1~20位氨基酸残基) 经历了很大的构型变化,这可能与其对LC3的去脂化作用有关,因为只有当ATG4B处于游离状态时更倾向于与膜上脂化形式的LC3-PE相结合[15]。
目前,自噬被报导与多种疾病相关,包括肿瘤、神经退行性疾病、感染性疾病、心血管和代谢性疾病等[16]。而ATG4B作为自噬过程中的一个关键性靶标,通过调控ATG4B的活性可以调控整个自噬的过程。因此,作为一个潜在的生物标记物及疾病治疗的靶标,特异性ATG4B小分子抑制剂的发现至关重要。
本研究旨在建立一种虚拟筛选的方法,并利用该方法筛选出特异性ATG4B小分子抑制剂 (Fig 4B)。我们基于游离态ATG4B的晶体结构2CY7,综合计算机虚拟筛选以及体外生物活性测试,得到具有明显ATG4B抑制活性的小分子化合物AG-690/10400046。AG-690/10400046,其IC50为36.8 μmol·L-1,优于文献报道的化合物NSC185058[8]。AG-690/10400046对半胱氨酸家族另一成员caspase-3无酶切抑制作用,说明该化合物为选择性ATG4B小分子抑制剂而不是广泛的半胱氨酸蛋白酶抑制剂。以上结果表明,我们建立了一种可靠的ATG4B小分子抑制剂的虚拟筛选方法,且筛选到了高选择性ATG4B小分子抑制剂AG-690/10400046。这为进一步寻找或设计更为高效的ATG4B小分子抑制剂及研究ATG4B抑制剂在疾病中的作用奠定了基础。
| [1] | 杨根梦, 陈逊, 曾晓锋. 甲基苯丙胺诱导神经细胞自噬的研究进展[J]. 中国药理学通报, 2016, 32 (10): 1341-4. Yang G M, Chen X, Zeng X F. Research progress of methamphetamine inducing nerve cell′s autophagy[J]. Chin Pharmacol Bull, 2016, 32 (10): 1341-4. |
| [2] | 齐元麟, 陈富华, 任正肖, 等. 动脉平滑肌细胞的钙池操纵钙通道对细胞自噬的调[J]. 中国药理学通报, 2016, 32 (10): 1416-21. Qi Y L, Chen F H, Ren Z X, et al. Regulation of autophagy by store-operated calcium channel in arterial smooth muscle cells[J]. Chin Pharmacol Bull, 2016, 32 (10): 1416-21. |
| [3] | Feng Y, He D, Yao Z, et al. The machinery of macroautophagy[J]. Cell Res, 2014, 24 (1): 24-41. doi:10.1038/cr.2013.168 |
| [4] | Kuma A, Mizushima N, Ishihara N, et al. Formation of the approximately 350-kDa Apg12-Apg5^Apg16 multimeric complex, mediated by Apg16 oligomerization, is essential for autophagy in yeast[J]. J Biol Chem, 2002, 277 (21): 18619-25. doi:10.1074/jbc.M111889200 |
| [5] | Yu Z Q, Ni T, Hong B, et al. Dual roles of Atg8-PE deconjugation by Atg4 in autophagy[J]. Autophagy, 2012, 8 (6): 883-92. doi:10.4161/auto.19652 |
| [6] | Liu P F, Leung C M, Chang Y H, et al. ATG4B promotes colorectal cancer growth independent of autophagic flux[J]. Autophagy, 2014, 10 (8): 1454-65. doi:10.4161/auto.29556 |
| [7] | Rothe K, Lin H, Lin K B, et al. The core autophagy protein ATG4B is a potential biomarker and therapeutic target in CML stem/progenitor cells[J]. Blood, 2014, 123 (23): 3622-34. doi:10.1182/blood-2013-07-516807 |
| [8] | Akin D, Wang S K, Habibzadegah-Tari P, et al. A novel ATG4B antagonist inhibits autophagy and has a negative impact on osteosarcoma tumors[J]. Autophagy, 2014, 10 (11): 2021-35. doi:10.4161/auto.32229 |
| [9] | Li M, Chen X, Ye Q Z, et al. A high-throughput FRET-based assay for determination of Atg4 activity[J]. Autophagy, 2012, 8 (3): 401-12. doi:10.4161/auto.18777 |
| [10] | Li M, Khambu B, Zhang H, et al. Suppression of lysosome function induces autophagy via a feedback down-regulation of MTOR complex 1(MTORC1) activity[J]. J Biol Chem, 2013, 288 (50): 35769-80. doi:10.1074/jbc.M113.511212 |
| [11] | Kirisako T, Ichimura Y, Okada H, et al. The reversible modification regulates the membrane-binding state of Apg8/Aut7 essential for autophagy and the cytoplasm to vacuole targeting pathway[J]. J Cell Biol, 2000, 151 (2): 263-76. doi:10.1083/jcb.151.2.263 |
| [12] | Li M, Hou Y, Wang J, et al. Kinetics comparisons of mammalian Atg4 homologues indicate selective preferences toward diverse Atg8 substrates[J]. J Biol Chem, 2011, 286 (9): 7327-38. doi:10.1074/jbc.M110.199059 |
| [13] | Kumanomidou T, Mizushima T, Komatsu M, et al. The crystal structure of human Atg4b, a processing and de-conjugating enzyme for autophagosome-forming modifiers[J]. J Mol Biol, 2006, 355 (4): 612-8. doi:10.1016/j.jmb.2005.11.018 |
| [14] | Sugawara K, Suzuki N N, Fujioka Y, et al. Structural basis for the specificity and catalysis of human ATG4B responsible for mammalian autophagy[J]. J Biol Chem, 2005, 280 (48): 40058-65. doi:10.1074/jbc.M509158200 |
| [15] | Satoo K, Noda N N, Kumeta H, et al. The structure of ATG4B-LC3 complex reveals the mechanism of LC3 processing and delipidation during autophagy[J]. EMBO J, 2009, 28 (9): 1341-50. doi:10.1038/emboj.2009.80 |
| [16] | 陈菊英, 刘朝纯, 曾智, 等. 紫草素通过PI3K/Akt通路促进人乳腺癌MCF-7细胞自噬[J]. 中国药理学通报, 2013, 29 (2): 194-8. Chen J Y, Liu Z C, Zeng Z, et al. Shikonin promotes autophagy of MCF-7 human breast cancer cells through PI3K /Akt pathway[J]. Chin Pharmacol Bull, 2013, 29 (2): 194-8. |