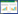文章信息
- 谢铱子, 詹少锋, 黄慧婷, 温武金, 刘小虹, 江勇
- XIE Yizi, ZHAN Shaofeng, HUANG Huiting, WEN Wujin, LIU Xiaohong, JIANG Yong
- 生物信息学联合机器学习鉴定重症登革热的预警标志物
- Bioinformatics combined with machine learning to identify early warning markers for severe dengue
- 中国医科大学学报, 2024, 53(7): 583-590
- Journal of China Medical University, 2024, 53(7): 583-590
-
文章历史
- 收稿日期:2023-08-25
- 网络出版时间:2024-06-26 14:27:20
2. 广州中医药大学第一临床医学院, 广州 510405;
3. 广州中医药大学第一附属医院呼吸与危重症医学科, 广州 510405;
4. 广州中医药大学岭南医学研究中心, 广州 510405;
5. 广东省中医临床研究院, 广州 510405
2. The First Clinical Medical College of Guangzhou University of Chinese Medicine, Guangzhou 510405, China;
3. Department of Respiratory and Critical Care Medicine, The First Affiliated Hospital of Guangzhou University of Chinese Medicine, Guangzhou 510405, China;
4. Lingnan Medical Research Center of Guangzhou University of Chinese Medicine, Guangzhou 510405, China;
5. Guangdong Clinical Research Academy of Chinese Medicine, Guangzhou 510405, China
登革热是由登革病毒(dengue virus,DENV)感染引起的、全球传播最广泛的蚊媒传染病之一[1]。据统计,登革热已于全球100多个国家流行,发病率在近几十年迅速升高。2000年各地向世界卫生组织报告了505 430例登革热,2019年增加到520万例[2]。临床上登革热分为普通登革热和重症登革热,普通登革热表现为发热、头痛、全身肌肉关节疼痛等症状,部分患者可进展为重症登革热,出现严重出血、四肢渗漏征、休克等,危及生命[1]。重症登革热的发生机制尚不清楚,目前认为可能和宿主的年龄、性别与基础疾病,病毒毒力,抗体依赖增强感染作用等多种因素有关[3]。尽早诊断与治疗是降低重症登革热致残率与病死率,提高患者生活质量的关键[3]。探讨重症登革热发生的风险基因、潜在生物过程及通路,有助于挖掘重症登革热预警的生物标志物和临床上对普通登革热患者的重症风险评估,同时有助于重症登革热疾病机制的阐明,也有利于特异性治疗药物的研发。
本研究基于生物信息学与机器学习,以登革热患者的基因芯片为基础,探讨与重症登革热预警相关的生物标志物,为临床重症登革热发生的风险评估提供参考。
1 材料与方法 1.1 数据来源2009年世界卫生组织把传统的登革热与登革热出血热/登革热休克综合征,更新为有或无预警指征的登革热和重症登革热[4]。我国也在《中华人民共和国卫生行业标准-登革热诊断(WS216-2018)》 [5]中进行了同样的修改,故本研究将登革热出血热、登革休克综合征归为重症登革热。
通过基因表达综合(Gene Expression Omnibus,GEO)(https://www.ncbi.nlm.nih.gov/geo/)下载基因芯片GSE96656和GSE25001。芯片以患者的全血为样本。GSE96656取7例登革热出血热患者为实验组,24例普通登革热患者为对照组。GSE25001取12例登革休克综合征患者为实验组,30例普通登革热患者为对照组。芯片间的批次效应去除则通过R4.1.2软件的“sva”包完成。
1.2 基因差异表达分析基于R4.1.2软件的“limma”包,以|log2FC| > 0.5,P < 0.05为标准,计算出重症登革热与普通登革热之间的差异表达基因,视为普通登革热发展为重症登革热的潜在基因。
1.3 富集分析通过微生信网站(https://www.bioinformatics.com.cn/)、“clusterProfiler”等R语言包对差异表达基因进行基因本体论(Gene Ontology,GO)功能富集分析,京都基因与基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析,其中GO功能富集分析包括分子功能(molecular function,MF)分析、细胞组分(cellular component,CC)分析、生物过程(biological process,BP)分析。设置P < 0.05,对结果作可视化展示。
1.4 筛选重症登革热预警基因以差异表达基因为基础,利用随机森林模型预测普通登革热发展为重症登革热的预警基因。采用R软件包“randomForest”构建随机森林模型。根据基因的重要性评分挑选出分数最高的5个基因,作为普通登革热发展为重症登革热的预警基因,对预警基因的表达水平进行可视化处理。
1.5 重症登革热预警基因的准确性验证采用R4.1.2软件的“pROC”包绘制受试者操作特征(receiver operating characteristic,ROC)曲线,评估普通登革热发展为重症登革热预警基因的准确性。
1.6 列线图的构建构建列线图模型以量化预警基因。通过预警基因的表达量预测普通登革热进展为重症登革热的风险。基于logistic回归模型,采用R4.1.2软件的“rms”包构建列线图模型,绘制校准曲线展现模型的预测符合度,并采用“rmda”R语言包绘制临床决策曲线分析(decision curve analysis,DCA)与临床影响曲线。
2 结果 2.1 基因差异表达分析对登革热基因进行差异表达分析,共得到817个差异表达基因,其中,上调基因404个,下调基因413个,见图 1。
|
| 图 1 重症登革热差异表达基因火山图 Fig.1 Volcano plot of differentially expressed genes of severe dengue |
2.2 富集分析
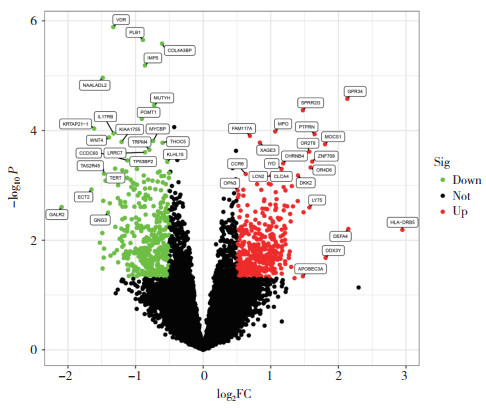
对差异表达基因进行GO功能富集分析,包括BP、CC、MF分析。P值越小,显著性越高。每个分析都挑选出显著性较高的条目进行柱状图展示,见图 2。GO富集分析主要涉及抗微生物体液反应、杀死其他生物的细胞、体液免疫反应、分泌颗粒腔、细胞质泡腔、丝氨酸型肽酶活性、丝氨酸水解酶活性、细胞-细胞黏附介质活性,作为受体作用于过氧化物等。
|
| 图 2 重症登革热的差异表达基因的GO功能富集分析 Fig.2 GO functional enrichment analysis on differentially expressed genes of severe dengue |
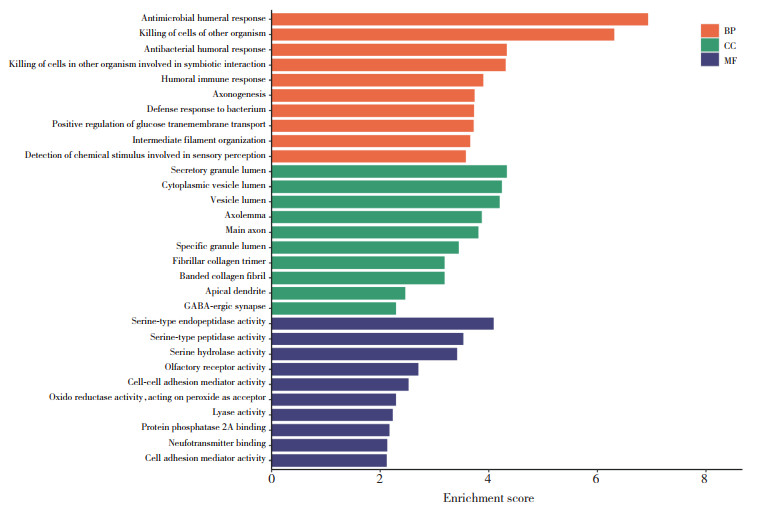
对差异表达基因进行KEGG通路富集分析,选取显著性较高的条目进行展示,见图 3。KEGG富集分析结果提示,催乳素信号通路、糖鞘脂生物合成-乳糖和新内酯系列、内分泌和其他因素调节钙重吸收、花生四烯酸代谢等与普通登革热发展为重症登革热相关。
|
| 图 3 重症登革热的差异表达基因的KEGG通路富集分析 Fig.3 KEGG pathway enrichment analysis on differentially expressed genes of severe dengue |
2.3 重症登革热预警基因筛选
对差异表达基因进行随机森林模型构建,得到随机森林树的数量与模型误差之间的相关图,接着根据基尼系数法计算每个基因的重要性得分,评分越高,基因越重要(图 4)。重要性评分最高的5个基因(AZU1、PDCD4、COL4A3BP、TRPM4、ATP4A)作为普通登革热进展为重症登革热的预警基因。
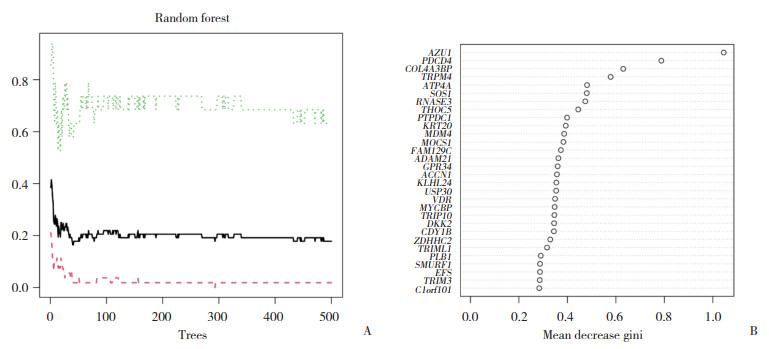
|
| A, random forest model; B, score chart of gene importance. The horizontal coordinates indicate the number of trees. The vertical coordinates indicate the cross-validation error. The green and red curves represent the errors of the control and experimental groups, respectively. The black curve represents the errors of all the samples. 图 4 重症登革热的预警基因筛选 Fig.4 Screening of warning genes of severe dengue |
2.4 预警基因表达情况
比较普通登革热发展为重症登革热的预警基因的表达量(图 5)。结果显示,相比于普通登革热患者,ATP4A、COL4A3BP、TRPM4在重症登革热患者呈现低表达状态;AZU1、PDCD4在重症登革热患者中的表达量偏高,提示ATP4A、COL4A3BP、TRPM4可能通过基因表达水平的下调影响重症登革热的发生,而AZU1、PDCD4则相反。
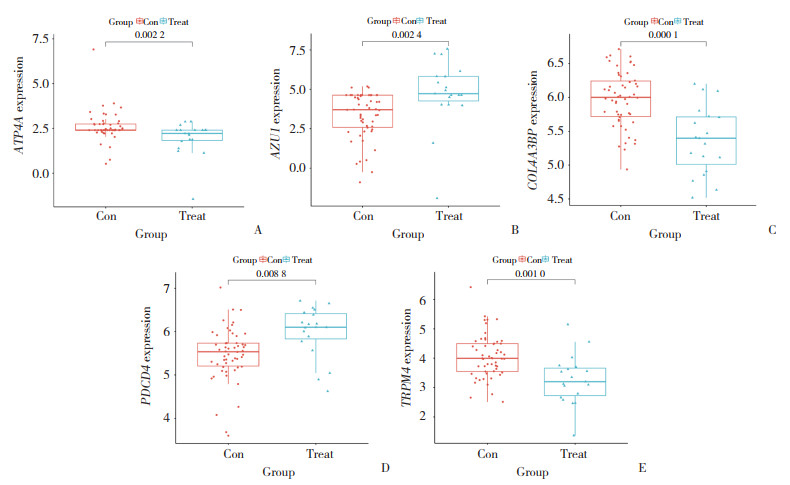
|
| A, ATP4A; B, AZU1; C, COL4A3BP; D, PDCD4; E, TRPM4. 图 5 重症登革热预警基因表达情况 Fig.5 Expression of warning genes of severe dengue |
2.5 预警基因准确性验证
为进一步验证上述5个基因作为普通登革热发展为重症登革热预警基因的准确性,对其进行ROC曲线分析。结果显示,5个基因的AUC均在0.7以上,准确性良好,见图 6。

|
| A, ATP4A; B, AZU1; C, COL4A3BP; D, PDCD4; E, TRPM4. 图 6 重症登革热预警基因的ROC曲线 Fig.6 ROC curves of warning genes of severe dengue |
2.6 列线图
进一步量化5个预警基因对普通登革热发展为重症登革热的诊断价值,针对5个基因构建列线图模型(图 7)。通过Bootstrap法对列线图作内部验证,以原始数据重复抽样1 000次,结果显示,平均绝对误差为0.046,预测曲线和标准曲线拟合度良好,表明模型预测准确度良好。DCA曲线结果显示,当列线图的预测概率为0.10~0.99时,列线图的临床获益率良好。临床影响曲线结果显示,高风险阈值> 0.8,预测事件发生与实际事件发生高度重叠,提示列线图的临床有效性较好。因此,检测普通登革热患者ATP4A、COL4A3BP、TRPM4、AZU1和PDCD4基因的表达量以评估该患者发展为重症登革热的风险,具有一定的可行性。
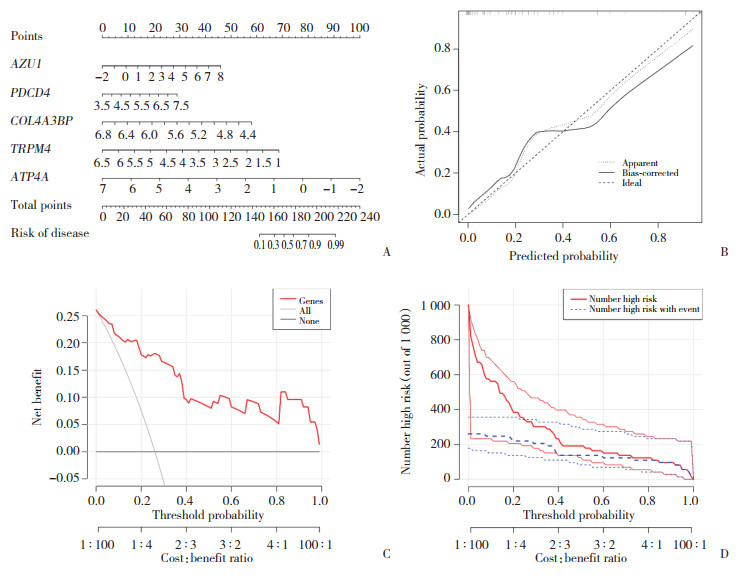
|
| A, nomograms; B, calibration curve; C, clinical decision curve; D, clinical impact curve. 图 7 基于预警基因的重症登革热风险预测模型 Fig.7 Risk prediction model for severe dengue based on warning genes |
3 讨论
本研究挖掘了重症登革热风险评估模型。结果显示,ATP4A、COL4A3BP、TRPM4在重症登革热中呈低表达,而AZU1、PDCD4相反。AZU1又称肝素结合蛋白,可促进炎症、诱导血管渗漏及水肿形成,进而造成器官功能障碍,人体在发生低血压或器官功能障碍前的数小时内,血浆AZU1水平升高[6]。而血管渗漏、严重脏器损伤与休克是重症登革热的主要表现[1]。细胞凋亡是DENV感染后的宿主反应之一,病毒复制会引起细胞紊乱,触发细胞凋亡相关的信号通路或特定传感器[7]。PDCD4与细胞凋亡有关,参与细胞周期调节、衰老和炎症反应等生物过程[8]。抑制PDCD4介导的细胞凋亡能够缓解病毒性心肌炎[9]。在重症登革热中,PDCD4水平上调可能与病毒性心肌损害有关。COL4A3BP又称神经酰胺转移蛋白,可将神经酰胺从内质网转移至高尔基体,促进鞘磷脂合成[10]。而COL4A3BP失活与高尔基体拆卸均有助于促凋亡应激期间鞘磷脂合成的减少[11]。TRPM7下调可明显减少T细胞迁移数量及平均速度[12]。TRPM7可能通过T细胞影响宿主免疫反应,进而影响登革热的发生发展。ATP4A表达下调与结肠炎有关,药物可减少ATP4A下调引起的缺氧诱导糖酵解,从而改善结肠腔pH值、缓解结肠黏膜上皮细胞缺氧诱导的糖酵解[13]。DENV可诱导糖酵解途径以支持有效的病毒复制[14]。故ATP4A下调可能通过诱导糖酵解来支持DENV复制。综上,AZU1、PDCD4、COL4A3BP、TRPM4、ATP4A均可能与重症登革热密切相关,这为重症登革热风险预测模型提供了理论支持。
GO富集分析提示细胞质泡腔、丝氨酸型肽酶活性、抗微生物体液反应、体液免疫反应、丝氨酸水解酶活性等与重症登革热的发生有关。适应性免疫系统中的体液免疫由B细胞与中和抗体介导[15]。B细胞激活后,可生成病毒特异性抗体,部分抗体与病毒包膜蛋白结合并中和病毒粒子,阻止病毒进入靶细胞[16]。DENV丝氨酸蛋白酶在病毒复制中起着关键作用,已被视为DENV感染的关键药物靶标[17]。在普通登革热进展为重症登革热时,上述生物过程可能发挥重要作用。
KEGG富集分析结果主要涉及花生四烯酸代谢等通路。研究[18]发现,2型DENV感染人神经母细胞瘤细胞后,由磷脂酶A2激活、花生四烯酸升高、超氧化物阴离子生成与核因子κB(nuclear factor kappa-B,NF-κB)激活组成的通路会被活化,最终导致细胞凋亡。因此,花生四烯酸代谢通路可能通过细胞凋亡影响重症登革热的发生发展。
综上所述,本研究提出了AZU1、PDCD4、COL4A3BP、TRPM4、ATP4A的重症登革热风险评估模型。GO和KEGG富集分析结果显示,丝氨酸型肽酶、丝氨酸水解酶以及花生四烯酸代谢等可能与DENV密切相关。但本研究结果为计算机分析所得,仍需进一步的动物实验与临床研究加以验证。
| [1] |
中华医学会感染病学分会, 中华医学会热带病与寄生虫学分会, 中华中医药学会急诊分会. 中国登革热临床诊断和治疗指南[J]. 传染病信息, 2018, 31(5): 385-392. DOI:10.3969/j.issn.1007-8134.2018.05.001 |
| [2] |
WHO. Dengue and severe dengue[EB/OL]. (2023-03-17)[2023-05-01]. https://www.who.int/news-room/fact-sheets/detail/dengue-and-severe-dengue.
|
| [3] |
邱爽, 张复春, 洪文昕, 等. 重症登革热临床特点及危险因素分析[J]. 中国现代药物应用, 2021, 15(10): 86-88. DOI:10.14164/j.cnki.cn11-5581/r.2021.10.031 |
| [4] |
WHO/TDR. Dengue: guidelines for diagnosis, treatment, prevention and control: new edition[M]. Geneva: World Health Organization, 2009: 1-147.
|
| [5] |
国家卫生和计划生育委员会. 登革热诊断: WS 216—2018[S]. 北京: 中国标准出版社, 2018.
|
| [6] |
FISHER J, LINDER A. Heparin-binding protein: a key player in the pathophysiology of organ dysfunction in sepsis[J]. J Intern Med, 2017, 281(6): 562-574. DOI:10.1111/joim.12604 |
| [7] |
COURAGEOT MP, CATTEAU A, DESPRÈS P. Mechanisms of Dengue virus-induced cell death[M]. Advances in Virus Research. Amsterdam: Elsevier, 2003: 157-186. DOI: 10.1016/s0065-3527(03)60005-9.
|
| [8] |
CHEN G, LI PH, HE JY, et al. Molecular cloning, inducible expression with SGIV and Vibrio alginolyticus challenge, and functional analysis of Epinephelus coioides PDCD4[J]. Dev Comp Immunol, 2021, 119: 104013. DOI:10.1016/j.dci.2021.104013 |
| [9] |
HE J, YUE Y, DONG CS, et al. MiR-21 confers resistance to CVB3-induced myocarditis by inhibiting PDCD4-mediated apoptosis[J]. Clin Invest Med, 2013, 36(2): E103-E111. DOI:10.25011/cim.v36i2.19573 |
| [10] |
YAMAJI T, KUMAGAI K, TOMISHIGE N, et al. Two sphingolipid transfer proteins, CERT and FAPP2, are involved in sphingolipid metabolism[J]. IUBMB Life, 2008, 60(8): 511-518. DOI:10.1002/iub.83 |
| [11] |
CHANDRAN S, MACHAMER C. Inactivation of ceramide transfer protein during proapoptotic stress by Golgi disassembly and caspase cleavage[J]. Biochem J, 2012, 442(2): 391-401. DOI:10.1042/bj20111461 |
| [12] |
KURAS Z, YUN YH, CHIMOTE AA, et al. KCa3.1 and TRPM7 channels in the uropod regulate the migration of activated human T cells[J]. PLoS One, 2012, 7(8): e43859. DOI:10.1371/journal.pone.0043859 |
| [13] |
HE W, PAN HF, TAO P, et al. Panax notoginseng attenuated hypoxia-induced glycolysis in colonic mucosal epithelial cells in DSS-induced colitis[J]. Ann Transl Med, 2022, 10(4): 218. DOI:10.21037/atm-22-566 |
| [14] |
FONTAINE KA, SANCHEZ EL, CAMARDA R, et al. Dengue virus induces glycolysis for optimal replication[J]. J Virol, 2015, 89(4): 2358-2366. DOI:10.1128/JVI.02309-14 |
| [15] |
LEE MF, VOON GZ, LIM HX, et al. Innate and adaptive immune evasion by the dengue virus[J]. Front Cell Infect Microbiol, 2022, 12: 1004608. DOI:10.3389/fcimb.2022.1004608 |
| [16] |
KING CA, WEGMAN AD, ENDY TP. Mobilization and activation of the innate immune response to the dengue virus[J]. Front Cell Infect Microbiol, 2020, 10: 574417. DOI:10.3389/fcimb.2020.574417 |
| [17] |
DWIVEDI VD, BHARADWAJ S, AFROZ S, et al. Evaluation of anti-dengue activity of bioflavonoids from Azadirachta indica by dengue virus serine protease inhibition[J]. J Biomol Struct Dyn, 2021, 39(4): 1417-1430. DOI:10.1080/07391102.2020.1734485 |
| [18] |
JAN JT, CHEN BH, MA SH, et al. Potential dengue virus-triggered apoptotic pathway in human neuroblastoma cells: arachidonic acid, superoxide anion, and NF-kappaB are sequentially involved[J]. J Virol, 2000, 74(18): 8680-8691. DOI:10.1128/jvi.74.18.8680-8691.2000 |
 2024, Vol. 53
2024, Vol. 53