文章信息
- 杨丛语, 邓青春, 刘嘉铭
- YANG Congyu, DENG Qingchun, LIU Jiaming
- 基底膜基因在子宫内膜癌中的表达及其临床意义
- Expression and clinical significance of basement membrane genes in endometrial carcinoma
- 中国医科大学学报, 2024, 53(5): 451-457
- Journal of China Medical University, 2024, 53(5): 451-457
-
文章历史
- 收稿日期:2023-08-29
- 网络出版时间:2024-05-14 14:25:58
2. 海南医学院第二附属医院妇科,海口 570311;
3. 四川大学华西医院泌尿外科,成都 610041
2. Department of Gynecology, The Second Affiliated Hospital of Hainan Medical University, Haikou 570311, China;
3. Department of Urology, West China Hospital of Sichuan University, Chengdu 610041, China
子宫内膜癌是常见的女性恶性肿瘤,在全球癌症病例中占很大比例[1]。近年来,由于生活方式的改变和雌激素替代疗法的增加,子宫内膜癌的患病率急剧上升,对女性健康构成了严重威胁。以手术治疗为主、放化疗和激素治疗为辅的方案疗效仍不理想,约1/5的患者会术后复发。大多数子宫内膜癌患者的死因都与未能早期识别和治疗癌症有关。因此,确定子宫内膜癌的新靶点对其诊断和治疗非常重要[2]。
基底膜的主要组成成分是层粘连蛋白和胶原Ⅳ,它是位于上皮下并环绕大多数组织的片状结构,是细胞外基质的致密层,在组织形成和功能发挥中起重要作用[3]。基底膜将上皮、内皮、神经、脂肪与下层组织分离,为其覆盖的细胞提供丰富的物理和生化信号,其作用是维持组织结构的完整,保障细胞信号转导,发挥膜屏障功能。LAMBERT等[4]认为,90%的癌症死亡与肿瘤细胞通过基底膜入侵间质、淋巴结和血管进而转移有关。LUO等[5]认为,基底膜的完整性是乳腺癌和结直肠癌的重要预后指标。子宫内膜癌细胞通过附着在基底膜层粘连蛋白上,产生降解细胞外基质的蛋白水解酶并跨基底膜迁移,进而实现远距离转移和对宿主组织的侵袭,此过程的关键环节是癌细胞与层粘连蛋白的相互作用。基质金属蛋白酶等在细胞外基质中通过降解基底膜胶原Ⅳ,在恶性肿瘤的早期侵袭中发挥重要作用,并参与子宫内膜癌的发展[6]。这说明基底膜的2种主要成分均与子宫内膜癌密切相关,为数据分析提供了生物学理论基础。
JAYADEV等[7]发现了224个与人类疾病紧密相关的基底膜基因,揭示了基底膜基因在癌症中的重要作用。目前,尚无研究证实基底膜基因在子宫内膜癌中发挥的作用。本研究应用基因组学转录本和临床数据,通过生物信息学分析联合细胞实验,探讨这些基底膜基因在子宫内膜癌中的表达情况和预后价值。
1 材料与方法 1.1 数据来源从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中下载子宫内膜癌的转录数据以及35个正常子宫内膜样本和552个癌症样本的临床信息(包括年龄、FIGO分期、肿瘤分级),剔除部分信息缺失的样本后,纳入548例子宫内膜癌患者的临床信息用于后续分析。从JAYADEV等[7]的研究中直接获取224个癌症相关基底膜基因。
1.2 筛选差异基因和构建预后模型采用R语言的limma包筛选差异基因,标准为错误发现率(false discovery rate,FDR)<0.05且|log2FC|>1。采用R语言的ggplot2包识别上调和下调的基因,绘制火山图。采用R语言中的scale函数将TCGA的表达数据进行集中化和标准化处理后计算风险评分,风险评分=
将差异基因上传到David数据库(https://david.ncifcrf.gov),选择FDR值最小的生物过程、细胞成分、分子功能通路各3条,采用R语言的GOplot包进行基因本体(Gene Ontology,GO)分析,采用R语言的Cluster-Profile包进行京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析。
1.4 荧光定量PCR采用人子宫内膜上皮细胞ECS(武汉赛奥斯生物科技有限公司)和人子宫内膜癌细胞Ishikawa(武汉尚恩生物技术有限公司)进行实验。磷酸盐缓冲液冲洗细胞,加入TRIzol(武汉赛奥斯生物科技有限公司)反复吹打,直到细胞完全裂解后提取细胞裂解液中的总RNA。应用反转录试剂盒(武汉尚恩生物技术有限公司)将RNA逆转录为cDNA,将其稀释后经变性、退火共45个循环得到标准曲线。选择比较阈值法,以GAPDH为内参,采用2-△△CT法计算目的基因mRNA的相对表达量。引物序列:ACHE,正向5’-GAGGGCTCGTATTTTCTGGTTTAC-3’,反向5’-CCAGGTCACTTACCTGGGGA-3’;CD44,正向5’- CACACCCTCCCCTCATTCAC -3’,反向5’- TGGATGGCTGGTATGAGCTG -3’;COL12A1,正向5’- TCCCACTCAGATCCTAG -3’,反向5’- CTCCCTCCCGGTGCATT -3’;GAPDH,正向5’- CACCACCAACTGCTTAGCC -3’,反向5’- AGTGGATGCAGGGATGATGT -3’。
1.5 统计学分析采用R语言(4.3.1版)和GraphPad Prism10进行统计学分析。采用单因素和多因素Cox回归分析评估预后模型的预测价值;采用Kaplan-Meier生存曲线和log-rank检验预测患者的总生存时间。计量资料用x±s表示,2组间的比较采用独立样本t检验。P<0.05为差异有统计学意义。
2 结果 2.1 筛选差异基因和构建预后模型经筛选得到91个差异基因,选择差异最显著的前30个基因,并在热图中展示。见图 1。
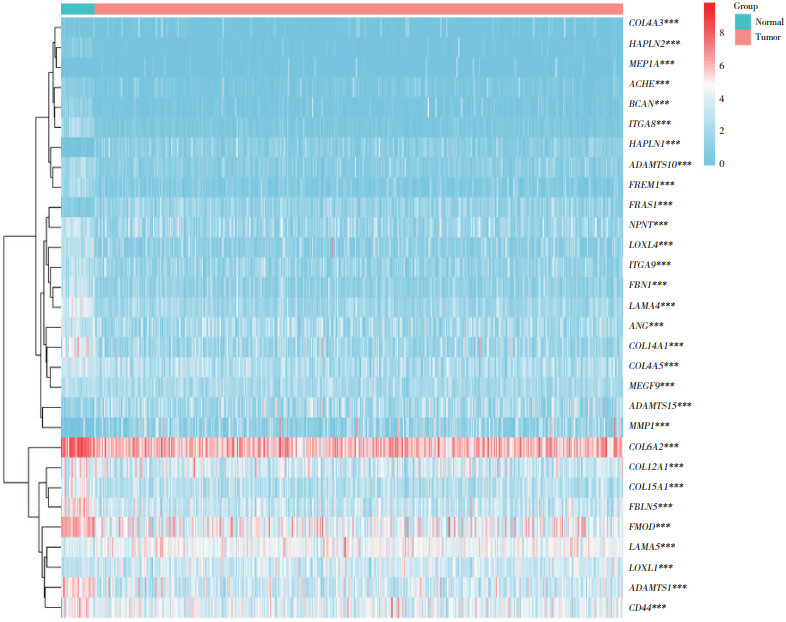
|
| *** P<0.001. 图 1 子宫内膜癌和正常子宫内膜间前30个差异基底膜基因的热图 Fig.1 Heatmap of the top 30 differential basement membrane genes compared in endometrial carcinoma and normal endometrium |
单因素Cox回归初步筛选出9个基因(ADAMTS16、COL4A6、ADAMTS15、ACHE、ANG、ADAM17、COL12A1、CD44、COL4A5),多因素Cox回归分析筛选出3个预后基因(ACHE、CD44、COL12A1)并纳入建模(图 2A、2B)。根据中位风险评分将患者平均分为高风险组和低风险组,可见患者的死亡率随风险评分的增加而增加。ROC曲线显示,患者1、3、5年的生存率分别为0.631、0.687、0.690,提示该模型具有良好的预测能力。Kaplan-Meier生存曲线显示,高风险组患者的生存率更低(P<0.001)。主成分分析表明,高风险组和低风险组2组内散点聚集较密集且2组间散点的富集区域明显不同,说明2组内各样本的相似度和2组间的区分度都很高,数据的可靠性很强;低风险组患者的无进展生存期明显更长,说明其带瘤生存期更长。见图 2C~2H。
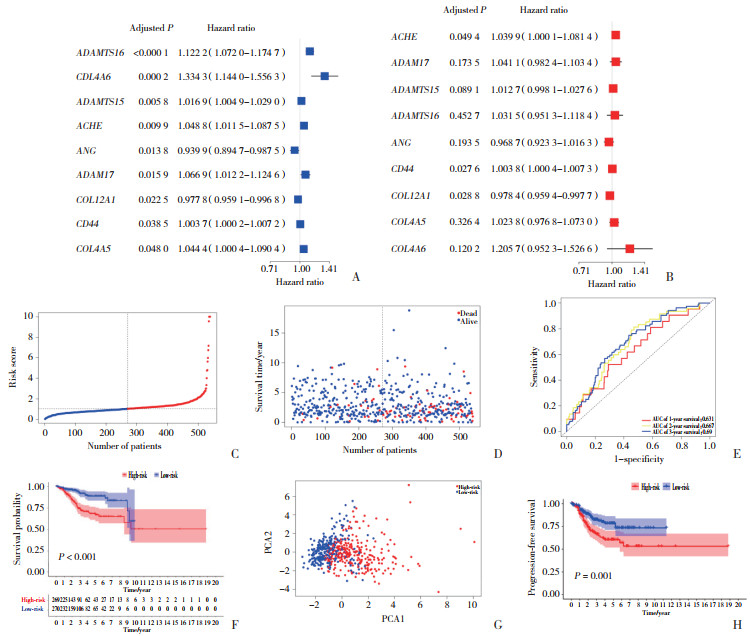
|
| A,univariate Cox regression analyses;B,multivariate Cox regression analyses;C and D,distribution of survival status based on the median risk score;E,ROC analysis to predict survival probability;F,Kaplan-Meier analysis;G,PCA analysis;H,progression-free survival analysis. 图 2 基底膜基因预后模型的构建与评价 Fig.2 Construction and evaluation of the basement membrane gene prognostic model |
2.2 预后分析
采用Cox回归模型进行独立预后分析。单因素Cox回归分析显示,年龄(P = 0.002 1)、肿瘤分级(P<0.000 1)、FIGO分期(P<0.000 1)、风险评分(P<0.000 1)均与患者的预后显著相关。多因素Cox回归分析显示,年龄(P= 0.018 1)、肿瘤分级(P = 0.022 2)、FIGO分期(P<0.001)、风险评分(P<0.05)仍与预后显著相关,故该预后模型可以准确预测子宫内膜癌患者的预后(图 3A、3B)。采用χ2检验进行临床预后分析,结果显示,年龄、肿瘤分级、FIGO分期的二分类变量间有统计学差异(P<0.001),年龄≥65岁、肿瘤分级高、肿瘤分期Ⅲ+Ⅳ期患者的风险评分明显更高,即预后更差(图 3C~3E)。
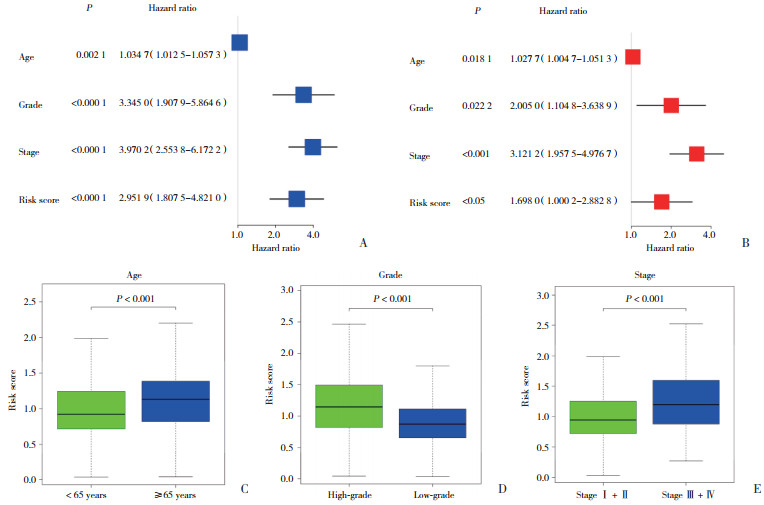
|
| A,univariate Cox regression;B,multivariate Cox regression;C-E,differences in age (<65 years and ≥65 years),grade (high- and low-grade),and stage (stage Ⅰ + Ⅱ and stage Ⅲ + Ⅳ) between the two subgroups. 图 3 结合临床因素的预后分析 Fig.3 Prognostic analyses combined with clinical factors |
2.3 功能富集分析
GO和KEGG分析能够揭示基底膜基因的生物学功能和作用途径。GO富集分析显示,基底膜基因主要参与的生物过程是构建细胞外基质和胶原纤维组织;主要富集在基底膜、细胞外基质、细胞外间隙;发挥的分子功能主要是构建细胞外基质、结合整合素和胶原(图 4A)。KEGG分析提示,基底膜基因主要参与细胞外基质受体相互作用、黏着斑、人乳头瘤病毒感染、PI3K-Akt信号通路、蛋白质的消化和吸收、肥大性心肌病等信号通路中(图 4B)。
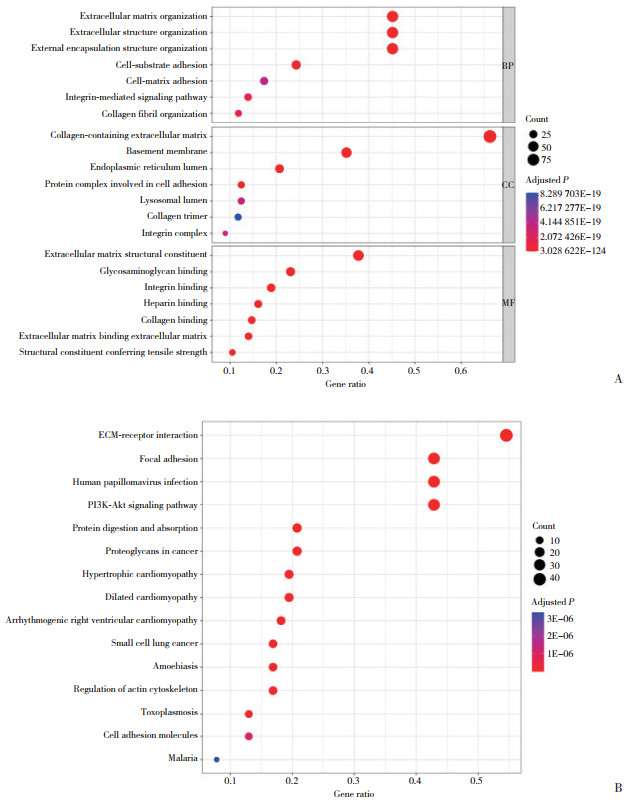
|
| A,GO analysis;B,KEGG analysis. 图 4 功能富集分析 Fig.4 Functional enrichment analysis |
2.4 预后基因在子宫内膜癌细胞系中的表达水平
荧光定量PCR结果显示,ECS细胞和Ishikawa细胞中,ACHE mRNA的表达量分别为1.47±0.09和3.24±0.34,COL12A1 mRNA的表达量分别为1.08±0.17和0.41±0.14,CD44 mRNA的表达量分别为1.08±0.14和0.41±0.11。与ECS细胞相比,Ishikawa细胞中ACHE mRNA的表达量显著增加(t = 8.848,df=4,P<0.001),COL12A1 mRNA的表达量显著降低(t = 5.435,df=4,P<0.01),CD44 mRNA的表达量无统计学差异(t = 0.644 2,df=4,P>0.05)。
3 讨论
子宫内膜癌是一种预后较差的妇科恶性肿瘤,传统的病理分类方法不利于深入认识其多样性特征。由于影响基因表达的因素众多,故基于单基因预测癌症的预后不具有说服力;相反,由多基因构建的组合模型可以更精确地预测癌症预后[8],这对患者的个体化治疗非常重要。因此,本研究构建了基底膜基因的子宫内膜癌预后模型。
CD44、COL12A1、ACHE是通过生物信息学分析筛选出的3个预后基因。CD44是一种单跨膜糖蛋白,在胚胎干细胞、结缔组织和骨髓上均不同程度地表达。它的主要配体是在间质细胞和癌细胞中高表达的透明质酸,当它结合CD44的配体结构域时会诱导构象改变,激活各种信号通路,导致细胞增殖、黏附、迁移和侵袭[9]。这表明CD44与癌症和细胞外基质具有重要关联。COL12A1位于人类染色体6q12-q13上,属于断裂的三螺旋胶原纤维蛋白家族成员,在胃癌、结直肠癌、乳腺癌中高表达,并可促进肿瘤的迁移、侵袭和转移[10]。与既往研究不同的是,本研究发现COL12A1在子宫内膜癌中属于保护性因素,与正常组织相比,子宫内膜癌细胞中其表达量显著降低,未来应对COL12A1在不同肿瘤中表达量的差异进行深入研究。乙酰胆碱参与细胞增殖分化和免疫应答等多种生物学过程,它积累在肿瘤微环境中,能支持肿瘤细胞的生长[11]。乙酰胆碱酯酶在胆碱能系统中发挥重要作用,其失调与多种人类疾病有关[12]。水解乙酰胆碱可降低肝癌的发病风险,因此有研究[11]提出将ACHE确定为肝癌的预后标志物。利用转铁蛋白修饰脂质体靶向肝癌细胞表面递送ACHE基因,可作为一种潜在的肝癌疗法[12]。有关ACHE基因在癌症中的分子机制的研究很少。本研究结果提示,ACHE基因与子宫内膜癌存在某种关联,其分子机制值得进一步探索。
本研究使用多种方法评价模型的预测能力。Kaplan-Meier生存曲线表明,高风险组与低风险组患者的生存时间具有显著差异;ROC曲线和PCA证明该模型具有良好的预测能力;独立预后分析表明,风险评分是子宫内膜癌的预后指标;临床预后分析提示,该模型与子宫内膜癌患者的年龄、肿瘤分级、FIGO分期显著相关,说明了该预后模型与临床因素具有良好的契合度。PCR结果显示,子宫内膜癌细胞中ACHE mRNA表达量显著高于正常细胞,提示其作为危险因素可以增加子宫内膜癌的发病风险;COL12A1在子宫内膜癌细胞中的表达量显著降低,提示其作为保护因素可以降低子宫内膜癌的发病风险;然而,基于生物信息学方法筛选出的CD44基因在子宫内膜癌细胞和正常子宫内膜细胞间的表达量无统计学差异,推测它可能是联合其他基因在预测子宫内膜癌的预后中起到辅助作用,这种作用可能会加强或减弱ACHE和COL12A1的表达,但CD44本身表达水平的高低与子宫内膜癌的患病风险并无直接关联。
综上所述,本研究基于3个基底膜基因建立了预后模型,分析了其与临床因素的相关性,论证了该预后模型可以预测子宫内膜癌患者的预后,同时从功能通路的角度描述了基因的各项特征,为深层次的研究提供了依据,为临床治疗提供了潜在靶点。
| [1] |
HUVILA J, PORS J, THOMPSON EF, et al. Endometrial carcinoma: molecular subtypes, precursors and the role of pathology in early diagnosis[J]. J Pathol, 2021, 253(4): 355-365. DOI:10.1002/path.5608 |
| [2] |
ZHOU C, LI C, YAN F, et al. Identification of an immune gene signature for predicting the prognosis of patients with uterine corpus endometrial carcinoma[J]. Cancer Cell Int, 2020, 20(1): 541. DOI:10.1186/s12935-020-01560-w |
| [3] |
POZZI A, YURCHENCO PD, IOZZO RV. The nature and biology of basement membranes[J]. Matrix Biol, 2017, 57-58: 1-11. DOI:10.1016/j.matbio.2016.12.009 |
| [4] |
LAMBERT AW, PATTABIRAMAN DR, WEINBERG RA. Emerging biological principles of metastasis[J]. Cell, 2017, 168(4): 670-691. DOI:10.1016/j.cell.2016.11.037 |
| [5] |
LUO X, FONG ELS, ZHU C, et al. Hydrogel-based colorectal cancer organoid co-culture models[J]. Acta Biomater, 2021, 132: 461-472. DOI:10.1016/j.actbio.2020.12.037 |
| [6] |
BERGER AA, KAWALER EA, DAO F, et al. The role of CTNNB1 mutations and matrix metalloproteinases (MMPs) in anti-angiogenesis treatment of endometrial carcinoma[J]. Gynecol Oncol, 2022, 167(2): 323-333. DOI:10.1016/j.ygyno.2022.09.013 |
| [7] |
JAYADEV R, MORAIS MRPT, ELLINGFORD JM, et al. A basement membrane discovery pipeline uncovers network complexity, regulators, and human disease associations[J]. Sci Adv, 2022, 8(20): eabn2265. DOI:10.1126/sciadv.abn2265 |
| [8] |
HU Y, ZHENG M, ZHANG D, et al. Identification of the prognostic value of a 2-gene signature of the WNT gene family in UCEC using bioinformatics and real-world data[J]. Cancer Cell Int, 2021, 21(1): 516. DOI:10.1186/s12935-021-02215-0 |
| [9] |
XU H, NIU M, YUAN X, et al. CD44 as a tumor biomarker and therapeutic target[J]. Exp Hematol Oncol, 2020, 9(1): 36. DOI:10.1186/s40164-020-00192-0 |
| [10] |
XIANG Z, LI J, SONG S, et al. A positive feedback between IDO1 metabolite and COL12A1 via MAPK pathway to promote gastric cancer metastasis[J]. J Exp Clin Cancer Res, 2019, 38(1): 314. DOI:10.1186/s13046-019-1318-5 |
| [11] |
AVCI ME, KESKUS AG, TARGEN S, et al. Development of a novel zebrafish xenograft model in ache mutants using liver cancer cell lines[J]. Sci Rep, 2018, 8: 1570. DOI:10.1038/s41598-018-19817-w |
| [12] |
WANG K, SHANG F, CHEN D, et al. Protein liposomes-mediated targeted acetylcholinesterase gene delivery for effective liver cancer therapy[J]. J Nanobiotechnology, 2021, 19(1): 31. DOI:10.1186/s12951-021-00777-9 |
 2024, Vol. 53
2024, Vol. 53




