文章信息
- 任敬秦, 周常慧, 刘成清, 娜孜拉·赛提尼亚孜, 杨楠, 李榕
- REN Jingqin, ZHOU Changhui, LIU Chengqing, SAITINIYAZI Nazila, YANG Nan, LI Rong
- 基于生物信息学构建宫颈癌预后相关ceRNA网络
- Bioinformatics-based construction of a cervical cancer prognosis-associated competing endogenous RNA network
- 中国医科大学学报, 2024, 53(5): 385-391
- Journal of China Medical University, 2024, 53(5): 385-391
-
文章历史
- 收稿日期:2023-06-27
- 网络出版时间:2024-05-14 14:19:21
2. 新疆特殊环境与健康研究重点实验室,乌鲁木齐 830000;
3. 中亚高发病成因与防治国家重点实验室,乌鲁木齐 830000
2. Key Laboratory of Special Environment and Health Research in Xinjiang, College of Public Health, Xinjiang Medical University, Urumqi 830000, China;
3. State Key Laboratory of Pathogenesis, Prevention and Treatment of High Incidence Diseases in Central Asia, Urumqi 830000, China
宫颈癌是常见的妇科肿瘤之一,也是女性癌症死亡的主要原因,严重影响女性健康。寻找潜在的标志物,有助于提高患者预后。竞争性内源RNA(competitive endogenous RNA,ceRNA)网络是指非编码RNA(non-coding RNA,ncRNA)通过竞争性结合微RNA(microRNA,miRNA)进而影响mRNA编码蛋白能力的调节机制[1],部分环状RNA(circle RNA,circRNA)、长链非编码RNA(long non-coding RNA,lncRNA)、mRNA可竞争性结合miRNA,形成ceRNA调控网络。ceRNA网络可调控多种生物学过程,其在多种疾病特别是肿瘤中的作用已引起广泛关注[2]。宫颈癌中circRNA-miRNA-mRNA和lncRNA-miRNA-mRNA之间的ceRNA网络研究较多,但目前尚未有circRNA-lncRNA-miRNA-mRNA的研究。本研究利用癌症基因组图谱(The Cancer Genome Atlas,TCGA)和基因表达综合(Gene Expression Omnibus,GEO)数据库构建ceRNA网络、预后评估模型和Nomogram预测模型。
1 材料与方法 1.1 数据获取在TCGA数据库下载RNA-seq数据,共包含309例,其中3例正常样本和306例宫颈癌样本。GEO数据库下载GSE102686、GSE44001、GSE63514、GSE9750数据,其中GSE102686包含5对宫颈癌及癌旁样本,GSE44001包含300例宫颈鳞癌样本,GSE63514包含24例正常样本和28例宫颈癌样本,GSE9750包含24例正常样本和33例宫颈癌样本。
应用R语言中的“edgeR”包筛选TCGA数据库中宫颈癌样本及正常样本中的差异表达(differential expression,DE)lncRNAs、DEmRNAs(|log2FC| > 2,校正P < 0.01)。采用在线工具GEO2R筛选GSE102686中的DEcircRNAs(|log2FC| > 1,校正P < 0.05)。
1.2 靶向关系预测采用miRcode数据库[3]预测DElncRNAs靶向的miRNAs。使用CircBase数据库[4]进行DEcircRNAs的ID转换。使用CircBank数据库[5]预测DEcircRNAs靶向的miRNAs。使用Targetscan数据库[6]预测miRNAs靶向的mRNAs。使用Cystoscape软件[7]进行数据可视化。
1.3 预后模型构建采用单因素Cox分析、LASSO分析及多因素Cox分析进而确定核心mRNAs。继而使用“Survminer”包计算最佳临界值,将样本分为高危组和低危组。通过使用“survival”包进行K-M生存分析,使用“survival ROC”包构建受试者操作特征(receiver operating characteristic,ROC)曲线。同时使用GSE44001数据进行外部验证。
1.4 Nomogram构建采用“rms”包对TCGA数据库进行Nomogram预后模型构建。患者的1、3、5、10年生存率及复发率可通过模型预测。使用校准曲线以及C指数显示此预后模型的准确性。
1.5 实时PCR收集2021年至2023年新疆医科大学第一附属医院病理科宫颈癌组织和正常宫颈组织各6例。本研究经新疆医科大学第一附属医院伦理委员会批准(S220120-20),患者均知情同意。
采用TRIzol试剂(美国赛默飞世尔科技有限公司)从组织中提取总RNA,并使用PrimeScript® RT试剂盒(中国宝日医生物技术有限公司)进行逆转录。实时PCR在QuantStudio™ 1 Plus实时PCR检测系统(美国赛默飞世尔科技有限公司)上进行,使用SYBR Green Supermix试剂盒(中国宝日医生物技术有限公司)按照说明书进行操作。引物序列如下:GTPBP2,正向5’-TGTTCCATGCCACCACCTTCC-3’,反向5’-CCACTGCCTTCTCGCCTGTC-3’;HELLS,正向5’-TTTCAACAACCAAAGCACTTCACTG-3’,反向5’-TTCCCAAAGCATCCTAAGCCATTC-3’;ROR1,正向5’-ACCGCACCGTCTATATGGAGTC-3’,反向5’-AAGGAATGGCGAACTGAGAACAC-3’;HLF,正向5’-AAGAGAACCAGATCGCCATCCG-3’,反向5’-CCTTCCTCAAGTCAGCCACCTC-3’;SBDS,正向5’-CAACCAGATCCGCCTAACCAATG-3’,反向5’-CGCCAGCCGACGACCTTG-3’。
2 结果 2.1 筛选DElncRNAs、DEmRNAs和DEcircRNAsTCGA数据库共筛选出634个DElncRNAs,其中,上调基因491个,下调基因143个(图 1A);2 618个DEmRNAs,其中,上调基因1 585个,下调基因1 033个(图 1B)。GSE102686数据集中筛选出175个DEcircRNAs,其中,上调基因64个,下调基因111个(图 1C)。
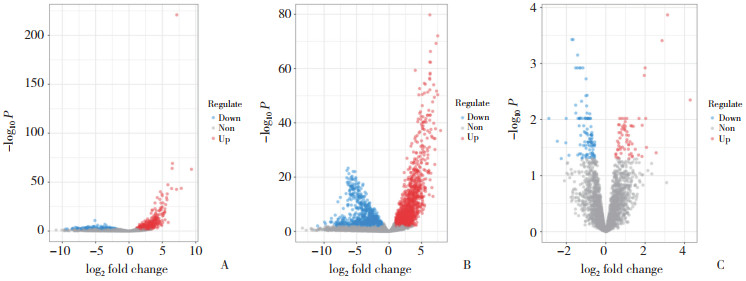
|
| A,volcano maps of DElncRNAs from TCGA;B,volcano maps of DEmRNAs from TCGA;C,volcano maps of DEcircRNAs from GSE102686. 图 1 筛选差异基因 Fig.1 Identification of differentially expressed genes |
2.2 预测DElncRNAs和DEcircRNAs靶向的miRNAs
miRcode数据库预测发现30个DElncRNA和206个miRNA之间存在靶向关系;CircBank数据库鉴定出173个DEcircRNA和2 365个miRNA之间存在靶向关系。将DElncRNAs和DEcircRNAs靶向的miRNAs进行交集分析得到49个miRNA,见图 2。
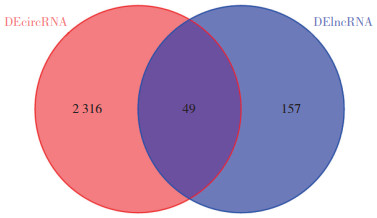
|
| 图 2 预测靶向miRNAs Fig.2 Prediction of miRNAs |
2.3 预测miRNA靶向的mRNA并与DEmRNAs进行交集分析
Targetscan数据库预测出836个mRNA。将miRNA靶向的mRNA和DEmRNAs进行交集分析得到138个mRNA,见图 3。
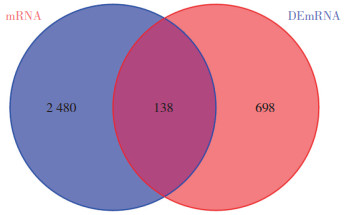
|
| 图 3 预测靶向mRNAs Fig.3 Prediction of mRNAs |
2.4 构建预后模型
通过单因素Cox回归分析筛选出15个mRNA;进一步进行LASSO回归鉴定得出8个mRNA(图 4),经过多因素Cox回归选择5个mRNA进行预后模型构建(表 1)。5个mRNA分别为GTPBP2、HELLS、ROR1、HLF和SBDS。
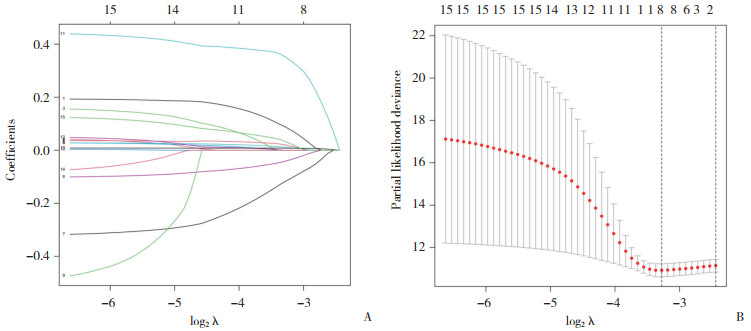
|
| A,trajectories for each independent variable,with the horizontal axis representing the logarithmic value of the independent variable λ and the vertical axis representing the coefficients of the independent variables;B,λ is calculated from the partial likelihood deviation of the tenfold cross-validation. The dashed line represents the best value using the minimum and 1-SE criteria. 图 4 LASSO回归结果分析 Fig.4 Analysis of LASSO regression results |
| Gene | Univariate Cox analysis | Multivariate Cox analysis | |||
| HR | P | HR | P | ||
| BOLA3 | 1.288 486 | 0.010 402 | - | - | |
| CORO2B | 1.179 890 | 0.048 600 | - | - | |
| DLG4 | 1.279 814 | 0.023 266 | - | - | |
| DNAJB4 | 1.042 719 | 0.036 688 | - | - | |
| GTPBP2 | 1.025 780 | 0.044 727 | 1.029 668 | 0.025 7 | |
| HELLS | 0.869 707 | 0.009 259 | 0.891 381 | 0.032 9 | |
| HLF | 0.743 416 | 0.008 762 | 0.721 200 | 0.006 7 | |
| LATS2 | 1.131 212 | 0.008 278 | - | - | |
| LDLRAD2 | 2.596 836 | 0.047 522 | - | - | |
| OAF | 1.024 483 | 0.029 295 | - | - | |
| ROR1 | 1.589 864 | 0.000 610 | 1.744 559 | 0.000 2 | |
| RSPO3 | 1.051 224 | 0.029 249 | - | - | |
| SBDS | 1.014 065 | 2.01E-05 | 1.009 061 | 0.018 9 | |
| SVEP1 | 1.135 977 | 0.030 003 | - | - | |
| ZHX3 | 1.217 257 | 0.024 160 | - | - | |
根据最佳截断值,将样本分为高危组和低危组并进行K-M生存分析(图 5A),结果显示2组患者的生存率有统计学差异(P < 0.05)。为验证模型的准确性,使用ROC曲线进行评估预后模型的准确性,1年、2年、3年总生存期(overall survival,OS)预测风险评分的曲线下面积(area under the curve,AUC)分别为0.71、0.71、0.70(图 5B),说明该预后模型具有较高的灵敏度和特异度。
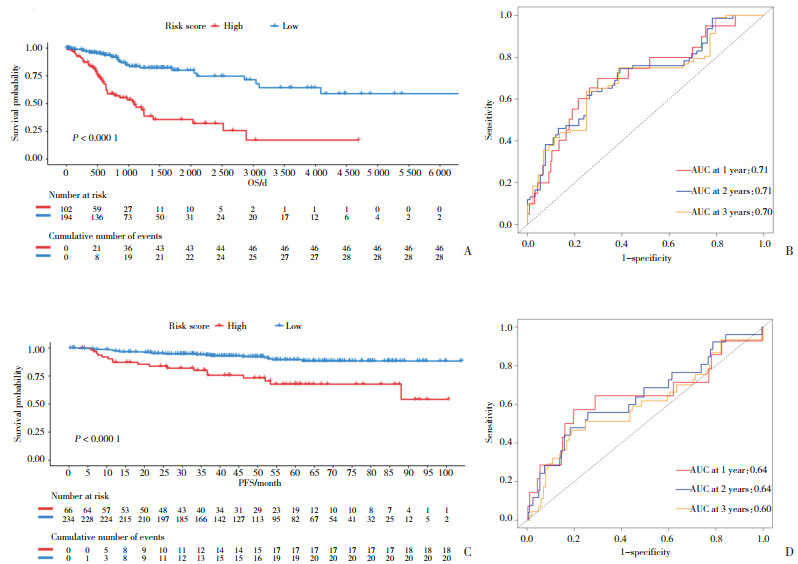
|
| A,K-M survival analysis of TCGA;B,time-dependent ROC curves for the two groups of patients in TCGA;C,K-M survival analysis of GSE44001;D,time-dependent ROC curves for the two groups of patients in GSE44001. 图 5 生存分析结果 Fig.5 Survival analysis results |
与此同时,使用GSE44001数据集进行预后模型的外部验证。根据最佳截断值,将样本分为高危组和低危组并进行K-M生存分析(图 5C),结果显示,2组患者的生存率有统计学差异(P < 0.05)。1年、2年、3年OS预测风险评分的AUC分别为0.64、0.64、0.60(图 5D)。
2.5 Nomogram模型构建基于风险评分、figo分期、T分期、是否原发等独立预测因素,构建OS预测的个体化预测模型(图 6A)。采用个体化预测模型可估计宫颈癌患者(1、2、3、5和10年)的生存率。校准曲线中的Nomogram和实际观测结果显示出良好的重叠,表明其具有最佳的预测精度(图 6B)。该Nomogram模型的C指数为0.707,高于其他预测模型(图 6C)。
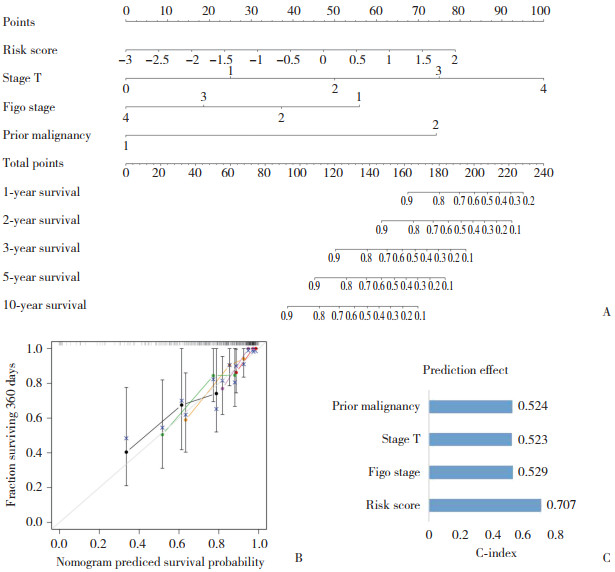
|
| A,Nomogram model was constructed using clinical data from TCGA;B,calibration to verify the fit of the nomogram model fit throughout time periods 1,2,3,5,and 10;C,the C-index was used to evaluate the predictive effectiveness of the model. 图 6 构建及验证Nomogram模型 Fig.6 Nomogram model construction and validation |
2.6 构建circRNA-lncRNA-miRNA-mRNA的ceRNA网络
根据ceRNA网络中的上述关系,共构建了包含5个mRNA、12个miRNA、27个lncRNA和39个circRNA的预后circRNA-lncRNAmiRNA-mRNA ceRNA网络。
2.7 实时PCR验证核心mRNA的表达采用实时荧光定量PCR技术检测宫颈癌组织及正常宫颈组织中核心mRNA的表达差异(图 7),结果发现GTPBP2和HELLS在宫颈癌组织中的表达显著高于正常宫颈组织,ROR1、HLF和SBDS在宫颈癌组织的表达显著低于正常宫颈组织(P < 0.05)。
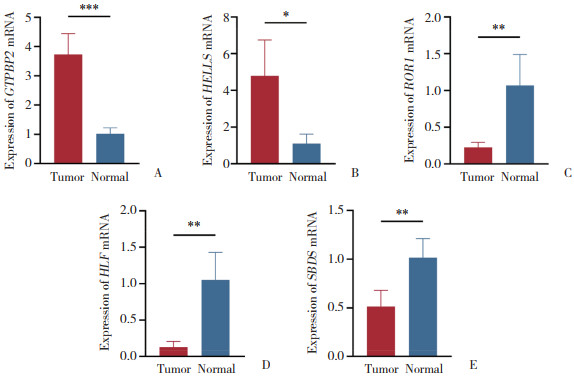
|
| A,GTPBP2 expression in cervical cancer;B,HELLS expression in cervical cancer;C,ROR1 expression in cervical cancer;D,HLF expression in cervical cancer;and E,SBDS expression in cervical cancer. *P < 0.01;**,P < 0.001,***P < 0.000 1. 图 7 实时PCR验证核心mRNA的表达 Fig.7 Verification of hub mRNA expression using real-time PCR |
3 讨论
宫颈癌是全球第四大常见癌症,同时也是女性死亡的第四大原因[8]。宫颈癌的发病机制复杂,就诊时多处于中晚期,常伴有肺转移,预后较差,目前仍缺乏可靠的预后预测指标。因此,了解宫颈癌的异常基因表达调控网络,寻找可能的预后标志物,对实现早期诊断和治疗至关重要。
ncRNAs可调控基因表达并参与炎症反应等生物学过程[9],在肿瘤领域受到越来越多的关注,例如肝细胞癌[10]、非小细胞肺癌[11]、宫颈癌[12]等。ceRNA调控网络理论指出,ncRNAs具有共同的miRNA应答元件(microRNA response element,MRE)能够竞争性结合miRNA,并达到互相调控的效果[13]。然而,以往关于宫颈癌ceRNA调控网络的研究仅探讨了circRNA[14]和lncRNA[15]的作用,尚未同时全面筛选circRNA、lncRNA、miRNA和mRNA构建宫颈癌预后的ceRNA网络。本研究分析了多个数据库,并通过多次筛选和验证,建立了枢纽式预后ceRNA调控网络。
本研究采用TCGA和GEO数据库筛选了差异表达的mRNA、lncRNA和circRNA。随后根据lncRNA-miRNA、circRNA-miRNA和miRNA-mRNA之间的靶向关系,通过单因素Cox、LASSO回归、多因素Cox进一步筛选出5个生存相关的核心mRNA,构建了宫颈癌预后模型。本研究以5种mRNA(GTPBP2、HELLS、ROR1、HLF、SBDS)为核心基础,构建更全面的与circRNA、lncRNA、miRNA、mRNA相关的ceRNA调控网络,可作为宫颈癌预后的生物标志物。
为验证核心mRNA在宫颈癌中的表达差异,本研究利用实时PCR检测临床组织样本中mRNA的表达,结果显示,5种核心mRNA在宫颈癌组织和正常宫颈组织中存在表达差异,此结果与前期数据库结果一致。后续将进行核心mRNA在宫颈癌中的功能验证及基础实验探讨核心mRNA在宫颈癌中的具体作用及机制。
宫颈癌患者早期复发的监测对临床实践具有重要意义。如果能够构建个体化的早期复发预测模型,并具有较高的准确性,就有可能做出更好的临床决策,从而改善患者的预后。为促进预后模型的临床应用,本研究构建了Nomogram预测模型。由于Nomogram模型中其他预测因素为常规检查项目,因此该预测模型具有一定的适用性。
综上所述,本研究首次构建了宫颈癌的circRNA-lncRNA-miRNA-mRNA的ceRNA网络及个体化预测模型,可为临床患者预后及个体化诊疗护理提供新的理论依据。
| [1] |
QI X, LIN YX, CHEN JJ, et al. Decoding competing endogenous RNA networks for cancer biomarker discovery[J]. Brief Bioinform, 2020, 21(2): 441-457. DOI:10.1093/bib/bbz006 |
| [2] |
KARRETH FA, PANDOLFI PP. ceRNA cross-talk in cancer: when ce-bling rivalries go awry[J]. Cancer Discov, 2013, 3(10): 1113-1121. DOI:10.1158/2159-8290.CD-13-0202 |
| [3] |
JEGGARI A, MARKS DS, LARSSON E. miRcode: a map of putative microRNA target sites in the long non-coding transcriptome[J]. Bioinformatics, 2012, 28(15): 2062-2063. DOI:10.1093/bioinformatics/bts344 |
| [4] |
GLAŽAR P, PAPAVASILEIOU P, RAJEWSKY N. circBase: a database for circular RNAs[J]. RNA, 2014, 20(11): 1666-1670. DOI:10.1261/rna.043687.113 |
| [5] |
LIU M, WANG Q, SHEN J, et al. Circbank: a comprehensive database for circRNA with standard nomenclature[J]. RNA Biol, 2019, 16(7): 899-905. DOI:10.1080/15476286.2019.1600395 |
| [6] |
MCGEARY SE, LIN KS, SHI CY, et al. The biochemical basis of microRNA targeting efficacy[J]. Science, 2019, 366(6472): eaav1741. DOI:10.1126/science.aav1741 |
| [7] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [8] |
SUNG H, FERLAY J, SIEGEL RL, et al. Global cancer statistics 2020:globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [9] |
LI LJ, LENG RX, FAN YG, et al. Translation of noncoding RNAs: focus on lncRNAs, pri-miRNAs, and circRNAs[J]. Exp Cell Res, 2017, 361(1): 1-8. DOI:10.1016/j.yexcr.2017.10.010 |
| [10] |
CAO XL, ZHANG GP, LI T, et al. LINC00657 knockdown suppresses hepatocellular carcinoma progression by sponging miR-424 to regulate PD-L1 expression[J]. Genes Genomics, 2020, 42(11): 1361-1368. DOI:10.1007/s13258-020-01001-y |
| [11] |
ZHANG R, NIU ZQ, PEI HH, et al. Long noncoding RNA LINC00657 induced by SP1 contributes to the non-small cell lung cancer progression through targeting miR-26b-5p/COMMD8 axis[J]. J Cell Physiol, 2020, 235(4): 3340-3349. DOI:10.1002/jcp.29222 |
| [12] |
ZHANG D, ZHANG YY, SUN XY. LINC01133 promotes the progression of cervical cancer via regulating miR-30a-5p/FOXD1[J]. Asia Pac J Clin Oncol, 2021, 17(3): 253-263. DOI:10.1111/ajco.13451 |
| [13] |
SALMENA L, POLISENO L, TAY Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?[J]. Cell, 2011, 146(3): 353-358. DOI:10.1016/j.cell.2011.07.014 |
| [14] |
XU TY, SONG XF, WANG YL, et al. Genome-wide analysis of the expression of circular RNA full-length transcripts and construction of the circRNA-miRNA-mRNA network in cervical cancer[J]. Front Cell Dev Biol, 2020, 8: 603516. DOI:10.3389/fcell.2020.603516 |
| [15] |
WANG J, ZHANG C. Identification and validation of potential mRNA- microRNA- long-noncoding RNA (mRNA-miRNA-lncRNA) prognostic signature for cervical cancer[J]. Bioengineered, 2021, 12(1): 898-913. DOI:10.1080/21655979.2021.1890377 |
 2024, Vol. 53
2024, Vol. 53




