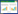文章信息
- 曾媛媛, 常莉, 池晴佳, 封顺, 田菲菲
- ZENG Yuanyuan, CHANG Li, CHI Qingjia, FENG Shun, TIAN Feifei
- 结合铁死亡建立遗传算法优化的反向传播神经网络脓毒症预后模型
- Combined machine learning with ferroptosis to establish the prognosis model for sepsis based on backward propagation neural network optimized using genetic algorithm
- 中国医科大学学报, 2024, 53(4): 295-301
- Journal of China Medical University, 2024, 53(4): 295-301
-
文章历史
- 收稿日期:2023-03-27
- 网络出版时间:2024-04-10 18:54:02
2. 武汉理工大学理学院工程结构与力学系,武汉 430070
2. Department of Engineering Structure and Mechanics, School of Science, Wuhan University of Technology, Wuhan 430070, China
脓毒症被定义为微生物感染引起的全身炎症反应并伴有多器官功能障碍[1],其预后问题是脓毒症患者和临床医生面临的重要挑战[2]。铁死亡是一种新的程序性细胞死亡形式[3]。研究[4]发现,铁死亡可被脓毒症诱导,参与脓毒症的发病机制。已有临床研究[5]表明,铁死亡会影响脓毒症的发展进程,铁死亡抑制剂可抵抗脓毒症引起的多器官功能障碍和炎症,提高脓毒症小鼠的存活率。因此,结合铁死亡研究有助于发现脓毒症新的预后生物标志物。本研究结合铁死亡建立了一个基于遗传算法优化的反向传播网络(genetic algorithm optimized backward propagation neural network,GA-BPNN)四基因预后模型,并利用外部数据验证了该模型的预后能力。
1 材料与方法 1.1 数据收集本研究从高通量基因表达数据库(gene expression omnibus,GEO)中下载了3个数据集GSE65682、GSE95233和GSE33119,删除缺失和健康的样本。GSE65682中479例脓毒症样本的表达谱数据和临床随访信息作为训练集,GSE95233中51例和GSE33119中40例脓毒症样本的表达谱数据和临床随访信息作为验证集。3个队列的临床特征总结见表 1。
| Characteristic | GSE65682(n = 479) | GSE95233(n = 51) | GSE33119(n = 40) |
| Age [n(%)] | |||
| < 60 years | 183(38.2) | 20(39.2) | 10(25.0) |
| ≥60 years | 296(61.8) | 31(60.8) | 30(75.0) |
| Gender [n(%)] | |||
| Female | 207(43.2) | 20(39.2) | 16(40.0) |
| Male | 272(56.8) | 31(60.8) | 24(60.0) |
| Timing of mortality(d) | 28 | 28 | 28 |
| Mortality [n(%)] | 114(23.7) | 17(33.3) | 20(50.0) |
1.2 铁死亡基因集富集分析(gene set enrichment analysis,GSEA)以及基因功能富集分析
通过GSEA数据库下载铁死亡相关基因集。用“gsea”R包、“clusterProfiler”R包和“GSVA”R包进行铁死亡通路分析、铁死亡基因富集得分分析以及基因功能富集分析[6-7]。
1.3 加权基因共表达网络分析(weighted gene coexpression network analysis,WGCNA)为了评估加权基因共表达网络及其模块成员[8],通过WGCNA构建共表达网络。用R.4.1.2中的WGCNA包,在WGCNA算法中选择软阈值构建网络。通过动态树切算法将共表达基因分配到模块中。
1.4 蛋白质-蛋白质相互作用(protein-protein interaction,PPI)分析利用R.4.1.2中的“STRINGdb”包分析模块基因,基于中介中心性筛选关键基因,高于中介中心性中位值的基因被认为是枢纽基因,利用Cytoscape软件构建PPI网络并进行显示[9]。
1.5 短时间序列表达式挖掘(short time series expression miner,STEM)分析STEM是实现用于聚类和比较短时间序列基因表达数据的独特算法,结合其可视化能力能较直观地表现出在疾病发展过程中上调和下调的基因。使用基于皮尔逊相关性的距离度量,将每个基因分配到最近的剖面,以确定给定转录组谱的显著性水平,显著聚类的标准为P < 0.05[10]。
1.6 遗传算法(genetic algorithm,GA)-反向传播神经网络(backwark propagation neural network,BPNN)BPNN是目前最流行的神经网络方法之一,但BPNN容易出现局部问题最优解。相对于其他优化算法而言,GA模仿自然选择的过程解决优化问题,可有效避免算法陷入局部最优,从而弥补BPNN的不足,提高模型的分类效果[11]。本研究将GA优化后的权值与阈值导入BPNN中,建立了一种脓毒症患者预后模型。
1.7 预后关键基因与免疫细胞的相关性CIBERSORT是一种基于线性支持向量回归原理的反卷积算法[12],采用P < 0.05对结果进行过滤。利用CIBERSORT作为反卷积算法,本研究分别检测了脓毒症发展相关的4个关键基因的表达与浸润免疫细胞水平的相关性。
2 结果 2.1 脓毒症中铁死亡相关模块基因筛选本研究中,为了确定脓毒症中铁死亡相关模块基因,首先在GSE65682脓毒症患者的存活样本和未存活样本之间进行GSEA分析,以P < 0.05为显著性判断标准。结果发现,不论是在训练集还是验证集GSE95233中,脓毒症患者的生存状态与铁死亡通路之间存在显著的相关性(图 1A、1B)。利用GSVA对每个样本进行铁死亡相关基因集富集评分,结果发现,在数据集GSE65682中,脓毒症存活组和未存活组中铁死亡相关基因集富集评分差异显著,且脓毒症未存活组铁死亡相关基因的评分明显上调(图 1C)。之后将每个样本的铁死亡富集评分作为临床数据,在GSE65682中,对这11 760个基因进行WGCNA分析,共识别到8个模块。其中,红色模块与脓毒症中铁死亡通路的相关系数(-0.63)绝对值最大(图 1D),模块中共包含472个基因。因此,将红色模块中的基因作为脓毒症中铁死亡相关模块基因进行进一步分析。
|
| A, activity difference of ferroptosis related gene set in training set GSE65682;B, activity difference of ferroptosis related gene set in validation set GSE95233;C, ferroptosis enrichment score of GSVA in GSE65682 sepsis survival group and nonsurviving group; D, the relevance of modules to clinical features. Red shows a positive correlation and blue shows a negative correlation. The rows represent module genes and the columns are ferroptosis enrichment scores. In the module, the first row is the correlation coefficient, the second row is the P value. 图 1 脓毒症中铁死亡相关模块基因筛选 Fig.1 Screening of ferroptosis related module genes in sepsis |
2.2 脓毒症中铁死亡相关基因的共表达网络
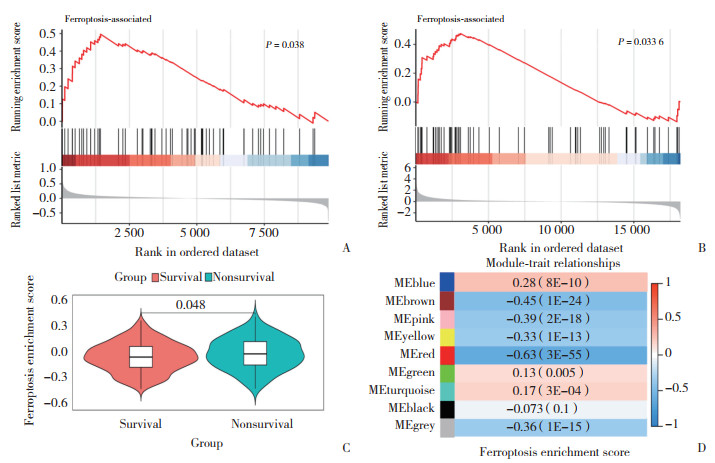
对红色模块中的472个基因进行PPI分析,共筛选出97个枢纽基因。将这97个枢纽基因导入Cytoscape软件中,获得PPI关系图(图 2A)。PPI网络图表明,97个铁死亡相关枢纽基因在脓毒症中存在复杂的相互作用。为进一步了解这些基因的功能,对这97个枢纽基因进行基因本体论(gene ontology,GO)和京都基因和基因组数据库(Kyoto encyclopedia of genes and genomes,KEGG)分析,就生物过程(biological processes,BP)而言,这些基因主要富集在对中性粒细胞介导的免疫,NF-κB激酶和NF-κB信号分子活化的调节等。同时,KEGG分析显示这些基因富集在人乳头瘤病毒感染、Notch信号通路、ErbB信号通路、HIF-1信号通路等通路中(P < 0.05,图 2B、2C)。其中,NF-κB的过度激活在抑制炎症反应中起重要作用,激活NF-κB途径会促进肠脓毒症的发展[13]。这些基因主要集中在细胞信号通路和免疫中,为脓毒症治疗提供了新的潜在治疗靶点。
|
| A, STRING database was used to construct PPI network of hub genes, the darker the color, the higher the connectivity; B, GO enrichment analysis of hub genes; C, KEGG enrichment analysis of hub genes. BP, biological processes; CC, cell components; MF, molecular function. 图 2 脓毒症中铁死亡相关基因的共表达网络 Fig.2 Co-expression network of ferroptosis related genes in sepsis |
2.3 脓毒症生存预后关键基因
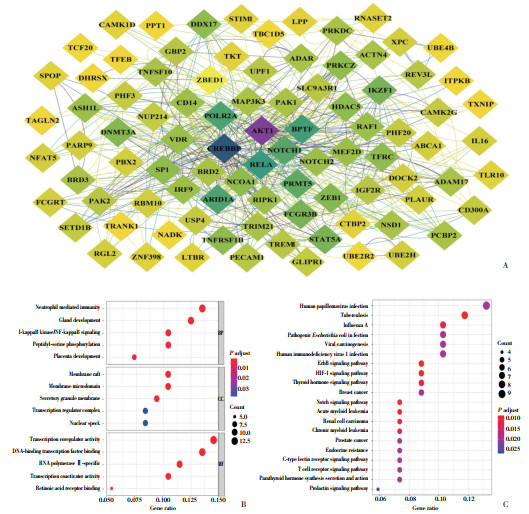
为了确定脓毒症到死亡的发展过程中基因表达的变化,按健康、脓毒症存活、脓毒症未存活分组,利用STEM软件分析GSE65682中的基因。最终确定了3个在脓毒症存活和未存活个体中差异显著的集群,共191个基因(P < 0.05)。将STEM中得到的脓毒症发展进程中表达差异显著的基因与2.2中脓毒症铁死亡相关基因取交集,获得4个关键基因(TFRC,CPPED1,PECAM1,TRANK1)。为了进一步探究这4个关键基因与生存之间的关系,比较分析了脓毒症存活组和未存活组之间这4个基因的表达情况,发现这4个基因在脓毒症存活组和未存活组之间均差异表达(P < 0.05)(图 3A)。根据这4个基因的表达情况,使用Kaplan-Meier(K-M)生存分析方法对脓毒症患者28 d生存情况进行生存分析,结果显示,CPPED1、PECAM1和TRANK1高表达组患者的存活概率明显低于低表达组,TFRC高表达组的患者存活概率明显高于低表达组(P < 0.05,图 3B~3E)。关键基因的表达分析和K-M生存分析都进一步证明这4个基因均与脓毒症患者的生存状态显著相关。脓毒症的病理生理机制复杂,目前公认的发病机制之一是宿主免疫反应失衡[14],关键基因与脓毒症患者浸润免疫细胞的相关分析结果显示,不论是训练集GSE65682还是验证集GSE95233中,4个关键基因都与多种免疫细胞显著相关(P < 0.05,图 3F、3G)。因此,推测这4个关键基因可能参与了脓毒症免疫微环境的稳态,从而影响脓毒症的发展进程。
|
| A, analysis of the expression of four key genes; B to E, survival analysis and statistics were based on the expression groups of CPPED1, TFRC, PECAM1, and TRANK1, respectively. The overall survival rate of patients with high gene expression and patients with low gene expression was statistically analyzed by log-rank test (P < 0.05);F, G, correlation analysis between the key genes and immune cells in the training set GSE65682 (F) and validation set GSE95233 (G). 图 3 关键基因的表达分析、Kaplan-Meier生存分析和免疫浸润分析 Fig.3 Expression analysis of key genes, Kaplan-Meier survival analysis, and immune infiltration analysis |
2.4 四基因预后模型建立与验证
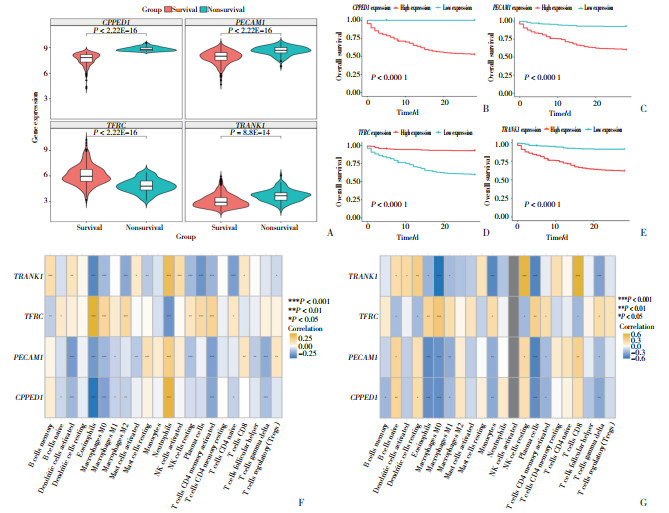
根据4个关键基因建立脓毒症预后GA-BPNN模型,结果显示,在训练集中,回归图显示了BPNN训练的预测模型的输出与目标之间的关系。回归系数R均接近于1,说明预后模型训练良好,预测几乎等于目标(图 4A)。训练集GSE65682中曲线下面积(area under the curve,AUC)为0.98(图 4B)。为进一步证明GA-BPNN模型的分类效果和泛化能力的优越性,使用相同的外部数据作为验证集对GA-BPNN与支持向量机(support vector machine,SVM)、BPNN和随机森林(random forest,RF)进行比较,应用受试者操作特征(receiver operating characteristic,ROC)曲线如图 4C、4D所示。与传统模型相比,本研究提出的GA-BPNN模型具有更好的拟合性和更高的预测精度。不管是在训练集还是验证集中,该模型的AUC值都在0.9以上,提示该分类器对于脓毒症患者28 d生存情况有良好的预测效果及泛化能力。
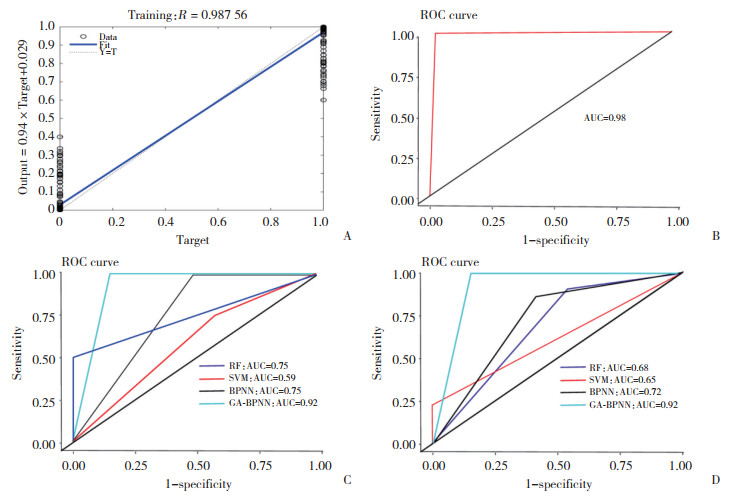
|
| A, training set GA-BPNN prediction results; B, ROC curve of training set GA-BPNN; C, comparison of the ROC curves between the GA-BPNN model and the traditional model in set GSE33119;D, comparison of the ROC curves between the GA-BPNN model and the traditional model in set GSE95233. 图 4 四基因模型的回归分析和ROC曲线图 Fig.4 Regression analysis and ROC curve of the 4-gene model |
3 讨论
目前,关于脓毒症的预后研究很多,包括临床严重程度评分、序贯器官衰竭评分以及分子生物标志物等,但在信号通路层面的研究不足[15-16]。鉴于铁死亡对脓毒症发生和发展的潜在影响,识别新的铁死亡相关的生物标志物可能提供重要的预后信息和治疗靶点。本研究基于存活和死亡脓毒症患者的铁死亡通路表达差异,筛选出与脓毒症预后相关的关键基因TFRC、CPPED1、PECAM1、TRANK1,并进一步探讨了这4个关键基因与免疫细胞浸润的相关性。本研究还利用GA-BPNN开发了一种新的四基因模型,用于预测脓毒症患者28 d生存情况,结果表明,该GA-BPNN模型具有良好的分类效果和泛化能力。
脓毒症的发病过程不仅与铁死亡有关,还涉及免疫和炎症反应[17]。本研究在铁死亡相关基因富集分析中发现,相关基因主要富集在中性粒细胞介导的免疫和NF-κB信号通路等生物学功能。中性粒细胞胞外诱捕网及其诱导的铁死亡可促进脓毒症的病理进程[18]。NF-κB转录因子是炎症和免疫稳态的主要调节因子,参与炎症反应和脓毒症病理生理的基因表达调控[19]。研究[20]发现,调节NF-κB可抑制炎症反应,抑制脓毒症。本研究对关键基因的免疫浸润分析结果发现,PECAM1与中性粒细胞呈显著相关。PECAM1可在细胞黏附分子的作用下调节人中性粒细胞的迁移过程[21],PECAM1还参与NF-κB信号通路的激活[22]。K-M生存分析发现,PECAM1低表达组生存状态显著优于高表达组。因此,推测PECAM1可能通过影响脓毒症中免疫和炎症反应成为脓毒症的治疗靶点。
综上所述,本研究揭示了铁死亡通路在脓毒症发展进程中的作用,并基于GA-BPNN提出了四基因模型用于预测脓毒症患者的预后,结果可靠。本研究对脓毒症铁死亡提出了新的见解,并为脓毒症治疗提供了新的作用靶点。
| [1] |
CHEN Q, LIANG X, WU TZ, et al. Integrative analysis of metabolomics and proteomics reveals amino acid metabolism disorder in sepsis[J]. J Transl Med, 2022, 20(1): 123. DOI:10.1186/s12967-022-03320-y |
| [2] |
LIU AC, PATEL K, VUNIKILI RD, et al. Sepsis in the era of data-driven medicine: personalizing risks, diagnoses, treatments and prognoses[J]. Brief Bioinform, 2020, 21(4): 1182-1195. DOI:10.1093/bib/bbz059 |
| [3] |
JIANG XJ, STOCKWELL BR, CONRAD M. Ferroptosis: mechanisms, biology and role in disease[J]. Nat Rev Mol Cell Biol, 2021, 22(4): 266-282. DOI:10.1038/s41580-020-00324-8 |
| [4] |
LIU YT, TAN SC, WU YB, et al. The emerging role of ferroptosis in sepsis[J]. DNA Cell Biol, 2022, 41(4): 368-380. DOI:10.1089/dna.2021.1072 |
| [5] |
CAO ZZ, QIN HQ, HUANG YH, et al. Crosstalk of pyroptosis, ferroptosis, and mitochondrial aldehyde dehydrogenase 2-related mechanisms in sepsis-induced lung injury in a mouse model[J]. Bioengineered, 2022, 13(3): 4810-4820. DOI:10.1080/21655979.2022.2033381 |
| [6] |
WU TZ, HU EQ, XU SB, et al. clusterProfiler 4.0: a universal enrichment tool for interpreting omics data[J]. Innovation (Camb), 2021, 2(3): 100141. DOI:10.1016/j.xinn.2021.100141 |
| [7] |
HÄNZELMANN S, CASTELO R, GUINNEY J. GSVA: gene set variation analysis for microarray and RNA-seq data[J]. BMC Bioinform, 2013, 14: 7. DOI:10.1186/1471-2105-14-7 |
| [8] |
LANGFELDER P, HORVATH S. WGCNA: an R package for weigh-ted correlation network analysis[J]. BMC Bioinform, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 |
| [9] |
SZKLARCZYK D, GABLE AL, NASTOU KC, et al. The STRING database in 2021: customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets[J]. Nucleic Acids Res, 2021, 49(D1): D605-D612. DOI:10.1093/nar/gkaa1074 |
| [10] |
ERNST J, BAR-JOSEPH Z. STEM: a tool for the analysis of short time series gene expression data[J]. BMC Bioinform, 2006, 7: 191. DOI:10.1186/1471-2105-7-191 |
| [11] |
JOHNSON KL, CARNEGIE NB. Calibration of an adaptive genetic algorithm for modeling opinion diffusion[J]. Algorithms, 2022, 15(2): 45. DOI:10.3390/a15020045 |
| [12] |
NEWMAN AM, LIU CL, GREEN MR, et al. Robust enumeration of cell subsets from tissue expression profiles[J]. Nat Methods, 2015, 12(5): 453-457. DOI:10.1038/nmeth.3337 |
| [13] |
CAO YY, WANG Z, WANG ZH, et al. Inhibition of miR-155 alleviates sepsis-induced inflammation and intestinal barrier dysfunction by inactivating NF-κB signaling[J]. Int Immunopharmacol, 2021, 90: 107218. DOI:10.1016/j.intimp.2020.107218 |
| [14] |
VAN DER POLL T, SHANKAR-HARI M, WIERSINGA WJ. The immunology of sepsis[J]. Immunity, 2021, 54(11): 2450-2464. DOI:10.1016/j.immuni.2021.10.012 |
| [15] |
LEE HJ, KO BS, RYOO SM, et al. Modified cardiovascular SOFA score in sepsis: development and internal and external validation[J]. BMC Med, 2022, 20(1): 1-15. DOI:10.1186/s12916-022-02461-7 |
| [16] |
QU ZY, ZHU YB, WANG MP, et al. Prognosis and risk factors of sepsis patients in Chinese ICUs: a retrospective analysis of a cohort database[J]. Shock, 2021, 56(6): 921-926. DOI:10.1097/SHK.0000000000001784 |
| [17] |
NEDEVA C. Inflammation and cell death of the innate and adaptive immune system during sepsis[J]. Biomolecules, 2021, 11(7): 1011. DOI:10.3390/biom11071011 |
| [18] |
ZHANG H, LIU JL, ZHOU YL, et al. Neutrophil extracellular traps mediate m6A modification and regulates sepsis-associated acute lung injury by activating ferroptosis in alveolar epithelial cells[J]. Int J Biol Sci, 2022, 18(8): 3337-3357. DOI:10.7150/ijbs.69141 |
| [19] |
YU H, LIN LB, ZHANG ZQ, et al. Targeting NF-κB pathway for the therapy of diseases: mechanism and clinical study[J]. Signal Transduct Target Ther, 2020, 5(1): 209. DOI:10.1038/s41392-020-00312-6 |
| [20] |
LUO J, WANG J, ZHANG J, et al. Nrf2 deficiency exacerbated CLP-induced pulmonary injury and inflammation through autophagy- and NF-κB/PPARγ-mediated macrophage polarization[J]. Cells, 2022, 11(23): 3927. DOI:10.3390/cells11233927 |
| [21] |
WINNEBERGER J, SCHÖLS S, LESSMANN K, et al. Platelet endothelial cell adhesion molecule-1 is a gatekeeper of neutrophil transendothelial migration in ischemic stroke[J]. Brain Behav Immun, 2021, 93: 277-287. DOI:10.1016/j.bbi.2020.12.026 |
| [22] |
LIU YH, ZHANG ZY, LI WJ, et al. PECAM1 combines with CXCR4 to trigger inflammatory cell infiltration and pulpitis progression through activating the NF-κB signaling pathway[J]. Front Cell Dev Biol, 2020, 8: 593653. DOI:10.3389/fcell.2020.593653 |
 2024, Vol. 53
2024, Vol. 53