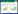文章信息
- 邵冬雪, 张晨阳, 叶苗苗, 陈帆, 郝丽英
- SHAO Dongxue, ZHANG Chenyang, YE Miaomiao, CHEN Fan, HAO Liying
- 长QT综合征相关钙调蛋白突变体E141G的C末端片段载体构建和蛋白制备
- Vector construction and protein preparation of long QT syndrome-related C-terminal lobe of calmodulin mutant E141G
- 中国医科大学学报, 2024, 53(11): 967-971
- Journal of China Medical University, 2024, 53(11): 967-971
-
文章历史
- 收稿日期:2024-01-19
- 网络出版时间:2024-11-21 11:14:35
电压门控钙通道(voltage gated calcium channel,VGCC)是细胞内Ca2+内流的最主要途径,介导细胞内多种Ca2+依赖性调控过程的发生,如兴奋产生、肌肉收缩、激素和神经递质的分泌及基因表达等[1-3]。VGCC受2种钙调蛋白(calmodulin,CaM)介导的Ca2+依赖性的反馈机制调节,即Ca2+依赖性易化(Ca2+-dependent facilitation,CDF)和Ca2+依赖性失活(Ca2+-dependent inactivation,CDI)过程,从而维持细胞内钙稳态[4-10]。对于心肌CaV1.2钙通道,CaM的结合位点包括位于通道C末端的IQ和preIQ基序,Ⅰ-Ⅱ环以及通道N末端[9-11]。近期研究[12-16]表明一系列CaM关键结构域内位点突变引发可伴随心源性猝死的长QT综合征(long QT syndrome,LQTS),有研究[12-17]表明这可能与CaM突变后改变与CaV钙通道作用引发CDI异常有关。
CaM是主要的Ca2+传感器之一,为哑铃形构象,有2个球形末端[N末端片段(N-terminal lobe,N-lobe;a.a. 3-81)和C末端片段(C-terminal lobe,C-lobe;a.a.77-149)],两端结构域有48%相同序列和75%同源序列[18]。N-lobe和C-lobe虽然结构相似,但两端的不同序列也决定了它们对Ca2+的亲和力不同,与CaV钙通道上不同结合位点的亲和力也不同,两者对CaV钙通道起到特异性调节作用[19-21]。有趣的是,全外显子组测序相继发现的与LQTS相关的多种CaM致病突变体均位于CaM的C-lobe上,这可能与N-lobe和C-lobe对CaVs钙通道调节的特异性有关,但具体机制尚不明确。
本研究首次构建LQTS相关CaM突变体E141G的C-lobe(C-lobe of CaM mutant E141G,C-lobeE141G),以排除CaM的N-lobe对通道的调节,可清晰观察突变对CaM的C-lobe作用的影响,旨在阐明突变对CaM的C-lobe特异性调节CaV钙通道的影响,进而为CaM的C-lobe突变引发的LQTS的发生机制的研究提供材料基础。
1 材料与方法 1.1 实验材料和动物pGEX-6p-3/C-lobe质粒由日本鹿儿岛大学KAMEYAMA教授惠赠,pGEX-6p-3/C-lobeE141G由江苏溥博生物科技有限公司以C-lobe为模版采用点突变试剂盒制备。6~8周龄、体重220~320 g的SD大鼠由中国医科大学实验动物部提供。动物饲养和实验原则遵照《中国医科大学实验动物福利与伦理审查委员会规定》进行。
1.2 方法 1.2.1 质粒的转化及鉴定取大肠杆菌BL21感受态细胞100 μL,加入pGEX-6p-3/C-lobeE141G质粒10 ng,冰中放置30 min后于42 ℃水浴中精确热击60 s,然后快速转移至冰中冷却2 min,加入预热好的37 ℃ SOC培养基1 mL,37 ℃摇震荡培养1 h,4 000 r/min离心2 min后吸除部分上清培养液,剩余约200 μL培养液重悬菌体,均匀涂布在含氨苄青霉素(0.1 mg/mL)的琼脂平板培养基37 ℃过夜筛选。挑取单克隆菌种接种于装有LB培养液(含氨苄青霉素0.05 mg/mL)的试管中震摇培养12~16 h,采用碱裂解法提取质粒。质粒经BamHⅠ和NotⅠ双酶切后进行琼脂糖凝胶电泳,并进行DNA测序,鉴定质粒。
1.2.2 融合蛋白的表达及纯化取适量pGEX-6p-3/C-lobeE141G重组质粒转化BL21菌种,加入装有400 mL LB培养液(含AMP 50 μg/mL)的锥形瓶中,在37 ℃水浴锅中以120 r/min速度震摇培养12~16 h。当波长595时的吸光度(A595)值达到0.6~1.0时,加入异丙基硫代-β-D半乳糖苷(isopropyl thiobeta-D galactoside,IPTG)使其终浓度为1 mmol/L,37 ℃、120 r/min条件下诱导谷胱甘肽-S-转移酶(glutathione-S-transferase,GST)-C-lobeE141G融合蛋白表达4 h。菌液以5 000 r/min、4 ℃离心10 min后弃上清,用20 mL PBS重悬沉淀菌体,加入溶菌酶至终浓度为0.1 mg/mL,冰浴处理30 min。在冰浴中以200 W、超声3 s间隔7 s的条件共超声20 min,打破菌体后4 ℃、16 000 r/min离心10 min。收集上清与两管300 μL GS-4B beads混合后置10 r/min的旋转培养器4 ℃旋转过夜。PBS清洗3次后每管加入495 μL PBS和5 μL蛋白酶切酶,混匀后室温剧烈震摇酶切GST标签5 h。600 r/min,离心3 min,收集上清即获得目的蛋白C-lobeE141G,并于-20 ℃冻存。
preIQ重组质粒经LB培养基培养后,IPTG诱导GST融合蛋白表达,采用GS-4B beads进行分离纯化后得到GST-preIQ融合蛋白[20]。
1.2.3 蛋白的纯度鉴定及浓度测定常规15% SDS-PAGE电泳检测纯化后的C-lobeE141G蛋白纯度及相对分子量。参照BCA蛋白浓度测定试剂盒方法测定纯化后的C-lobeE141G蛋白浓度。
1.2.4 GST pull-down实验取连接于GS-4B beads上的GST-preIQ融合蛋白40 μL于2 mL EP管中,加入C-lobeE141G使其终浓度分别为0.1、0.35、0.7、1.4、2.1、3.5 μmol/L,加入[Ca2+]为2 mmol/L的Tris缓冲液至总体系为300 μL。4 ℃低转速旋转孵育4 h。800 r/min4 ℃离心3 min。弃掉上清后取Tris缓冲液300 μL,800 r/min 4 ℃轻柔清洗2次,每管加入15 μL 1×SDS样品缓冲液洗脱。取上清,采用常规15% SDS-PAGE电泳检测纯化蛋白。
1.2.5 单个大鼠心室肌细胞的制备SD大鼠经苯巴比妥腹腔注射麻醉后,在人工呼吸机支持下进行主动脉插管,分离心脏。然后利用Langendorff灌流装置,采用胶原酶消化大鼠心室肌细胞来获得单个心肌细胞。
1.2.6 单通道钙电流的检测应用膜片钳单通道记录模式监测钙通道活性。首先形成细胞贴附模式,然后将膜片自细胞游离建立膜内面向外模式,再将膜片小心移动入浴槽旁边充满测试溶液的凹槽中。用pClamp10.2软件记录和分析数据,由于I = N×Po×i,测定实验脉冲开始后5~105 ms期间的平均电流(I),并除以单通道电流幅值(i)可获得通道开放频率(NPo),其中N为膜片中的通道数目,Po为通道的平均时间开放概率。
2 结果 2.1 重组质粒的鉴定重组质粒pGEX-6p-3/C-lobeE141G经BamHⅠ和NotⅠ双酶切后的琼脂糖凝胶电泳图见图 1。目的片段C-lobeE141G约为240 bp,与图 1显示明显亮带位置相符,说明C-lobeE141G目的片段扩增成功。DNA测序结果于NCBI数据库中进行Blast比对,结果显示,与Homo sapiens calmodulin 2(CALM2)相比,141位谷氨酸(E)对应的GAG碱基突变为甘氨酸(G)对应的GGG碱基(图 2),其他碱基比对结果一致。证明质粒构建成功。
|
| 图 1 重组质粒的酶切鉴定琼脂糖电泳图 Fig.1 Agarose electrophoresis of recombination plasmids digested by restriction endonuclease |

|
| 图 2 C-lobe和C-lobeE141G氨基酸序列及其各自对应的碱基序列图 Fig.2 Amino acid sequences and the corresponding base sequences of C-lobe和C-lobeE141G |
2.2 C-lobeE141G蛋白纯度和浓度的测定
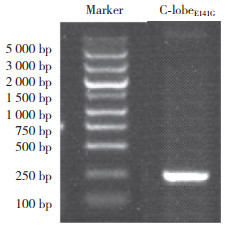
结果如图 3所示,通过15% SDS-PAGE凝胶电泳检测纯化后的C-lobeE141G蛋白,条带位置与预期结果大概一致,且杂带较少即纯度较高。采用BCA试剂盒测定蛋白浓度为0.98 g/L。
|
| 图 3 纯化后的C-lobeE141G蛋白SDS-PAGE图 Fig.3 SDS-PAGE of the purified C-lobeE141G |
以上结果表明重组质粒能够高表达GST-C-lobeE141G融合蛋白,纯化后获得高纯度、高浓度C-lobeE141G蛋白。
2.3 C-lobeE141G蛋白与CaV1.2钙通道preIQ片段的结合GST pull-down结果显示,纯化后的C-lobeE141G在2 mmol/L [Ca2+]条件下可与CaV1.2钙通道的preIQ片段浓度依赖性结合,表明本实验纯化的蛋白具有与CaV1.2钙通道结合的生物活性。见图 4。

|
| 图 4 C-lobeE141G与GST-preIQ结合的SDS-PAGE图 Fig.4 SDS-PAGE of C-lobeE141G binding to GST-preIQ |
2.4 C-lobeE141G蛋白对心肌钙通道的作用
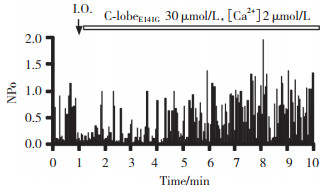
膜片钳单通道膜内向外(inside-out)模式下记录通道电流时,由于人为改变细胞内环境,通道活性会随着记录时间下降,即“run-down”现象。CaM及ATP是恢复钙通道“run-down”的必要因子[22]。结果显示,纯化后C-lobeE141G蛋白可恢复已“run-down”的心肌钙通道活性至169%,表明本研究纯化的蛋白具有维持钙通道开放的电生理活性。见图 5。
|
| 图 5 C-lobeE141G对CaV1.2的电生理作用 Fig.5 Electrophysiological effects of C-lobeE141G on CaV1.2 |
3 讨论
研究[13]表明,编码相同CaM的3个基因(CALM1、CALM2和CALM3)中的任一个基因突变都会导致罕见的危及生命的心律失常综合征,统称为CaM病,包括LQTS、儿茶酚胺诱发的心动过速(catecholaminergic polymorphic ventricular tachycardia,CPVT),特发性心室颤动(idiopathic ventricular fibrillation,IVF)及重叠特征表型等。2016年BOCZEK等[23]首次报道CaM突变体E141G(CaME141G)导致C-lobe结构域的Ca2+亲和力降低,N-lobe的Ca2+亲和力没有显著变化;这不仅扰乱了CaV1.2通道的CDI作用,也可以使NaV 1.5后期电流增强,是第1个显示出会影响成年心脏钠通道亚型的CaM突变体。当共表达野生型CaM与突变体CaME141G时,NaV1.5晚钠电流被修复,然而,3倍过量的野生型CaM却无法修复突变体CaME141G导致的CaV1.2通道CDI异常[23]。SU等[17]进一步研究发现,和CaM相比,CaME141G与CaV1.2通道的结合减少;但是在低钙状态下CaME141G与CaM相比变化并不明显。
2013年以来,通过全外显子组测序相继发现了与LQTS相关的多种CaM致病突变体[12-16]。有趣的是,已知的这些突变位点均位于CaM的C-lobe上,除F142为紧邻EF-hand4钙结合环的第1个氨基酸,其余均是EF-hand钙结合环内与钙共价结合的位点。JENSEN等[24]利用Gnom AD数据库对138 632例基因筛查发现的28个CaM突变位点散在分布于整个CaM上,并且只有1个位点位于C-lobe的钙结合位点上,这些突变体未有LQTS表型。说明并不是CaM的所有突变都会导致LQTS,只有突变发生在C-lobe上,并且影响C-lobe结合钙的能力,才可能导致LQTS,而此现象背后的机制尚未阐明。
本课题组前期研究[20]显示,CaM的N-lobe和C-lobe均可钙依赖性与CaV1.2通道结合而调节通道功能;当以CaM全长为研究对象研究CaM突变体E141G对CaV1.2功能的影响时,N-lobe仍保持一定的生理活性,尤其干扰在低钙条件下观察C-lobe突变对通道结合的影响。采用CaM截短蛋白C-lobeE141G,可排除CaM的N-lobe对通道的调节,直接观察突变对CaM的C-lobe作用的影响。
综上所述,本研究成功构建LQTS相关C-lobeE141G载体,并制备高浓度高纯度C-lobeE141G蛋白,可作为研究LQTS相关的CaM突变位点集中于C-lobe结构域这一现象,以及CaM突变体通过其C环干扰CaV1.2通道CDI作用机制的基础。CaM突变引发的严重LQTS,临床上治疗的传统药物或方法(β受体阻断剂、苯妥英、螺内酯、钾以及单腔起搏器等)效果并不理想,因此寻找新的治疗靶点具有重要的临床意义。本研究为探究CaM突变引发的LQTS的发生机制及寻找新的治疗靶点提供了材料基础。
| [1] |
PALLIEN T, KLUSSMANN E. New aspects in cardiac L-type Ca2+ channel regulation[J]. Biochem Soc Trans, 2020, 48(1): 39-49. DOI:10.1042/BST20190229 |
| [2] |
PITT GS, MATSUI M, CAO CK. Voltage-gated calcium channels in nonexcitable tissues[J]. Annu Rev Physiol, 2021, 83: 183-203. DOI:10.1146/annurev-physiol-031620-091043 |
| [3] |
VERGNOL A, TRAORÉ M, PIETRI-ROUXEL F, et al. New insights in CaVβ subunits: role in the regulation of gene expression and cellular homeostasis[J]. Front Cell Dev Biol, 2022, 10: 880441. DOI:10.3389/fcell.2022.880441 |
| [4] |
BEN-JOHNY M, YUE DT. Calmodulin regulation (calmodulation) of voltage-gated calcium channels[J]. J Gen Physiol, 2014, 143(6): 679-692. DOI:10.1085/jgp.201311153 |
| [5] |
CATTERALL WA. Structure and regulation of voltage-gated Ca2+ channels[J]. Annu Rev Cell Dev Biol, 2000, 16: 521-555. DOI:10.1146/annurev.cellbio.16.1.521 |
| [6] |
HOFMANN F, FLOCKERZI V, KAHL S, et al. L-type CaV1.2 calcium channels: from in vitro findings to in vivo function[J]. Physiol Rev, 2014, 94(1): 303-326. DOI:10.1152/physrev.00016.2013 |
| [7] |
BENMOCHA GUGGENHEIMER A, ALMAGOR L, TSEMAKHOVICH V, et al. Interactions between N and C termini of α1C subunit regulate inactivation of CaV1.2 L-type Ca (2+) channel[J]. Channels, 2016, 10(1): 55-68. DOI:10.1080/19336950.2015.1108499 |
| [8] |
HAN DY, MINOBE E, WANG WY, et al. Calmodulin- and Ca2+- dependent facilitation and inactivation of the Cav1.2 Ca2+ channels in guinea-pig ventricular myocytes[J]. J Pharmacol Sci, 2010, 112(3): 310-319. DOI:10.1254/jphs.09282fp |
| [9] |
AMES JB. L-type Ca2+ channel regulation by calmodulin and CaBP1[J]. Biomolecules, 2021, 11(12): 1811. DOI:10.3390/biom11121811 |
| [10] |
GAUBERG J, ELKHATIB W, SMITH CL, et al. Divergent Ca2+/calmodulin feedback regulation of CaV1 and CaV2 voltage-gated calcium channels evolved in the common ancestor of Placozoa and Bilateria[J]. J Biol Chem, 2022, 298(4): 101741. DOI:10.1016/j.jbc.2022.101741 |
| [11] |
PATE P, MOCHCA-MORALES J, WU Y, et al. Determinants for calmodulin binding on voltage-dependent Ca2+ channels[J]. J Biol Chem, 2000, 275(50): 39786-39792. DOI:10.1074/jbc.M007158200 |
| [12] |
URRUTIA J, AGUADO A, MUGURUZA-MONTERO A, et al. The crossroad of ion channels and calmodulin in disease[J]. Int J Mol Sci, 2019, 20(2): 400. DOI:10.3390/ijms20020400 |
| [13] |
CROTTI L, SPAZZOLINI C, TESTER DJ, et al. Calmodulin mutations and life-threatening cardiac arrhythmias: insights from the International Calmodulinopathy Registry[J]. Eur Heart J, 2019, 40(35): 2964-2975. DOI:10.1093/eurheartj/ehz311 |
| [14] |
ADLER A, NOVELLI V, AMIN AS, et al. An international, multicentered, evidence-based reappraisal of genes reported to cause congenital long QT syndrome[J]. Circulation, 2020, 141(6): 418-428. DOI:10.1161/CIRCULATIONAHA.119.043132 |
| [15] |
HUSSEY JW, LIMPITIKUL WB, DICK IE. Calmodulin mutations in human disease[J]. Channels, 2023, 17(1): 2165278. DOI:10.1080/19336950.2023.2165278 |
| [16] |
WANG KQ, BROHUS M, HOLT C, et al. Arrhythmia mutations in calmodulin can disrupt cooperativity of Ca2+ binding and cause misfolding[J]. J Physiol, 2020, 598(6): 1169-1186. DOI:10.1113/JP279307 |
| [17] |
SU JY, GAO QH, YU LF, et al. The LQT-associated calmodulin mutant E141G induces disturbed Ca2+-dependent binding and a flicke-ring gating mode of the CaV1.2 channel[J]. Am J Physiol Cell Phy- siol, 2020, 318(5): C991-C1004. DOI:10.1152/ajpcell.00019.2020 |
| [18] |
CHIN D, MEANS AR. Calmodulin: a prototypical calcium sensor[J]. Trends Cell Biol, 2000, 10(8): 322-328. DOI:10.1016/s0962-8924(00)01800-6 |
| [19] |
LIANG HY, DEMARIA CD, ERICKSON MG, et al. Unified mechanisms of Ca2+ regulation across the Ca2+ channel family[J]. Neuron, 2003, 39(6): 951-960. DOI:10.1016/s0896-6273(03)00560-9 |
| [20] |
SHAO DX, ZHAO MM, XU JJ, et al. The individual N- and C-lobes of calmodulin tether to the Cav1.2 channel and rescue the channel activity from Run-down in ventricular myocytes of guinea-pig heart[J]. FEBS Lett, 2014, 588(21): 3855-3861. DOI:10.1016/j.febslet.2014.09.029 |
| [21] |
BANERJEE R, YODER JB, YUE DT, et al. Bilobal architecture is a requirement for calmodulin signaling to CaV1.3 channels[J]. Proc Natl Acad Sci USA, 2018, 115(13): E3026-E3035. DOI:10.1073/pnas.1716381115 |
| [22] |
KAMEYAMA M, MINOBE E, SHAO DX, et al. Regulation of cardiac Cav1.2 channels by calmodulin[J]. Int J Mol Sci, 2023, 24(7): 6409. DOI:10.3390/ijms24076409 |
| [23] |
BOCZEK NJ, GOMEZ-HURTADO N, YE D, et al. Spectrum and prevalence of CALM1-, CALM2-, and CALM3-encoded calmodulin variants in long QT syndrome and functional characterization of a novel long QT syndrome-associated calmodulin missense variant, E141G[J]. Circ Cardiovasc Genet, 2016, 9(2): 136-146. DOI:10.1161/CIRCGENETICS.115.001323 |
| [24] |
JENSEN HH, BROHUS M, NYEGAARD M, et al. Human calmodulin mutations[J]. Front Mol Neurosci, 2018, 11: 396. DOI:10.3389/fnmol.2018.00396 |
 2024, Vol. 53
2024, Vol. 53