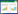文章信息
- 唐明政, 李晓凤, 荣耀, 吴志航, 马国榕, 刘松华, 蔡辉
- TANG Mingzheng, LI Xiaofeng, RONG Yao, WU Zhihang, MA Guorong, LIU Songhua, CAI Hui
- 双硫死亡相关lncRNA建立胰腺癌新的预后特征并预测免疫治疗反应
- Disulfidptosis-related lncRNAs establish new prognostic features and predict immunotherapeutic response in pancreatic cancer
- 中国医科大学学报, 2024, 53(1): 20-26
- Journal of China Medical University, 2024, 53(1): 20-26
-
文章历史
- 收稿日期:2023-05-06
- 网络出版时间:2024-01-04 19:36:32
2. 甘肃省人民医院普外科临床医学中心, 兰州 730000;
3. 甘肃省人民医院甘肃省外科肿瘤分子诊断与精准医学重点实验室, 兰州 730000
2. Clinical Medical Center for General Surgery, Gansu Provincial Hospital, Lanzhou 730000, China;
3. Key Laboratory of Molecular Diagnostics and Precision Medicine for Surgical Oncology in Gansu Province, Gansu Provincial Hospital, Lanzhou 730000, China
胰腺癌是一种预后不良、死亡率高的恶性肿瘤,近几十年来其5年总生存率几乎没有改善。预计在未来10年,胰腺癌将成为西方国家癌症相关死亡的第二大原因[1]。大多数癌症治疗失败和死亡是由于癌细胞的远处器官转移[2]。由于大多数胰腺癌早期症状不典型,发现时已为晚期,因此对胰腺癌早期诊断进行深入研究十分重要。
多项研究[3]已经证实胰腺癌与多种细胞死亡关系紧密,如铁死亡、铜死亡、失巢死亡和坏死性凋亡等。2023年LIU等[4]首次提出了一种新型细胞死亡方式,即由细胞内过量胱氨酸积累引起的二硫化物应激导致的快速死亡方式,并将这种全新的细胞死亡方式命名为双硫死亡;其研究揭示了二硫化物应激诱导细胞死亡的机制,即过量积累的二硫化物分子使肌动蛋白细胞骨架蛋白间的异常二硫化物键合,干扰其组织,最终导致肌动蛋白网络崩溃和细胞死亡。目前已经发现的双硫死亡相关基因有24个,分别为SLC7A11、GYS1、NDUFS1、NDUFA11、NUBPL、NCKAP1、LRPPRC、SLC3A2、RPN1、ACTN4、ACTB、CD2AP、CAPZB、DSTN、FLNA、FLNB、INF2、IQGAP1、MYH10、MYL6、MYH9、PDLIM1、TLN1和OXSM。
长链非编码RNA(long non-coding RNA,lncRNA)作为广泛的肿瘤标志物,可用于癌症的早期检测、诊断、预后以及药物治疗反应的预测。不同的lncRNA能通过调控表观遗传学或转录交替调节基因的表达,进而参与肿瘤的发生、转移,并影响化学耐药性的过程[5]。
本研究基于癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中的胰腺癌组织和癌旁正常组织样本数据构建双硫死亡相关的lncRNA风险评分模型。通过列线图预测胰腺癌患者的预后,通过基因本体(gene ontology,GO)、基因集富集分析(gene set enrichment analysis,GSEA)和免疫分析探究生物学功能。此外,通过肿瘤突变负荷(tumor mutation burden,TMB)评估免疫治疗反应。通过进一步了解胰腺癌中双硫死亡相关lncRNA的功能,为精确治疗和个体化管理提供新的方法,为患者提供新的潜在治疗策略、临床用药和抗肿瘤靶点,并为改善治疗选择提供了新的见解和观点。
1 材料与方法 1.1 数据的获取从TCGA数据库(https://portal.gdc.cancer.gov)中获取胰腺癌组织和癌旁正常组织样本的转录组RNA-seq数据集(包括HTSeq-seq和FPKM值),共获得179例胰腺癌样本和4例癌旁正常组织样本。
1.2 双硫死亡相关lncRNA的鉴定使用24个双硫死亡相关基因构建双硫死亡相关lncRNA。通过GENCODE注释文件和GTEx数据(http://cancergenome.nih.gov/abouttcga和 http://www.gtexportal.org)鉴定出TCGA中与双硫死亡相关的16 877个lncRNA。采用R语言DESeq2包筛选差异表达lncRNA,标准为|log2FC| > 1,错误发现率(false discovery rate,FDR) < 0.05,P < 0.05,并应用Pearson相关性分析鉴定双硫死亡相关基因lncRNA。
1.3 预后风险评分模型的建立和验证通过单变量Cox回归分析确定不同预后分类的lncRNA,随后通过LASSO回归分析过滤双硫死亡相关lncRNA,进行10倍交叉验证。利用LASSO方法筛选的双硫死亡相关lncRNA,建立多变量Cox回归风险评分模型。风险评分=系数1×lncRNA1的表达+ 系数2×lncRNA2的表达+……+系数n×lncRNAn的表达。根据表达值和系数计算风险评分,根据风险评分中位数将患者分为低风险组和高风险组。采用χ2检验,验证临床特征与风险评分的相关性。通过R语言glmnet、rms、survminer和timeROC包进行自变量评估,包括单因素和多因素Cox回归分析,最后应用受试者操作特征(receiver operator characteristic,ROC)曲线和一致性指数(C-index)测量模型的精准度。
1.4 预测列线图的构建和校准采用R语言rms包设置列线图,评估1年、3年和5年总生存率的风险评分的预测有效性。利用校准曲线检测列线图模型的准确性。
1.5 主成分分析(principal component analysis,PCA)、GO分析和GSEA采用PCA对胰腺癌样本双硫死亡相关lncRNA的表达模式进行分类,并绘制三维散点图以显示样本的空间分布。为了探究相关的生物学过程,进行GO分析和GSEA,以显示高风险组和低风险组中差异表达的生物学功能途径。采用R语言org.Hs.eg.db、clusterProfiler和enrichplot包进行分析。显著富集的生物过程和途径的阈值为P < 0.05,FDR < 0.25。
1.6 低风险和高风险人群的肿瘤微环境采用CIBERSORT算法评估风险评分与肿瘤浸润免疫细胞的相关性[6]。通过R语言estimate包计算低风险组和高风险组的肿瘤微环境评分,包括基质评分和免疫评分。
1.7 TMB从TCGA网站获取体细胞突变文件(TCGA.BRCA.varscan.DR-10.0.somatic),应用R语言maftools包,按风险评分中位数分为高风险组和低风险组,计算TMB。
2 结果 2.1 双硫死亡相关lncRNA在胰腺癌中的表达改变从TCGA数据库的胰腺癌样本中提取了24个双硫死亡相关基因和16 877个lncRNA。为了鉴定与双硫死亡相关基因相关的lncRNA,通过Pearson相关性分析和P < 0.005进行共表达分析,鉴定出171个与双硫死亡相关的lncRNA。
2.2 预后风险评分模型的构建与验证使用单因素Cox回归分析选取了23个显著差异表达的lncRNA(图 1A)。通过LASSO分析筛选出3个lncRNA(图 1B)。基于这些lncRNA,使用多因素Cox回归公式构建风险评分模型。风险评分=-0.5837× AC007292.1 + -1.5899×AC025165.1 + 0.3233×AC106045.1。AC106045.1在高风险组中高表达(图 1C)。与低风险组患者相比,高风险组患者总生存率显著降低(图 1D)。在肿瘤Ⅰ期和Ⅱ期患者中,与低风险组患者相比,高风险组患者生存率更低(图 1E)。风险评分模型对胰腺癌患者的1年、3年、5年的生存率有较好的诊断能力(图 1F)。ROC曲线分析显示,与其他临床病理特征相比,风险评分模型具有更强的生存预测能力(AUC=0.729)(图 1G)。
|
| A, univariate Cox regression analysis; B, correlation between lncRNAs and disulfidptosis-related genes in the risk score model; C, expression of disulfidptosis-related lncRNAs in the high-and low-risk groups; D, survival status of pancreatic cancer prognostic characteristics in the risk score model in the high-and low-risk groups; E, Kaplan-Meier survival curves of high-and low-risk groups at stagesⅠandⅡ; F, ROC curves at 1, 3, and 5 years; G, ROC curves for risk score model and clinicopathological features. 图 1 双硫死亡相关lncRNA的预后风险评分模型的构建与验证 Fig.1 Construction and validation of a prognostic risk score model for disulfidptosis-related lncRNA |
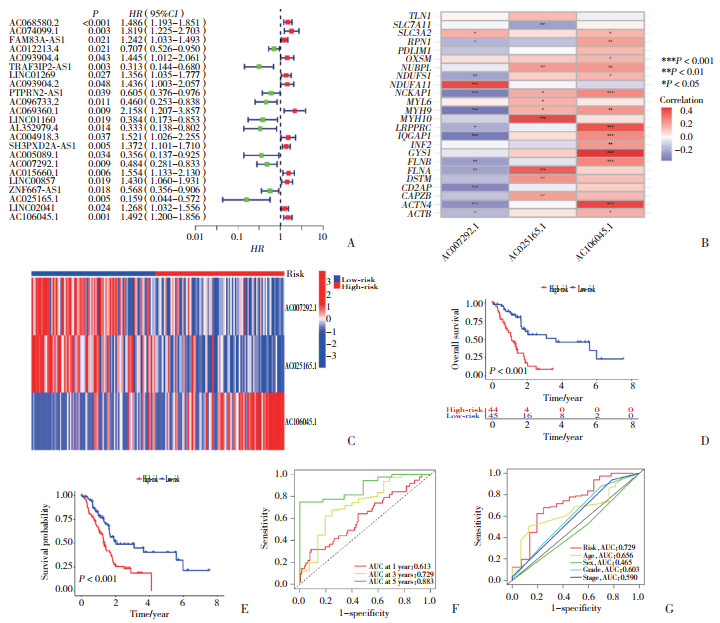
2.3 列线图的构建
根据风险评分、年龄和临床病理因素,开发列线图,预测1年、3年和5年的总生存率(图 2A)。使用校准图证明列线图预测的准确性,结果显示,与实际预测结果具有很好的一致性(图 2B)。
|
| A, nomogram to predict overall survival; B, calibration curves for overall survival at 1, 3, and 5 years. 图 2 模型的列线图和校准曲线 Fig.2 Nomogram and calibration curve of the model |
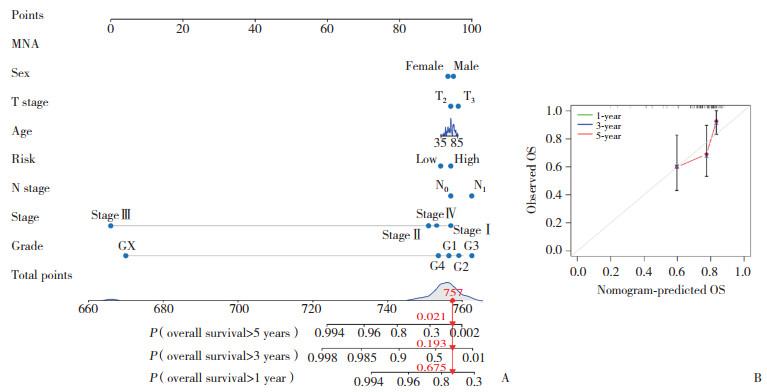
2.4 PCA和生物途径分析
使用PCA比较风险评分不同的样本,结果显示,按风险评分分组的样本有明显的聚集特征(图 3A)。GO分析结果显示,与跨突触信号的调节、信号释放和膜电位调节等有关(图 3B)。GSEA结果显示,低风险组和高风险组的生物学功能不同,如细胞周期、DNA复制和蛋白酶体等(图 3C、3D)。
|
| A, focused features of disulfidptosis-related lncRNAs in the risk score model; B, GO analysis; C and D, GSEA. BP, biological process; CC, cellular component; MF, molecular function. 图 3 高风险和低风险患者的PCA 3D散点图和功能分析 Fig.3 3D scatter plot and functional analysis of PCA in patients with high- and low-risk |
2.5 高危人群免疫特征的调查
将胰腺癌样本按风险评分从低到高排序,以显示各种免疫细胞的比例(图 4A)。高风险组患者的基质评分、免疫评分和ESTIMATE评分均低于低风险组患者(图 4B)。
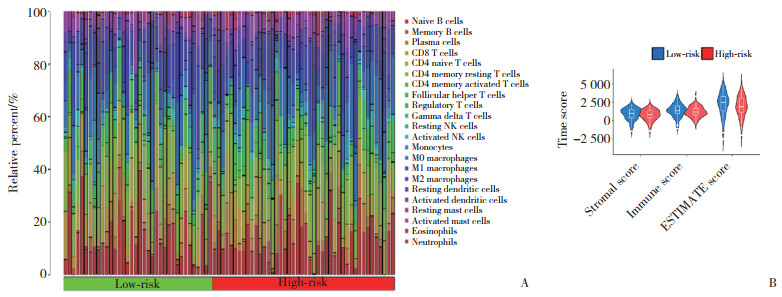
|
| A, relative proportions of infiltrating immune cells in different risk subgroups; B, stromal score, immune score, and estimate score for high-and low-risk groups. 图 4 具有不同风险评分的胰腺癌组织的免疫微环境 Fig.4 Immune microenvironment of pancreatic cancer tissues with different risk scores |
2.6 TMB
体细胞突变分析发现,高风险组和低风险组中突变率最高的5个基因是KRAS、TP53、SMAD4、CDKN2A和TTN。高风险组患者的TP53突变频率明显较高(图 5A、5B),且高风险组的TMB也明显较高(图 5C)。与低风险组相比,高风险组的高TMB患者的预后最差(图 5D)。
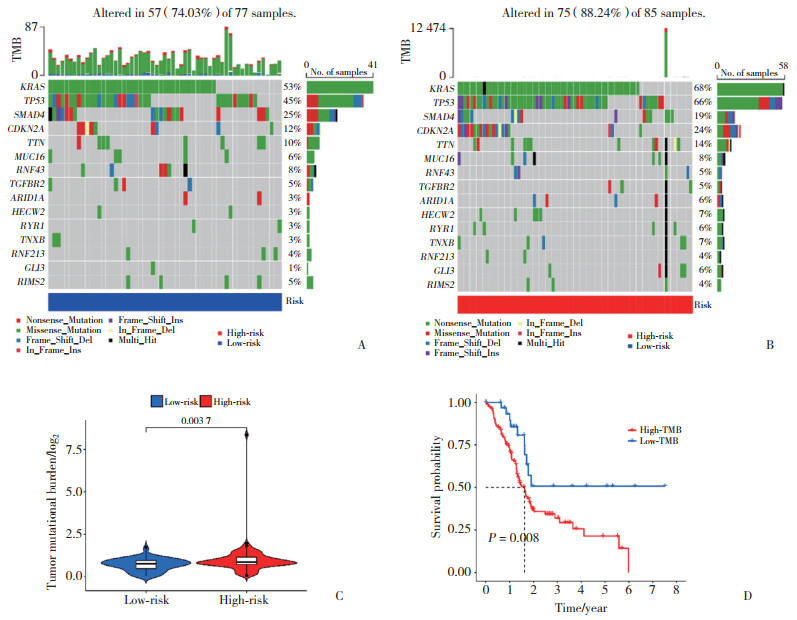
|
| A, waterfall plot of somatic mutations in tumors with low-risk score; B, waterfall plot of somatic mutations in tumors with high-risk score; C, TMB between low-and high-risk groups; D, Kaplan-Meier survival curves between high-and low-TMB groups. 图 5 TMB及其预后分析 Fig.5 TMB and its prognostic analysis |
3 讨论
胰腺癌是全球最致命的恶性肿瘤之一[7]。近年来在胰腺癌的筛查方面取得了一些进展,但仍不能准确识别和筛查高危人群。由于胰腺是腹膜后深层器官,早期胰腺癌患者可无任何症状,一旦发现可能已为晚期,导致胰腺癌的预后极差。因此,早期准确识别和发现生物标志物以预测胰腺癌是一个重大挑战[8]。
LIU等[4]发现,经诱导葡萄糖饥饿后,过表达SLC7A11的癌细胞死亡。这种死亡方式与之前的细胞死亡类型不同,其特征表现为不能被抑制细胞死亡的抑制剂恢复,并且敲低铁死亡关键基因ACSL4不能抑制这种细胞死亡。SLC7A11高表达在一定程度上恢复了葡萄糖饥饿导致的ATP降低。深入研究后发现,这种细胞死亡过程中胱氨酸累积并没有产生结晶,而胱氨酸是溶解度最差的氨基酸。以上研究结果提示,SLC7A11促进葡萄糖饥饿诱导的细胞死亡,并不是由于ATP耗竭或胱氨酸结晶毒性,而是硫醇氧化试剂diamide等显著增强葡萄糖饥饿条件下SLC7A11导致的细胞死亡。这种细胞死亡被命名为双硫死亡[4]。
lncRNA参与多种癌症相关的信号通路并发挥重要作用,在癌症的诊断和预后预测方面具有良好的潜力[9]。目前的研究旨在建立新颖有效的lncRNA风险评分模型,以及寻找恶性肿瘤患者的分子特征和潜在治疗靶点[10]。有研究[11]表明,lncRNA PVT1通过调节miR-619-5p/Pygo2和miR-619-5p/ATG14轴激活Wnt/β-连环蛋白和自噬途径,增强胰腺癌对吉西他滨的耐药性。另外,lncRNA PSMB8-AS1通过调节miR-382-3p/STAT1/PD-L1轴促进胰腺癌进展[12]。但是,胰腺癌中双硫死亡相关lncRNA风险评分模型的构建和预后预测的潜在能力尚未阐明。
本研究建立了一个包含3种双硫死亡相关lncRNA的风险评分模型,编号分别为AC007292.1、AC025165.1和AC106045.1。基于风险评分中位数,将患者分为高风险组和低风险组。GO分析结果显示,双硫死亡相关基因在跨突触信号的调节、信号释放以及膜电位调节等生物过程中具有重要作用。GSEA结果显示了双硫死亡相关基因富集程度不同的通路,包括细胞周期、DNA复制以及蛋白酶体等。ROC和C-index曲线验证了风险评分模型的可靠性。通过构建列线图预测胰腺癌患者预后,发现风险评分可能是预测预后的标准。
本研究采用ESTIMATE方法进行免疫分析,发现高风险组的免疫评分和基质评分均显著低于低风险组,从而推测该方法可能有助于预测胰腺癌患者的免疫细胞浸润和肿瘤微环境。体细胞突变分析结果显示,高风险组中TP53的突变频率更高,这说明TP53突变有可能提高胰腺癌对免疫治疗的活性。然而,并非每个胰腺癌患者都能从免疫疗法中受益,因为胰腺癌是免疫抵抗力最强的肿瘤类型之一,这与其独特的基因组景观有关[13]。因此,对于免疫治疗更敏感的患者,必须要选择合适的生物标志物。TMB与免疫检查点阻断治疗的效果有关[14]。本研究中,高风险组患者表现出更高的TMB,表明高风险组患者对免疫检查点阻断治疗的反应更好[15]。
本研究存在一些局限性。首先,所有胰腺癌的临床数据均从公共数据库获得,因此数据可能存在一些偏差。其次,虽然本研究表明双硫死亡相关lncRNA与免疫之间存在联系,但详细的机制尚不清楚。再有,建立的风险评分模型是否有效,还需更大的样本量来验证。最后,本研究结果基于生物信息学,未来需要更多的细胞实验和动物实验来验证。
综上所述,本研究通过构建新的lncRNA标记和列线图预测胰腺癌患者的预后,基于风险评分模型的分子表征为胰腺癌的进展和治疗提供了新的视角。免疫细胞浸润表达的差异可能是胰腺癌患者预后和治疗的重要指征。这些发现为评估胰腺癌预后和制订最佳治疗策略提供了新的途径。本研究结果提示,胰腺癌和多种细胞死亡均存在联系,如铁死亡、铜死亡、失巢死亡和坏死性凋亡等。双硫死亡相关的lncRNA介导胰腺癌免疫微环境的潜在机制研究大有前景。双硫死亡这一新兴的细胞死亡形式,未来应对其与胰腺癌的关系进行更多且更深层次的研究。
| [1] |
KLEIN AP. Pancreatic cancer epidemiology: understanding the role of lifestyle and inherited risk factors[J]. Nat Rev Gastroenterol Hepatol, 2021, 18(7): 493-502. DOI:10.1038/s41575-021-00457-x |
| [2] |
NOVAK U, JI H, KANAGASUNDARAM V, et al. STAT3 forms stable homodimers in the presence of divalent cations prior to activation[J]. Biochem Biophys Res Commun, 1998, 247(3): 558-563. DOI:10.1006/bbrc.1998.8829 |
| [3] |
SANTOFIMIA-CASTAÑO P, IOVANNA J. Combating pancreatic cancer chemoresistance by triggering multiple cell death pathways[J]. Pancreatology, 2021, 21(3): 522-529. DOI:10.1016/j.pan.2021.01.010 |
| [4] |
LIU X, NIE L, ZHANG Y, et al. Actin cytoskeleton vulnerability to disulfide stress mediates disulfidptosis[J]. Nat Cell Biol, 2023, 25(3): 404-414. DOI:10.1038/s41556-023-01091-2 |
| [5] |
RAMILOWSKI JA, YIP CW, AGRAWAL S, et al. Functional annotation of human long noncoding RNAs via molecular phenotyping[J]. Genome Res, 2020, 30(7): 1060-1072. DOI:10.1101/gr.254219.119 |
| [6] |
HUANG W, LI J, ZHOU S, et al. Discovering a four-gene prognostic model based on single-cell data and gene expression data of pancreatic adenocarcinoma[J]. Front Endocrinol (Lausanne), 2022, 13: 883548. DOI:10.3389/fendo.2022.883548 |
| [7] |
LI S, XU HX, WU CT, et al. Angiogenesis in pancreatic cancer: current research status and clinical implications[J]. Angiogenesis, 2019, 22(1): 15-36. DOI:10.1007/s10456-018-9645-2 |
| [8] |
SINGHI AD, KOAY EJ, CHARI ST, et al. Early detection of pancreatic cancer: opportunities and challenges[J]. Gastroenterology, 2019, 156(7): 2024-2040. DOI:10.1053/j.gastro.2019.01.259 |
| [9] |
SNYDER AG, HUBBARD NW, MESSMER MN, et al. Intratumoral activation of the necroptotic pathway components RIPK1 and RIPK3 potentiates antitumor immunity[J]. Sci Immunol, 2019, 4(36): eaaw2004. DOI:10.1126/sciimmunol.aaw2004 |
| [10] |
GUO Y, QU Z, LI D, et al. Identification of a prognostic ferroptosis-related lncRNA signature in the tumor microenvironment of lung adenocarcinoma[J]. Cell Death Discov, 2021, 7(1): 190. DOI:10.1038/s41420-021-00576-z |
| [11] |
ZHOU C, YI C, YI Y, et al. LncRNA PVT1 promotes gemcitabine resistance of pancreatic cancer via activating Wnt/β-catenin and autophagy pathway through modulating the miR-619-5p/Pygo2 and miR-619-5p/ATG14 axes[J]. Mol Cancer, 2020, 19(1): 118. DOI:10.1186/s12943-020-01237-y |
| [12] |
ZHANG H, ZHU C, HE Z, et al. LncRNA PSMB8-AS1 contributes to pancreatic cancer progression via modulating miR-382-3p/STAT1/PD-L1 axis[J]. J Exp Clin Cancer Res, 2020, 39(1): 179. DOI:10.1186/s13046-020-01687-8 |
| [13] |
BEAR AS, VONDERHEIDE RH, O'HARA MH. Challenges and opportunities for pancreatic cancer immunotherapy[J]. Cancer Cell, 2020, 38(6): 788-802. DOI:10.1016/j.ccell.2020.08.004 |
| [14] |
WANG C, WANG Z, ZHAO Y, et al. Tumor mutation burden-related long non-coding RNAs is predictor for prognosis and immune response in pancreatic cancer[J]. BMC Gastroenterol, 2022, 22(1): 495. DOI:10.1186/s12876-022-02535-z |
| [15] |
KEENAN TE, BURKE KP, VAN ALLEN EM. Genomic correlates of response to immune checkpoint blockade[J]. Nat Med, 2019, 25(3): 389-402. DOI:10.1038/s41591-019-0382-x |
 2024, Vol. 53
2024, Vol. 53