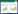文章信息
- 刘雅峰, 鲁继斌
- LIU Yafeng, LU Jibin
- 铜死亡相关配对长链非编码RNA肺腺癌预后模型的构建
- Cuproptosis-related long noncoding RNA pairs as the prognostic signature in lung adenocarcinoma
- 中国医科大学学报, 2023, 52(7): 638-643, 661
- Journal of China Medical University, 2023, 52(7): 638-643, 661
-
文章历史
- 收稿日期:2022-09-30
- 网络出版时间:2023-07-06 15:15:47
肺癌是全球癌症死亡的最主要原因。在我国,其发病率和死亡率亦位于恶性肿瘤之首[1]。肺腺癌属于非小细胞肺癌,是肺癌最常见的病理类型,约占肺癌整体的40%[2]。分子靶向治疗和免疫治疗已经成为肺腺癌的常规治疗手段,使其预后有所改善,但我国肺癌患者总体5年生存率仍小于20%。因此,寻找预测肺癌预后的生物标志物十分必要。
铜是生物体必需的微量元素。但当细胞内的铜离子浓度超过稳态阈值时,会表现出细胞毒性。铜死亡是一种新型细胞程序性死亡,其原理为过量的铜离子直接与三羧酸循环中的脂酰化成分结合,诱导脂酰化蛋白质聚集和铁硫簇蛋白质丢失,激发蛋白质毒性应激,从而导致细胞死亡[3]。已有研究[4]表明,铜离子与肿瘤的生长、增殖和抗血管生成等密切相关。
长链非编码RNA(long noncoding RNA,lncRNA)是一种长度超过200个核苷酸的非编码RNA。许多研究[5-6]证实,lncRNA可以调控肺腺癌的生物学行为,还可以作为预测肺腺癌预后等的潜在生物标志物。本研究旨在开发新型铜死亡相关配对lncRNA的预后标志物,以期提高预测肺腺癌患者预后的准确性以及评估其药物敏感性。
1 材料与方法 1.1 数据的获取和确定铜死亡相关差异lncRNA(differentially expressed cuproptosis-related lncRNA,DEcrlncRNA)由癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库获取肺腺癌患者转录组数据、基因突变信息和对应的临床信息。使用Ensembl数据库来源的注释文件,注释其中的lncRNA。从已有文献中获取共10个铜死亡相关基因,包括FDX1、LIAS、LIPT1、DLD、DLAT、PDHA1、PDHB、MTF1、GLS和CDKN2A。以相关系数|r| > 0.4且P < 0.000 1为标准进行Pearson相关性分析,得到铜死亡相关lncRNA(cuproptosisrelated lncRNA,crlncRNA)。使用R语言limma包,设定阈值为|log2FC| > 1且P < 0.05进行差异分析,获得DEcrlncRNA。
1.2 鉴定铜死亡相关配对lncRNA对DEcrlncRNA进行两两循环配对分析。根据lncRNA的相对表达水平,若lncRNA1与lncRNA2的表达水平之比 > 1,标记该配对lncRNA为1,反之记录为0,从而构造出包含1或0的表达矩阵。仅保留0或1在所有样本中比率在0.2~0.8之间的配对lncRNA。
1.3 风险模型的构建和检验使用单因素Cox回归分析确定与患者总生存时间(overall survival,OS)相关的配对DEcrlncRNA。为了防止模型过度拟合,应用LASSO回归分析进行进一步收敛。然后采用多因素Cox回归分析,构建风险模型并计算风险评分。根据风险评分的中位数,将肺腺癌患者分为高风险组和低风险组。使用Kaplan-Meier生存分析验证高风险组和低风险组间患者的生存差异。此外,通过绘制受试者操作特征(receiver operating characteristic,ROC)曲线,以曲线下面积(area under curve,AUC)为标准,评估风险评分预测1年、3年和5年生存情况的准确性。绘制风险评分与临床参数的ROC曲线,以对比预测效能。最后,对风险评分和常见的临床参数进行单因素和多因素Cox回归分析,以验证构建的风险模型是否可以独立于性别、年龄和肿瘤分期等临床参数,有效预测肺腺癌患者的预后。
1.4 基因功能富集分析应用R语言clusterProfiler包,并基于基因本体(gene ontology,GO)以及京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG),探索影响铜死亡风险评分高低的生物学功能机制,分析潜在通路。
1.5 基因突变分析为了进一步分析不同铜死亡相关风险分组的基因突变图景,使用R语言maftools包处理由TCGA数据库获取的肺腺癌患者基因突变信息,分别分析并展示高风险组和低风险组基因突变的频率和特点。
1.6 药物反应预测为了更好的评估风险模型对肺腺癌患者的临床应用价值,使用癌症基因组计划数据库来源的药物处理信息,应用R语言pRRophetic包评估常见肺腺癌化疗药物的半数抑制浓度(half maximal inhibitory concentration,IC50)。比较高风险组和低风险组间IC50的差异。
1.7 统计学分析采用R语言4.1.0版本进行统计学分析。采用Pearson相关性分析评估相关系数。采用t检验确定组间差异。采用Kaplan-Meier法进行生存分析,采用对数秩检验确定差异显著性。P < 0.05为差异有统计学意义。
2 结果 2.1 确定DEcrlncRNA和配对DEcrlncRNA 2.1.1 DEcrlncRNA的筛选纳入TCGA数据库中临床信息完整的481个肺腺癌样本和59个癌旁组织样本的转录组数据。提取其中的lncRNA数据,与铜死亡相关基因进行相关性分析后,共得到267个crlncRNA。crlncRNA经差异分析后共筛选出145个DEcrlncRNA,其中上调lncRNA 74个,下调lncRNA 71个。
2.1.2 配对DEcrlncRNA的确定对145个DEcrlncRNA循环配对并计算0/1矩阵,再次筛选后得到有效配对DEcrlncRNA,共3 647对。
2.2 配对DEcrlncRNA的肺腺癌预后模型的构建与评估 2.2.1 预后模型的构建采用单因素Cox回归分析筛选出OS相关配对DEcrlncRNA共178个,行Lasso回归分析。之后使用逐步回归法,得到用于构建风险模型的6个配对DEcrlncRNA(表 1)。基于以上配对DEcrlncRNA,通过多因素Cox回归分析计算风险评分。
| DEcrlncRNA pairs | Univariate Cox | Multivariate Cox | |||
| HR(95%CI) | P | HR(95%CI) | P | ||
| TDRKH-AS1|OGFRP1 | 0.916(0.886-0.947) | < 0.001 | 0.950(0.920-0.990) | 0.009 | |
| AC092809.3|LINC02518 | 0.929(0.898-0.962) | < 0.001 | 0.960(0.930-1.000) | 0.032 | |
| CRIM1-DT|AC009065.5 | 1.079(1.042-1.117) | < 0.001 | 1.070(1.030-1.110) | < 0.001 | |
| EMSLR|AC009065.5 | 1.075(1.034-1.118) | < 0.001 | 1.050(1.010-1.100) | 0.011 | |
| AC026401.3|CARD8-AS1 | 1.102(1.052-1.154) | < 0.001 | 1.100(1.050-1.160) | < 0.001 | |
| AC024075.1|OGFRP1 | 0.932(0.902-0.963) | < 0.001 | 0.960(0.930-0.990) | 0.022 | |
2.2.2 预后模型的评估
Kaplan-Meier生存分析曲线提示,高风险组预后更差,进一步证实了预后模型的有效性(图 1A)。预后模型1年、3年和5年生存率ROC曲线的AUC范围为0.69~0.76(图 1B)。此外,比较预后模型与常见临床相关因素对3年生存率的预测能力,发现风险评分具有最大AUC(图 1C),这些结果充分说明了预后风险模型的优异性。对风险评分与常见临床因素进行单因素和多因素Cox回归分析,结果显示,仅风险评分和临床分期为肺腺癌患者的独立预后因素(图 1D、1E),可见风险评分具有良好的预后预测价值。
|
| A, survival outcomes between high-and low-risk patients based on Kaplan-Meier survival analysis; B, 1-, 3-, and 5-year survival rate ROC curves and AUC values of the signature; C, comparison of ROC curves of prognostic signature with other clinical features; D, univariate Cox regression analysis of risk score and clinical features, revealed that the risk score was an independent risk factor; E, multivariate Cox regression analysis of risk score and clinical features, revealed that the risk score was an independent risk factor. 图 1 预后模型的评估 Fig.1 Validation of the prognostic signature |
2.3 高风险组和低风险组间差异基因的功能富集分析
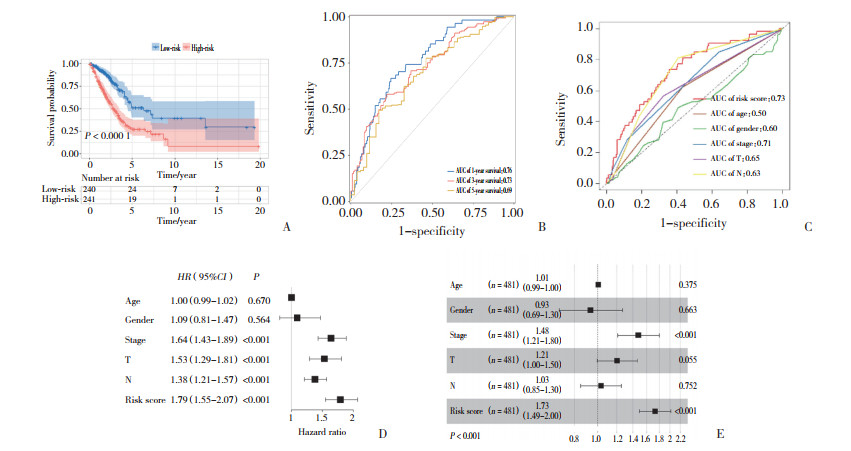
为深入探究不同风险组间潜在的生物学机制,进行了基于高风险组和低风险组间差异基因的GO和KEGG分析。GO分析结果(图 2A)显示,风险评分差异基因主要作用于生长因子的结合、细胞外基质构成和分泌颗粒等生物学过程。KEGG通路分析结果(图 2B)提示,差异基因主要集中于p53信号通路、肿瘤的转录调控通路和白细胞介素-17信号通路等。
|
| A, GO;B, KEGG. BP, biological process; CC, cell component; MF, molecular function. 图 2 功能富集分析 Fig.2 Functional enrichment analysis |
2.4 铜死亡相关预后模型的基因突变分析
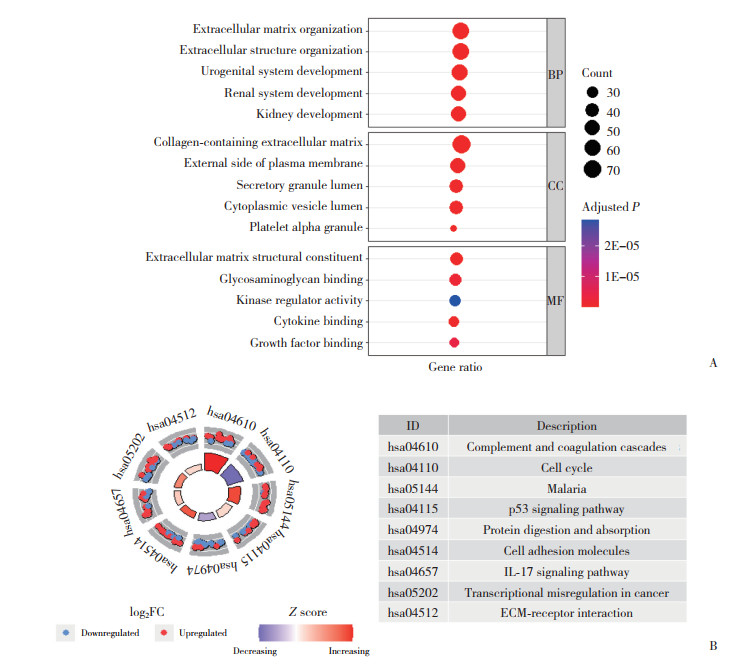
使用基因突变全景分析,对理解基因突变层面的高风险组和低风险组的差别意义重大。高风险组患者基因突变频率更高,TP53与TTN为2组患者最常见的突变基因(图 3)。此外,多数的2组间共有基因,在低风险组中突变频率显著降低。COL1A1等基因突变频率较高,仅发生于高风险组,这可能与高风险组更差的预后相关。
|
| A, the landscape of mutation of patients in high-risk groups; B, the landscape of mutation of patients in low-risk groups. 图 3 基因突变瀑布图 Fig.3 The waterfall plot of gene mutation |
2.5 预后模型预测化疗药物敏感性
鉴于高风险组和低风险组间生存差异显著,对肺腺癌患者常用的化疗药物进行筛选、分析,以期进一步指导临床决策。比较高风险组与低风险组化疗药物的IC50,发现在高风险组的患者中,顺铂、紫杉醇、吉他西滨、多西他赛、长春瑞滨和依托泊苷的IC50均相对较低(图 4)。这些结果表明,更高的风险评分与化疗药物敏感性的增加相关,证实风险模型是指导临床个体化治疗的优秀指标。

|
| 图 4 药物敏感性分析 Fig.4 Drug sensitivity analysis |
3 讨论
肺腺癌是肺癌的最常见亚型。近年来,应用低剂量CT进行肺癌筛查逐渐普及,显著降低了肺腺癌的死亡率。分子靶向药物和免疫治疗不断进步,明显改善了肺腺癌患者的预后。然而,肺腺癌具有异质性,不同人群对治疗的反应程度及其生存时间有很大的差异性。此外,肺癌的总体预后仍然较差。由此可见,目前仍迫切需要开发肺腺癌相关的预后模型,发现新的肺腺癌治疗靶点,并探索其潜在的生物学机制。
细胞程序性死亡是最具潜力的抗肿瘤机制之一。最近,QU等[6]提出了铜死亡的概念,它是一种不同于细胞凋亡、铁死亡和焦亡等的全新的铜依赖性细胞程序性死亡。铜死亡的发现,进一步丰富了抗肿瘤的潜在机制。目前已有研究[7-8]证实,铜死亡相关的特征模型对肺腺癌患者预后具有预测价值或免疫治疗相关性,其中部分研究[8]建立了基于lncRNA的模型。考虑到测序平台、检测时间和标准化处理等的差异带来的批次效应,可能影响基于表达量进行预后预测的准确性。受既往研究[9]启发,本研究采用一种新算法,即根据lncRNA相对表达量而非具体的表达量,来构建预后模型。这种模型既削弱了批次效应,还可以利用如免疫组织化学、PCR、转录组数据和基因芯片等各种来源的lncRNA表达量数据,具有较高的临床应用价值。
本研究中,预后风险模型共包含10个lncRNA(基于6个配对DEcrlncRNA),其中5个lncRNA已被证实与肺腺癌或其他肿瘤的发生、发展和预后密切相关。研究[10]证实,OGFRP1通过干扰miR-124-3p进而上调LYPD3表达来促进非小细胞肺癌进展。LIU等[11]发现,OGFRP1还可通过miR-4640-5p/eIF5A轴在肺腺癌中发挥癌基因作用。CARD8-AS1表达量降低与肺腺癌较差的预后相关,其机制可能是CARD8-AS1下调后,通过靶向miR-650进而下调Bax表达实现[12]。JIAO等[13]证明,TDRKH-AS1还可通过激活Wnt/β-catenin信号通路促进结直肠癌细胞的增殖和侵袭,且其与结直肠癌预后呈负相关。LINC02518和AC026401.3被证实与肝细胞癌预后呈负相关[14-15],其中AC026401.3通过与OCT1相互作用来增加E2F2的转录,以此增强肝细胞癌对索拉非尼和仑伐替尼的耐药性。以上研究为构建的模型提供了理论支持。
本研究存在一定的局限性。首先,本研究为回顾性研究,未来需要多中心前瞻性的真实世界临床数据来进一步证实结论。其次,本研究仅采用来自于TCGA数据库的转录组数据,需要更多的实验数据来探究以上lncRNA调节铜死亡和影响铜死亡等方面的潜在机制,以进一步支持这些结论。最后,受临床数据和药物数据库所限,本研究仅能分析部分肺腺癌常用化疗药物(如顺铂、紫杉醇和依托泊苷等)的IC50,缺乏对靶向药和免疫治疗药物敏感性的分析数据。
综上所述,本研究确立了一种由配对DEcrlncRNA构建的新模型,它可以有效预测肺腺癌患者的预后情况,不受lncRNA精确表达量和数据来源的限制,并且有助于预测治疗敏感性,这可能有利于筛选出相应的潜在受益人群。
| [1] |
XIA C, DONG X, LI H, et al. Cancer statistics in China and United States, 2022:profiles, trends, and determinants[J]. Chin Med J (Engl), 2022, 135(5): 584-590. DOI:10.1097/CM9.0000000000002108 |
| [2] |
ZAPPA C, MOUSA SA. Non-small cell lung cancer: current treatment and future advances[J]. Transl Lung Cancer Res, 2016, 5(3): 288-300. DOI:10.21037/tlcr.2016.06.07 |
| [3] |
TSVETKOV P, COY S, PETROVA B, et al. Copper induces cell death by targeting lipoylated TCA cycle proteins[J]. Science, 2022, 375(6586): 1254-1261. DOI:10.1126/science.abf0529 |
| [4] |
GE EJ, BUSH AI, CASINI A, et al. Connecting copper and cancer: from transition metal signalling to metalloplasia[J]. Nat Rev Cancer, 2022, 22(2): 102-113. DOI:10.1038/s41568-021-00417-2 |
| [5] |
DONG HX, WANG R, JIN XY, et al. LncRNA DGCR5 promotes lung adenocarcinoma (LUAD) progression via inhibiting hsa-mir-22-3p[J]. J Cell Physiol, 2018, 233(5): 4126-4136. DOI:10.1002/jcp.26215 |
| [6] |
QU S, JIAO Z, LU G, et al. PD-L1 lncRNA splice isoform promotes lung adenocarcinoma progression via enhancing c-Myc activity[J]. Genome Biol, 2021, 22(1): 104. DOI:10.1186/s13059-021-02331-0 |
| [7] |
ZHANG G, CHEN X, FANG J, et al. Cuproptosis status affects treatment options about immunotherapy and targeted therapy for patients with kidney renal clear cell carcinoma[J]. Front Immunol, 2022, 13(1): 954440-954460. DOI:10.3389/fimmu.2022.954440 |
| [8] |
YANG X, WANG X, SUN X, et al. Construction of five cuproptosisrelated lncRNA signature for predicting prognosis and immune activity in skin cutaneous melanoma[J]. Front Genet, 2022, 13: 972899. DOI:10.3389/fgene.2022.972899 |
| [9] |
LI B, CUI Y, DIEHN M, et al. Development and validation of an individualized immune prognostic signature in early-stage nonsquamous non-small cell lung cancer[J]. JAMA Oncol, 2017, 3(11): 1529-1537. DOI:10.1001/jamaoncol.2017.1609 |
| [10] |
TANG LX, CHEN GH, LI H, et al. Long non-coding RNA OGFRP1 regulates LYPD3 expression by sponging miR-124-3p and promotes non-small cell lung cancer progression[J]. Biochem Biophys Res Commun, 2018, 505(2): 578-585. DOI:10.1016/j.bbrc.2018.09.146 |
| [11] |
LIU X, NIU N, LI P, et al. LncRNA OGFRP1 acts as an oncogene in NSCLC via miR-4640-5p/eIF5A axis[J]. Cancer Cell Int, 2021, 21(1): 425. DOI:10.1186/s12935-021-02115-3 |
| [12] |
JI Y, ZHANG G, ZHANG X. Identification of lncRNA CARD8-AS1 as a potential prognostic biomarker associated with progression of lung adenocarcinoma[J]. Br J Biomed Sci, 2022, 79: 10498. DOI:10.3389/bjbs.2022.10498 |
| [13] |
JIAO Y, ZHOU J, JIN Y, et al. Long non-coding RNA TDRKH-AS1 promotes colorectal cancer cell proliferation and invasion through the β-catenin activated Wnt signaling pathway[J]. Front Oncol, 2020, 10: 639. DOI:10.3389/fonc.2020.00639 |
| [14] |
CUI W, HUANG J, WANG R, et al. Predictive value of a novel lnc-RNA LINC02518 in evaluating the prognosis of patients with hepatocellular carcinoma[J]. Biomark Med, 2021, 15(14): 1277-1288. DOI:10.2217/bmm-2020-0795 |
| [15] |
WANG Y, TAN K, HU W, et al. LncRNA AC026401.3 interacts with OCT1 to intensify sorafenib and lenvatinib resistance by activating E2F2 signaling in hepatocellular carcinoma[J]. Exp Cell Res, 2022, 420(1): 113335-113347. DOI:10.1016/j.yexcr.2022.113335 |
 2023, Vol. 52
2023, Vol. 52