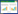文章信息
- 刘畅, 卞华, 许博
- LIU Chang, BIAN Hua, XU Bo
- 基于生物信息学探讨骨关节炎相关分子机制及免疫细胞浸润分析
- Molecular mechanism and immune cell infiltration of osteoarthritis based on bioinformatics
- 中国医科大学学报, 2023, 52(5): 385-391, 397
- Journal of China Medical University, 2023, 52(5): 385-391, 397
-
文章历史
- 收稿日期:2022-10-13
- 网络出版时间:2023-05-19 15:33:28
2. 南阳理工学院张仲景国医国药学院,河南省张仲景方药与免疫调节重点实验室,河南 南阳 473004
2. Zhang Zhongjing College of Traditional Chinese Medicine, Nanyang Institute of Technology, Henan Key Laboratory of Zhang Zhongjing Formulae and Herbs for Immunoregulation, Nanyang 473004, China
骨关节炎(osteoarthritis,OA)可影响任何滑膜关节,其中髋部、膝部、手部、足部和脊柱是最常受累的部位[1]。目前对影响OA发病的危险因素尚不清楚,多数研究[2]认为可能与年龄、性别、肥胖、遗传、饮食,以及关节损伤、错位和异常负荷有关。本研究基于基因表达图谱数据库芯片数据,通过与正常人群数据进行对比,探讨OA发病的分子机制。
1 材料与方法 1.1 数据收集以“osteoarthritis”为关键词,在基因表达综合(gene expression omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)中检索,选取编号为GSE51588和GSE98918的2组芯片,共包含52例OA和22例正常对照组实验数据。再根据GEO数据库中获得的探针注释文件,将每个数据集中的探针更改为基因符号,将这2个数据集合并到1个元数据队列中进行集成分析。当同一基因符号对应多个探针时,将探针的平均值作为该基因的表达值。然后选取包含12例OA和12例正常对照组样本的GSE117999芯片作为验证队列。
1.2 差异表达基因(differentially expressed gene,DEG)分析使用R软件中的“limma”包对元数据队列进行差异分析,采用“FDR”处理方法并设置过滤条件为logFCfilter > 1,校正P < 0.05,进行筛选得到DEG。然后使用R软件中“clusterProfiler”包以P(P value filter) < 0.05为过滤条件对DEG进行基因本体(gene ontology,GO)和疾病本体(disease ontology,DO)富集分析。
1.3 特征生物标志物筛选使用最小绝对收缩和选择算子(least absolute shrinkage and selection operator,LASSO)与支持向量机(support vector machine,SVM)2种机器学习算法预测特征生物标志物。首先利用R软件中的“glmnet”包,对DEG使用LASSO回归算法进行交叉验证并筛选特征基因。通过R软件中的“e1071”“kernlab”“caret”包采用支持向量机-递归特征消除(support vector machine-recursive feature elimination,SVM-RFE)方法,识别DEG中具有最高分辨能力的基因集。
1.4 特征生物标志物对OA的诊断与治疗价值判断为测试1.3中得到的特征基因价值,将验证组中所包含的12例OA和12例正常对照组样本数据导入R软件中,利用“limma”和“ggpubr”包绘制箱线图,判断LASSO与SVM-RFE算法所得交集基因的价值。通过R软件中的“pROC”包,使用相同的数据绘制受试者操作特征(receiver operating characteristic,ROC)曲线,用ROC曲线下面积(area under the curve,AUC)值确定OA的诊断有效性。
1.5 免疫细胞含量分析采用CIBERSORT算法对浸润免疫细胞进行相关分析和可视化。用R软件中的“corrplot”和“vioplot”包对DEG行进一步分析及可视化处理,得到OA免疫细胞之间的相关图以及代表差异大小的小提琴图。
1.6 已鉴定基因生物标志物与浸润性免疫细胞的相关性分析利用R软件中的Spearman等级相关分析,研究已鉴定的基因生物标志物与免疫细胞渗入水平的相关性,再用图表函数和R软件“ggplot2”包对二者的相关性进行可视化处理。
2 结果 2.1 DEG获取结果对GSE51588、GSE98918芯片中基因表达数据进行分析,结果显示,共获取OA组与正常对照组DEG 96个,其中上调基因47个,下调基因49个,包含显著上调基因20个,显著下调基因23个。见图 1。
|
| Red indicates up-regulation, green indicates down-regulation, the higher the position, the more obvious. 图 1 训练组差异表达火山图 Fig.1 The training group showed differentially expressed volcano map |
2.2 GO富集分析及DO富集分析结果
对DEG进行GO和DO富集分析,并绘制柱状图。GO富集分析结果显示,DEG主要集中在共生体相互作用、真菌等生物学进程和初级溶酶体、嗜苯胺蓝颗粒等细胞组分,以及肝素结合、受体配体活动等分子功能。DO富集结果显示,DEG多富集在动脉粥样硬化、动脉硬化性心血管疾病、动脉硬化等疾病。
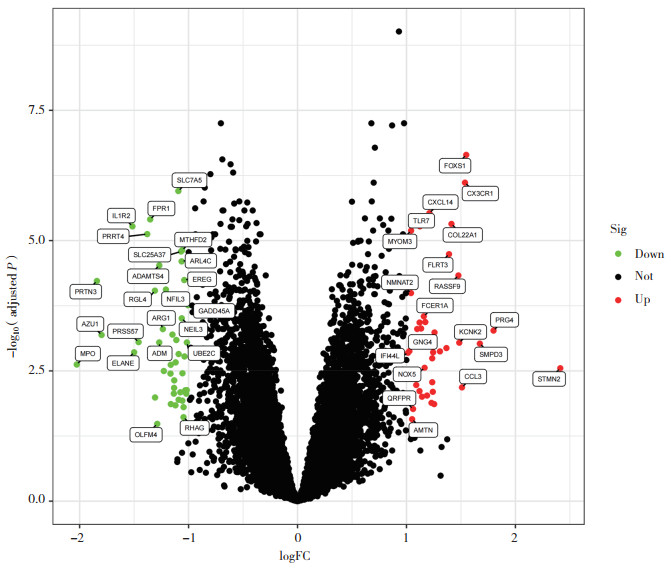
2.3 诊断特征生物标志物的识别和验证首先用LASSO算法缩小DEG的范围,最终确定了19个变量作为OA的诊断生物标志物(图 2A)。使用SVM-RFE算法确定了DEG中25个特征的子集(图 2B)。最终选择了这2种算法之间13个重叠特征基因(图 2C)。
|
| A, LASSO; B, SVM-RFE; C, Venn diagram of LASSO-SVM-RFE. 图 2 OA特征生物标志物的识别 Fig.2 Identification of OA characteristic biomarkers |
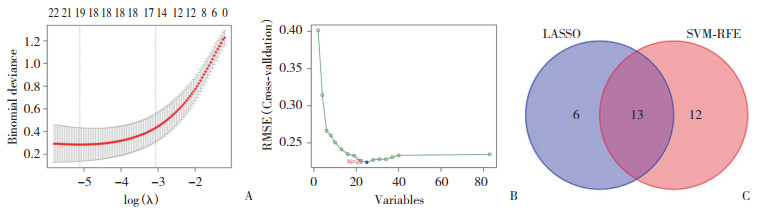
为了产生更准确可靠的结果,使用验证组GSE117999芯片验证这25个特征基因的表达水平,结果如图 3所示,样品中CSN1S1、CXCL14、MTHFD2、NMNAT2、TLR7表达水平在OA中差异显著(均P < 0.05),因此,这5个显著差异基因被用于logistic回归算法建立诊断模型。
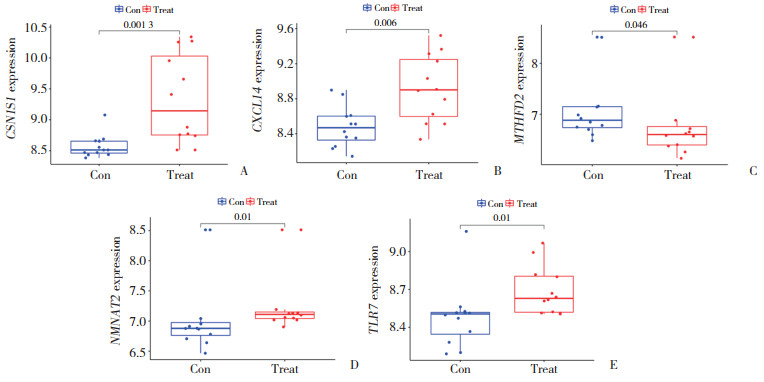
|
| A, CSN1S1; B, CXCL14; C, MTHFD2; D, NMNAT2; E, TLR7. 图 3 OA特征生物标志物的验证 Fig.3 Validation of OA characteristic biomarkers |
2.4 特征基因的诊断有效性
如图 4所示,CSN1S1、CXCL14、MTHFD2、NMNAT2、TXR7的AUC值分别为0.819(95%CI:0.721~0.906)、0.899(95%CI:0.817~0.962)、0.862(95%CI:0.775~0.935)、0.884(95%CI:0.800~0.952)、0.858(95%CI:0.751~0.940),提示这5种特征基因生物标志物对区分OA与正常对照组有统计学意义。
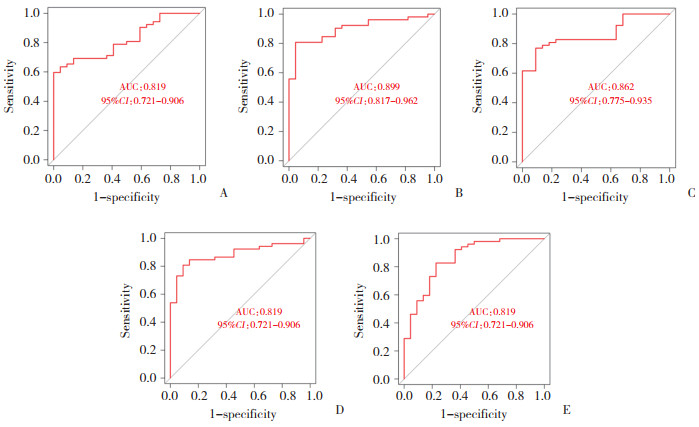
|
| A, CSN1S1; B, CXCL14; C, MTHFD2; D, NMNAT2; E, TLR7. 图 4 OA特征基因的诊断有效性 Fig.4 Diagnostic validity of OA characteristic gene |
2.5 免疫细胞浸润分析
OA组免疫细胞间相关性分析结果如图 5所示,正相关性较高的组合有静息记忆CD4+T细胞和幼稚B细胞、幼稚B细胞和单核细胞、嗜酸性粒细胞和激活的树突状细胞等。负相关性较高的组合有幼稚CD4+T细胞和单核细胞、MO巨噬细胞和CD8+T细胞、中性粒细胞和幼稚CD4+T细胞等。免疫细胞差异分析结果如图 6所示,正常对照组中,幼稚B细胞、单核细胞、激活的肥大细胞、中性粒细胞的表达明显增加(P < 0.05);OA组中,幼稚CD4+T细胞、滤泡辅助细胞、巨噬细胞M1、静息树突状细胞的表达明显增加(P < 0.05)。
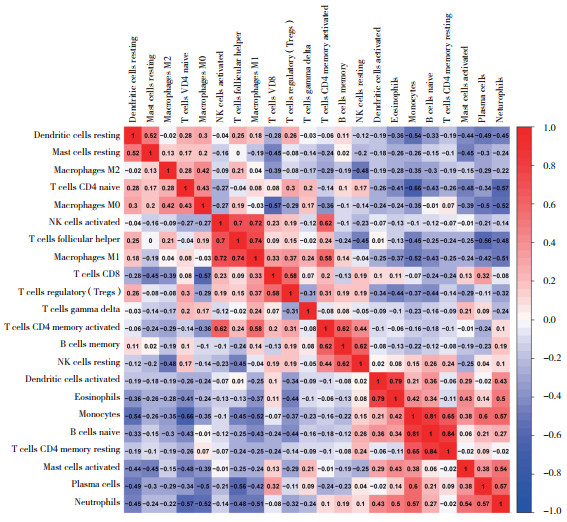
|
| The darker the red, the higher the positive correlation between the two, and the darker the blue, the higher the negative correlation between the two. 图 5 免疫细胞间的相关性 Fig.5 The correlation between immune cells |
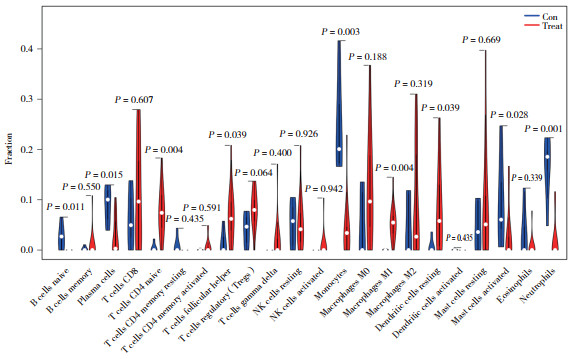
|
| 图 6 免疫细胞差异分析 Fig.6 Analysis of immune cell differences |
2.6 基因生物标志物与浸润性免疫细胞的相关性分析
相关性分析结果显示,CSN1S1与单核细胞、浆细胞呈负相关(图 7A);MTHFD2与静息肥大细胞呈正相关,与CD8+T细胞呈负相关(图 7B);NMNAT2与巨噬细胞M1、调节性T细胞呈正相关,与幼稚B细胞呈负相关(图 7C)。
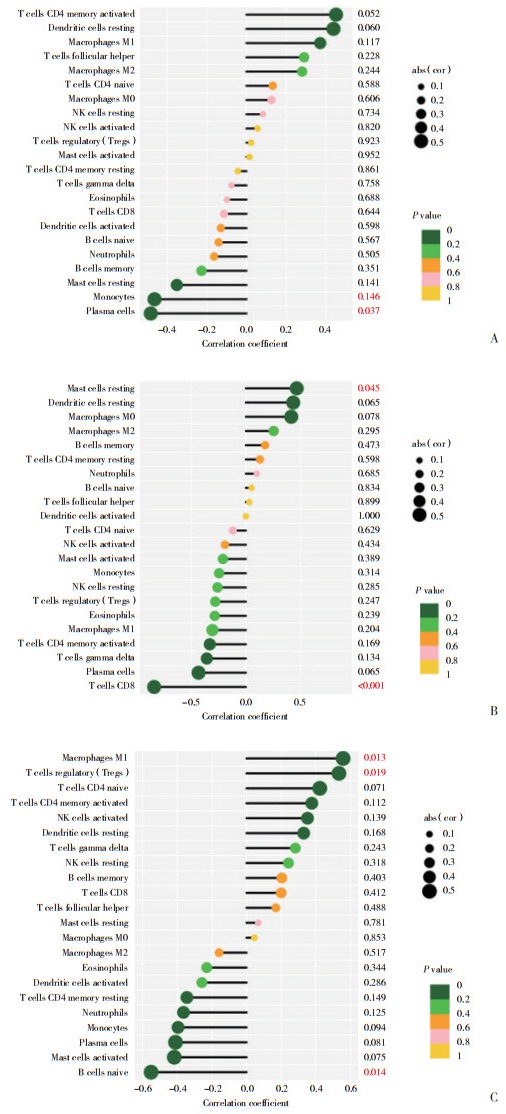
|
| A, CSN1S1; B, MTHFD2; C, NMNAT2. P < 0.05 Represents a significant correlation between immune cells and genes. 图 7 基因生物标志物与浸润性免疫细胞的相关性 Fig.7 Association of genetic biomarkers with invasive immune cells |
3 讨论
OA是一种慢性退行性疾病,常见于40岁以上人群[3],是致残的主要原因[4]。OA的病理特征是滑膜关节中关节软骨的局灶性缺失,与不同程度的骨赘形成、软骨下骨改变和滑膜炎相关[5]。虽然滑膜炎症与疼痛严重程度密切相关,但导致OA疼痛的分子机制尚不清楚。本研究通过生物信息学技术对比OA组与正常对照组后,共筛查出96个DEG。GO和DO富集分析显示,这些DEG涉及多种生物学进程、细胞组分、分子功能及疾病。
本研究中,基于LASSO与SVM这2种机器学习算法,还识别出CSN1S1、CXCL14、MTHFD2、NMNAT2、TLR7共5种特征基因。BROPHY等[6]通过生物信息学技术在OA和非OA样本之间鉴定出168个显著差异表达转录物,发现CSN1S1是OA半月板中升高较突出的转录物。LIU等[7]用激光捕获显微解剖1周龄小鼠胫骨关节软骨生长的浅层、中层和深层区域并分离软骨细胞,通过微阵列基因表达模式鉴定出中层关节软骨中含有CXCL14等分子标志物。ZHAO等[8]通过差异基因表达分析筛选出MTHFD2、TLR7、CX3CR1、SLC7A5、ARL4C 5种生物标志物,在软骨、软骨下骨和滑膜组织中表达差异显著,并证明使用这5种生物标志物通过逻辑回归合成的模型诊断OA的准确性优于单一生物标志物,可作为新型生物标志物提高膝OA诊断的准确度,改善临床疗效。HOSHIKAWA等[9]通过OA大鼠模型研究发现,滑膜组织释放的细胞外miR-21通过激活TLR7介导OA模型大鼠的膝关节疼痛,miR-21的镇痛作用可通过拮抗TLR7被阻断,TLR7拮抗剂对膝OA疼痛有持久的镇痛作用。TLR7参与免疫反应已在多种炎症性疾病中报道,OA患者的软骨组织和脂多糖诱导的软骨细胞中TLR7表达增加,TLR7下调可通过阻断p21介导的JAK2/STAT3途径,减弱软骨细胞的凋亡和损伤,表明TLR7可能是OA的一个治疗靶点[10]。研究[11]发现,TLR7通过不同免疫细胞、多种途径影响OA免疫微环境,并在OA患者中表达水平较高,可作为OA的诊断标志物。另有研究[12]通过SNaPshot测序技术证明,TLR7可能在中国汉族人群的膝OA中发挥易感性作用,并可能与膝OA的严重程度和膝OA积液滑膜炎的风险有关。
免疫细胞浸润分析结果显示,特征基因与单核细胞、巨噬细胞M1、浆细胞、CD8+T细胞、幼稚B细胞、调节性T细胞、静息肥大细胞等相关。单核细胞、巨噬细胞是健康关节的主要免疫细胞[13],参与调节骨转换,清除关节腔中的碎片,并与滑膜成纤维细胞一起形成保护屏障,是创伤后OA的治疗靶点。无论是巨噬细胞M1亚型还巨噬细胞M2亚型,均与OA的发病机制有关[14]。OA小鼠模型滑膜巨噬细胞M1极化部分通过分泌Rspo2和激活软骨细胞中的β-连环蛋白信号传导,导致小鼠OA恶化,提示巨噬细胞M1和Rspo2是OA的潜在治疗靶点[15-16]。果苷可通过抑制体外和体内巨噬细胞M1极化,降低白细胞介素(interleukin,IL)-6和TNF-α的分泌,延缓软骨和细胞外基质降解和软骨细胞肥大,从而减轻OA小鼠的滑膜炎症并延缓OA的发展。研究[17]显示,浆细胞和浆母细胞可产生如IL-17和IL-10等细胞因子,在自身免疫性疾病中发挥调节作用。另有研究[18]显示,富含血小板的血浆由于其软骨诱导特性和丰富的生长因子库而被广泛用于治疗软骨缺损和OA,临床上对OA患者有很明确的疗效。在OA发病过程中,CD8+T细胞被激活并在OA进展过程中扩增,抑制关节中TIMP-1的表达,从而延缓OA病情进展[19]。调节性T细胞通过促进抗炎反应之间的最佳平衡,产生免疫生理适应,包括减少全身促炎症介质产生,刺激由中性粒细胞介导的针对病原体的先天防御,即吞噬作用,实现最佳的先天反应[20]。肥大细胞可通过淋巴器官内的同源相互作用微调T细胞和B细胞活性,从而调节局部及全身炎症反应[21]。
综上所述,本研究发现,OA发病可能是通过调节CSN1S1、CXCL14、MTHFD2、NMNAT2、TLR7等特征基因表达,参与单核细胞、巨噬细胞M1、浆细胞、CD8+T细胞、幼稚B细胞、调节性T细胞、静息肥大细胞等实现。本研究存在数据库样本数量少、未行细胞分子学实验验证等缺点,可能存在误差。总之,本研究应用生物信息学技术探讨了OA发病的相关分子机制,为今后OA的治疗提供了新的靶点视角。
| [1] |
O'NEILL TW, MCCABE PS, MCBETH J. Update on the epidemiology, risk factors and disease outcomes of osteoarthritis[J]. Best Pract Res Clin Rheumatol, 2018, 32(2): 312-326. DOI:10.1016/j.berh.2018.10.007 |
| [2] |
JOHNSON VL, HUNTER DJ. The epidemiology of osteoarthritis[J]. Best Pract Res Clin Rheumatol, 2014, 28(1): 5-15. DOI:10.1016/j.berh.2014.01.004 |
| [3] |
COPE PJ, OURRADI K, LI Y, et al. Models of osteoarthritis: the good, the bad and the promising[J]. Osteoarthritis Cartilage, 2019, 27(2): 230-239. DOI:10.1016/j.joca.2018.09.016 |
| [4] |
VINA ER, KWOH CK. Epidemiology of osteoarthritis: literature update[J]. Curr Opin Rheumatol, 2018, 30(2): 160-167. DOI:10.1097/BOR.0000000000000479 |
| [5] |
DIEPPE PA, LOHMANDER LS. Pathogenesis and management of pain in osteoarthritis[J]. Lancet, 2005, 365(9463): 965-973. DOI:10.1016/S0140-6736(05)71086-2 |
| [6] |
BROPHY RH, ZHANG B, CAI L, et al. Transcriptome comparison of meniscus from patients with and without osteoarthritis[J]. Osteoarthritis Cartilage, 2018, 26(3): 422-432. DOI:10.1016/j.joca.2017.12.004 |
| [7] |
LIU JC, CHAU M, CHEN WP, et al. Spatial regulation of gene expression during growth of articular cartilage in juvenile mice[J]. Pediatr Res, 2015, 77(3): 406-415. DOI:10.1038/pr.2014.208 |
| [8] |
ZHAO YD, XIA Y, KUANG GY, et al. Cross-tissue analysis using machine learning to identify novel biomarkers for knee osteoarthritis[J]. Comput Math Methods Med, 2022, 2022: 9043300. DOI:10.1155/2022/9043300 |
| [9] |
HOSHIKAWA N, SAKAI A, TAKAI S, et al. Targeting extracellular miR-21-TLR7 signaling provides long-lasting analgesia in osteoarthritis[J]. Mol Ther Nucleic Acids, 2020, 19: 199-207. DOI:10.1016/j.omtn.2019.11.011 |
| [10] |
LIU D, LIU W, JIANG L, et al. Silencing of TLR7 protects against lipopolysaccharide-induced chondrocyte apoptosis and injury by blocking the p21-mediated JAK2/STAT3 pathway[J]. Am J Transl Res, 2021, 13(12): 13555-13566. |
| [11] |
HU Y, WU YT, GAN F, et al. Identification of potential therapeutic target genes in osteoarthritis[J]. Evid Based Complement Alternat Med, 2022, 2022: 8027987. DOI:10.1155/2022/8027987 |
| [12] |
XI XT, MEHMOOD A, NIU PY, et al. Association of X-linked TLR-7 gene polymorphism with the risk of knee osteoarthritis: a case-control study[J]. Sci Rep, 2022, 12(1): 7243. DOI:10.1038/s41598-022-11296-4 |
| [13] |
HAUBRUCK P, PINTO MM, MORADI B, et al. Monocytes, macrophages, and their potential niches in synovial joints -therapeutic targets in post-traumatic osteoarthritis?[J]. Front Immunol, 2021, 12: 763702. DOI:10.3389/fimmu.2021.763702 |
| [14] |
LIU B, ZHANG M, ZHAO J, et al. Imbalance of M1/M2 macrophages is linked to severity level of knee osteoarthritis[J]. Exp Ther Med, 2018, 16(6): 5009-5014. DOI:10.3892/etm.2018.6852 |
| [15] |
ZHANG HY, LIN CX, ZENG C, et al. Synovial macrophage M1 polarisation exacerbates experimental osteoarthritis partially through R-spondin-2[J]. Ann Rheum Dis, 2018, 77(10): 1524-1534. DOI:10.1136/annrheumdis-2018-213450 |
| [16] |
WANG H, ZHANG HY, FAN K, et al. Frugoside delays osteoarthritis progression via inhibiting miR-155-modulated synovial macrophage M1 polarization[J]. Rheumatology (Oxford), 2021, 60(10): 4899-4909. DOI:10.1093/rheumatology/keab018 |
| [17] |
WANG AA, GOMMERMAN JL, ROJAS OL. Plasma cells: from cytokine production to regulation in experimental autoimmune encephalomyelitis[J]. J Mol Biol, 2021, 433(1): 166655. DOI:10.1016/j.jmb.2020.09.014 |
| [18] |
VINOD E, AMIRTHAM SM, KACHROO U, et al. Articular chondroprogenitors in platelet rich plasma for treatment of osteoarthritis and osteochondral defects in a rabbit knee model[J]. Knee, 2021, 30: 51-62. DOI:10.1016/j.knee.2021.03.010 |
| [19] |
HSIEH JL, SHIAU AL, LEE CH, et al. CD8+ T cell-induced expression of tissue inhibitor of metalloproteinses-1 exacerbated osteoarthritis[J]. Int J Mol Sci, 2013, 14(10): 19951-19970. DOI:10.3390/ijms141019951 |
| [20] |
GÁLVEZ I, TORRES-PILES S, ORTEGA E. Effect of mud-bath therapy on the innate/inflammatory responses in elderly patients with osteoarthritis: a discussion of recent results and a pilot study on the role of the innate function of monocytes[J]. Int J Biometeorol, 2020, 64(6): 927-935. DOI:10.1007/s00484-019-01748-4 |
| [21] |
CARDAMONE C, PARENTE R, FEO GD, et al. Mast cells as effector cells of innate immunity and regulators of adaptive immunity[J]. Immunol Lett, 2016, 178: 10-14. DOI:10.1016/j.imlet.2016.07.003 |
 2023, Vol. 52
2023, Vol. 52