文章信息
- 王潇, 杨智勇
- WANG Xiao, YANG Zhiyong
- 基于GEO数据库的肥厚型心肌病差异表达基因分析
- Identification of differentially expressed genes of hypertrophic cardiomyopathy based on the Gene Expression Omnibus database
- 中国医科大学学报, 2023, 52(4): 313-317
- Journal of China Medical University, 2023, 52(4): 313-317
-
文章历史
- 收稿日期:2022-11-30
- 网络出版时间:2023-04-12 14:01:06
肥厚型心肌病是一种以心室壁和室间隔不对称性肥厚为主要特征的心肌疾病,多见于青壮年。目前认为肥厚型心肌病可能是一种常染色体显性遗传病,由于心室腔变小,左心室血液充盈受阻,可导致心悸、疲劳、劳力性呼吸困难、胸闷胸痛、晕厥甚至猝死等,是青年人运动性猝死的原因之一[1]。
目前,对于肥厚型心肌病的治疗以改善症状、减少并发症和预防猝死为主要目标,内科治疗主要通过应用β受体阻滞剂、非二氢吡啶类钙通道阻滞剂等增加心室充盈时间来减轻和缓解症状。对于室间隔过度肥厚、药物治疗无效、存在重度左心室流出道梗阻的患者,可行室间隔切除术;对于无法行室间隔切除术的患者,还可考虑行酒精室间隔消融术,以减轻左心室流出道与主动脉峰值的压力阶差,从而缓解梗阻[2-3]。但是,以上治疗手段可能导致心肌梗死、高度房室传导阻滞、急性二尖瓣关闭不全或室间隔穿孔等并发症,部分患者预后较差,可进展为终末期心力衰竭,还可能出现心房颤动、栓塞等。因此,寻找新的诊断和治疗手段是临床工作的关键[4-5]。
随着表观遗传学研究的不断深入,从基因水平探究疾病的相关机制受到越来越多研究者的关注。肥厚性心肌病具有复杂的遗传学病因,目前相关研究证实了能量代谢障碍、钙离子代谢紊乱、线粒体功能异常等均是肥厚型心肌病的分子遗传学病因[6-8]。基因表达综合(Gene Expression Omnibus,GEO)数据库是一个存储多种生物信息的信息管理系统,是一个在线的基因表达数据库,可以用于检索任何物种来源的遗传信息,通过提供基因表达测量数据信息平台,使更多的研究者能够进行更深入的数据挖掘。本研究采用GSE143786和GSE148602基因芯片,通过R语言对关键差异表达基因(differentially expressed gene,DEG)进行筛选和深度挖掘,并对其相关通路进行研究,通过肥厚型心肌病的DEG及其作用通路为临床诊断和治疗提供新思路。
1 材料与方法 1.1 数据来源以“hypertrophic cardiomyopathy”为关键词在GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)中检索肥厚型心肌病的基因芯片,选取GSE68316和GSE148602基因芯片,该芯片样本类型均为人类。GSE68316数据集中包含肥厚型心肌病7例和正常对照者5例,GSE148602数据集中包含肥厚型心肌病15例和正常对照者7例。
1.2 数据处理和DEG筛选应用GEO数据库在线分析工具GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/),定义分组后,分析、筛选各数据集中的DEG,以|log2FC|≥1和P < 0.05作为筛选标准筛选出DEG后,利用R语言,通过RStudio软件绘制火山图和热图。
1.3 京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)和基因本体论(gene ontology,GO)富集分析利用DAVID在线数据库(https://david.ncifcrf.gov)对DEG进行KEGG富集分析,包括生物过程、细胞组分和分子功能。利用R语言,通过RStudio软件实现KEGG和GO结果的可视化。
2 结果 2.1 DEG的筛选根据GSE68316和GSE148602芯片的分析结果绘制火山图(图 1)。在GSE68316数据集中共筛选出247个DEG,包括125个表达上调的DEG和122个表达下调的DEG;在GSE148602数据集中共筛选出157个DEG,包括44个表达上调的DEG和113个表达下调的DEG。
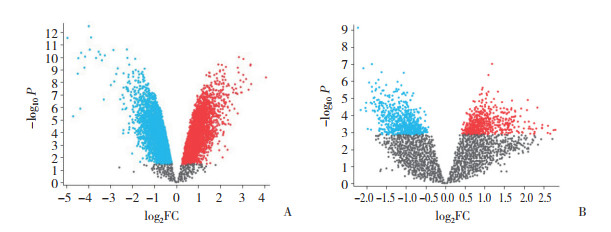
|
| A, GSE68316;B, GSE148602. 图 1 GSE68316和GSE148602中DEG的火山图 Fig.1 Volcano maps of DEGs in GSE68316 and GSE148602 |
2.2 显著上调和下调DEG的热图
分别取GSE68316和GSE148602芯片中差异表达最显著的上调和下调基因各10个,绘制这些基因的热图,见图 2。
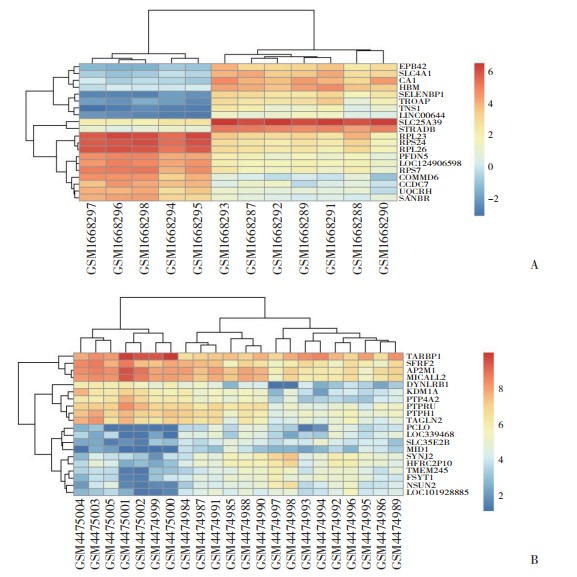
|
| A, GSE68316;B, GSE148602. 图 2 GSE68316和GSE148602中显著上调和下调DEG的热图 Fig.2 Heat maps of DEGs in GSE68316 and GSE148602 |
2.3 DEG的KEGG通路富集分析
取GSE68316和GSE148602基因芯片中所有有意义的DEG,共404个,进行KEGG分析。取主要富集通路(图 3),结果显示,主要在黏着斑激酶、免疫球蛋白Fc受体介导的吞噬作用、肌动蛋白细胞骨架的调节、多能干细胞调节的信号通路、泛素介导的蛋白质水解、感染、吞噬作用、沙门菌、谷胱甘肽的代谢、胆碱代谢等通路中存在富集。
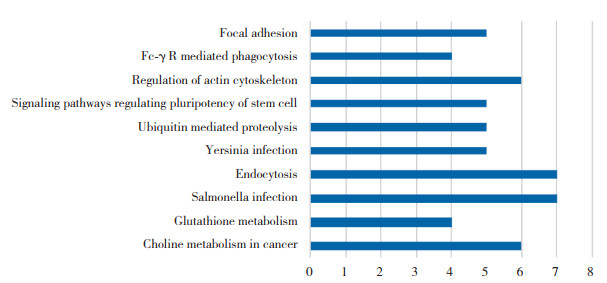
|
| 图 3 DEG的KEGG通路富集分析 Fig.3 KEGG pathway enrichment analysis of DEGs |
2.4 GO功能富集分析
取2个数据集中共同的DEG,共404个,进行GO分析。取其中前10个主要富集通路(图 4),结果显示,在生物过程中,主要在胞内蛋白质的靶向调控、定位调节和修饰以及糖代谢等过程中存在显著富集;在细胞组分中,主要在胞质内黏着斑激酶、细胞核及核质、细胞质核周区、刷状缘、皮质细胞骨架、肌动蛋白细胞骨架中存在显著富集;在分子功能中,主要在蛋白间的相互作用中存在显著富集,如肌动蛋白、钙黏蛋白、小分子GTP酶、低密度脂蛋白颗粒受体与其他蛋白的结合、转录激活因子、RNA聚合酶的活性改变等。
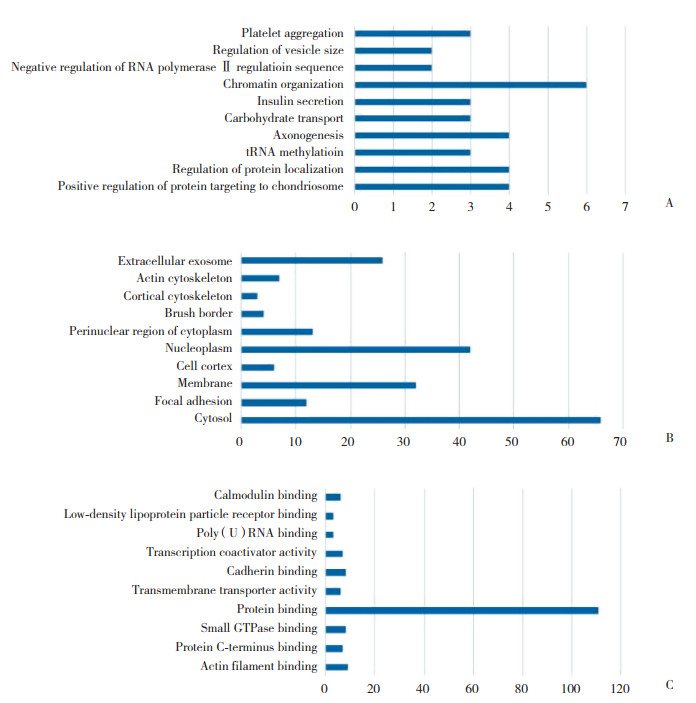
|
| A, biological process; B, cellular component; C, molecular function. 图 4 DEG的GO功能富集分析 Fig.4 GO enrichment analysis of DEGs |
3 讨论
肥厚型心肌病的发病机制较为复杂,其具体发病机制亟待研究。但肥厚型心肌病作为一种常染色体显性遗传病,应该从基因层面提示其发病机制。因此,从基因学和通路层面研究肥厚型心肌病的具体发病机制是目前的研究热点。
本研究通过对2个不同的基因数据集进行综合分析,发现在编码激动蛋白肌丝的基因(如MYH9、MYO1C、MYO9R等)中存在较多差异表达。本研究中,KEGG分析发现肌动蛋白细胞骨架的调节,GO分析发现钙黏蛋白结合相关通路中存在较多富集,由此推测肥厚型心肌病的发病机制可能与肌动蛋白的编码基因变异及其下游通路和蛋白表达发生改变相关。目前认为,编码心肌肌节的基因变异是各种心肌病的基础病因,了解其致病基因及其对下游通路的影响是研究的关键,有望成为心肌病治疗的靶点[9]。由于肌动蛋白相关基因的突变会影响其轻链间的相互作用,进而对ATP水解产生影响,无法使化学能转换为机械能。同时也会影响肌动蛋白与其他蛋白的相互联系,影响与钙离子的亲和力,使其对钙离子的敏感性出现异常,从而使ATP酶活性降低,影响蛋白激酶A的磷酸化,使横桥无法在心肌舒张期脱离下来,导致心肌肥厚的形成[10]。
本研究发现,小分子RNA在DEG中占较大比例,小分子RNA是一种内源性非编码单链RNA,它可以通过与mRNA以碱基互补配对的方式结合,发挥调控基因表达的作用以及影响转录翻译过程等。近年来,关于小分子RNA在心肌肥厚等方面中作用的研究越来越多,研究[11]发现,小分子RNA在心肌肥厚调节方面发挥重要的生物学效应。事实上,小分子RNA主要在基因调控方面起作用。本研究中,GO分析发现,tRNA甲基化、RNA聚合酶Ⅱ调节区序列特异性DNA结合区的负调控、RNA聚合酶的活性调节等通路中均存在较多富集,由此推测小分子RNA在肥厚型心肌病中的作用。OHBA等[12]通过利用靶向TRPC的小干扰RNA转染小鼠心肌细胞,结果发现,可阻止TRPC1表达诱导的心肌细胞肥大。CINGALANI等[13]将RNA干扰导入大鼠心肌细胞,通过活体和体外培养下调L型钙通道的β亚基,发现可以抑制L型钙通道,在不影响心肌细胞收缩的前提下减弱心肌细胞的肥大,最终证实小分子RNA参与了L型钙通道的调控。本研究中,根据KEGG和GO分析结果,发现在黏着斑激酶通路中均存在富集,该通路主要参与心肌细胞对压力变化的影响。CLEMENTE等[14]的研究中,通过导入特异性小干扰RNA使黏着斑激酶表达沉默,发现小鼠左心功能下降的情况变少,而肥厚的心肌细胞和成纤维细胞减少,表明黏着斑激酶通路在心肌细胞肥厚中存在一定影响,并且该通路可以受到小分子RNA的调节。
此外,线粒体相关基因突变、基因修饰和转录后调控、钾离子门控通道相关基因突变致肥厚型心肌病相关心律失常等也是目前提出的肥厚型心肌病相关机制,其具体过程尚不明确,仍需要大规模研究证实。本研究的GO分析结果发现在以上通路中存在富集,以期为肥厚型心肌病的治疗提供新策略。
| [1] |
苟永超, 储伟. 肥厚型心肌病左室肥厚合并预激疑似Fabry病1例[J]. 实用心电学杂志, 2020, 29(3): 226-228. DOI:10.13308/j.issn.2095-9354.2020.03.020 |
| [2] |
CHUMAKOVA OS. Hypertrophic cardiomyopathy in elderly: causes, diagnostic and treatment approaches[J]. Ter Arkh, 2020, 92(9): 63-69. DOI:10.26442/00403660.2020.09.000558 |
| [3] |
ARGIRÒ A, ZAMPIERI M, BERTEOTTI M, et al. Emerging medical treatment for hypertrophic cardiomyopathy[J]. J Clin Med, 2021, 10(5): 951. DOI:10.3390/jcm10050951 |
| [4] |
CHRISPIN J, MARINE JE. Atrial fibrillation and hypertrophic cardiomyopathy: more progress needed[J]. J Cardiovasc Electrophysiol, 2021, 32(3): 667-668. DOI:10.1111/jce.14874 |
| [5] |
PYDAH SC, MAUCK K, SHULTIS C, et al. Screening for hypertrophic cardiomyopathy[J]. JAAPA, 2021, 34(10): 23-27. DOI:10.1097/01.JAA.0000791464.54226.d8 |
| [6] |
RANJBARVAZIRI S, KOOIKER KB, ELLENBERGER M, et al. Altered cardiac energetics and mitochondrial dysfunction in hypertrophic cardiomyopathy[J]. Circulation, 2021, 144(21): 1714-1731. DOI:10.1161/CIRCULATIONAHA.121.053575 |
| [7] |
SZYGUŁA-JURKIEWICZ B, SZCZUREK-WASILEWICZ W, OSADNIK T, et al. Oxidative stress markers in hypertrophic cardiomyopathy[J]. Medicina (Kaunas), 2021, 58(1): 31. DOI:10.3390/medicina58010031 |
| [8] |
ZHANG JJ, WANG L, KAZMIERCZAK K, et al. Hypertrophic cardio-myopathy associated E22K mutation in myosin regulatory light chain decreases calcium-activated tension and stiffness and reduces myofi-lament Ca2+ sensitivity[J]. FEBS J, 2021, 288(15): 4596-4613. DOI:10.1111/febs.15753 |
| [9] |
VAN DER VELDEN J, STIENEN GJM. Cardiac disorders and pathophysiology of sarcomeric proteins[J]. Physiol Rev, 2019, 99(1): 381-426. DOI:10.1152/physrev.00040.2017 |
| [10] |
DVORNIKOV AV, SMOLIN N, ZHANG MJ, et al. Restrictive cardiomyopathy troponinⅠR145W mutation does not perturb myo-filament length-dependent activation in human cardiac sarcomeres[J]. J Biol Chem, 2016, 291(41): 21817-21828. DOI:10.1074/jbc.M116.746172 |
| [11] |
GRÄWE C, STELLOO S, VAN HOUT FAH, et al. RNA-centric methods: toward the interactome of specific RNA transcripts[J]. Trends Biotechnol, 2021, 39(9): 890-900. DOI:10.1016/j.tibtech.2020.11.011 |
| [12] |
OHBA T, WATANABE H, MURAKAMI M, et al. Upregulation of TRPC1 in the development of cardiac hypertrophy[J]. J Mol Cell Cardiol, 2007, 42(3): 498-507. DOI:10.1016/j.yjmcc.2006.10.020 |
| [13] |
CINGOLANI E, RAMIREZ CORREA GA, KIZANA E, et al. Gene therapy to inhibit the calcium channel beta subunit: physiological consequences and pathophysiological effects in models of cardiac hypertrophy[J]. Circ Res, 2007, 101(2): 166-175. DOI:10.1161/CIRCRESAHA.107.155721 |
| [14] |
CLEMENTE CF, TORNATORE TF, THEIZEN TH, et al. Targeting focal adhesion kinase with small interfering RNA prevents and reverses load-induced cardiac hypertrophy in mice[J]. Circ Res, 2007, 101(12): 1339-1348. DOI:10.1161/CIRCRESAHA.107.160978 |
 2023, Vol. 52
2023, Vol. 52




