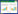文章信息
- 张娟, 刘依萍, 曹士盛, 李欣, 董晓曦, 李宏霄
- ZHANG Juan, LIU Yiping, CAO Shisheng, LI Xin, DONG Xiaoxi, LI Hongxiao
- 应用拉曼光谱技术对伴2型糖尿病慢性牙周炎患者龈下菌斑的研究
- Distinguishing subgingival plaques of patients with chronic periodontitis and type 2 diabetes mellitus based on Raman spectroscopy
- 中国医科大学学报, 2023, 52(12): 1113-1118
- Journal of China Medical University, 2023, 52(12): 1113-1118
-
文章历史
- 收稿日期:2023-01-19
- 网络出版时间:2023-12-07 11:15:08
2. 天津医科大学口腔医院牙周科,天津 300070;
3. 中国医学科学院生物医学工程研究所,天津 300192
2. Department of Periodontics, Hospital of Stomatology, Tianjin Medical University, Tianjin 300070, China;
3. Institute of Biomedical Engineering, Chinese Academy of Medical Science, Tianjin 300192, China
慢性牙周炎是由牙菌斑生物膜中的牙周致病菌引起的牙周组织慢性感染性疾病[1]。2型糖尿病是以胰岛素作用受损引起的高血糖为主要特征的代谢紊乱[2]。研究[3-4]表明,伴或不伴2型糖尿病的慢性牙周炎患者的龈下菌斑微生物水平存在差异。
拉曼光谱技术是一种非侵入性的分析技术,可检测生物流体、细胞和组织等复杂生物样品的化学成分。拉曼光谱技术与机器学习算法相结合,已广泛用于微生物学和医学研究[5-6]。本研究运用拉曼光谱技术获得伴或不伴2型糖尿病的慢性牙周炎患者及健康成人龈下菌斑的拉曼光谱数据,并通过机器学习算法构建分类模型,区分这3种类型受试者的龈下菌斑,验证龈下菌斑微生物在2型糖尿病中的监测预警功能,以期为2型糖尿病的早期发现提供新思路。
1 材料与方法 1.1 研究对象于2021年12月至2022年2月期间,在天津医科大学口腔医院、天津医科大学朱宪彝纪念医院招募志愿者,最终纳入受试者66例,包括确诊慢性牙周炎伴2型糖尿病患者20例(A组),确诊单纯慢性牙周炎(不伴2型糖尿病) 患者23例(B组),无2型糖尿病和慢性牙周炎的健康成人23例(C组)。纳入标准:(1) 年龄20~70岁;(2) 不限性别;(3) 口内天然牙≥14颗;(4) 3年内无吸烟史。排除标准:(1) 1型糖尿病或其他特殊类型糖尿病;(2) 伴有全身其他系统疾病,如高血压、心血管疾病、肝、肾功能异常、免疫疾病、骨质疏松症等;(3) 妊娠期或哺乳期;(4) 服用双磷酸盐、环孢霉素、苯妥英钠、硝苯地平、钙通道阻滞剂等药物;(5) 3个月内接受过牙周治疗、抗生素治疗或使用含有抗菌素的漱口水。慢性牙周炎纳入标准:(1) 口内天然牙≥14颗;(2) 临床附着丧失(clinical attachment loss,CAL) ≥1 mm牙齿占受检牙齿的30%以上。2型糖尿病诊断标准符合1999年WHO糖尿病诊断标准,已确诊≥1年,近3个月服药无变化。本研究获得天津医科大学伦理委员会批准(编号:TMUhMEC2019047),所有受试者均签署知情同意书。
1.2 研究方法 1.2.1 牙周检查使用UNC-15牙周探针(豪孚迪Hu-Friedy公司,美国) 检查并记录所有研究对象口内余留牙,每颗牙记录6个位点(近颊、正中、远颊、近舌、正中、远舌) 的牙周探诊深度(probing depth,PD)和CAL,记录每颗牙颊舌面的牙龈出血指数(bleeding index,BI) 和菌斑指数(plaque index,PLI)。
1.2.2 指数牙的龈下菌斑获取所有研究对象选取6颗指数牙16、11、26、31、36、46 (若指数牙缺失则以该区段其他牙齿代替)。用无菌棉球擦去龈上软垢,使用消毒后的Gracey刮治器(豪孚迪Hu-Friedy公司,美国) 于每颗牙刮取6个位点(近颊、正中、远颊、近舌、正中、远舌) 的龈下菌斑,置于装有1.5 mL PBS (北京索莱宝科技有限公司,中国) 的微量离心管中,冻存于-80 ℃备用。
1.2.3 拉曼光谱数据采集(1) 将奥谱天成拉曼光谱仪(ATR3110-785,奥谱天成公司,中国) 与电脑连接,显示在线状态;(2) 调整拉曼光谱仪参数,激光波长785 nm,激光强度200 mW,积分时间10 s,积分次数1次,光谱扫描范围200~2 600 cm-1,擦镜纸擦拭拉曼光谱仪光纤探头;(3) 使用校准样品乙腈作为测量样品,得到正确光谱曲线数据,确认可使用;(4) 对装在1.5 mL微量离心管中的菌斑样本进行测量(注意使光纤探头正对菌斑样本),每个样本重复测量10次。
1.3 统计学分析采用SPSS 25.0统计软件进行数据分析,计数资料采用Fisher’s精确检验或Pearson χ2检验比较;计量资料以x±s表示,2组比较采用独立样本t检验,3组比较采用单因素方差分析。P < 0.05为差异有统计学意义。
1.4 机器学习算法本研究使用拉曼光谱仪自带的扣除暗底功能,将扣除暗底基线平滑后的原始数据纳入研究。应用Auto-Sklearn工具包[7],使用8种机器学习算法构建模型,算法包括极限树(extra trees,ET)、自适应提升(AdaBoost,ADA)、梯度提升(gradient boosting,GB)、线性判别分析(linear discriminant analysis,LDA)、支持向量机(support vector machine,SVM)、多层感知器(multi-layer perceptron,MLP)、被动攻击分类器(passive-aggressive classifier,PAC) 和二次判别分析(quadratic discriminant analysis,QDA)。
本研究共得到20个A组龈下菌斑样本(200个光谱)、23个B组龈下菌斑样本(230个光谱) 和23个C组龈下菌斑样本(230个光谱) 3个数据集。验证过程采用3折交叉验证[8],3个数据集中的每一个均按照龈下菌斑样本编号随机划分为3个大小近似相等的子集,3次训练和验证过程中,每次都有3个类别的各1个子集作为验证集,其余2个子集作为训练集,重复3次后,每个子集都作为1次验证集和2次训练集参与了整个交叉验证过程。样本的类别标签由其10个光谱的预测标签采用多数投票制决定。
2 结果 2.1 3组一般特征比较如表 1所示,3组年龄比较有统计学差异(P < 0.05);3组性别比较无统计学差异(P = 0.11);A组与B组体质量指数(body mass index,BMI) 比较无统计学差异(P = 0.06),A组、B组与C组BMI比较有统计学差异(P < 0.01)。
| Group | Age (year) | Sex (male/female) | BMI (kg/m2) |
| A | 54.00±11.63 | 15/5 | 25.97±4.31 |
| B | 35.09±9.41) | 14/9 | 23.91±3.82 |
| C | 25.26±1.661),2) | 10/13 | 19.95±1.881),2) |
| F/χ2 | 62.11 | 4.44 | 17.07 |
| P | < 0.01 | 0.11 | < 0.01 |
| BMI,body mass index. 1) compare with group A,P < 0.05;2) compare with group B,P < 0.05. | |||
2.2 3组牙周状况比较
如表 2所示,A组与B组PD比较无统计学差异(P = 0.14),A组、B组与C组比较有统计学差异(P < 0.05);A组与B组CAL比较有统计学差异(P < 0.05);A组与B组BI比较无统计学差异(P = 0.52),A组、B组与C组比较有统计学差异(P < 0.05);A组与B组PLI比较无统计学差异(P = 0.95),A组、B组与C组比较有统计学差异(P < 0.05)。
| Group | PD (mm) | CAL (mm) | BI | PLI |
| A | 3.06±0.77 | 3.08±1.11 | 2.96±0.40 | 2.04±0.37 |
| B | 3.35±0.75 | 1.63±0.531) | 2.87±0.60 | 2.05±0.49 |
| C | 2.37±0.231),2) | - | 2.37±0.421),2) | 1.56±0.391),2) |
| F | 14.91 | 31.32 | 9.46 | 9.82 |
| P | 0.001 | < 0.001 | 0.001 | 0.001 |
| PD,probing depth;CAL,clinical attachment loss;BI,bleeding index;PLI,plaque index. 1) compare with group A,P < 0.05;2) compare with group B,P < 0.05. | ||||
2.3 3组龈下菌斑的拉曼光谱分析
如图 1所示,3组龈下菌斑的平均拉曼光谱中,较为明显的拉曼高峰为代表鸟嘌呤的397 cm-1[9],代表蛋白质S-S二硫键拉伸的524 cm-1[10],代表磷酸盐离子相互作用的806/807 cm-1[10],代表脯氨酸和羟脯氨酸C-C拉伸的839 cm-1[10],代表蛋白质和核酸的υ (C-C) 摆动的971 cm-1[10],代表脂质υ (C-C) 的1 151 cm-1[10],代表核酸嘌呤碱基中CH3/CH2摆动模式的1 328 cm-1[10],代表核酸模式的1 458 cm-1[10]。
|
| 图 1 3组龈下菌斑的平均拉曼光谱 Fig.1 Average Raman spectra of the three groups of subgingival plaques |
2.4 机器学习算法鉴别3组龈下菌斑 2.4.1 三分类的最优模型
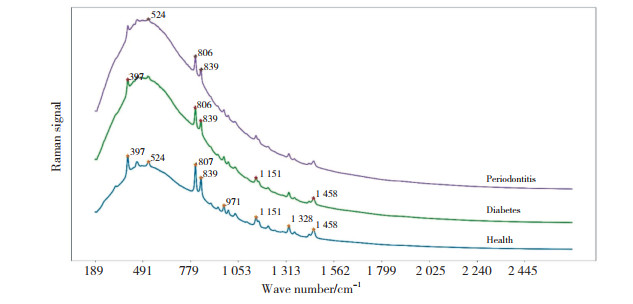
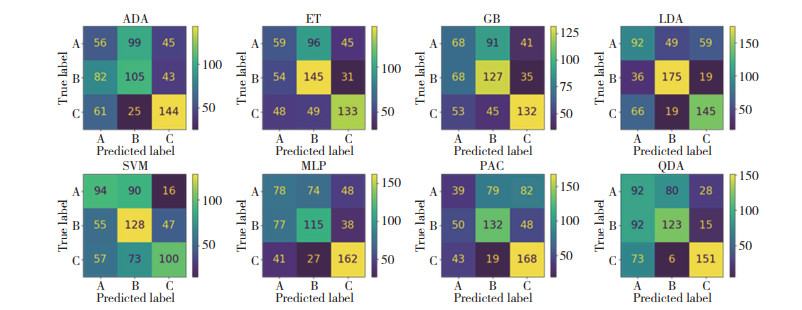
图 2所示为测试的8种算法模型在光谱级别的3折交叉验证结果。该准确率是3组子集分别作为验证集时的准确率均值。区分3组龈下菌斑在光谱级别的最优模型是LDA,准确率为62.5%。图 3所示为测试的8种算法模型在样本级别的3折交叉验证准确率,最优模型是LDA,准确率为60.6%。
|
| ADA, AdaBoost; ET, extra trees; GB, gradient boosting; LDA, linear discriminant analysis; SVM, support vector machine; MLP, multi-layer perceptron; PAC, passive-aggressive classifier; QDA, quadratic discriminant analysis. The values in each cell indicate the number of spectrums. 图 2 测试的8种算法模型在光谱级别的3折交叉验证结果汇总的混淆矩阵 Fig.2 Confusion matrix of the 3-fold cross-validation results at the spectral level for the eight tested models |
|
| The values in each cell indicate the number of samples. 图 3 测试的8种算法模型在样本级别的3折交叉验证结果汇总的混淆矩阵 Fig.3 Confusion matrix of the 3-fold cross-validation results at the sample level for the eight tested models |
2.4.2 二分类的最佳模型
为了探索任意2组龈下菌斑拉曼光谱之间的差异,分别在3组二分类数据上训练了8个机器学习算法模型。图 4中条形图显示了分类器在光谱级别和样本级别区分2组(A组vs. B组、A组vs. C组、B组vs. C组) 龈下菌斑的准确率。在光谱级别,3组二分类数据的最佳模型分别来自LDA算法(A组vs. B组,准确率75.7%),ET和SVM算法(A组vs. C组,准确率70.9%),LDA算法(B组vs. C组,准确率86.9%)。在样本级别,3组二分类数据的最佳模型分别来自LDA算法(A组vs. B组,准确率79.3%),ET和QDA算法(A组vs. C组,准确率74.2%) 和LDA算法(B组vs. C组,准确率87.2%)。8个模型中有7个模型区分“B组vs. C组”比区分“A组vs. C组”准确率更高,区分“A组vs. C组”比区分“A组vs. B组”准确率更高。
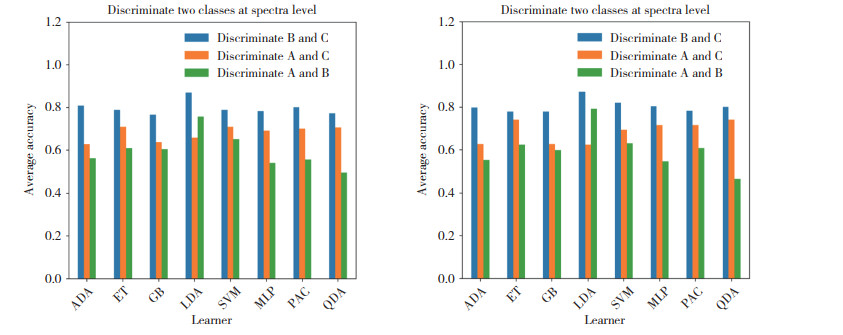
|
| The horizontal axis represents learner algorithms. The vertical axis represents accuracy. The labels in the legend display the two discriminated classes. 图 4 机器学习算法在样本或光谱级别区分龈下菌斑的准确率 Fig.4 Accuracy rate of machine learning algorithm in differentiating subgingival plaque at the sample or spectral level |
3 讨论
2型糖尿病可能会在一定程度上影响龈下牙周致病菌的水平,与健康对照组相比,2型糖尿病患者的龈下菌斑样本中牙周致病菌的检出率更高[4],2型糖尿病患者比单纯慢性牙周炎患者具有更多龈下菌斑细菌计数[3],龈下菌斑微生物具有提示2型糖尿病的潜力。
既往对不同人群龈下菌斑的研究常用常规和实时PCR及16s rRNA基因测序技术,需要复杂的样品制备、昂贵的实验试剂和很高的技术要求。本研究使用拉曼光谱技术结合机器学习算法构建模型,可区分不同人群的龈下菌斑,不需要复杂的样本制备及昂贵的试剂,甚至可以椅旁操作,具有简便、快速的优点。
本研究应用拉曼光谱技术结合机器学习算法构建模型,对伴或不伴2型糖尿病的慢性牙周炎患者及健康成人的龈下菌斑进行区分,准确率最高达62.5% (LDA算法)。当本研究对3组龈下菌斑进行两两比较时,模型分类准确率有了一定的提高,模型分类准确率最高可达70.9%~87.2% (LDA算法和ET算法)。如图 4所示,在3组龈下菌斑的二分类模型中,8个模型中有7个模型区分单纯慢性牙周炎和健康成人的准确率比区分慢性牙周炎伴2型糖尿病和健康成人更高,区分慢性牙周炎伴2型糖尿病和健康成人的准确率比区分慢性牙周炎伴2型糖尿病和单纯慢性牙周炎更高。说明本研究中伴或不伴2型糖尿病慢性牙周炎患者的龈下菌斑差异要小于健康成人和慢性牙周炎患者的龈下菌斑差异。本研究发现,LDA算法在三分类模型和二分类模型中分类准确率高且性能良好,推荐用于类似的鉴别工作。
综上所述,拉曼光谱技术与机器学习算法结合的方案在识别3种人群龈下菌斑和探究不同人群龈下菌斑成分方面显示出广阔的发展前景,未来可作为筛查或诊断工具与临床实践相结合。该模型还具有改进的空间,今后可通过增加样本量,丰富训练和测试数据,在更大的人群筛查中进行2型糖尿病诊断时,将获得更高的灵敏度和特异度。
| [1] |
CHANG Y, LEE JS, LEE KJ, et al. Improved oral hygiene is associated with decreased risk of new-onset diabetes: a nationwide population-based cohort study[J]. Diabetologia, 2020, 63(5): 924-933. DOI:10.1007/s00125-020-05112-9 |
| [2] |
BALMASOVA IP, LOMAKIN YA, BABAEV EA, et al. Shielding of cytokine induction by the periodontal microbiome in patients with periodontitis associated with type 2 diabetes mellitus[J]. Acta Naturae, 2019, 11(4): 79-87. DOI:10.32607/20758251-2019-11-4-79-87 |
| [3] |
MONTEVECCHI M, VALERIANI L, GATTO MR, et al. Subgingival pathogens in chronic periodontitis patients affected by type 2 diabetes mellitus: a retrospective case-control study[J]. J Periodontal Implant Sci, 2021, 51(6): 409. DOI:10.5051/jpis.2100180009 |
| [4] |
AL-OBAIDA MI, AL-NAKHLI AM, ARIF IA, et al. Molecular identification and diversity analysis of dental bacteria in diabetic and non-diabetic females from Saudi Arabia[J]. Saudi J Biol Sci, 2020, 27(1): 358-362. DOI:10.1016/j.sjbs.2019.10.005 |
| [5] |
PEZZOTTI G. Raman spectroscopy in cell biology and microbiology[J]. J Raman Spectrosc, 2021, 52(12): 2348-2443. DOI:10.1002/jrs.6204 |
| [6] |
KONG K, KENDALL C, STONE N, et al. Raman spectroscopy for medical diagnostics — from in-vitro biofluid assays to in-vivo cancer detection[J]. Adv Drug Deliv Rev, 2015, 89: 121-134. DOI:10.1016/j.addr.2015.03.009 |
| [7] |
HUTTER F, KOTTHOFF L, VANSCHOREN J. Automated machine learning: methods, systems, challenges[M]. Cham: Springer, 2019.
|
| [8] |
WONG TT, YEH PY. Reliable accuracy estimates from k-fold cross validation[J]. IEEE Trans Knowl Data Eng, 2020, 32(8): 1586-1594. DOI:10.1109/TKDE.2019.2912815 |
| [9] |
DE GELDER J, DE GUSSEM K, VANDENABEELE P, et al. Refe-rence database of Raman spectra of biological molecules[J]. J Raman Spectrosc, 2007, 38(9): 1133-1147. DOI:10.1002/jrs.1734 |
| [10] |
MOVASAGHI Z, REHMAN S, REHMAN IU. Raman spectroscopy of biological tissues[J]. Appl Spectrosc Rev, 2007, 42(5): 493-541. DOI:10.1080/05704920701551530 |
 2023, Vol. 52
2023, Vol. 52