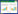文章信息
- 刘虹汝, 宁静华, 张鑫, 赵严红, 屈润, 张钰哲
- LIU Hongru, NING Jinghua, ZHANG Xin, ZHAO Yanhong, QU Run, ZHANG Yuzhe
- m6A RNA甲基化调节因子与前列腺癌预后的关系
- Relationship between m6A RNA methylation regulators and prognosis of prostate cancer
- 中国医科大学学报, 2023, 52(12): 1098-1105
- Journal of China Medical University, 2023, 52(12): 1098-1105
-
文章历史
- 收稿日期:2023-03-27
- 网络出版时间:2023-12-07 13:10:57
2. 云南抗病原药用植物筛选重点实验室,云南 大理 671000;
3. 云南省昆虫生物医药重点实验室,云南 大理 671000
2. Yunnan Key Laboratory of Antipathogenic Medicinal Plant Screening, Dali 671000, China;
3. Yunnan Provincial Key Laboratory of Insect Biomedicine, Dali 671000, China
前列腺癌是指发生在前列腺上皮的恶性肿瘤,在全球男性肿瘤统计报告中位居第2位,每年有超过120万例新诊断病例,相关死亡人数超过35万[1-5]。影响前列腺癌发病的风险因素包括年龄、遗传、高脂饮食、种族、地区、宗教信仰等。迄今为止,前列腺癌的发病机制仍未完全阐明,全基因组DNA测序、mRNA测序、蛋白质组分析和前列腺特异性抗原筛查为前列腺癌分型及其病理学的遗传基础提供重要见解[6]。前列腺癌发病机制中表观遗传分子途径是目前的研究热点,研究认为,其表观遗传改变比遗传改变发生得更早、更频繁。全基因组关联研究已确定超过170个与前列腺癌发病率相关的单核苷酸多态性位点,单核苷酸多态性不仅可检测早发性和家族性前列腺癌,还可用于计算遗传风险评分,从而进行靶向筛查。同时,有研究[7]表明,微RNA可通过降解RNA或抑制蛋白质翻译来控制蛋白质表达,其将成为前列腺癌诊断、预后和治疗选择的有潜在价值的标志物,以及前列腺癌治疗的潜在药物。
N6-甲基腺嘌呤(N6-methyladenosine,m6A) 是真核细胞中最丰富的RNA修饰[8],目前已在所有生物体的RNA中鉴定出150多种转录后修饰。广泛的RNA碱基修饰统称为表观转录组,与翻译控制、RNA剪接和许多癌症类型有关。m6A甲基化由m6A甲基转移酶(包括METTL14、METTL3、KIAA1429、RBM15、ZC3H13和WTAP)、m6A去甲基酶(包括FTO和ALKBH5)、能与甲基化结合位点结合的特殊基
因(包括YTHDC1、YTHDC2、YTHDF1、YTHDF2和HNRNPC) 共同参与调控,其参与RNA代谢的诸多方面,如mRNA前体剪接、3’末端加工、出核转运、翻译调节、mRNA降解和非编码RNA加工等。m6A甲基化异常会导致肿瘤发生和发育迟缓等,并对基因表达、调控产生深远的影响[9]。虽然近年来m6A已成为肿瘤研究的热点,但m6A修饰在前列腺癌发生、发展中的分子机制仍未被深入探索。因此,本研究旨在探寻m6A RNA甲基化调节因子(以下简称m6A调节因子) 在前列腺癌组织中的差异表达情况,并探讨其与预后的关系。
1 材料与方法 1.1 数据下载和整理从癌症基因组图谱(The Cancer Genome Atlas,TCGA) 数据库下载416例前列腺癌组织样本和80例癌旁组织样本(共496例) 的相关RNA-seq转录组数据和相关临床病理数据,包括年龄、T分期(原发肿瘤)、N分期(淋巴结转移)、生存时间、生存状态。
1.2 m6A调节因子的筛选和比较本研究使用的12个m6A调节因子包括5个m6A甲基转移酶(METTL3、METTL14、WTAP、RBM15和ZC3H13),5个能与甲基化结合位点结合的特殊基因(YTHDC1、YTHDC2、YTHDF1、YTHDF2和HNRNPC),2个m6A去甲基酶(FTO和ALKBH5)。从TCGA数据库中下载这些调节因子在前列腺癌组织和癌旁组织中的mRNA表达量,使用R软件Pheatmap包以及Wilcoxon秩和检验,比较前列腺癌组织和癌旁组织中12个m6A调节因子的表达水平。
1.3 前列腺癌样本的无监督聚类分析使用R软件Consensus Cluster Plus包和χ2检验,对前列腺癌组织进行无监督聚类分组,确定前列腺癌样本分层聚类分析的最佳样本组数,绘制Kaplan-Meier生存曲线,分析各组间的预后差异,探究m6A调节因子与前列腺癌预后的潜在关联。
1.4 构建m6A调节因子的多因素Cox回归模型及绘制生存曲线通过R软件对m6A调节因子进行多因素Cox回归分析,筛选出差异表达的m6A调节因子,应用survival包绘制高风险组和低风险组的Kaplan-Meier生存曲线,评估高风险和低风险对前列腺癌患者总生存率(overall survival,OS) 的影响。
1.5 临床病理因素的风险评分和多因素Cox回归模型对临床病理因素(包括年龄、T分期、N分期) 进行风险评分,通过R软件survival包对临床病理因素和总风险评分进行多因素Cox回归分析,绘制森林图,判断临床病理因素和总风险评分能否作为独立的预后指标。
1.6 列线图分析使用library (rms) 函数包进行由年龄、分期和风险评分组成的列线图分析,用于预测患者2年、3年和5年的OS。
1.7 免疫组织化学染色采用免疫组织化学染色,检测包含前列腺癌组织样本(n = 20) 和癌旁组织样本(n = 20) 的组织芯片中METTL14和FTO蛋白表达水平。组织芯片经脱蜡、水化后,采用枸橼酸缓冲液高压修复抗原,冷却至常温。再用3% H2O2处理,避光封闭15 min;PBS洗涤3次,每次5 min;滴加山羊血清,37 ℃封闭30 min;4 ℃过夜,孵育兔抗人METTL14(EPR27234-62,中国Abcam公司) 和FTO (ab94482,中国Abcam公司) 单克隆抗体,稀释浓度为1∶100;PBS洗涤3次,每次5 min,滴加兔通用IgG二抗,37 ℃孵育30 min;PBS洗涤3次,滴加辣根过氧化酶标记的三抗,37 ℃孵育30 min;PBS洗涤3次后,DAB显色;流水冲洗15 min,苏木精复染1 min后,流水冲洗30 min,脱水封片,在光学显微镜下观察并采集图像。采用半定量方法评价METTL14和FTO蛋白表达情况。METTL14和FTO蛋白染色强度:阴性,0分;弱阳性,1分;中阳性,2分;强阳性,3分。METTL14和FTO蛋白阳性染色百分比:< 5%,0分;5%~ < 25%,1分;25%~ < 50%,2分;50%~ < 75%,3分;≥75%,4分。每个组织标本METTL14和FTO蛋白的染色积分为强度得分×百分比得分,总分为0~12分,其中 < 9分为低表达,≥9分为高表达。
2 结果 2.1 数据下载和m6A调节因子的筛选与比较本研究的496例前列腺癌患者中,年龄 < 45岁223例,≥45岁273例;T分期:T2 187例,T3 291例,T4 11例;N分期:N0 343例,N1 80例;生存时间:> 5年84例,≤5年412例;生存状态:死亡9例,存活487例。
416例癌组织样本和80例癌旁组织样本的m6A调节因子的差异表达分析结果显示,8个m6A调节因子的表达水平有统计学差异(P < 0.05)。其中ALKBH5、FTO、METTL14、YTHDC1和ZC3H13在癌组织中下调,HNRNPC、METTL3和YTHDF1在癌组织中上调(图 1A)。比较12个m6A调节因子在不同T分期癌组织中的表达水平,结果显示,不同分期时HNRNPC、YTHDC2、YTHDF1和YTHDF2的表达水平有统计学差异(P < 0.05),其中HNRNPC的表达量随前列腺癌的进展出现显著持续升高(图 1B)。将前列腺癌样本按淋巴结转移分为转移组(N1) 和无转移组(N0),分析2组间12个m6A调节因子的表达差异。结果发现,N1组HNRNPC、RBM15、YTHDF1和YTHDF2 mRNA的表达水平明显高于N0组(图 1C)。上述结果表明,m6A调节因子与前列腺癌的发生和进展存在潜在关联,对前列腺癌预后标志物的筛选具有研究价值。

|
| A, expression levels of m6A regulators in prostate cancer and adjacent normal tissues; B, expression levels of m6A regulators in prostate cancer samples at different T stages; C, expression levels of m6A regulators in prostate cancer samples grouped according to N stage. * P < 0.05;**P < 0.01;***P < 0.001. 图 1 前列腺癌样本中m6A调节因子的表达情况 Fig.1 Expression of m6A regulators in prostate cancer samples |
2.2 前列腺癌样本无监督聚类分析
根据8个差异表达的m6A调节因子的mRNA表达水平对前列腺癌样本进行无监督聚类分析,确定最佳样本聚类数为3 (图 2A)。采用层次聚类方法将前列腺癌样本分为3组,分别为cluster 1、cluster 2、cluster 3 (图 2B)。Kaplan-Meier生存分析结果显示,3组间OS的差异有统计学意义(P < 0.05),cluster 2的10年OS较cluster 1和cluster 3高,表明cluster 2有较好的预后(图 2C)。以上结果表明,m6A调节因子对前列腺癌聚类结果与前列腺癌预后存在潜在关联。
|
| A, unsupervised clustering was applied to prostate cancer samples and this determined that the optimal number of clusters was three; B, prostate cancer samples were divided into three groups by hierarchical clustering; C, cluster 2 had a higher overall survival than Clusters 1 and 3. 图 2 前列腺癌样本的聚类和预后分析 Fig.2 Clustering and prognostic analysis of prostate cancer samples |
2.3 构建m6A调节因子的多因素Cox回归模型及绘制生存曲线
构建多因素Cox比例风险模型,进一步筛选出与前列腺癌OS相关的m6A调节因子,其中METTL14和FTO的差异有统计学意义(P < 0.05)。根据风险评分的HR和95%CI,确定METTL14为前列腺癌独立高风险影响因素(图 3A)。根据中位风险评分将样本分为高风险组和低风险组,与低风险组相比,高风险组前列腺癌患者OS较低,差异有统计学意义(P < 0.05,图 3B)。

|
| A, METTL14 is an independent high-risk factor for prostate cancer; B, prostate cancer patients in the high-risk group had a poor prognosis. 图 3 m6A调节因子与前列腺癌预后的关联 Fig.3 Association between m6A regulators and prostate cancer prognosis |
2.4 风险评分与临床病理因素的关系及多因素Cox回归模型
对不同前列腺癌患者的临床病理因素(包括年龄、T分期和N分期) 进行风险评分,结果显示,前列腺癌风险与临床病理因素无关(图 4A~4C)。通过构建多因素Cox回归模型对前列腺癌患者的风险评分、年龄、T分期和N分期进行分析,结果显示,风险评分可作为前列腺癌的独立预后指标(P < 0.05,图 4 D)。

|
| A, prostate cancer risk is independent of age; B, prostate cancer risk is not related to T stage; C, prostate cancer risk is not related to N stage; D, the risk score is an independent prognostic indicator of prostate cancer. 图 4 风险评分与前列腺癌临床病理因素的关系 Fig.4 Association of risk score with clinicopathological factors in prostate cancer samples |
2.5 列线图的构建和验证
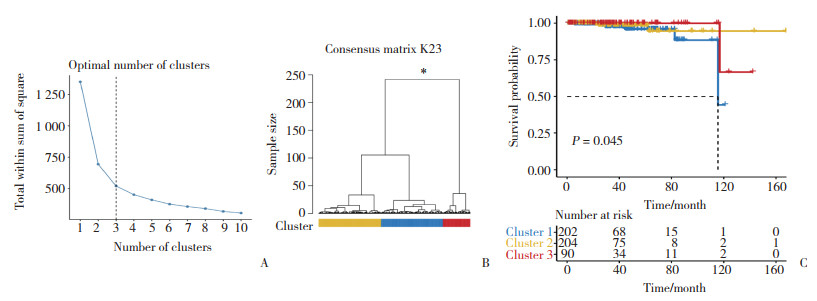
通过多变量Cox回归分析中包含的重要因素,即年龄、T分期、N分期和风险评分,发现列线图构建的模型能够较好地预测患者2、3和5年的OS (图 5)。
|
| 图 5 年龄、分期和风险评分组成的列线图预测前列腺癌患者2年、3年和5年的OS Fig.5 A nomogram of age, stage, and risk score predicts 2-, 3-, and 5-year OS for prostate cancer patients. |
2.6 免疫组织化学结果判断
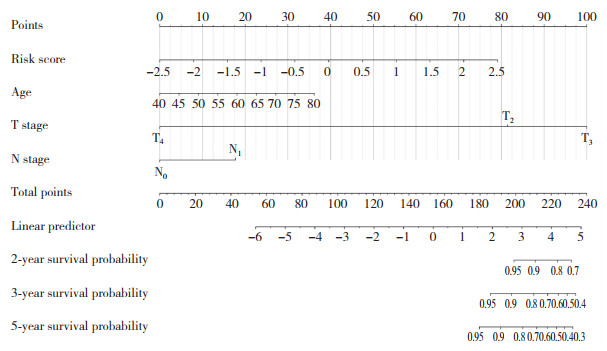
每张切片由2名病理科医师进行独立判断并评分,两者不一致时采取重新评估原则。每张玻片在高倍镜视野下进行多处观察,同一范围采100个细胞。结果显示,包含前列腺癌组织样本和癌旁组织样本的组织芯片中,METTL14和FTO蛋白阳性标记为棕黄色染色颗粒并弥漫分布于细胞核,前列腺癌组织与癌旁正常组织均表达METTL14和FTO。前列腺癌组织和癌旁正常组织中FTO蛋白阳性表达率分别为76% (76/100) 和28% (28/100),染色积分分别为12分(高表达) 和4分(低表达);METTL14蛋白阳性表达率分别为54% (54/100) 和11% (11/100),染色积分分别为9分(高表达) 和2分(低表达)。邻近正常组织中FTO和METTL14蛋白阳性表达率均明显低于前列腺癌组织(均P < 0.05)。见图 6。
|
| A, protein expression(×100) and statistical boxplot of FTO; B, protein expression(×100) and statistical boxplot of METTL14. * P < 0.05. 图 6 前列腺癌组织和癌旁正常组织中FTO和METTL14蛋白的表达 Fig.6 Expression of FTO and METTL14 protein in prostate cancer tissues and adjacent normal tissues |
3 讨论
在人类基因组表观遗传学修饰中,已发现超过7 000多种mRNA和300多种非编码RNA中存在m6A表观遗传学修饰 [10],并且越来越多的证据表明m6A这种高丰度修饰在肿瘤的发生和转移中均有重要作用[11]。目前,对于前列腺癌转移机制的研究和治疗尚未取得突破性进展,迫切需要确定前列腺癌治疗的新靶点,进而为前列腺癌的早期诊断和预后提供科学依据。因此,在前列腺癌中鉴定m6A甲基化调控基因可能提供有价值的治疗新靶点。
本研究从TCGA数据库下载了年龄、T分期、N分期等数据完整的496例样本的临床病理资料及mRNA相关数据,获取了METTL3、METTL14、WTAP、RBM15、ZC3H13、YTHDC1、YTHDC2、YTHDF1、YTHDF2、HNRNPC、FTO和ALKBH5共12种m6A调节因子,筛选出5个下调(ALKBH5、FTO、METTL14、YTHDC1和ZC3H13) 和3个上调(HNRNPC、METTL3和YTHDF1) 调节因子;不同T分期的前列腺癌中HNRNPC、YTHDC2、YTHDF1和YTHDF2 mRNA的表达水平不同,并且HNRNPC的表达量随着前列腺癌的进展出现显著持续升高;淋巴结转移组RBM15、HNRNPC、YTHDF2和YTHDF1 mRNA的表达水平明显高于淋巴结未转移组。对8个差异表达的m6A调节因子进行无监督聚类分析,将样本分成cluster 1、cluster 2和cluster 3,并且发现3组间OS存在统计学差异。多因素Cox回归分析发现,m6A调节因子METTL14和FTO与前列腺癌患者的预后密切相关。构建LASSO回归模型对前列腺癌患者进行风险评分,并分为高、低风险组,发现2组间OS有统计学差异,且风险评分可作为独立预后指标,用于预测患者的生存情况。前列腺癌组织样本和癌旁正常组织样本的免疫组织化学染色结果显示,METTL14和FTO蛋白水平在前列腺癌组织中高表达。值得注意的是,由于前列腺患者的死亡率低,且临床数据中T4分期的样本过少,导致根据T分期预测患者的生存情况结果欠准确,但这并不影响列线图预测整体的准确性。未来可以通过扩大样本量,进一步提高T分期在预测中的准确性。
研究发现,m6A甲基化结合位点结合的特殊基因中的HNRNPC可参与RNA的剪接、非特异性RNA输出、RNA表达、3’末端加工和翻译,并在黑色素瘤、肝癌、肺癌、胶质母细胞瘤等多种肿瘤中表达上调。研究 [12]表明,沉默HNRNPC可减少胶质母细胞瘤细胞增殖并增强依托泊苷诱导的细胞凋亡,表明HNRNPC可作为胶质母细胞瘤预后和治疗的潜在标志物。研究 [13]指出,过表达HNRNPC时可促进化学抗性,通过小干扰RNA敲低HNRNPC可逆转化学抗性,HNRNPC过表达与接受化疗药物治疗的胃癌患者的生存率呈负相关,这表明HNRNPC与胃癌预后不良有关。研究 [14]发现,HNRNPC过表达可通过上皮-间质转化促使口腔鳞状细胞发生癌变。本研究发现,HNRNPC的表达水平与前列腺T分期相关,HNRNPC的表达随着前列腺癌的进展而显著持续升高。从而推测,HNRNPC可能为前列腺癌提供新的治疗靶点。
METTL14是m6A甲基转移酶复合物的重要组成成分,在体外甲基化转移能力较高,且在多种肿瘤中表达异常,参与多种肿瘤的恶性表型调控。FTO是第1个发现的m6A去甲基化酶,可以影响相邻基因转录,消除mRNA的m6A修饰。FTO在急性髓系白血病、胶质母细胞瘤干细胞自我更新、宫颈鳞状细胞癌化疗、放疗耐药性中起关键作用。此外,FTO高表达促进胃癌细胞迁移、侵袭和淋巴结转移,与不良预后密切相关。METTL14和FTO尽管已经被证明与多种癌症的发展有关,但其在前列腺中的作用目前仍然不明确,且m6A调节因子在前列腺癌中的作用机制相关研究较少。
综上所述,本研究发现,METTL14和FTO可成为独立预测前列腺癌患者预后的关键调控基因,强调了RNA修饰在前列腺癌发生、发展中的重要作用,并为治疗方法的选择提供了潜在的标志物。
| [1] |
REBELLO RJ, OING C, KNUDSEN KE, et al. Prostate cancer[J]. Nat Rev Dis Primers, 2021, 7(1): 9. DOI:10.1038/s41572-020-00243-0 |
| [2] |
SIEGEL RL, MILLER KD, FUCHS HE, et al. Cancer statistics, 2021[J]. CA Cancer J Clin, 2021, 71(1): 7-33. DOI:10.3322/caac.21654 |
| [3] |
SIEGEL RL, MILLER KD, JEMAL A. Cancer statistics, 2018[J]. CA Cancer J Clin, 2018, 68(1): 7-30. DOI:10.3322/caac.21442 |
| [4] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [5] |
WANG G, ZHAO D, SPRING DJ, et al. Genetics and biology of prostate cancer[J]. Genes Dev, 2018, 32(17/18): 1105-1140. DOI:10.1101/gad.315739.118 |
| [6] |
STOPSACK KH, NANDAKUMAR S, WIBMER AG, et al. Oncogenic genomic alterations, clinical phenotypes, and outcomes in metastatic castration-sensitive prostate cancer[J]. Clin Cancer Res, 2020, 26(13): 3230-3238. DOI:10.1158/1078-0432.CCR-20-0168 |
| [7] |
LI D, HAO X, SONG Y. Identification of the key microRNAs and the miRNA-mRNA regulatory pathways in prostate cancer by bioinformatics methods[J]. Biomed Res Int, 2018, 2018: 6204128. DOI:10.1155/2018/6204128 |
| [8] |
UMUHIRE JURU A, HARGROVE AE. Frameworks for targeting RNA with small molecules[J]. J Biol Chem, 2021, 296: 100191. DOI:10.1074/jbc.REV120.015203 |
| [9] |
MA Z, JI J. N6-methyladenosine (m6A) RNA modification in cancer stem cells[J]. Stem Cells, 2020, 38(12): 1511-1519. DOI:10.1002/stem.3279 |
| [10] |
DENG X, SU R, FENG X, et al. Role of N6-methyladenosine modification in cancer[J]. Curr Opin Genet Dev, 2018, 48: 1-7. DOI:10.1016/j.gde.2017.10.005 |
| [11] |
ZHANG S, ZHAO BS, ZHOU A, et al. m6A demethylase ALKBH5 maintains tumorigenicity of glioblastoma stem-like cells by sustaining FOXM1 expression and cell proliferation program[J]. Cancer Cell, 2017, 31(4): 591-606.e6. DOI:10.1016/j.ccell.2017.02.013 |
| [12] |
PARK YM, HWANG SJ, MASUDA K, et al. Heterogeneous nuclear ribonucleoprotein C1/C2 controls the metastatic potential of glioblastoma by regulating PDCD4[J]. Mol Cell Biol, 2012, 32(20): 4237-4244. DOI:10.1128/MCB.00443-12 |
| [13] |
HUANG H, HAN Y, ZHANG C, et al. HNRNPC as a candidate biomarker for chemoresistance in gastric cancer[J]. Tumor Biol, 2016, 37(3): 3527-3534. DOI:10.1007/s13277-015-4144-1 |
| [14] |
HUANG GZ, WU QQ, ZHENG ZN, et al. M6A-related bioinformatics analysis reveals that HNRNPC facilitates progression of OSCC via EMT[J]. Aging, 2020, 12(12): 11667-11684. DOI:10.18632/aging.103333 |
 2023, Vol. 52
2023, Vol. 52