文章信息
- 高吴陆怡, 王静艳, 舒岑皓, 尚德淑, 韩锁, 商宇
- GAO Wuluyi, WANG Jingyan, SHU Cenhao, SHANG Deshu, HAN Suo, SHANG Yu
- 基于网络药理学与分子对接的丹皮酚治疗结直肠癌的分子机制研究
- Assessment of the molecular mechanism of paeonol in colorectal cancer treatment based network pharmacology and molecular docking
- 中国医科大学学报, 2023, 52(11): 992-998
- Journal of China Medical University, 2023, 52(11): 992-998
-
文章历史
- 收稿日期:2022-12-06
- 网络出版时间:2023-11-02 13:59:45
2. 佳木斯大学附属第一医院康复医学科, 黑龙江 佳木斯 154003;
3. 中国医科大学生命科学学院发育细胞生物学教研室, 教育部医学细胞生物学重点实验室, 沈阳 110122;
4. 哈尔滨医科大学附属第四医院中心导管室, 哈尔滨 150001
2. Department of Rehabilitation Medicine, First Affiliated Hospital of Jiamusi University, Jiamusi 154003, China;
3. Department of Developmental Cell Biology, School of Life Sciences, China Medical University, Key Laboratory of Medical Cell Biology, Ministry of Education, Shenyang 110122, China;
4. Central Cath Laboratory, Fourth Affiliated Hospital of Harbin Medical University, Harbin 150001, China
结直肠癌(colorectal cancer,CRC)是消化系统常见的癌症类型之一。根据国际癌症研究机构的统计结果,2020年全球CRC发病人数约为190万,死亡人数约为93.5万,其发病率和死亡率均居于全球前三位[1]。CRC的发病具有隐匿性,而中晚期CRC的手术与常规辅助治疗往往无法获得理想的治疗效果[2]。因此,探索新的有效的CRC辅助治疗手段具有重大意义。
丹皮酚又名牡丹酚、芍药醇,是中药牡丹皮和徐长卿的活性成分[3]。丹皮酚的药理作用包括抗炎、抗菌、镇痛以及抗凝血等[4]。在临床上,丹皮酚片被用于头痛、类风湿关节炎等疾病的治疗,丹皮酚软膏被用于湿疹、皮炎等皮肤病的治疗。丹皮酚能够在多种癌症(如胃癌[5]、膀胱癌[6]以及食管癌[7]等)中抑制癌细胞增殖,促进癌症细胞凋亡。丹皮酚在CRC中发挥治疗作用。LI等[8]发现,丹皮酚通过抑制PGE2的生成和COX-2蛋白的表达,促进CRC细胞的凋亡。LIU等[9]发现丹皮酚通过抑制Wnt/β-catenin途径来抑制CRC细胞的增殖,并诱导其发生细胞周期阻滞。
本研究基于网络药理学探索了丹皮酚治疗CRC的核心药物靶点与关键信号通路,旨在为丹皮酚在CRC治疗方面的临床转化提供更加充分的证据。
1 材料与方法 1.1 数据库检索及丹皮酚药物靶点的筛选在Pubchem数据库获得丹皮酚的Smiles信号。将Smilesk号输入到SwissTargetPrediction与TargetNet 2个数据库中检索丹皮酚的药物靶点。SwissTargetPrediction数据库中可能性值(Probability)排序前15名的基因被认为是丹皮酚的可靠药物靶点[10];TargetNet数据库中可能性值> 0的基因被认为是丹皮酚的可靠药物靶点[11]。将检索2个数据库的靶点合并与去除重复后即为丹皮酚的药物靶点。
1.2 数据库检索及CRC疾病靶点的筛选从数据库Genecards、Mendelian Inheritance in Man(OMIM)与Therapeutic Target Database(TTD)获取CRC的疾病靶点。Genecards数据库中Score > 50的基因被作为疾病靶点保留。OMIM与TTD数据库检索到的基因均被做为疾病靶点保留。将数据汇总并删除重复项后即为检索到的CRC相关靶点。
1.3 丹皮酚治疗CRC药物靶点的收集利用Venny在线工具(https://bioinfogp.cnb.csic.es/tools/venny/)对丹皮酚的药物靶点与CRC的药物靶点取交集并作图,获得的共同靶点为丹皮酚治疗CRC的靶点。
1.4 药物靶点的富集分析通过Metascape平台对丹皮酚治疗CRC的靶点进行基因本体(gene ontology,GO)富集分析与京都基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)信号通路富集分析[12]。方法为输入相交靶点蛋白的“Gene name”,物种选择为“H.sapiens”,且选择Custom Analysis的分析方式,在P≤0.01情况下分别进行GO的生物过程分析和KEGG信号通路分析。
1.5 蛋白质-蛋白质相互作用网络(protein-protein interaction networks,PPI)的构建利用STRING平台(https://www.string-db.org/)与Cytoscape软件(版本3.6.0)完成PPI分析[13]。将药物靶点导入STRING平台的检索栏,设置置信度为0.4,获得PPI结果并下载数据。将PPI数据导入Cytoscape软件进行可视化并获得互作节点评分。以连通性(Degree)为主要指标对节点蛋白排序,连通性大于均值的蛋白为相互作用的枢纽节点。
1.6 对丹皮酚与枢纽节点蛋白进行分子对接用2款基于不同算法的软件LeDock与CB-Dock2分别对丹皮酚与枢纽节点蛋白进行分子对接[14-15]。通过其LePro模块自动处理受体蛋白并生成对接配置文件。用PyMOL软件(版本号2.2.0)获得受体蛋白的对接口袋,并修改LeDock对接配置文件的口袋位置[16]。导入丹皮酚分子结构后,通过其LeDock模块完成受体蛋白与丹皮酚的对接。上传经PyMOL软件删除配体与水分子的受体蛋白结构文件与丹皮酚结构文件,使用其Auto Blind Docking模块自动探测受体口袋与完成对接。
2 结果 2.1 丹皮酚治疗CRC的药物靶点从SwissTargetPrediction数据库筛选得到15个丹皮酚药物靶点,从TargetNet数据库筛选到了221个药物靶点。药物靶点经汇总与删除重复项后,最终获得了203个药物靶点。从Genecards等数据库获得的CRC疾病靶点经汇总与删除重复项后共有710个疾病靶点。药物靶点与疾病靶点进行重复交集比对后最终得到21个丹皮酚治疗CRC的药物靶点,见图 1。这些药物靶点包括AR、ATP4A、CHEK2、CNR2、ESR1、KDR、MDM2、MET、MMP9、PDGFRA、PLA2G2A、PPARG、PTPN22、RARA、RET、SIRT1、SRC、TERT、TLR9、VCAM1以及EP300。部分药物靶点的可能性值以及数据来源等信息见表 1。
|
| 图 1 丹皮酚靶点与结直肠癌靶点韦恩图 Fig.1 Venn diagram of paeonol and colorectal cancer targets |
| Gene | Protein | Probability | Data source |
| ESR1 | Estrogen receptor | 0.997 | TargetNet |
| MMP9 | Matrix metalloproteinase-9 | 1.000 | TargetNet |
| PDGFRA | Platelet-derived growth factor receptor alpha | 0.858 | TargetNet |
| PPARG | Peroxisome proliferator-activated receptor gamma | 0.997 | TargetNet |
| PTPN22 | Tyrosine-protein phosphatase non-receptor type 22 | 0.117 | SwissTargetPrediction |
| RARA | Retinoic acid receptor alpha | 0.979 | TargetNet |
| TLR9 | Toll-like receptor 9 | 0.996 | TargetNet |
| VCAM1 | Vascular cell adhesion protein 1 | 0.180 | TargetNet |
| EP300 | Histone acetyltransferase p300 | 0.125 | SwissTargetPrediction |
2.2 交集靶点的富集分析
GO富集分析结果显示,丹皮酚治疗CRC的药物靶点富集于性腺发育、防御反应的调节、上皮管的形态变化、对缺氧的反应以及激酶活性的调节等生物过程。根据P值进行排序,排名前20的生物过程见图 2。在KEGG信号通路分析中,药物靶点被富集于雌激素信号通路、Ras信号通路、Rap1信号通路、EGFR酪氨酸激酶抑制剂耐药以及癌症中的中心碳代谢等33条通路。P值排名前20的信号通路绘图见图 3。
|
| 图 2 GO生物过程富集分析结果 Fig.2 Bioprocess enrichment analysis results |
|
| 图 3 KEGG信号通路富集分析结果 Fig.3 KEGG signaling pathway enrichment analysis results |
2.3 PPI网络分析
STRING平台分析结果显示,输入的21个基因共包含有72种相互作用。Cytoscape重新绘图后,去除无效的2个节点后最终得到19个节点,见图 4。Cytoscape计算PPI网络发现连通性(Degree)的均值为0.758,高于0.758的节点蛋白有9个,包括SRC、MMP9、PPARG、ESR1、TERT、SIRT1、MET、EP300、MDM2,说明以上9种蛋白是PPI网络中的枢纽蛋白,见表 2。
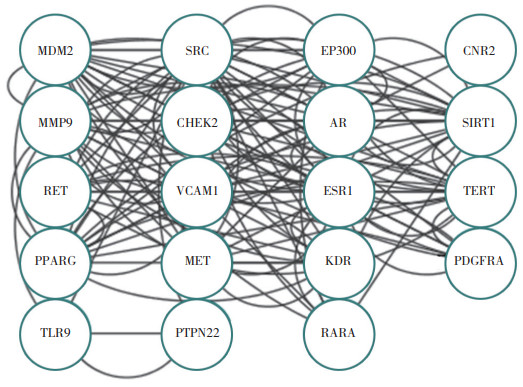
|
| 图 4 丹皮酚靶点蛋白的相互作用网络 Fig.4 Interaction network of paeonol target proteins |
| Protein | Degree | Betweenness centrality |
| SRC | 16 | 0.252 |
| ESR1 | 13 | 0.075 |
| MDM2 | 12 | 0.053 |
| SIRT1 | 11 | 0.047 |
| MMP9 | 10 | 0.055 |
| TERT | 10 | 0.035 |
| PPARG | 10 | 0.136 |
| EP300 | 9 | 0.021 |
| MET | 8 | 0.011 |
2.4 丹皮酚与枢纽蛋白的分子对接
分子对接结果显示,丹皮酚与9个枢纽节点均具有结合潜力。在LeDock预测结果中,与丹皮酚亲和力最高的5种蛋白依次为PPARG、ESR1、MMP9、EP300和SRC。CB-Dock2的预测结果中亲和力最高的5种蛋白依次为MMP9、TERT、EP300、SIRT1和SRC,见表 3。综合2种软件的预测结果,SRC、EP300和MMP9在2款软件中均表现出了良好的结合能力,可能为丹皮酚的核心药物靶点,见图 5A。SRC、EP300和MMP9通过LeDock预测的最高亲和力结合模式,见图 5B。SRC、EP300和MMP9通过CB-Dock2预测的最高亲和力结合模式,见图 5C。
| niport ID | Protein | Ledock’s molecular docking results | CB-Dock2’s molecular docking results(kcal/mol) | |||
| Binding energy | Combining the center coordinates of the cavity(X,Y,Z) | Vina score(kcal/mol) | Combining the center coordinates of the cavity(X,Y,Z) | |||
| P14780 | MMP9 | -3.42 | 27,47,47 | -7.1 | 43,53,39 | |
| P37231 | PPARG | -3.63 | 41,1,84 | -5.5 | 44,6,82 | |
| P12931 | SRC | -3.36 | 15,24,57 | -6.1 | 27,45,80 | |
| P03372 | ESR1 | -3.56 | 16,32,22 | -5.7 | 10,27,11 | |
| O14746 | TERT | -2.85 | -17,31,5 | -6.5 | -24,48,32 | |
| Q96EB6 | SIRT1 | -3.16 | -17,66,13 | -6.3 | -68,94,1 | |
| P08581 | MET | -3.28 | 21,18,57 | -5.9 | 4,22,79 | |
| Q09472 | EP300 | -3.37 | -34,-18,-22 | -6.4 | -35,-19,12 | |
| Q00987 | MDM2 | -2.04 | 23,-25,-8 | -4.5 | 28,-22,-6 | |
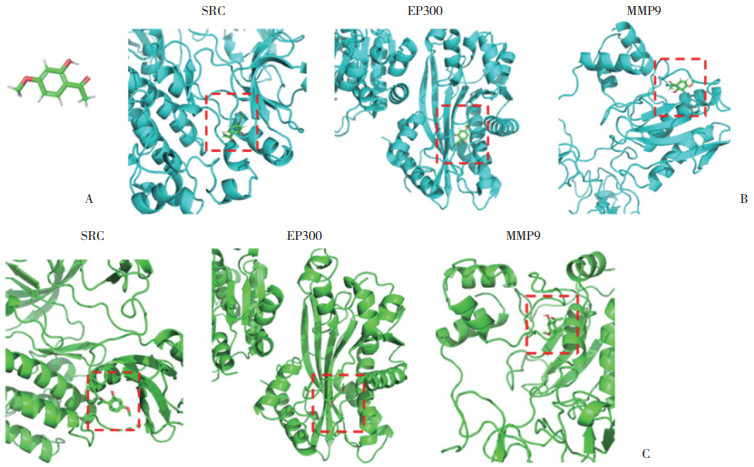
|
| A, 3D molecular structure of paeonol; B, diagram of molecular docking patterns of paeonol with SRC, EP300, and MMP9 proteins detected using the Le-Dock software; C, diagram of molecular docking patterns of paeonol with SRC, EP300, and MMP9 proteins detected using CB-Dock2. 图 5 丹皮酚与重要靶点蛋白SRC、EP300和MMP9的分子对接模式 Fig.5 Molecular docking patterns of paeonol with key target proteins SRC, EP300, and MMP9 |
3 讨论
丹皮酚是中药材牡丹皮的主要活性成分,具有显著的抗氧化和消炎作用,广泛应用于医疗与保健[17]。丹皮酚能够通过多种分子途径抑制细胞增殖、侵袭以及诱导细胞凋亡和周期停滞,表现出广泛的抗癌作用[18]。研究[8-9]证实丹皮酚具有抗CRC的作用。本研究基于网络药理学方法,从整体出发分析丹皮酚抗CRC的具体作用机制。
本研究结果显示,共21个蛋白涉及丹皮酚的抗CRC作用。通过KEGG富集分析发现这21个蛋白富集于Ras、雌激素和Rap1等信号通路。RAS基因(KRAS与NRAS)的致癌突变在CRC中十分常见,其不仅驱动癌症进展,还影响放疗、化疗与靶向治疗的治疗效果[19]。雌激素在维持结肠上皮的结构和生理完整性中起到重要作用,雌激素及其受体与CRC的患病风险和癌症转移呈负相关[20]。GO富集分析结果显示,丹皮酚对CRC的治疗作用主要涉及激酶活性的调节、MAPK级联的调控、对缺氧的反应等生物过程。缺氧是肿瘤微环境的特征之一,其诱导的HIF信号通路激活与CRC病情进展和预后不良明显相关[21]。由此可见,丹皮酚对CRC相关信号通路与生物过程具有广泛的调控作用。
通过PPI分析和分子对接,从21个靶点中筛选出SRC、EP300以及MMP9作为丹皮酚的核心靶点。SRC为非受体酪氨酸激酶,可以通过不同的激酶途径(MAPK,PI3K/Akt/mTOR、JAK/STAT3、PEAK1和Rho/ROCK等)进行胚胎发育和细胞生长调节,并传导下游信号以参与癌细胞的细胞增殖、血管生成、迁移、侵袭和转移[22]。SRC在CRC的患者中呈显著高表达,并且与肿瘤的转移和生存预后密切相关[23]。MMP9是基质金属蛋白酶家族成员,肿瘤细胞中基质金属蛋白酶的早期表达有助于细胞外基质的重塑,并释放膜结合生长因子,为肿瘤发生奠定微环境。MMP9在血管生成、转移和癌症侵袭中的作用已被证实,并且与癌细胞的存活和扩散相关[24],其在CRC中高表达并且参与癌细胞的转移[25]。EP300是组蛋白乙酰转移酶,其通过影响染色质重塑调控基因表达。EP300在CRC癌组织中表达异常,其可能通过调控BAX的表达影响CRC的疾病进程[26]。SRC、EP300和MMP9广泛参与到显著富集的信号通路与生物过程。例如,在排名前20的KEGG信号通路中,SRC参与其中11条通路,MMP9参与其中10条通路,SRC与MMP9共同参与的信号通路有6条。以上的结果表明SRC、EP300以及MMP9可能通过多种途径调控CRC细胞的发展进程,有望成为新的治疗靶点。
综上所述,丹皮酚能够靶向SRC、EP300与MMP9,通过调控Ras、Rap1等信号通路与激酶活性、对缺氧的反应等生物过程,发挥治疗结直肠癌的作用。本研究的不足之处是没有进行体内与体外的实验验证,应该在未来的研究中继续探索与补充。
| [1] |
SUNG H, FERLAY J, SIEGEL RL, et al. Global cancer statistics 2020:globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
SIEGEL RL, MILLER KD, JEMAL A. Cancer statistics, 2020[J]. CA ACancer J Clin, 2020, 70(1): 7-30. DOI:10.3322/caac.21590 |
| [3] |
蒋鹤飞, 杨小四. 丹皮酚抗炎作用实验研究进展[J]. 中国医药科学, 2022, 12(5): 53-56. DOI:10.3969/j.issn.2095-0616.2022.05.014 |
| [4] |
刘阳欣, 赵锋, 康秉涛, 等. 丹皮酚药理学研究进展[J]. 陕西中医, 2020, 41(4): 550-552. DOI:10.3969/j.issn.1000-7369.2020.04.045 |
| [5] |
LI J, ZHANG G, WU GQ. Effect of paeonol on proliferation, apoptosis, migration, invasion and glutamine of gastric cancer cells via circSFMBT2/miR-665 axis[J]. Cell Mol Biol (Noisy-le-grand), 2020, 66(8): 33-40. DOI:10.14715/cmb/2020.66.8.6 |
| [6] |
ZHANG JJ, CAI LJ, PANG K, et al. Paeonol inhibits proliferation and induces cell apoptosis of human T24 and 5637 bladder cancer cells in vitro and in vivo[J]. Clin Transl Oncol, 2021, 23(3): 601-611. DOI:10.1007/s12094-020-02455-y |
| [7] |
DU JK, SONG DB, LI JW, et al. Paeonol triggers apoptosis in HeLa cervical cancer cells: the role of mitochondria-related caspase pathway[J]. Psychopharmacology, 2022, 239(7): 2083-2092. DOI:10.1007/s00213-021-05811-0 |
| [8] |
LI M, TAN SY, WANG XF. Paeonol exerts an anticancer effect on human colorectal cancer cells through inhibition of PGE2 synthesis and COX-2 expression[J]. Oncol Rep, 2014, 32(6): 2845-2853. DOI:10.3892/or.2014.3543 |
| [9] |
LIU LH, SHI RJ, CHEN ZC. Paeonol exerts anti-tumor activity against colorectal cancer cells by inducing G0/G1 phase arrest and cell apoptosis via inhibiting the Wnt/β-catenin signaling pathway[J]. Int J Mol Med, 2020, 46(2): 675-684. DOI:10.3892/ijmm.2020.4629 |
| [10] |
DAINA A, MICHIELIN O, ZOETE V. SwissTargetPrediction: updated data and new features for efficient prediction of protein targets of small molecules[J]. Nucleic Acids Res, 2019, 47(W1): W357-W364. DOI:10.1093/nar/gkz382 |
| [11] |
YAO ZJ, DONG J, CHE YJ, et al. TargetNet: a web service for predicting potential drug-target interaction profiling via multi-target SAR models[J]. J Comput Aided Mol Des, 2016, 30(5): 413-424. DOI:10.1007/s10822-016-9915-2 |
| [12] |
ZHOU YY, ZHOU B, PACHE L, et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J]. Nat Commun, 2019, 10(1): 1523. DOI:10.1038/s41467-019-09234-6 |
| [13] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [14] |
XU SQ, WANG L, PAN XC. An evaluation of combined strategies for improving the performance of molecular docking[J]. J Bioinform Comput Biol, 2021, 19(2): 2150003. DOI:10.1142/s0219720021500037 |
| [15] |
LIU Y, YANG XC, GAN JH, et al. CB-Dock2:improved protein-ligand blind docking by integrating cavity detection, docking and homologous template fitting[J]. Nucleic Acids Res, 2022, 50(W1): W159-W164. DOI:10.1093/nar/gkac394 |
| [16] |
LIU WN, CHEN WJ, HU MT, et al. Bioinformatics analysis combined with molecular dynamics simulation validation to elucidate the potential molecular mechanisms of Jianshen Decoction for treatment of osteoporotic fracture[J]. Medicine (Baltimore), 2023, 102(16): e33610-e33625. DOI:10.1097/MD.0000000000033610 |
| [17] |
WU M, YU ZL, LI XY, et al. Paeonol for the treatment of atherosclerotic cardiovascular disease: a pharmacological and mechanistic overview[J]. Front Cardiovasc Med, 2021, 8: 690116. DOI:10.3389/fcvm.2021.690116 |
| [18] |
WANG Y, LI BS, ZHANG ZH, et al. Paeonol repurposing for cancer therapy: from mechanism to clinical translation[J]. Biomed Pharmacother, 2023, 165: 115277. DOI:10.1016/j.biopha.2023.115277 |
| [19] |
SEREBRIISKII IG, CONNELLY C, FRAMPTON G, et al. Comprehensive characterization of RAS mutations in colon and rectal cancers in old and young patients[J]. Nat Commun, 2019, 10: 3722. DOI:10.1038/s41467-019-11530-0 |
| [20] |
MAINGI JW, TANG S, LIU SR, et al. Targeting estrogen receptors in colorectal cancer[J]. Mol Biol Rep, 2020, 47(5): 4087-4091. DOI:10.1007/s11033-020-05414-6 |
| [21] |
XU K, ZHAN YP, YUAN ZT, et al. Hypoxia induces drug resistance in colorectal cancer through the HIF-1α/miR-338-5p/IL-6 feedback loop[J]. Mol Ther, 2019, 27(10): 1810-1824. DOI:10.1016/j.ymthe.2019.05.017 |
| [22] |
TELI G, PAL R, MAJI L, et al. Medicinal chemistry perspectives on recent advances in src kinase inhibitors as a potential target for the development of anticancer agents: biological profile, selectivity, structure-activity relationship[J]. Chem Biodivers, 2023, e202300515. DOI:10.1002/cbdv.202300515 |
| [23] |
JIN W. Regulation of src family kinases during colorectal cancer development and its clinical implications[J]. Cancers, 2020, 12(5): 1339. DOI:10.3390/cancers12051339 |
| [24] |
MONDAL S, ADHIKARI N, BANERJEE S, et al. Matrix metalloproteinase-9(MMP-9) and its inhibitors in cancer: a minireview[J]. Eur J Med Chem, 2020, 194: 112260. DOI:10.1016/j.ejmech.2020.112260 |
| [25] |
LU PF, CHEN JJ, YAN LH, et al. RasGRF2 promotes migration and invasion of colorectal cancer cells by modulating expression of MMP9 through Src/Akt/NF-κB pathway[J]. Cancer Biol Ther, 2019, 20(4): 435-443. DOI:10.1080/15384047.2018.1529117 |
| [26] |
KOWALCZYK AE, KRAZINSKI BE, GODLEWSKI J, et al. Expression of the EP300, TP53 and BAX genes in colorectal cancer: correlations with clinicopathological parameters and survival[J]. Oncol Rep, 2017, 38(1): 201-210. DOI:10.3892/or.2017.5687 |
 2023, Vol. 52
2023, Vol. 52