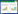文章信息
- 张乐, 宁静华, 张鑫, 王振, 刘虹汝, 张钰哲
- ZHANG Le, NING Jinghua, ZHANG Xin, WANG Zhen, LIU Hongru, ZHANG Yuzhe
- 综合分析和鉴定m6A RNA甲基化调节因子对前列腺癌进展及预后的影响
- Comprehensive analysis and identification of m6A RNA methylation regulators in the progression and prognosis of prostate cancer
- 中国医科大学学报, 2023, 52(11): 971-978
- Journal of China Medical University, 2023, 52(11): 971-978
-
文章历史
- 收稿日期:2022-12-31
- 网络出版时间:2023-11-02 13:31:19
2. 云南抗病原药用植物筛选重点实验室, 云南 大理 671000;
3. 云南省昆虫生物医药重点实验室, 云南 大理 671000
2. Yunnan Key Laboratory of Antipathogenic Medicinal Plant Screening, Dali 671000, China;
3. Yunnan Provincial Key Laboratory of Insect Biomedicine, Dali 671000, China
前列腺癌是中老年男性常见的恶性肿瘤,已成为威胁人类健康的公共问题[1]。目前,前列腺癌的治疗方法包括手术、内分泌治疗、放化疗、冷冻治疗和免疫治疗等[2],但由于人们对前列腺癌的早期筛查不够重视,通常确诊时已为晚期或转移。前列腺癌患者即使进行积极的雄激素剥夺治疗,但由于长时间的药物治疗,患者会产生耐药性,从而演变为去势抵抗性前列腺癌[3]。因此,寻找更为有效的早期诊断标志物和治疗方案势在必行。
N6-甲基腺苷(N6-methyladenosine,m6A)是最常见的RNA甲基化修饰。m6A修饰与肿瘤的发生、分化、增殖、侵袭和转移有关[4-7]。本研究基于以往研究最广泛报道的13种m6A RNA甲基化调节因子(以下简称m6A调节因子),利用癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中的RNA测序数据,系统分析了498例前列腺癌中这13种m6A调节因子的差异表达与临床病理特征的关联,评估m6A甲基化与前列腺癌进展和生存的相关性,为从甲基化修饰角度理解前列腺癌的发生和发展提供参考。
1 材料与方法 1.1 数据采集从TCGA数据库(https://cancergenome.nih.gov/)中获取TCGA-PRAD数据,下载前列腺癌和癌旁样本的基因表达数据。排除同一患者的重复样本后,共有498例前列腺癌组织样本和52例正常前列腺组织样本。
1.2 m6A调节因子的选择根据之前的研究[8-10],筛选出最广泛报道的13种m6A调节因子:METTL3、METTL14、WTAP、KIAA1429、RBM15、ZC3H13、YTHDC1、YTHDC2、YTHDF1、YTHDF2、HNRNPC、FTO、ALKBH5。提取13种基因的表达矩阵和临床样本的信息。
1.3 生物信息学分析 1.3.1 差异基因分析为了研究m6A调节因子在前列腺癌中的作用,应用R语言(4.2.1)limma包,分析13种m6A调节因子在前列腺癌和正常前列腺组织中的差异表达。
1.3.2 利用m6A调节因子在癌症中的表达水平进行无监督聚类分析为了确定m6A调节因子是否与前列腺癌的预后相关,将498例前列腺癌样本进行分组。使用R语言Consensus Cluster Plus包执行,根据集群和项目一致性原理,输出图形。集群数量通过累积分布函数曲线确定,为了验证根据13种m6A调节因子表达量进行的聚类分组在所有基因中的准确性,进行主成分分析,验证聚类分组效果。
1.3.3 m6A调节因子预后风险模型的构建为了研究聚类分析与临床病理特征的相关性,从TCGA中下载前列腺癌患者的临床信息,使用limma包分析13种m6A调节因子在Cluster 1和Cluster 2中的表达情况,并研究这13种m6A调节因子与前列腺癌患者的临床病理特征之间的相关性。随后筛选出Cluster 1与Cluster 2中的差异基因,对这些基因进行后续分析。利用基因本体论(gene ontology,GO)以及京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析,对差异基因进行功能注释。对差异基因进行单因素Cox回归分析,选择与生存密切相关的基因,应用LASSO Cox回归算法构建强大的预后特征,并计算每个基因的系数[11]。计算每例患者的风险评分,为每个基因评分的总和。根据前列腺癌患者风险评分的中位数,将前列腺癌分为高风险组和低风险组,绘制受试者操作特征曲线,计算曲线下面积(area under the curve,AUC),获得预后特征的灵敏度和特异度。
1.4 风险因子的表达水平进行单因素Cox回归分析,从297个差异表达基因中选出P < 0.01的基因,利用LASSO Cox算法选取误差最小的点,获得6个风险基因并构建风险模型。
1.5 基因表达和免疫浸润分析使用癌症基因组分析平台(http://bioinfo.life.hust.edu.cn/web/GSCALite/),对ROMO1在前列腺免疫微环境中的免疫浸润情况进行分析。为了探讨ROMO1的表达与前列腺癌免疫细胞的关系,使用癌症基因组分析平台中的Immune工具,分析ROMO1表达与前列腺癌免疫细胞浸润水平的关系。
2 结果 2.1 前列腺癌中m6A调节因子的关联性分析与正常前列腺组织相比,前列腺癌患者中METTL3、METTL14、WTAP、KIAA1429、RBM15、ZC3H13、YTHDC1、YTHDC2、YTHDF1、YTHDF2、HNRNPC、FTO、ALKBH5的表达水平较高(图 1A、1B),超过半数(11/13)的m6A调节因子在前列腺癌中高表达。m6A调节因子与前列腺癌的TNM分期、年龄以及生存状态相关,尤其与T3、M0、N0和年龄≤65岁患者的临床特征显著相关(图 1C)。
|
| A, expression levels of 13 m6A regulators in prostate cancer (red indicates upregulation and green indicates downregulation); B, expression of m6A regulators in prostate cancer (normal tissue is shown in blue and prostate cancer tissue is shown in red); C, expression of m6A regulators in prostate cancers with different clinicopathological features. ** P < 0.01;*** P < 0.001. 图 1 前列腺癌中m6A调节因子的分布 Fig.1 Distribution of m6A regulators in prostate cancer |
2.2 m6A调节因子的共识聚类鉴定2种不同的前列腺癌亚型
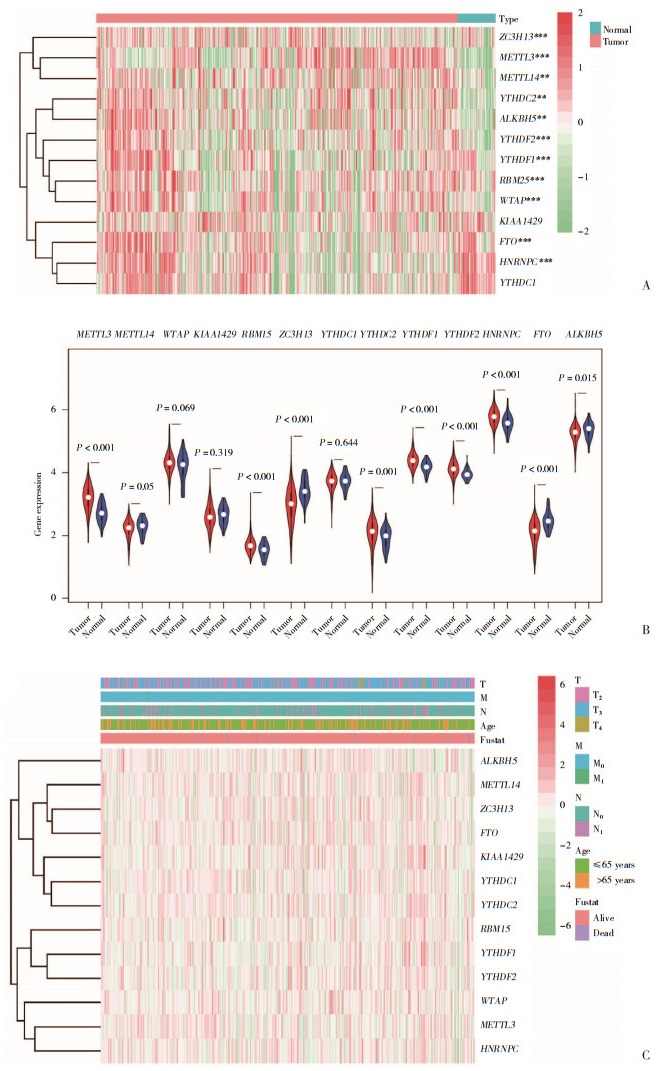
为了提供更为准确的分型,通过498例前列腺癌组织样本中13个m6A调节因子的mRNA表达共识,将前列腺癌分为几个集群。聚类指数k=2时,是聚类间最大差异的最佳点。从m6A调节因子的表达相似性来看,选择一致累积分布函数(cumulative distribution function,CDF)坡度较小的曲线对应的k值为最佳。结果发现,k=3时,似乎具有较小的CDF值(图 2A、2B),说明将前列腺癌组织样本分为3个聚类最佳。但将其分为3组后,组间相关性较高,样本数量较少,因此分为2组(Cluster 1和Cluster 2,图 2C)。为了验证聚类的分组效果以及说明利用m6A调节因子进行的聚类分组在所有基因中的适用性,进行主成分分析。结果显示,可以将Cluster 1与Cluster 2区分开来(图 2D),说明利用m6A调节因子的分类方法合理且准确。
|
| A, cumulative distribution function (CDF) of consistent clustering at k=2-9;B, relative change in area under the CDF curve when k=2-9;C, consistent clustering matrix at k=2;D, principal component analysis of total RNA expression profiles in the TCGA dataset. Cluster 1 subgroups are shown in red and Cluster 2 subgroups are shown in blue. 图 2 m6A调节因子一致聚类的识别 Fig.2 Identification of consistent clusters of m6A regulators |
2.3 通过共识聚类确定的类别与临床病理特征密切相关
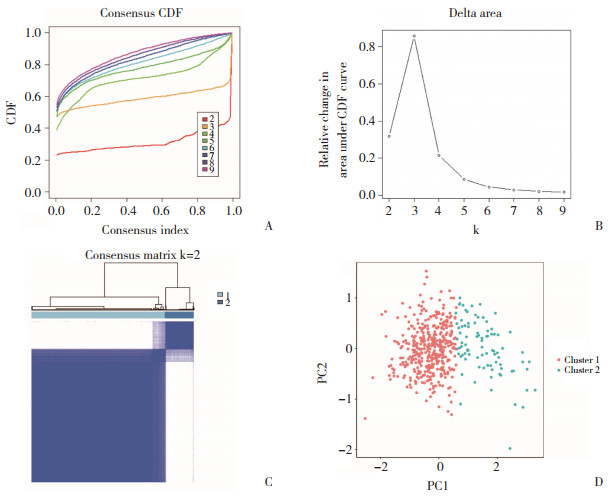
在TCGA数据库下载前列腺癌患者的临床信息,对患者的分型和临床病理特征进行分析,发现大部分m6A调节因子在Cluster 1中高表达(图 3)。与Cluster 2相比,Cluster 1与诊断时的年龄(≤65岁)、TNM分期(T3、M0、N0)、生存状态(存活)显著相关,聚类结果与前列腺癌的恶性程度密切相关。
|
| Clinicopathological characterization of the two clusters (Clusters 1 and 2) was defined by consensus expression of m6A regulators (red indicates upregulation and green indicates downregulation). 图 3 Cluster 1、Cluster 2与前列腺癌临床病理特征的关系 Fig.3 Relationship between Clusters 1 and 2 and the clinicopathological features of prostate cancer |
2.4 聚类间差异基因分析
从Cluster 1和Cluster 2中筛选出297个差异基因。GO分析结果表明,差异基因主要富集在恶性肿瘤的细胞基质黏附、黏附连接组织、线粒体呼吸链复合体组装、磷脂酰丝氨酸代谢过程、上皮细胞迁移的正向调节、细胞连接组织、氧化磷酸化作用等经典途径(图 4A、4B)。KEGG结果显示,差异基因富集于肿瘤的蛋白多糖、细胞外基质受体相互作用、转化生长因子-β信号通路、细胞骨架作用的调节、黏着斑、黏着连接(图 4C、4D)。
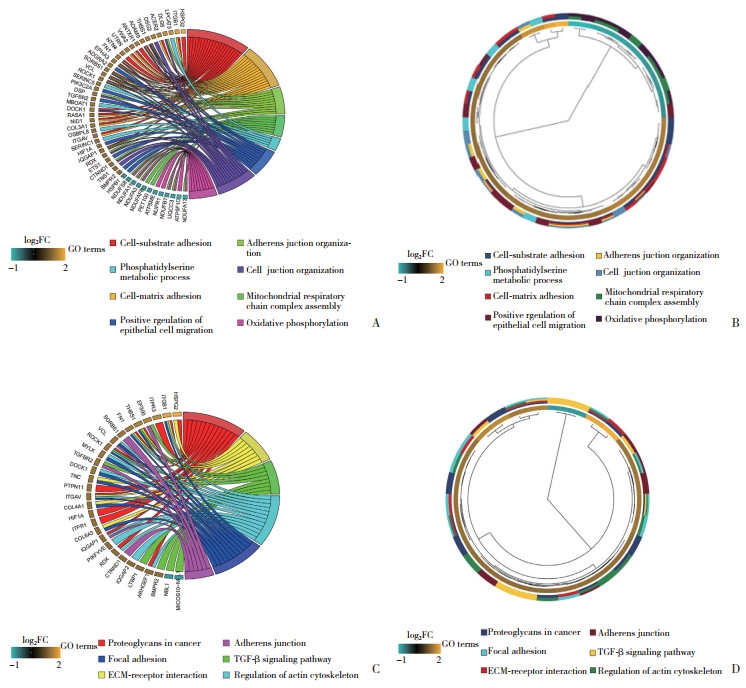
|
| A, B, GO analysis; C, D, KEGG pathway analysis. * P < 0.05;** P < 0.01. 图 4 利用生物过程GO分析和KEGG通路分析对Cluster 1亚群中表达较高的基因进行功能注释 Fig.4 Functional annotation of highly expressed genes in the Cluster 1 subgroup using bioprocess GO and KEGG pathway analyses |
2.5 m6A调节因子的风险特征与预后价值
从297个显著差异表达基因中,选出在单因素Cox回归分析中P < 0.01的基因,绘制森林图(图 5A)。风险比(hazard ratio,HR) > 1时,说明该基因是高风险基因;HR < 1时,说明该基因是低风险基因。结果表明,SNORD60、SNHG25、SNORD104、C4orf48、OAS3、NME3、ROMO1、SNHG9、TMEM238、MRPL41、SNHG19这些基因均HR > 1,说明以上基因表达水平较高时,前列腺癌患者的生存期较差。而SMIM22和MYOF均HR < 1,说明这2种基因表达水平较高时,前列腺癌患者有更好的生存期。后续研究中,由这些基因代替m6A调节因子。
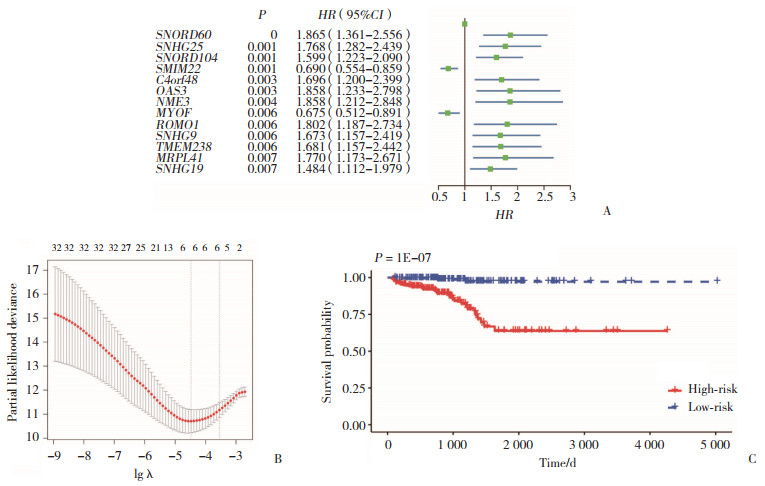
|
| A, risk assessment of 13 m6A regulators selected using Univariate Cox regression analysis; B, the six risk genes obtained during the analysis using LASSO Cox regression were used and models were constructed; C, survival curves for patients categorized into high-and low-risk groups based on risk scores in TCGA data. 图 5 6个m6A调节因子的风险特征 Fig.5 Risk profiles of six m6A regulators |
进行多因素Cox回归分析,利用LASSO Cox算法得到交叉验证的误差,选取误差最小的点并获得6个风险基因(图 5B),构建风险模型来预测样本的风险得分。为了研究风险模型预后作用,根据LASSO Cox算法得到的风险评分,利用风险评分的中位数,将TCGA数据集中的前列腺癌患者分为高风险组和低风险组,进行生存分析,结果显示,6个基因(OAS3、MYOF、SMIM22、SNORD60、ROMO1、MRPL41)构建的风险模型对前列腺癌的临床预后有较好参考价值(P = 1E-07)。提示高风险组前列腺癌患者的生存期较差(图 5C)。
2.6 风险评分与前列腺癌的临床病理特征密切相关6个风险基因与前列腺癌患者的年龄、分级、分期、T分期、M分期、N分期以及生存状态存在相关性(图 6A)。通过计算AUC,表明风险特征(6个基因)预测的准确率,AUC值越高,模型预测生存率越高。结果表明,风险评分可以预测前列腺癌的生存率(AUC=0.727)(图 6B)。单因素Cox回归分析结果显示,T分期、N分期、风险评分均可以作为高风险因素与风险评分相关(图 6C)。多因素Cox回归分析结果显示,风险评分可作为独立预后因子(图 6D)。以上证据表明,风险评分与前列腺癌临床病理特征密切相关,并且SNORD60、ROMO1、MRPL41与前列腺癌的恶性程度相关,特别是在高风险的前列腺癌中ROMO1表达水平更高。
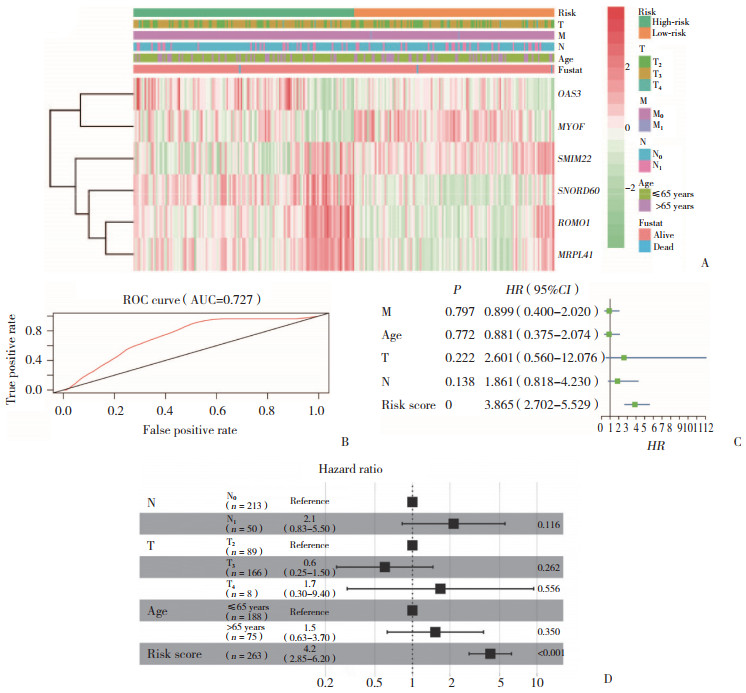
|
| A, expression levels of six m6A regulators in patients with prostate cancer in the high-and low-risk groups and the distribution of clinicopathological features in the two groups were compared; B, the ROC curve shows the accuracy of the prediction of the risk profile; C, univariate Cox regression analysis between clinicopathologic factors (including risk scores) and patient overall survival in TCGA data set; D, multivariate Cox regression analysis between clinicopathologic factors (including risk scores) and overall patient survival in the TCGA dataset. 图 6 高低风险组中风险评分和临床病理特征的关系 Fig.6 Relationship between risk scores and clinicopathological characteristics in high- and low-risk groups |
2.7 ROMO1在前列腺癌中的免疫浸润情况
结果发现,ROMO1与前列腺癌的CD8+T细胞的免疫浸润相关性强(图 7),这也揭示了ROMO1与前列腺癌免疫浸润有一定关系。
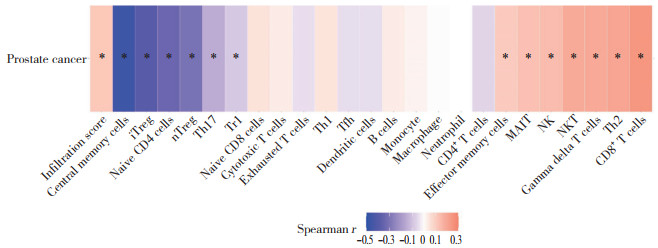
|
| Negative and positive correlations are shown in blue and red, respectively. iTeg, induced regulatory T cells; nTeg, natural regulatory T cells; Th17, T helper 17 cells; Tr1, type 1 regulatory T cells; Th1, T helper 1 cells; Tfh, T follicular helper cells; MAIT, mucosal-associated invariant T cells; NK, natural killer cells; NKT, natural killer T cells; Th2, T helper 2 cells. * P < 0.05. 图 7 ROMO1在前列腺癌免疫微环境中的免疫浸润情况 Fig.7 Immune infiltration by ROMO1 in the prostate cancer immune microenvironment |
3 讨论
本研究结果显示,m6A调节因子的表达与前列腺癌的临床病理特征高度相关;使用6种基因构建的临床风险模型可以作为预后指标;ROMO1与高风险组前列腺癌的恶性程度相关性较高;ROMO1与前列腺癌CD8+T细胞的免疫浸润相关,其有望独立作为前列腺癌临床预后的指标。
ROMO1可以通过干扰活性氧的产生,影响细胞的状态[12]。通过释放细胞色素c激活细胞凋亡途径,最终导致细胞死亡,从而引发癌症。从正常细胞转化为癌细胞,ROMO1作为一种活性氧调节蛋白,已在多种肿瘤细胞中被发现,并在肿瘤的发生、发展中起作用[13]。有研究[14]发现,ROMO1在前列腺癌组织中高表达,且与前列腺癌的免疫细胞浸润水平和免疫途径相关,与本研究结果一致,说明了本研究结果的准确性。本研究发现,ROMO1与前列腺癌的CD8+T细胞间存在免疫浸润关系,这也许为前列腺癌的免疫疗法提供了新的治疗方案。在机体正常情况下,体内的T细胞可以通过识别异常的癌细胞,启动抗肿瘤的免疫反应,T细胞越多,人体发动免疫反应抵抗癌细胞的效果就越好。CD8+T细胞对肿瘤细胞具有较强的杀伤功能。因此,从免疫治疗的角度考虑,可能通过调节ROMO1的表达导致前列腺癌免疫细胞浸润的改变,从而影响前列腺癌微环境的改变,进而导致前列腺癌异质性,从而达到精准化个体治疗的目的。
综上所述,本研究结果系统展示了m6A调节因子在前列腺癌中的表达、潜在功能和预后价值。通过分析筛选出的目标基因ROMO1与前列腺癌恶性程度高度相关,有望作为前列腺癌临床预后的独立指标,为去势抵抗性前列腺癌的治疗提供理论基础。
| [1] |
CAO W, CHEN HD, YU YW, et al. Changing profiles of cancer burden worldwide and in China: a secondary analysis of the global cancer statistics 2020[J]. Chin Med J (Engl), 2021, 134(7): 783-791. DOI:10.1097/CM9.0000000000001474 |
| [2] |
SCHATTEN H. Brief overview of prostate cancer statistics, grading, diagnosis and treatment strategies[J]. Adv Exp Med Biol, 2018, 1095: 1-14. DOI:10.1007/978-3-319-95693-0_1 |
| [3] |
GALLETTI G, LEACH BI, LAM L, et al. Mechanisms of resistance to systemic therapy in metastatic castration-resistant prostate cancer[J]. Cancer Treat Rev, 2017, 57: 16-27. DOI:10.1016/j.ctrv.2017.04.008 |
| [4] |
CAI J, YANG F, ZHAN H, et al. RNA m6A methyltransferase METTL3 promotes the growth of prostate cancer by regulating hedgehog pathway[J]. Oncotargets Ther, 2019, 12: 9143-9152. DOI:10.2147/ott.s226796 |
| [5] |
YUAN Y, DU Y, WANG L, et al. The M6A methyltransferase METTL3 promotes the development and progression of prostate carcinoma via mediating MYC methylation[J]. J Cancer, 2020, 11: 3588-3595. DOI:10.7150/jca.42338 |
| [6] |
LI S, CAO L. Demethyltransferase FTO alpha-ketoglutarate dependent dioxygenase (FTO) regulates the proliferation, migration, invasion and tumor growth of prostate cancer by modulating the expression of melanocortin 4 receptor (MC4R)[J]. Bioengineered, 2021, 13: 5598-5612. DOI:10.1080/21655979.2021.2001936 |
| [7] |
LI J, XIE H, YING Y, et al. YTHDF2 mediates the mRNA degradation of the tumor suppressors to induce AKT phosphorylation in N6-methyladenosine-dependent way in prostate cancer[J]. Mol Cancer, 2020, 19(1): 152. DOI:10.1186/s12943-020-01267-6 |
| [8] |
LIU ZX, LI LM, SUN HL, et al. Link between m6A modification and cancers[J]. Front Bioeng Biotechnol, 2018, 6: 89. DOI:10.3389/fbioe.2018.00089 |
| [9] |
OERUM S, MEYNIER V, CATALA M, et al. A comprehensive review of m6A/m6A mRNA methyltransferase structures[J]. Nucleic Acids Res, 2021, 49(13): 7239-7255. DOI:10.1093/nar/gkab378 |
| [10] |
WANG T, KONG S, TAO M, et al. The potential role of RNA N6-methyladenosine in cancer progression[J]. Mol Cancer, 2020, 19(1): 88. DOI:10.1186/s12943-020-01204-7 |
| [11] |
LIANG JY, WANG DS, LIN HC, et al. A novel ferroptosis-related gene signature for overall survival prediction in patients with hepatocellular carcinoma[J]. Int J Biol Sci, 2020, 16(13): 2430-2441. DOI:10.7150/ijbs.45050 |
| [12] |
SRINIVAS US, TAN BWQ, VELLAYAPPAN BA, et al. ROS and the DNA damage response in cancer[J]. Redox Biol, 2019, 25: 101084. DOI:10.1016/j.redox.2018.101084 |
| [13] |
AMINI MA, TALEBI SS, KARIMI J. Reactive oxygen species modulator 1(ROMO1), a new potential target for cancer diagnosis and treatment[J]. Chonnam Med J, 2019, 55(3): 136-143. DOI:10.4068/cmj.2019.55.3.136 |
| [14] |
WANG L, LIU X, LIU Z, et al. Network models of prostate cancer immune microenvironments identify ROMO1 as heterogeneity and prognostic marker[J]. Sci Rep, 2022, 12(1): 192. DOI:10.1038/s41598-021-03946-w |
 2023, Vol. 52
2023, Vol. 52