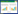文章信息
- 马俊杰, 安韶光, 梅静宇, 张磊, 张浩轩, 陆进
- MA Junjie, AN Shaoguang, MEI Jingyu, ZHANG Lei, ZHANG Haoxuan, LU Jin
- 肾透明细胞癌中拓扑异构酶Ⅱα与免疫浸润的关系及相关基因分析
- Analysis of immune infiltration and topoisomerase Ⅱα and related genes in renal clear cell carcinoma
- 中国医科大学学报, 2023, 52(10): 871-878
- Journal of China Medical University, 2023, 52(10): 871-878
-
文章历史
- 收稿日期:2022-11-23
- 网络出版时间:2023-10-16 18:37:56
2. 蚌埠医学院检验医学院2019级, 安徽 蚌埠 233030;
3. 蚌埠医学院第二附属医院肿瘤外科, 安徽 蚌埠 233030;
4. 蚌埠医学院基础医学院人体解剖学教研室, 安徽 蚌埠 233030
2. Grade 2019, School of Laboratory Medicine, Bengbu Medical College, Bengbu 233030, China;
3. Department of Oncology, The Second Affiliated Hospital of Bengbu Medical College, Bengbu 233030, China;
4. Department of Human Anatomy, School of Basic Medicine, Bengbu Medical College, Bengbu 233030, China
肾细胞癌(renal cell carcinoma,RCC)是泌尿生殖系统常见的恶性肿瘤之一,占成人肾脏肿瘤的85% 以上[1]。2020年RCC新发病例达43万,死亡患者约18万例[2]。肾透明细胞癌(kidney renal clear cell carcinoma,KIRC)是RCC的主要组织学亚型,约占80%[3]。目前,对于早期KIRC患者,手术切除仍然是主要和最有效的治疗方法,但发生远处转移的KIRC患者手术和放化疗效果不明显,5年生存率 < 10%,且复发风险较高[4]。因此,挖掘KIRC的新型生物标志物对于患者的诊断与治疗具有积极作用。
拓扑异构酶Ⅱα(topoisomerase Ⅱα,TOP2A)属于DNA解链因子,在细胞增殖、DNA复制以及染色体浓缩和分离等多种生物学过程(biological process,BP)中发挥关键作用,其表达具有明显的细胞周期依赖性[5]。TOP2A表达或结构异常时可导致染色体不稳定以及肿瘤的发生,如在卵巢癌和肺癌中TOP2A表达异常导致患者不良预后[6-7],但其在KIRC中的作用目前仍待商榷。因此,本研究旨在通过生物信息学方法分析TOP2A在KIRC中的表达、预后和免疫学等有关分子机制,为KIRC的诊断和治疗探索新的生物分子标志物。
1 材料与方法 1.1 数据来源KIRC单细胞测序数据来自基因表达综合数据库(Gene Expression Omnibus,GEO)GSE111360队列的GSM3029087(p87)、GSM3029088(p88)和GSM 3029091(p91)3个样本,根据以下标准过滤样本中的细胞和基因:(1)基因表达量200~50 000;(2)线粒体基因百分比 < 15%;(3)表达基因数量200~5 000;(4)≥3个细胞中表达的基因。其余分析所使用的数据均为各数据库默认来源。
1.2 TOP2A差异表达与预后分析TOP2A基因差异表达、临床分期及总生存期(overall survival,OS)的Kaplan-Meier(K-M)曲线通过GEPIA(http://gepia.cancer-pku.cn/)在线工具分析;TOP2A在正常肾组织和KIRC组织中的免疫组化结果应用HPA数据库进行获取。
1.3 TOP2A单细胞分析单细胞测序数据通过R包“Seurat”进行分析,首先通过NormalizeData函数对数据进行标准化,再通过FindIntegrationAnchors函数对3个患者样本识别锚点进行整合,然后通过RunPCA函数计算数据的主成分,选取前20个主成分通过K-最邻近分布进行聚类分析,细胞类型根据标记基因通过CellMarker数据库(http://bio-bigdata.hrbmu.edu.cn/CellMarker/)和文献收集进行注释。
1.4 TOP2A免疫分析TOP2A基因表达与KIRC免疫细胞浸润水平之间的相关性通过TIMER2.0(http://timer.cistrome.org/)分析;利用Sangerbox探究TOP2A基因表达与免疫和基质评分的关系,利用GEPIA2021的EPIC算法分析CD8+T细胞(cut-off=80%)、CD4+T细胞(cut-off=60%)、B细胞(cut-off=84%)、NK细胞(cut-off=85%)、巨噬细胞(cut-off=70%)和调节T(Tregs)细胞(cut-off=50%)的浸润水平与KIRC患者OS的关系。
1.5 TOP2A列线图将TOP2A基因和患者的临床特征纳入Aclbi进行单因素Cox回归分析,将P < 0.05的因素纳入模型进行列线图的构建,并通过校准曲线和受试者操作特征(receiver operating characteristic,ROC)曲线下面积(area under the curve,AUC)进行验证。
1.6 TOP2A相关基因筛选通过STRING数据库检索TOP2A基因的200个相关表达基因。然后,通过GEPIA2.0筛选TCGA-KIRC数据集中表达上调的基因(logFC≥1且q < 0.05)和TOP2A相关基因,共得到586个表达上调基因(以q值由小到大排序)和1 000个TOP2A相关基因(按相关性由大到小排序);最后,各取前200个基因进行韦恩图绘制,得到11个与TOP2A相关的基因。
1.7 TOP2A相关基因富集分析将获得的TOP2A及相关基因导入Aclbi分析其相关性;然后,通过Sangerbox探究TOP2A及相关基因的基因本体论(Gene Ontology,GO)和京都基因与基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集;最后,通过Sangerbox以KEGG通路基因集为参考,根据TOP2A基因表达中位值分为高、低表达组,对不同表达组进行基因集富集分析(gene set enrichment analysis,GSEA)。
1.8 TOP2A相关基因预后风险模型构建将TOP2A及相关基因导入Aclbi在线分析工具进行LASSO回归分析,将筛选出的基因进行风险评分(风险评分=基因表达量×系数)计算,根据风险评分的中位值将患者分为高、低风险组进行下游分析。
1.9 统计学分析单细胞数据通过R4.2.1软件进行分析,其余分析为各数据库默认软件。采用单向方差分析检验TOP2A基因在KIRC和正常组织差异表达,不同肿瘤分期表达采用F检验比较差异,免疫细胞和上皮细胞差异表达采用wilcoxon检验,采用K-M法和log-rank检测进行生存分析,采用Spearman相关性分析基因表达与免疫细胞浸润的水平相关性;P < 0.05为差异有统计学意义。
2 结果 2.1 TOP2A差异表达TOP2A在KIRC组织中表达上调,且TOP2A基因的表达水平随患者临床分期的增高而增加(P < 0.05,图 1A、1B)。TOP2A基因的高表达水平不利于患者的OS(图 1C);GSEA分析结果显示,高表达的TOP2A主要富集在细胞周期、不匹配修复和孕酮介导的卵母细胞成熟等与细胞增殖相关通路(图 1D)。同样,HPA数据库结果显示,TOP2A在肾癌组织中的蛋白质表达上调(图 1E)。
|
| A, differential expression of TOP2A in KIRC and normal tissues (*P < 0.05);B, relationship between TOP2A expression and clinical stages of patients; C, relationship between TOP2A expression and prognosis of KIRC patients; D, GSEA analysis of TOP2A;E, protein expression of TOP2A (left, normal tissue; right, KIRC tissue). 图 1 TOP2A差异表达、预后和GSEA分析 Fig.1 TOP2A differential expression, prognosis and GSEA analysis |
2.2 TOP2A基因单细胞表达分析
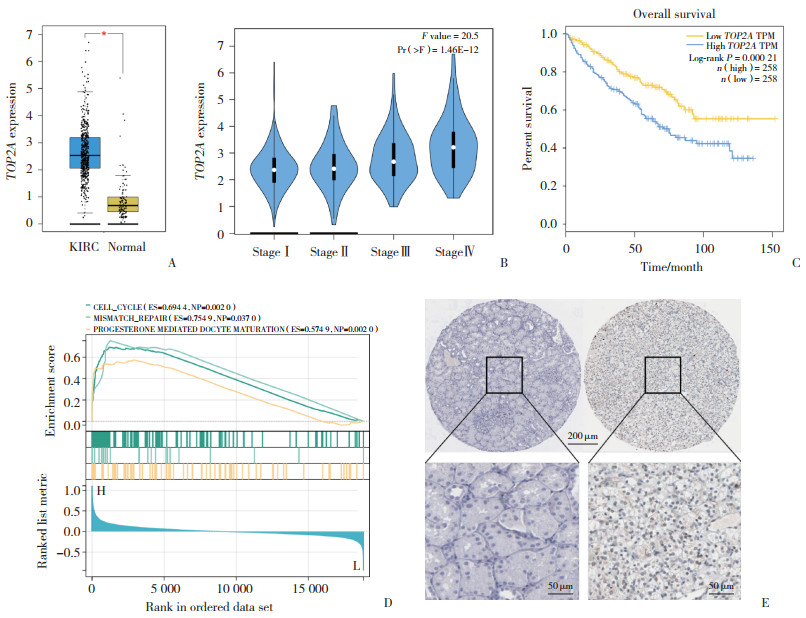
根据各聚类的标记基因可将各细胞分为上皮细胞和免疫细胞2种类型(图 2A、2B),且TOP2A基因在免疫细胞中的表达量高于上皮细胞(图 2C)。
|
| A, scatterplot of immune and epithelial cell clusters; B, expression ratio and expression level of marker genes in epithelial and immune cells; C, expression levels of TOP2A in epithelial and immune cells (* P < 0.001);D, scatterplot of the clustering of each immune cell; E, expression ratio and expression level of marker genes for each immune cell cluster; F, expression level of TOP2A in each immune cell. 图 2 TOP2A在不同细胞内的表达 Fig.2 Expression of TOP2A in different cells |
由于TOP2A基因主要在免疫细胞中表达,进一步对免疫细胞亚型进行聚类。根据各亚型的标记基因,共得到6种细胞类型NK细胞、CD8+T细胞、常规T细胞、Tregs、B细胞和肥大细胞(图 2D、2E)。TOP2A在多种免疫细胞中均有表达(图 2F)。
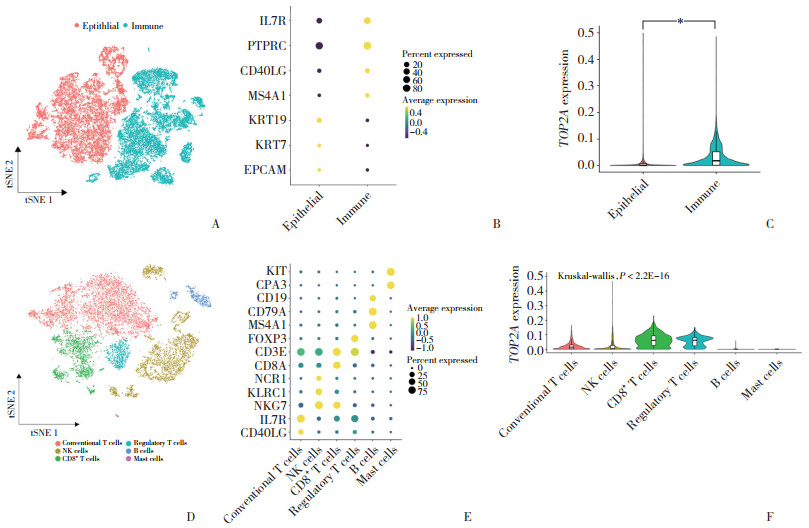
2.3 TOP2A免疫相关分析TIMER2.0数据库对TOP2A基因表达与各免疫细胞浸润水平的相关性分析结果显示,TOP2A与大多数免疫细胞浸润呈正相关(图 3A)。GEPIA2021数据库结果显示,CD8+T细胞和CD4+T细胞的高浸润水平与KIRC患者较好的预后相关,而B细胞、巨噬细胞、NK细胞及Tregs细胞在KIRC高浸润水平均为KIRC患者预后的危险因素(图 3B)。
|
| A, relationship between TOP2A gene expression and the level of immune cell infiltration; B, relationship between the level of immune cell infiltration and patient prognosis. 图 3 免疫细胞浸润水平与TOP2A基因表达及患者预后的关系 Fig.3 Relationship between immune cell infiltration level and TOP2A gene expression and patient prognosis |
2.4 TOP2A预后列线图
将TOP2A及患者的临床特征纳入单因素Cox回归分析,结果显示,TOP2A基因可做为KIRC患者预后影响因素(图 4A),TOP2A基因及患者临床特征所构建的列线图可预测KIRC患者的1、3和5年的生存状况,且随着列线图得分的增高,患者的生存率降低(图 4B)。校准曲线显示其具有较好的预测准确性(图 4C),且1年AUC为0.86、3年AUC为0.80、5年AUC为0.75,表明列线图可良好地预测患者的生存状况(图 4D)。
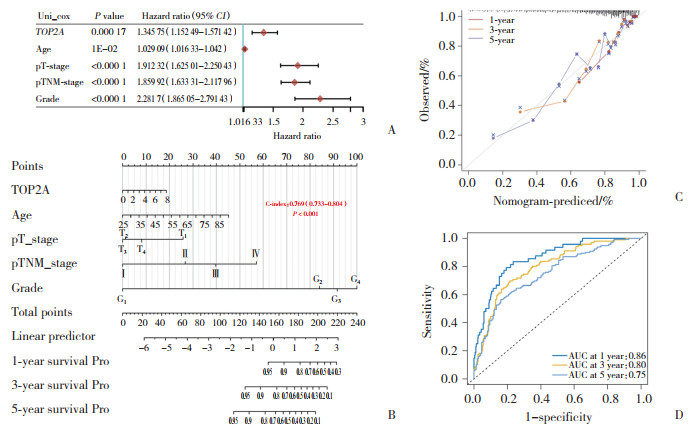
|
| A, forest plot of TOP2A gene and clinical characteristics of patients; B, nomogram of TOP2A gene and clinical characteristics of patients; C, calibration curves for nomogram for 1-, 3-, and 5-year survival rates; D, ROC curves for nomogram for 1-, 3-, and 5-year survival rates. 图 4 TOP2A风险预后列线图 Fig.4 Risk and prognostic factor nomogram for TOP2A |
2.5 TOP2A相关基因分析
TOP2A基因的相关基因进行韦恩图分析,共筛选出12个基因,且TOP2A基因及其相关基因的相关性较高(图 5A)。对包括TOP2A在内的12个基因进行GO功能注释和KEGG富集分析,结果显示,其KEGG通路主要富集在细胞周期、p53信号通路和细胞衰老等(图 5B);分子功能(molecular function,MF)主要包括腺苷酸结合、蛋白激酶活性和微管结合等;细胞构成(cellular component,CC)主要包括微管细胞骨架、染色体和微管组织中心等;BP主要包括细胞周期相变的调节、微管细胞骨架组织和细胞器调节等(图 5C)。
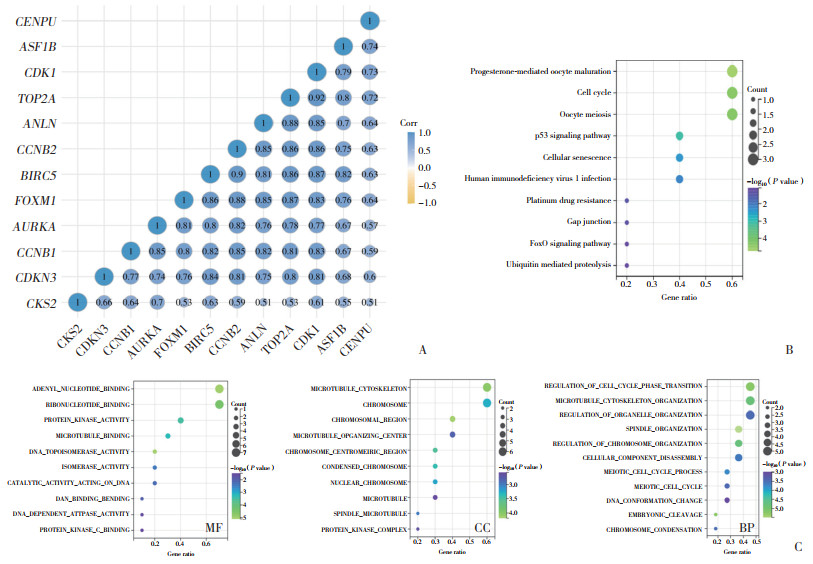
|
| A, correlation heatmap of TOP2A and its related genes; B, KEGG enrichment analysis; C, GO enrichment analysis. 图 5 TOP2A及其相关基因的富集分析 Fig.5 Enrichment analysis of TOP2A and its related genes |
2.6 TOP2A及其相关基因的风险评分模型
将TOP2A及其相关基因导入LASSO回归模型进行10倍交叉验证,得到最小λ值为0.013 8,筛选出包括TOP2A在内的9个特征基因,再根据基因表达量及相关系数构建风险评分(图 6A)。根据风险评分中位值将患者分为高、低风险组,结果显示,随着风险评分的增高患者的死亡率增高(图 6B),与低风险组相比,高风险组的OS显著缩短(图 6C)。且该模型预测患者OS ROC的AUC值1年为0.685、3年为0.655以及5年为0.659,具有较好的预测能力(图 6D)。
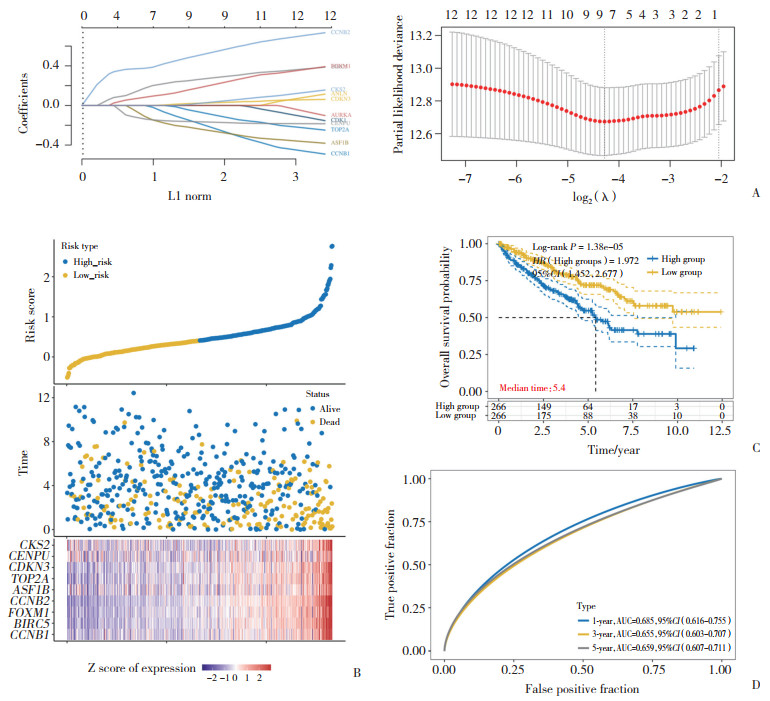
|
| A, LASSO regression model for TOP2A and its related genes; B, scatterplot of risk score versus patient overall survival and gene expression heatmap; C, K-M curves for patients in different risk groups; D, ROC curves for risk scores at 1, 3, and 5 years. 图 6 预后风险评分构建及验证 Fig.6 Construction and validation of prognostic risk scores |
3 讨论
KIRC是目前肾脏恶性肿瘤中最常见的组织学亚型,由于肿瘤的高度异质性,KIRC在不同患者中呈现多样化的临床表现,且晚期患者对于常见的化疗药物敏感性不高[8-9]。因此,迫切需要新的分子标志物评估患者临床的治疗和预后。目前,TOP2A已被证实与多种恶性肿瘤的进展和不良预后有关[10-11]。但其在KIRC中的相关研究甚少。
TOP2A基因在KIRC组织中的表达显著上调,并且与患者的不良预后和临床分期增高有关。此外,高表达的TOP2A主要富集在细胞周期和错配修复等过程,SMITH等[12]研究发现在肿瘤细胞由静止期进入细胞周期时,TOP2A的合成激活促进肿瘤细胞的增殖,这可能是其促进肿瘤发生发展的原因之一。差异分析和富集分析结果显示,TOP2A的表达可能与KIRC的发生、发展和转移关系密切。
对KIRC患者的单细胞测序数据分析发现,TOP2A基因在多种免疫细胞中表达,且与多种免疫细胞浸润水平呈正相关,与XU等[13]在肾乳头状细胞癌中的研究结果一致。不同的是在KIRC中,各免疫细胞的浸润水平长期增高反而不利于患者的预后。研究[14]显示,正常人体中CD4/CD8细胞比例处于特定的范围,二者的比例失调可导致机体免疫功能的异常。此外,巨噬细胞和B细胞也可能由于其在肿瘤中被诱导产生不同分化表型导致肿瘤细胞的增殖或肿瘤免疫逃避的产生[15-16]。TOP2A的表达不仅有利于KIRC的发生与转移,还可激活机体的免疫系统。肿瘤微环境是由肿瘤细胞与肿瘤间质细胞、免疫细胞、细胞外基质、血管组织和信号分子等相互作用形成的动态系统,各成分的相对比例改变也可能导致肿瘤侵袭性增高及患者的不良预后[17-18],这可能是TOP2A基因诱导KIRC患者不良预后的原因之一。
总之,本研究基于生物信息学方法探究了TOP2A基因在KIRC的表达和免疫浸润等有关分子机制,建立了预后风险模型,并应用列线图预测了TOP2A基因在KIRC中的有关预后作用,表明TOP2A可作为KIRC的预后分子标志物,对临床治疗和预后提供参考。本研究还需要相关的基础和临床实验进行验证,以便更加准确地探究有关分子机制及预后意义。
| [1] |
HUANG YF, WANG QZ, TANG Y, et al. Identification and validation of a cigarette smoke-related five-gene signature as a prognostic biomarker in kidney renal clear cell carcinoma[J]. Sci Rep, 2022, 12(1): 2189. DOI:10.1038/s41598-022-06352-y |
| [2] |
ZHANG FT, HOU T, CHEN L, et al. Comprehensive analysis of lower mitochondrial complex I expression is associated with cell metastasis of clear cell renal cell carcinoma[J]. Transl Cancer Res, 2022, 11(6): 1488-1502. DOI:10.21037/tcr-22-242 |
| [3] |
ZHANG H, ZHANG H, HU YM, et al. Diagnostic and prognostic role of basic leucine zipper transcription factor in kidney renal clear cell carcinoma[J]. Transl Androl Urol, 2022, 11(2): 238-252. DOI:10.21037/tau-21-1130 |
| [4] |
ZENG W, XIONG GG, HUA L, et al. APOA1 mRNA and protein in kidney renal clear cell carcinoma correlate with the disease outcome[J]. Sci Rep, 2022, 12(1): 12406. DOI:10.1038/s41598-022-16434-6 |
| [5] |
WANG XB, WANG JH, LYU L, et al. Oncogenic role and potential regulatory mechanism of topoisomeraseⅡα in a pan-cancer analysis[J]. Sci Rep, 2022, 12(1): 11161. DOI:10.1038/s41598-022-15205-7 |
| [6] |
LIU P, LI H, LIAO CF, et al. Identification of key genes and biological pathways in Chinese lung cancer population using bioinformatics analysis[J]. Peer J, 2022, 10: e12731. DOI:10.7717/peerj.12731 |
| [7] |
WEI M, BAI XF, DONG QM. Identification of novel candidate genes and small molecule drugs in ovarian cancer by bioinformatics strategy[J]. Transl Cancer Res, 2022, 11(6): 1630-1643. DOI:10.21037/tcr-21-2890 |
| [8] |
ZONG XF, FU JX, WANG ZY, et al. The diagnostic and prognostic values of HOXA gene family in kidney clear cell renal cell carcinoma[J]. J Oncol, 2022, 2022: 1762637. DOI:10.1155/2022/1762637 |
| [9] |
KONG GC, WANG YX, HUANG YJ, et al. Identification and verification of tumor immune microenvironment-related prognostic genes in kidney renal clear cell carcinoma[J]. Biomed Res Int, 2022, 2022: 5563668. DOI:10.1155/2022/5563668 |
| [10] |
TSYGANOV MM, IBRAGIMOVA MK, GARBUKOV EY, et al. Predictive and prognostic significance of mRNA expression and DNA copies aberrations of ERCC1, RRM1, TOP1, TOP2A, TUBB3, TYMS, and GSTP1 genes in patients with breast cancer[J]. Diagnostics, 2022, 12(2): 405. DOI:10.3390/diagnostics12020405 |
| [11] |
WANG J, WANG CY, YANG LQ, et al. Identification of the critical genes and miRNAs in hepatocellular carcinoma by integrated bioinformatics analysis[J]. Med Oncol, 2022, 39(2): 21. DOI:10.1007/s12032-021-01622-7 |
| [12] |
SMITH PJ, MCKEOWN SR, PATTERSON LH. Targeting DNA topoisomeraseⅡα (TOP2A) in the hypoxic tumour microenvironment using unidirectional hypoxia-activated prodrugs (uHAPs)[J]. IUBMB Life, 2023, 75(1): 40-54. DOI:10.1002/iub.2619 |
| [13] |
XU H, ZHENG XN, ZHANG SY, et al. Tumor antigens and immune subtypes guided mRNA vaccine development for kidney renal clear cell carcinoma[J]. Mol Cancer, 2021, 20(1): 159. DOI:10.1186/s12943-021-01465-w |
| [14] |
ZHANG L, ZHOU GZ, FENG WY, et al. Immune status and chemokine C receptor 7 expression in primary in patients with immune thrombocytopenia[J]. Turk J Haematol, 2022, 39(1): 29-37. DOI:10.4274/tjh.galenos.2021.2021.0281 |
| [15] |
JIN JX, YU GP. Hypoxic lung cancer cell-derived exosomal miR-21 mediates macrophage M2 polarization and promotes cancer cell proliferation through targeting IRF1[J]. World J Surg Oncol, 2022, 20(1): 241. DOI:10.1186/s12957-022-02706-y |
| [16] |
李慧婷, 李桂源, 徐平, 等. B细胞与肿瘤免疫逃逸[J]. 中南大学学报(医学版), 2022, 47(3): 358-363. DOI:10.11817/j.issn.1672-7347.2022.210275 |
| [17] |
WANG Y, SONG TJ, LI K, et al. Heparanase is a prognostic biomarker independent of tumor purity and hypoxia based on bioinformatics and immunohistochemistry analysis of esophageal squamous cell carcinoma[J]. World J Surg Onc, 2022, 20(1): 236. DOI:10.1186/s12957-022-02698-9 |
| [18] |
QIAN B, SUN J, ZUO PC, et al. Verification of genetic differences and immune cell infiltration subtypes in the neuroblastoma tumour microenvironment during immunotherapy[J]. World J Surg Onc, 2022, 20(1): 169. DOI:10.1186/s12957-022-02641-y |
 2023, Vol. 52
2023, Vol. 52