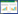文章信息
- 郭嘉, 卞华, 陈爽, 单雨, 卞博
- GUO Jia, BIAN Hua, CHEN Shuang, SHAN Yu, BIAN Bo
- 基于生物信息学分析硬皮病的发病机制
- Analysis of the pathogenesis of scleroderma based on bioinformatics
- 中国医科大学学报, 2022, 51(9): 776-782
- Journal of China Medical University, 2022, 51(9): 776-782
-
文章历史
- 收稿日期:2021-05-25
- 网络出版时间:2022-08-15 9:17
2. 南阳理工学院张仲景国医国药学院,河南省张仲景方药与免疫调节重点实验室,河南 南阳 473004
2. Zhang Zhongjing College of Traditional Chinese Medicine, Nanyang Institute of Technology, Henan Key Laboratory of Zhang Zhongjing Formulae and Herbs for Immunoregulation, Nanyang 473004, China
硬皮病也称系统性硬化症,其特征是广泛的血管损伤和功能障碍,血管生成受损,免疫失调和皮肤、内脏器官的进行性纤维化[1]。纤维化的发病机制主要涉及缺血再灌注、氧化应激以及纤溶和凝血功能失衡[2]。硬皮病发病有多种免疫异常,包括T细胞、B细胞和巨噬细胞[3]共同参与。硬皮病早期纤维性病变伴有血管周围炎症细胞浸润,主要由T淋巴细胞和单核细胞共同参与[2]。研究[2, 4]显示,超过90%的硬皮病患者能检测到激活的血管、上皮和免疫细胞产生促纤维化的细胞因子、趋化因子、生长因子、脂质介质和自身抗体(抗DNA拓扑异构酶Ⅰ、抗着丝粒和抗RNA聚合酶抗体)。目前,多数研究认为硬皮病发病与基因遗传、环境诱导、病毒感染有关。本研究基于基因表达图谱(gene expression omnibus,GEO)数据库芯片数据,并与正常人群进行比较,探讨硬皮病发病的分子机制。
1 材料与方法 1.1 GEO芯片数据及硬皮病差异表达基因(differentially expressed genes,DEGs)获取登录GEO数据库(https://www.ncbi.nlm.nih.gov/geo/),以“scleroderma”为关键词进行检索;获取以GPL14951-11332为平台文件的GSE117928芯片数据[5]。该芯片使用illumina BeadChips方法对18例硬皮病患者(硬皮病组)和19例正常对照者(正常组)进行全基因组mRNA转录和整体DNA甲基化分析。使用R软件中的“limma”包进行t检验,过滤条件为|logFC| > 1,调整P < 0.05,筛选硬皮病的DEGs;并通过R软件中“pheatmap”包绘制热图。
1.2 基因本体(gene ontology,GO)富集分析及蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络核心靶点获取使用R软件中的“colorspace”“stringi”“org.Hs.eg.db”“ggplot2”包对DEGs进行GO富集分析。将获取的DEGs输入String数据库(https://www.string-db.org)进行PPI分析,物种设置为“Homo sapiens”,最低互动要求分数设置为0.9。下载相关数据,使用Cytoscape 3.8.0软件,根据中介中心度、接近中心度、程度中心性、特征向量中心度、基于局部平均连接度的方法、信息中心度以及网络中心性,设置过滤条件为每项皆大于中位值,阶梯式过滤2次,最终获取核心靶点。
1.3 基因集富集分析(gene set enrichment analysis,GSEA)通路分析GSEA是用来确定一组先验定义的基因是否在两种生物状态之间显示出统计上显著、一致的差异的计算方法。将下载的GEO矩阵文件整理并分组为硬皮病组和正常组。使用GSEA 4.1.0软件,基因功能数据库选择c2.cp.kegg.v7.4.symbols.gmt[Curated],检验次数设置为1 000。一般认为|NES| > 1,P < 0.05,FDR q < 0.25的通路存在显著差异[6]。
1.4 免疫细胞浸润(immune cell infiltration,ICI)分析将获取的GSE117928芯片数据进行ICI分析。采用CIBERSORT标准,perm=1 000,P < 0.05为过滤条件,获取硬皮病组和正常组中20类免疫细胞表达的相对含量。分析2组免疫细胞的差异和硬皮病组内各免疫细胞的相关性。
2 结果 2.1 DEGs获取结果结果显示,共获取硬皮病组与正常组DEGs 722个,其中上调基因298个,下调基因424个。见图 1。
|
| 图 1 正常组与硬皮病组DEGs热图 Fig.1 Heat map of differentially expressed genes between normal and scleroderma groups |
2.2 GO富集分析及PPI核心靶点获取结果
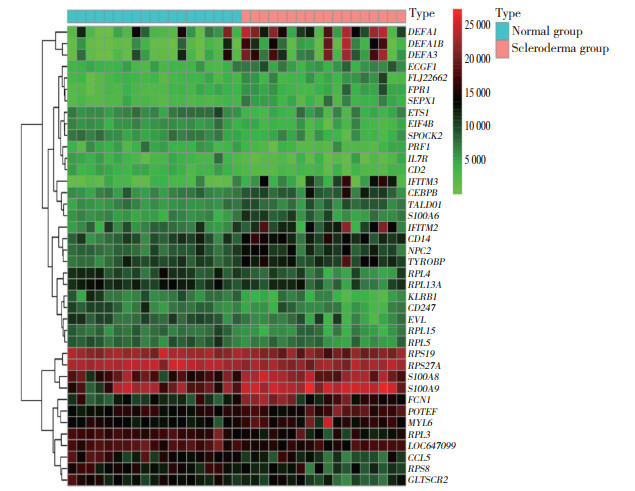
GO富集分析结果显示,DEGs多富集在中性粒细胞活化参与免疫反应、嗜中性粒细胞脱颗粒、髓系细胞分化、对脂多糖反应、积极调节防御反应、对细菌源分子的反应、先天免疫反应的调节、正向调节细胞因子的产生、对生物刺激反应的调节、核糖核酸分解过程等生物功能,见表 1。登录String数据库,输入DEGs构建PPI网络。下载相关数据,导入Cytoscape软件,使用CytoNCA插件对其进行分析,获取核心靶点包括RPS27A、BTRC、FBXO32、ZBTB16、FBXO21、VHL、ATG7、CBLB、ANAPC1、COMMD9、CSNK1E、RPS8、MPHOSPH10、KRR1、RPL5,见图 2。
| ID | Description | Gene ratio | P(adjust) |
| GO:0002283 | Neutrophil activation involved in immune response | 55/581 | 4.00e-13 |
| GO:0043312 | Neutrophil degranulation | 54/581 | 6.60e-13 |
| GO:0030099 | Myeloid cell differentiation | 38/581 | 1.00e-05 |
| GO:0032496 | Response to lipopolysaccharide | 32/581 | 1.71e-05 |
| GO:0031349 | Positive regulation of defense response | 34/581 | 3.68e-05 |
| GO:0002237 | Response to molecule of bacterial origin | 32/581 | 5.14e-05 |
| GO:0045088 | Regulation of innate immune response | 29/581 | 6.34e-05 |
| GO:0001819 | Positive regulation of cytokine production | 36/581 | 8.76e-05 |
| GO:0002831 | Regulation of response to biotic stimulus | 34/581 | 8.76e-05 |
| GO:0006401 | RNA catabolic process | 33/581 | 0.000 329 |
|
| 图 2 PPI网络核心靶点 Fig.2 Core targets of PPI network |
2.3 GSEA通路分析
通过GSEA通路分析,根据P值大小,筛选2组排名前五位的通路绘图。结果显示,硬皮病组排名前五位的通路为氨基糖和核苷酸糖代谢、溶酶体代谢、谷胱甘肽代谢、糖酵解糖异生和半乳糖代谢通路。正常组排名前五位的通路为自然杀伤细胞介质细胞毒性、剪接体、泛素介导的蛋白水解、小细胞肺癌、T细胞受体信号通路。见表 2。
| Pathways | NES | P | FDR q |
| Scleroderma group | |||
| KEGG_AMINO_SUGAR_AND_NUCLEOTIDE_SUGAR_METABOLISM | 1.769 | 0.002 | 0.187 |
| KEGG_LYSOSOME | 1.831 | 0.002 | 0.197 |
| KEGG_GLUTATHIONE_METABOLISM | 1.731 | 0.004 | 0.175 |
| KEGG_GLYCOLYSIS_GLUCONEOGENESIS | 1.624 | 0.004 | 0.155 |
| KEGG_GALACTOSE_METABOLISM | 1.605 | 0.004 | 0.147 |
| Normal group | |||
| KEGG_SPLICEOSOME | -1.627 | 0.016 | 0.500 |
| KEGG_UBIQUITIN_MEDIATED_PROTEOLYSIS | -1.626 | 0.018 | 0.334 |
| KEGG_SMALL_CELL_LUNG_CANCER | -1.392 | 0.028 | 0.395 |
| KEGG_T_CELL_RECEPTOR_SIGNALING_PATHWAY | -1.596 | 0.028 | 0.322 |
2.4 ICI结果
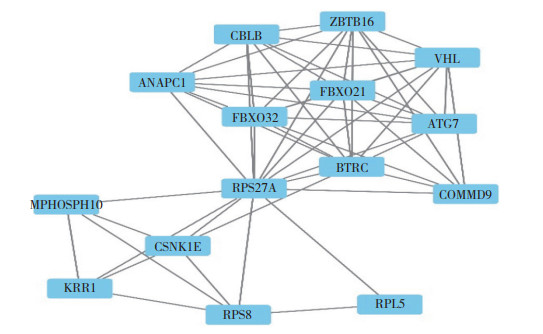
使用CIBERSORT筛选出20种免疫细胞表达的相对含量。结果显示,正常组CD8+T细胞、幼稚CD4+T细胞表达明显增加,而硬皮病组单核细胞表达明显增加,见图 3。
|
| 图 3 硬皮病组与正常组免疫细胞表达相对含量比较 Fig.3 Comparison of immune cell expression between normal and scleroderma groups |
硬皮病组各免疫细胞相关分析结果显示,浆细胞与巨噬细胞M1、激活的肥大细胞,巨噬细胞M1与激活的肥大细胞,巨噬细胞M2与激活的树突状细胞,巨噬细胞M0与静息的肥大细胞呈正相关;CD8+ T细胞与静息的CD4+T细胞、单核细胞,激活的CD4+ T细胞与滤泡辅助T细胞、单核细胞,调节性T细胞与单核细胞,静息的NK细胞与巨噬细胞M2呈负相关,见图 4。
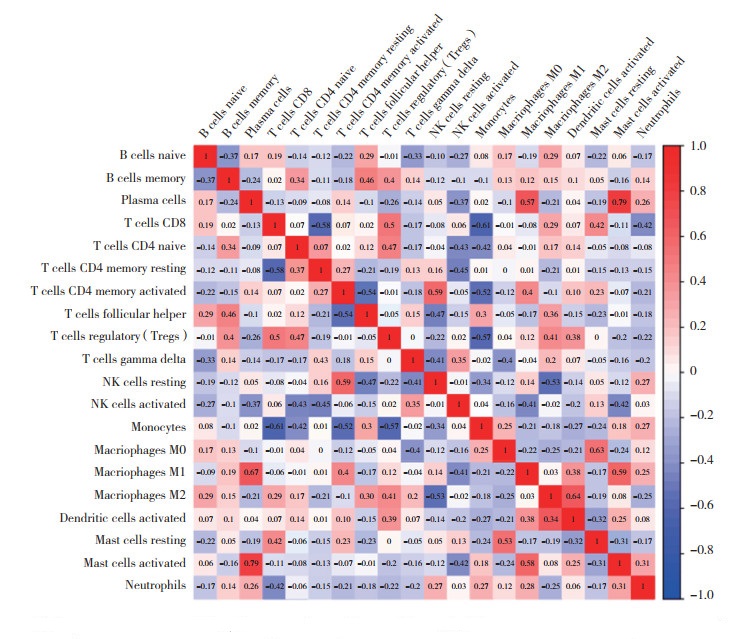
|
| Red represents a positive correlation,blue represents a negative correlation,and a darker color indicates a more significant correlation. 图 4 硬皮病组内免疫细胞表达相对含量相关性分析 Fig.4 Correlation analysis of immune cell expression in scleroderma group |
3 讨论
本研究结果显示,硬皮病组与正常组DEGs共722个,其中上调基因298个,下调基因424个。已有研究[7]显示,氧化应激发生与硬皮病的发病机制有关,已知中性粒细胞在硬皮病的发病过程中会产生大量活性氧。在中性粒细胞激活过程中,由于氧化应激,细胞释放被组蛋白、酶和其他抗菌蛋白覆盖的DNA细丝。这些细丝称为中性粒细胞外陷阱,参与细菌防御过程,但它们的形成在大多数情况下会导致中性粒细胞死亡[8]。中性粒细胞与血小板衍生的微粒相互作用,促进自噬和硬皮病患者净副产物的产生[9]。抗中性粒细胞胞质抗体(anti-neutrophil cytoplasmic antibodies,ANCA)是在单核细胞、中性粒细胞和溶酶体初级颗粒中发现的酶自身抗体,与小血管炎症的发病机制直接相关。研究[10]显示ANCA与硬皮病患者肺间质肺炎、肺栓塞患病率增加有关。先天免疫在硬皮病发病过程中占据着重要地位。先天性淋巴样细胞(innate lymphoid cells,ILCs)被发现于人类和小鼠的黏膜系统和内脏器官中,皮肤、肺和肠黏膜中含量丰富。ILCs存于胎儿肝脏和成人骨髓中,可以产生先天淋巴祖细胞以及B、T细胞系[11]。国际免疫学会联合将ILCs分为自然杀伤细胞、ILC1、ILC2、ILC3和淋巴组织诱导物5个分型[12]。研究[13]显示,ILC2在促纤维化的“2型”途径中处于中心地位,能够引发下游的一系列事件,通过这些事件使转化生长因子-β信号上调,使胶原过度沉积而导致异常纤维化。ILCs在协调免疫反应、维持代谢稳态和调节组织炎症方面发挥着重要作用[11]。GO富集分析显示,DEGs多富集在中性粒细胞活化参与免疫反应、嗜中性粒细胞脱颗粒、髓系细胞分化、对脂多糖反应、积极调节防御反应、对细菌源分子的反应、先天免疫反应的调节、正向调节细胞因子的产生、对生物刺激反应的调节、核糖核酸分解过程等生物功能,与已有研究相互佐证。
本研究结果显示,核心靶点包括RPS27A、BTRC、FBXO32、ZBTB16、FBXO21、VHL、ATG7、CBLB、ANAPC1、COMMD9、CSNK1E、RPS8、MPHOSPH10、KRR1、RPL5。现有研究[14]显示,FBXO32、过表达的miR-26可增加骨骼肌横截面积,降低FBXO32/ atrogin-1和TRIM63/MuRF1的上调,抑制了心肌纤维化病变。ZBTB16与代谢综合征关系密切,影响脂肪生成、心肌肥大和纤维化、血脂水平和胰岛素的敏感性[15]。细胞外纤维连接蛋白和胶原网络受VHL蛋白调节,VHL蛋白过表达会导致肺成纤维细胞增殖,促进纤维连接蛋白、胶原蛋白和细胞外纤维连接蛋白的表达。一项关于肾脏的研究[16]表明,受VHL和TGFb1调节的MXRA5蛋白具有抗炎和抗纤维化的功能。ZHOU等[17]研究发现VHL蛋白在博莱霉素诱导的肺纤维化中起着关键作用,可能是通过非依赖缺氧诱导因子的途径。ATG7是传统的大分子自噬的重要组成部分,它允许微管相关蛋白1A/1B轻链3B(LC3BI)通过磷脂酰乙醇胺偶联转化为LC3BII[18]。LC3BII在ATG12-ATG5:ATG16L1复合物的帮助下整合到吞噬体膜上,且ATG7也参与其中,导致自噬小体成熟;成熟的自噬小体与溶酶体结合,导致蛋白质和细胞器降解[19]。多项研究显示ATG7缺陷与肺纤维化[18]、肝纤维化[20]、肠纤维化[21]、腹膜纤维化[22]关系密切。
GSEA富集分析结果显示,硬皮病组排名前五位的通路包括氨基糖和核苷酸糖的代谢、溶酶体代谢、谷胱甘肽代谢、糖酵解糖质新生、半乳糖代谢。自噬-溶酶体是重要的细胞内自我降解系统,当自噬功能紊乱时对炎症疾病(感染、自身免疫和癌症、代谢紊乱、神经退行性变以及心血管和肝脏疾病[23])产生影响。研究[24]显示自噬-溶酶体途径与硬皮病硬化期皮肤纤维化密切相关。本研究结果显示,正常组排名前五位的通路包括自然杀伤细胞介导的细胞毒性、剪接体、泛素介导的蛋白质水解、凯格小细胞肺癌、T细胞受体信号通路。T细胞中Th17、Treg、Th2、Th9和Th22与硬皮病的纤维化病变密切相关[25]。
ICI结果显示,与正常组比较,硬皮病组单核细胞显著增多,而CD8+T细胞、静息CD4+T细胞减少,提示CD8+T细胞在硬皮病发病过程发挥重要作用。研究[26]显示,硬皮病患者外周血CD8+T细胞和活化/记忆性CD8+T细胞计数降低,与硬皮病的临床亚型、进展阶段、器官受累以及毛细支气管镜评估的微血管异常有关。但是,目前关于硬皮病患者外周血中T细胞异常的证据仍是相互矛盾的,一些研究[27]显示CD4+T和CD8+T细胞增加,另一些研究[28]则显示CD4+T和CD8+T细胞降低,还有研究[29]显示硬皮病患者和正常组之间没有显著差异,因此还需进一步论证。
综上所述,硬皮病发病可能是通过调节RPS27A、BTRC、FBXO32、ZBTB16、FBXO21、VHL、ATG7、CBLB、ANAPC1等基因,参与中性粒细胞活化参与免疫反应、嗜中性粒细胞脱颗粒等生物过程,上调氨基糖和核苷酸糖代谢、溶酶体代谢、谷胱甘肽代谢、糖酵解糖异生和半乳糖代谢等通路来实现的。硬皮病发病与单核细胞增多密切相关。本研究从生物信息学角度对硬皮病发病机制进行探究,为今后临床研究和基础实验提供了理论依据和数据支撑,但存在样本量少,未进行细胞分子学实验验证等不足之处,期待今后进一步研究论证。
| [1] |
DENTON CP, KHANNA D. Systemic Sclerosis[J]. Lancet, 2017, 390(10103): 1685-1699. DOI:10.1016/S0140-6736(17)30933-9 |
| [2] |
VARRICA C, DIAS HS, REIS C, et al. Targeted delivery in scleroderma fibrosis[J]. Autoimmun Rev, 2021, 20(2): 102730. DOI:10.1016/j.autrev.2020.102730 |
| [3] |
YOSHIZAKI A. Pathogenic roles of B lymphocytes in systemic sclerosis[J]. Immunol Lett, 2018, 195: 76-82. DOI:10.1016/j.imlet.2018.01.002 |
| [4] |
OKAMURA A, MATSUSHITA T, KOMURO A, et al. Adipose-derived stromal/stem cells successfully attenuated fibrosis in scleroderma mouse models[J]. Int J Rheum Dis, 2020, 23(2): 216-225. DOI:10.1111/1756-185X.13764 |
| [5] |
ZHU HL, ZHU CS, MI WT, et al. Integration of genome-wide DNA methylation and transcription uncovered aberrant methylation-regu- lated genes and pathways in peripheral blood mononuclear cells in systemic sclerosis[J]. Int J Rheumatol, 2018, 2018: 7342472. DOI:10.1155/2018/7342472 |
| [6] |
SUBRAMANIAN A, TAMAYO P, MOOTHA VK, et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles[J]. Proc Natl Acad Sci USA, 2005, 102(43): 15545-15550. DOI:10.1073/pnas.0506580102 |
| [7] |
VONA R, GIOVANNETTI A, GAMBARDELLA L, et al. Oxidative stress in the pathogenesis of systemic scleroderma: an overview[J]. J Cell Mol Med, 2018, 22(7): 3308-3314. DOI:10.1111/jcmm.13630 |
| [8] |
BRINKMANN V, ZYCHLINSKY A. Neutrophil extracellular traps: Is immunity the second function of chromatin?[J]. J Cell Biol, 2012, 198(5): 773-783. DOI:10.1083/jcb.201203170 |
| [9] |
MAUGERI N, CAPOBIANCO A, ROVERE-QUERINI P, et al. Platelet microparticles sustain autophagy-associated activation of neutrophils in systemic sclerosis[J]. Sci Transl Med, 2018, 10(451): eaao3089. DOI:10.1126/scitranslmed.aao3089 |
| [10] |
MOXEY J, HUQ M, PROUDMAN S, et al. Significance of anti-neutrophil cytoplasmic antibodies in systemic sclerosis[J]. Arthritis Res Ther, 2019, 21(1): 57. DOI:10.1186/s13075-019-1839-5 |
| [11] |
ZHOU SQ, LI QW, WU HJ, et al. Pathogenic role of innate lymphoid cells in autoimmune and inflammatory skin diseases[J]. Cell Mol Immunol, 2020, 17(4): 335-346. DOI:10.1038/s41423-020-0399-6 |
| [12] |
VIVIER E, ARTIS D, COLONNA M, et al. Innate lymphoid cells: 10 years[J]. Cell, 2018, 174(5): 1054-1066. DOI:10.1016/j.cell.2018.07.017 |
| [13] |
HORSBURGH S, TODRYK S, RAMMING A, et al. Innate lymphoid cells and fibrosis regulation[J]. Immunol Lett, 2018, 195: 38-44. DOI:10.1016/j.imlet.2017.08.022 |
| [14] |
WANG B, ZHANG AQ, WANG HD, et al. miR-26a limits muscle wasting and cardiac fibrosis through exosome-mediated microRNA transfer in chronic kidney disease[J]. Theranostics, 2019, 9(7): 1864-1877. DOI:10.7150/thno.29579 |
| [15] |
ŠEDA O, ŠEDOVÁ L, VČELÁK J, et al. ZBTB16 and metabolic syndrome: network perspective[J]. Physiol Res, 2017, 66. DOI:10.33549/physiolres.933730 |
| [16] |
POVEDA J, SANZ AB, FERNANDEZ-FERNANDZ B, et al. MXRA5 is a TGF-β1-regulated human protein with anti-inflammatory and antifibrotic properties[J]. J Cell Mol Med, 2017, 21(1): 154-164. DOI:10.1111/jcmm.12953 |
| [17] |
ZHOU QY, CHEN TJ, ZHANG W, et al. Suppression of von Hippel-Lindau protein in fibroblasts protects against bleomycin-induced pulmonary fibrosis[J]. Am J Respir Cell Mol Biol, 2016, 54(5): 728-739. DOI:10.1165/rcmb.2015-0111OC |
| [18] |
STOJANOVIĆ SD, FUCHS M, FIEDLER J, et al. Comprehensive bioinformatics analysis identified key miRNA players in ATG7-deficient lung fibroblasts[J]. Int J Mol Sci, 2020, 21(11): 4126. DOI:10.3390/ijms21114126 |
| [19] |
GALLUZZI L, GREEN DR. Autophagy-independent functions of the autophagy machinery[J]. Cell, 2019, 177(7): 1682-1699. DOI:10.1016/j.cell.2019.05.026 |
| [20] |
RUART M, CHAVARRIA L, CAMPRECIÓS G, et al. Impaired endothelial autophagy promotes liver fibrosis by aggravating oxidative stress responses during acute liver injury[J]. J Hepatol, 2019, 70(3): 458-469. DOI:10.1016/j.jhep.2018.10.015 |
| [21] |
MATHUR R, ALAM MM, ZHAO XF, et al. Induction of autophagy in Cx3cr1 + mononuclear cells limits IL-23/IL-22 axis-mediated intestinal fibrosis[J]. Mucosal Immunol, 2019, 12(3): 612-623. DOI:10.1038/s41385-019-0146-4 |
| [22] |
LI ST, PENG FF, GONG WQ, et al. Dimethylaminomicheliolide ameliorates peritoneal fibrosis by activating autophagy[J]. J Mol Med (Berl), 2019, 97(5): 659-674. DOI:10.1007/s00109-019-01757-1 |
| [23] |
DERETIC V. Autophagy in inflammation, infection, and immunometabolism[J]. Immunity, 2021, 54(3): 437-453. DOI:10.1016/j.immuni.2021.01.018 |
| [24] |
MORI T, TAMURA N, WAGURI S, et al. Autophagy is involved in the sclerotic phase of systemic sclerosis[J]. Fukushima J Med Sci, 2020, 66(1): 17-24. DOI:10.5387/fms.2019-28 |
| [25] |
ZHANG MJ, ZHANG S. T cells in fibrosis and fibrotic diseases[J]. Front Immunol, 2020, 11: 1142. DOI:10.3389/fimmu.2020.01142 |
| [26] |
ALMEIDA I, SILVA SV, FONSECA AR, et al. T and NK cell phenotypic abnormalities in systemic sclerosis: a cohort study and comprehensive literature review[J]. Clin Rev Allergy Immunol, 2015, 49(3): 347-369. DOI:10.1007/s12016-015-8505-8 |
| [27] |
ARTLETT CM, COX LA, RAMOS RC, et al. Increased microchimeric CD4+ T lymphocytes in the peripheral blood of women with systemic sclerosis[J]. Clin Immunol, 2002, 103(3 Pt 1): 303-308. DOI:10.1006/clim.2002.5222 |
| [28] |
GAMBICHLER T, TIGGES C, BURKERT B, et al. Absolute counts of T and B lymphocyte subsets are decreased in systemic sclerosis[J]. Eur J Med Res, 2010, 15(1): 44-46. DOI:10.1186/2047-783x-15-1-44 |
| [29] |
ERCOLE LP, MALVEZZI M, BOARETTI AC, et al. Analysis of lymphocyte subpopulations in systemic sclerosis[J]. J Investig Allergol Clin Immunol, 2003, 13(2): 87-93. DOI:10.1089/10799900360520450 |
 2022, Vol. 51
2022, Vol. 51